RNAサイレンシングにかかわるSiwiの結晶構造
松本直樹1・西増弘志1・塩見美喜子2・濡木 理1
(東京大学大学院理学系研究科 1生物科学専攻生物化学講座構造生命科学研究室,2生物科学専攻生物化学講座RNA生物学研究室)
email:西増弘志,塩見美喜子,濡木 理
DOI: 10.7875/first.author.2016.105
Crystal structure of silkworm PIWI-clade Argonaute Siwi bound to piRNA.
Naoki Matsumoto, Hiroshi Nishimasu, Kazuhiro Sakakibara, Kazumichi M. Nishida, Takamasa Hirano, Ryuichiro Ishitani, Haruhiko Siomi, Mikiko C. Siomi, Osamu Nureki
Cell, 167, 484-497.e9 (2016)
非コード小分子RNAのひとつであるpiRNAはPIWIタンパク質とpiRISCとよばれる複合体を形成し,生殖組織においてトランスポゾンの発現を抑制し遺伝情報を保護する役割をもつ.しかし,PIWIタンパク質の立体構造は不明であり,その作動機構の理解は遅れていた.今回,筆者らは,カイコのもつPIWIタンパク質のひとつであるSiwiについて,カイコの卵巣に由来する培養細胞からpiRNAと結合した状態で精製し,その結晶構造を決定することに成功した.結晶構造からSiwiによるpiRNAの認識機構が明らかにされた.さらに,SiwiとAGOタンパク質のひとつであるhAgo2との構造の比較により,両者の核酸結合チャネルに顕著な違いが見い出された.この違いはPIWIタンパク質とAGOタンパク質の機能の違いを反映すると考えられた.今回の研究の成果は,PIWIタンパク質によるトランスポゾンの抑制機構の解明にむけた第一歩となる.
非コード小分子RNAはArgonauteタンパク質と複合体を形成し,標的となる遺伝子の発現を制御する.この機構はRNAサイレンシングとよばれる.非コード小分子RNAはArgonauteタンパク質を相補的な標的RNAへと導き,切断や転写抑制あるいは翻訳抑制をひき起こす.Argonauteタンパク質はAGOタンパク質とPIWIタンパク質の2つのクレードに分類される.AGOタンパク質は全身の組織に広く発現し,約22塩基長のsiRNA(small interfering RNA)やmiRNA(microRNA)とRISC(RNA-induced silencing complex)を形成し多岐にわたる生命現象に関与する.一方,PIWIタンパク質は動物の生殖組織に発現し,23~33塩基長のpiRNA(PIWI-interacting RNA)とpiRISC(piRNA-induced silencing complex)を形成し,トランスポゾンに由来するRNAを抑制することにより遺伝情報を次世代に正確に伝達する役割を担う1).AGOタンパク質とPIWIタンパク質では複合体の形成の機構も異なる.AGOタンパク質においては2本鎖RNAが取り込まれたのち片方のRNA鎖が放出されることによりRISCが形成される.一方,PIWIタンパク質には長い1本鎖の前駆体RNAが取り込まれたのち,Zucchini 2)(新着論文レビュー でも掲載)などのRNA切断酵素やHen1 3) などのメチル化酵素により切断あるいは修飾されることによりpiRISCが形成される.これまでに,原核生物や真核生物に由来するAGOタンパク質の結晶構造解析により,4つの機能ドメインおよびリンカードメインから構成される全体構造,ガイドRNAの認識機構,および,標的RNAの認識機構および切断機構が明らかにされてきた4-8)(文献7) は(新着論文レビュー でも掲載).一方,PIWIタンパク質の構造情報はPAZドメインおよびMIDドメインにかぎられており,全長のPIWIタンパク質の立体構造は不明であった.
なお,真核生物に由来するAGOタンパク質の構造と機能については,佐々木 浩・泊 幸秀, 領域融合レビュー, 1, e001 (2012) も参照されたい.
一般的に,結晶構造解析には大腸菌や真核生物の細胞において大量に発現させた組換えタンパク質が用いられる.高等真核生物に由来するAGOタンパク質の構造解析には昆虫細胞から調製した組換え体が用いられてきたが6,8),PIWIタンパク質の組換え体の調製は困難であり構造解析の障壁となっていた.一方,カイコの卵巣に由来するBmN4培養細胞には2種類のPIWIタンパク質,SiwiおよびBmAgo3が発現しており9),抗Siwi抗体を利用して精製されたSiwiを用いて生化学的な解析が進められてきた10).そこで,BmN4細胞から内在性のSiwiを精製し結晶化に用いることを考えた.しかし,結晶化のためには解決しなければならない2つの問題が残されていた.まず,生化学的な解析には抗Siwi抗体ビーズに結合した状態のSiwiが用いられてきたが,結晶化にはSiwiのみを単離する必要があった.さらに,SiwiのN末端側に存在する約100残基の天然変性領域は結晶化を阻害することが予想された.先行研究において,ヒトに由来するAGOタンパク質であるhAgo2はガイドRNAとRISCを形成するとプロテアーゼであるサーモリシンに対し抵抗性をもつと報告されていた8).さらに,BmN4細胞に発現するSiwiは多様な配列をもつ約28塩基長のpiRNAと結合しpiRISCを形成していること,および,抗Siwi抗体はSiwiの天然変性領域を認識することがわかっていた10).これらの結果を総合し,プロテアーゼを用いてSiwiの天然変性領域を分解することにより,安定かつ結晶化する可能性の高いSiwiのコア領域を抗Siwi抗体から分離できるのではないかと考えた.
実際に,抗Siwi抗体を固定化したSepharoseビーズを用いてBmN4細胞の抽出液からSiwiを回収しサーモリシンを作用させたところ,Siwiのコア領域に対応する分子量のタンパク質がビーズから遊離した.N末端の解析の結果,これは天然変性領域が切除されたSiwiのコア領域であることが確かめられた.こうして得られたSiwiをカラムクロマトグラフィーにより高純度に精製した.変性ゲル電気泳動により,精製したSiwiには約28塩基長の内在性のpiRNAが結合しており,piRISCを形成していることが明らかにされた.
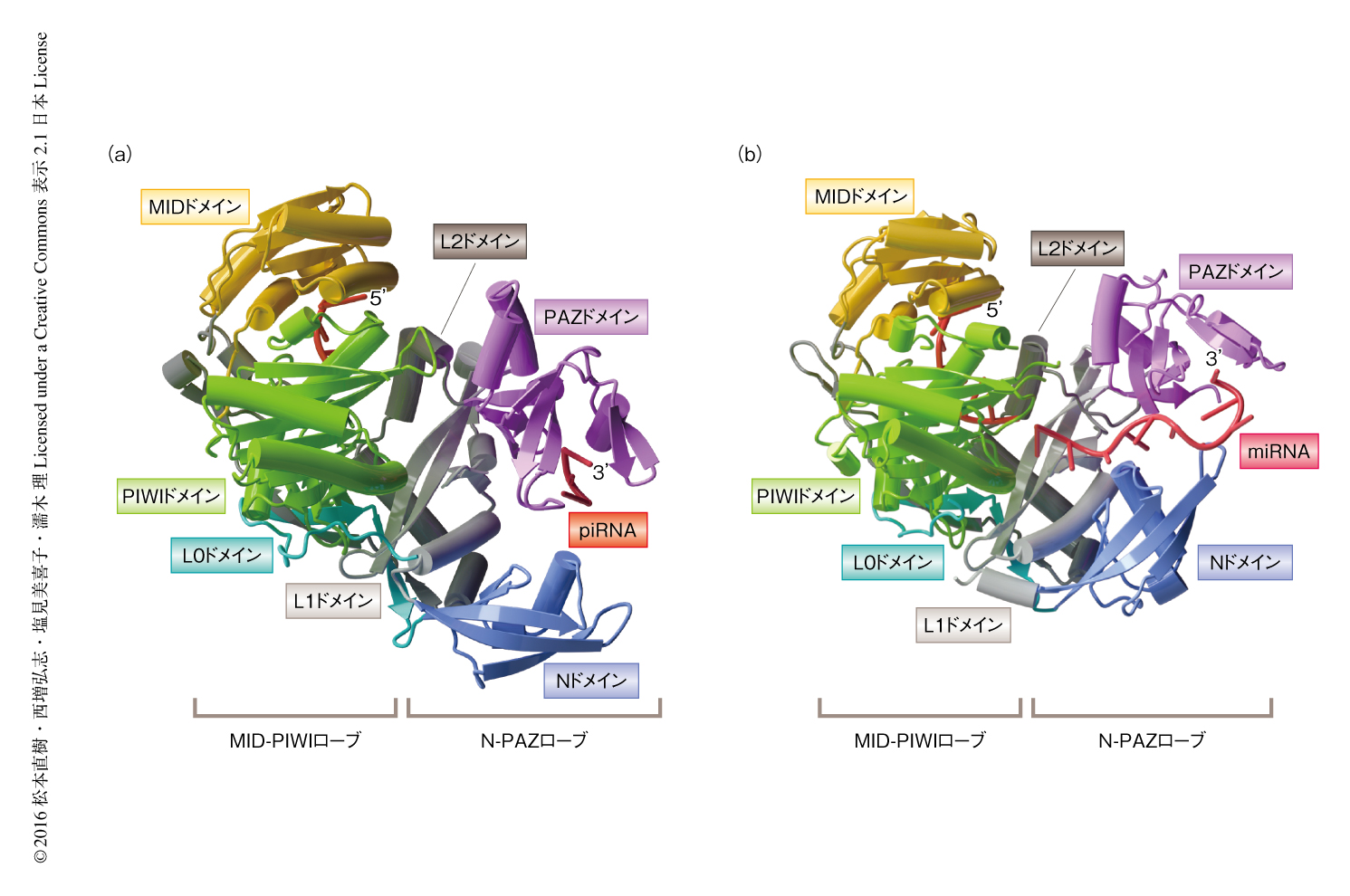
piRNAとSiwiからなるpiRISCの結晶構造を2.4Å分解能で決定した(PDB ID:5GUH,図1a).SiwiはNドメイン,PAZドメイン,MIDドメイン,PIWIドメインの4つの機能ドメインと,それらをつなぐL0ドメイン,L1ドメイン,L2ドメインのリンカードメインから構成され,N-PAZローブおよびMID-PIWIローブの2つのローブからなる構造をとっていた.piRNAの5’末端および3’末端はそれぞれMIDドメイン/PIWIドメインおよびPAZドメインと結合していた.Siwiは1位にUをもつpiRNAを好む9-12) ことと一致して,5’末端にはUに対応する電子密度が観察された.一方,piRNAの3’末端は2’-O-メチル化修飾される3,12) ことと一致して,3’末端には2’-O-メチル基に対応する電子密度が観察された.piRNAの中央領域の約20ヌクレオチドの電子密度は不明瞭だった.したがって,配列や長さが不均一な内在性のpiRNAの中央領域は柔軟な構造をとることが示唆された.
PIWIタンパク質であるSiwiの構造と,AGOタンパク質であるhAgo2の構造5)(PDB ID:4W5N,図1b)とを比較したところ,おのおののドメインの構造は似ている一方,MID-PIWIローブに対するNドメイン/PAZドメインの配置が顕著に異なることが明らかにされた.この違いは,Nドメイン/PAZドメインとL1/L2ドメインとのあいだの疎水性相互作用の違いに起因していた.このドメイン間相互作用に関与するアミノ酸残基はほかのPIWIタンパク質においても保存されていたことから,ほかのPIWIタンパク質もSiwiと類似した全体構造をもつことが示唆された.Nドメイン/PAZドメインはAGOタンパク質においてRISCの形成に関与することから,PIWIタンパク質とAGOタンパク質とのあいだのNドメイン/PAZドメインの配向の違いは両者のRISCの形成機構の違いを反映すると考えられた.
piRNAの5’末端領域はMIDドメインとPIWIドメインのあいだに結合していた.真核生物のAGOタンパク質は保存されたLysを用いてガイドRNAの5’末端のリン酸基を認識する一方,原核生物のAGOタンパク質はMg2+を介して5’末端のリン酸基を認識する4-8).予想外に,SiwiはMg2+を介してpiRNAの5’末端のリン酸基を認識していた.PIWIタンパク質において保存されたGlnがMg2+に配位していたことから,5’末端のリン酸基の認識機構はPIWIタンパク質において保存されていることが示唆された.これらの観察により,5’末端のリン酸基の認識機構に関して,PIWIタンパク質は原核生物のAGOタンパク質と似ていることが明らかにされた.piRNAの1位のUはMIDドメインと塩基に特異的な水素結合を形成しており,1位のA,G,CはMIDドメインと立体障害あるいは電荷的な反発を起こすことが予想された.これらの構造的な特徴から,piRNAの1位のUに対するSiwiの嗜好性9-12) が説明された.AGOタンパク質と結合したmiRNAと同様に,piRNAの2位~5位のシード領域はA型らせん様の構造をとり塩基を溶媒の側にむけてSiwiと結合していた.したがって,AGOタンパク質の形成するRISCと同様に,Siwiの形成するpiRISCにおいてもシード領域は標的RNAとの塩基対の形成の起点としてはたらくことが示唆された.piRNAの3’末端領域はPAZドメインにより認識され,3’末端の2’-O-メチル基は疎水性のアミノ酸残基が形成するポケットに収容されていた.変異体の解析により,piRNAの5’末端および3’末端の認識はSiwiとの結合に必須であることが明らかにされた.
AGOタンパク質のPIWIドメインはRNase Hフォールドをとり,DEDXテトラッド(XはAspあるいはHis)が形成する活性部位をもつ4,6-8).真核生物のAGOタンパク質においては,ガイドRNAとの結合により触媒テトラッドが形成される6-8).一方,高度好熱細菌Thermus thermophilusに由来するAGOタンパク質においては,ガイドDNAと標的RNAあるいは標的DNAとのあいだの塩基対の形成にともないGluが構造変化を起こし触媒テトラッドが完成する4).SiwiのもつPIWIドメインもRNase Hフォールドをとっており触媒テトラッドを形成するアミノ酸残基は保存されていた.さらに,変異体の解析により,Siwiにおいても触媒テトラッドは標的RNAの切断に必須であることが確かめられた.しかし,Siwiの結晶構造において,触媒テトラッドを形成するGluは一定の構造を形成せず活性部位から離れた位置に存在していた.したがって,T. thermophilusのAGOタンパク質と同様に,Siwiの触媒テトラッドは標的RNAとの結合にともない完成すると考えられた.
Siwiは同じくPIWIタンパク質であるBmAgo3とともにピンポンサイクルとよばれるpiRNA経路にかかわる10,11)(図2a).SiwiおよびBmAgo3はトランスポゾンに由来する転写産物を切断したのち,一方の切断産物を互いに受けわたすことによりトランスポゾンの抑制およびpiRNAの増幅を共役する.AGOタンパク質の切断産物が自発的に放出されるのとは対照的に(図2b),Siwiの切断産物は自発的には放出されず,RNAヘリカーゼであるVasaのはたらきによりBmAgo3へと効率的に受けわたされる10,11)(図2).この違いは,AGOタンパク質の切断産物は単なる分解産物にすぎないのに対し,Siwiの切断産物はpiRNAの前駆体RNAとして利用されることを反映すると考えられた.
切断産物の放出機構の違いを理解するため,SiwiあるいはAGOタンパク質のひとつhAgo2とガイドRNAと標的RNAからなる2本鎖RNAをドッキングしたモデルを作製した.ドッキングモデルから,hAgo2と結合した2本鎖RNAはNドメインと立体障害を起こすことが示唆された.したがって,hAgo2が2本鎖RNAを取り込むにはN-PAZローブが外側に開く必要があり,このN-PAZローブの外向きの動きが標的RNAの切断ののち内向きの力の生じる原動力となり,自発的な切断産物の放出を促進する可能性が考えられた.一方,SiwiのNドメインは2本鎖RNAと立体障害を起こさず相互作用することが示唆された.したがって,Siwiは切断産物と安定に結合し,切断産物の効率的な放出にはVasaのRNAヘリカーゼ活性を必要とすると推測された.
2007年,筆者らは,当時は未解明だった真核生物のArgonauteタンパク質の構造解析に着手した.いくつかの生物種に由来するAGOタンパク質およびPIWIタンパク質について昆虫細胞を用いて組換えタンパク質の調製を試みたが,高純度の試料を得ることができず結晶化はあきらめた.そのような状況のなか,2012年に真核生物のAGOタンパク質の結晶構造が3つの研究グループにより報告された6-8).3つの研究グループとも,昆虫細胞や大腸菌から調製した組換え体を結晶化しており,PIWIタンパク質の結晶構造が決定されるのも時間の問題だろうと思われた.しかし,これまで,PIWIタンパク質の組換え体の結晶構造が報告されることはなかった.今回,抗Siwi抗体とプロテアーゼによる限定分解を組み合わせることにより内在性のSiwiを精製するというトリッキーな方法がPIWIタンパク質の構造決定のブレークスルーになった.今後は,均一なガイドRNAとの複合体,ガイドRNA-標的RNAからなる2本鎖RNAとの複合体,Vasaなどの相互作用タンパク質との複合体の構造の決定が期待される.さらに,抗PIWI抗体とプロテアーゼを組み合わせた精製法はほかのPIWIタンパク質にも適用が可能と考えられる.今後,異なる生物種に由来するPIWIタンパク質の構造解析により,トランスポゾンの抑制機構の理解が進むことが期待される.
略歴:2016年 東京大学大学院理学系研究科博士課程 修了.
関心事:人体の正常と異常.
西増 弘志(Hiroshi Nishimasu)
略歴:2007年 東京大学大学院農学生命科学研究科 修了,2013年より東京大学大学院理学系研究科 助教,科学技術振興機構 さきがけ研究者 兼任.
研究テーマ:RNA-guided nucleaseの構造と機能.
関心事:減量.
塩見 美喜子(Mikiko C. Siomi)
東京大学大学院理学系研究科 教授.
研究室URL:http://www-siomilab.biochem.s.u-tokyo.ac.jp
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究室URL:http://www.nurekilab.net/
© 2016 松本直樹・西増弘志・塩見美喜子・濡木 理 Licensed under CC 表示 2.1 日本
(東京大学大学院理学系研究科 1生物科学専攻生物化学講座構造生命科学研究室,2生物科学専攻生物化学講座RNA生物学研究室)
email:西増弘志,塩見美喜子,濡木 理
DOI: 10.7875/first.author.2016.105
Crystal structure of silkworm PIWI-clade Argonaute Siwi bound to piRNA.
Naoki Matsumoto, Hiroshi Nishimasu, Kazuhiro Sakakibara, Kazumichi M. Nishida, Takamasa Hirano, Ryuichiro Ishitani, Haruhiko Siomi, Mikiko C. Siomi, Osamu Nureki
Cell, 167, 484-497.e9 (2016)
要 約
非コード小分子RNAのひとつであるpiRNAはPIWIタンパク質とpiRISCとよばれる複合体を形成し,生殖組織においてトランスポゾンの発現を抑制し遺伝情報を保護する役割をもつ.しかし,PIWIタンパク質の立体構造は不明であり,その作動機構の理解は遅れていた.今回,筆者らは,カイコのもつPIWIタンパク質のひとつであるSiwiについて,カイコの卵巣に由来する培養細胞からpiRNAと結合した状態で精製し,その結晶構造を決定することに成功した.結晶構造からSiwiによるpiRNAの認識機構が明らかにされた.さらに,SiwiとAGOタンパク質のひとつであるhAgo2との構造の比較により,両者の核酸結合チャネルに顕著な違いが見い出された.この違いはPIWIタンパク質とAGOタンパク質の機能の違いを反映すると考えられた.今回の研究の成果は,PIWIタンパク質によるトランスポゾンの抑制機構の解明にむけた第一歩となる.
はじめに
非コード小分子RNAはArgonauteタンパク質と複合体を形成し,標的となる遺伝子の発現を制御する.この機構はRNAサイレンシングとよばれる.非コード小分子RNAはArgonauteタンパク質を相補的な標的RNAへと導き,切断や転写抑制あるいは翻訳抑制をひき起こす.Argonauteタンパク質はAGOタンパク質とPIWIタンパク質の2つのクレードに分類される.AGOタンパク質は全身の組織に広く発現し,約22塩基長のsiRNA(small interfering RNA)やmiRNA(microRNA)とRISC(RNA-induced silencing complex)を形成し多岐にわたる生命現象に関与する.一方,PIWIタンパク質は動物の生殖組織に発現し,23~33塩基長のpiRNA(PIWI-interacting RNA)とpiRISC(piRNA-induced silencing complex)を形成し,トランスポゾンに由来するRNAを抑制することにより遺伝情報を次世代に正確に伝達する役割を担う1).AGOタンパク質とPIWIタンパク質では複合体の形成の機構も異なる.AGOタンパク質においては2本鎖RNAが取り込まれたのち片方のRNA鎖が放出されることによりRISCが形成される.一方,PIWIタンパク質には長い1本鎖の前駆体RNAが取り込まれたのち,Zucchini 2)(新着論文レビュー でも掲載)などのRNA切断酵素やHen1 3) などのメチル化酵素により切断あるいは修飾されることによりpiRISCが形成される.これまでに,原核生物や真核生物に由来するAGOタンパク質の結晶構造解析により,4つの機能ドメインおよびリンカードメインから構成される全体構造,ガイドRNAの認識機構,および,標的RNAの認識機構および切断機構が明らかにされてきた4-8)(文献7) は(新着論文レビュー でも掲載).一方,PIWIタンパク質の構造情報はPAZドメインおよびMIDドメインにかぎられており,全長のPIWIタンパク質の立体構造は不明であった.
なお,真核生物に由来するAGOタンパク質の構造と機能については,佐々木 浩・泊 幸秀, 領域融合レビュー, 1, e001 (2012) も参照されたい.
1.Siwiの精製
一般的に,結晶構造解析には大腸菌や真核生物の細胞において大量に発現させた組換えタンパク質が用いられる.高等真核生物に由来するAGOタンパク質の構造解析には昆虫細胞から調製した組換え体が用いられてきたが6,8),PIWIタンパク質の組換え体の調製は困難であり構造解析の障壁となっていた.一方,カイコの卵巣に由来するBmN4培養細胞には2種類のPIWIタンパク質,SiwiおよびBmAgo3が発現しており9),抗Siwi抗体を利用して精製されたSiwiを用いて生化学的な解析が進められてきた10).そこで,BmN4細胞から内在性のSiwiを精製し結晶化に用いることを考えた.しかし,結晶化のためには解決しなければならない2つの問題が残されていた.まず,生化学的な解析には抗Siwi抗体ビーズに結合した状態のSiwiが用いられてきたが,結晶化にはSiwiのみを単離する必要があった.さらに,SiwiのN末端側に存在する約100残基の天然変性領域は結晶化を阻害することが予想された.先行研究において,ヒトに由来するAGOタンパク質であるhAgo2はガイドRNAとRISCを形成するとプロテアーゼであるサーモリシンに対し抵抗性をもつと報告されていた8).さらに,BmN4細胞に発現するSiwiは多様な配列をもつ約28塩基長のpiRNAと結合しpiRISCを形成していること,および,抗Siwi抗体はSiwiの天然変性領域を認識することがわかっていた10).これらの結果を総合し,プロテアーゼを用いてSiwiの天然変性領域を分解することにより,安定かつ結晶化する可能性の高いSiwiのコア領域を抗Siwi抗体から分離できるのではないかと考えた.
実際に,抗Siwi抗体を固定化したSepharoseビーズを用いてBmN4細胞の抽出液からSiwiを回収しサーモリシンを作用させたところ,Siwiのコア領域に対応する分子量のタンパク質がビーズから遊離した.N末端の解析の結果,これは天然変性領域が切除されたSiwiのコア領域であることが確かめられた.こうして得られたSiwiをカラムクロマトグラフィーにより高純度に精製した.変性ゲル電気泳動により,精製したSiwiには約28塩基長の内在性のpiRNAが結合しており,piRISCを形成していることが明らかにされた.
2.Siwiの結晶構造
piRNAとSiwiからなるpiRISCの結晶構造を2.4Å分解能で決定した(PDB ID:5GUH,図1a).SiwiはNドメイン,PAZドメイン,MIDドメイン,PIWIドメインの4つの機能ドメインと,それらをつなぐL0ドメイン,L1ドメイン,L2ドメインのリンカードメインから構成され,N-PAZローブおよびMID-PIWIローブの2つのローブからなる構造をとっていた.piRNAの5’末端および3’末端はそれぞれMIDドメイン/PIWIドメインおよびPAZドメインと結合していた.Siwiは1位にUをもつpiRNAを好む9-12) ことと一致して,5’末端にはUに対応する電子密度が観察された.一方,piRNAの3’末端は2’-O-メチル化修飾される3,12) ことと一致して,3’末端には2’-O-メチル基に対応する電子密度が観察された.piRNAの中央領域の約20ヌクレオチドの電子密度は不明瞭だった.したがって,配列や長さが不均一な内在性のpiRNAの中央領域は柔軟な構造をとることが示唆された.
3.AGOタンパク質との構造の比較
PIWIタンパク質であるSiwiの構造と,AGOタンパク質であるhAgo2の構造5)(PDB ID:4W5N,図1b)とを比較したところ,おのおののドメインの構造は似ている一方,MID-PIWIローブに対するNドメイン/PAZドメインの配置が顕著に異なることが明らかにされた.この違いは,Nドメイン/PAZドメインとL1/L2ドメインとのあいだの疎水性相互作用の違いに起因していた.このドメイン間相互作用に関与するアミノ酸残基はほかのPIWIタンパク質においても保存されていたことから,ほかのPIWIタンパク質もSiwiと類似した全体構造をもつことが示唆された.Nドメイン/PAZドメインはAGOタンパク質においてRISCの形成に関与することから,PIWIタンパク質とAGOタンパク質とのあいだのNドメイン/PAZドメインの配向の違いは両者のRISCの形成機構の違いを反映すると考えられた.
4.piRNAの認識
piRNAの5’末端領域はMIDドメインとPIWIドメインのあいだに結合していた.真核生物のAGOタンパク質は保存されたLysを用いてガイドRNAの5’末端のリン酸基を認識する一方,原核生物のAGOタンパク質はMg2+を介して5’末端のリン酸基を認識する4-8).予想外に,SiwiはMg2+を介してpiRNAの5’末端のリン酸基を認識していた.PIWIタンパク質において保存されたGlnがMg2+に配位していたことから,5’末端のリン酸基の認識機構はPIWIタンパク質において保存されていることが示唆された.これらの観察により,5’末端のリン酸基の認識機構に関して,PIWIタンパク質は原核生物のAGOタンパク質と似ていることが明らかにされた.piRNAの1位のUはMIDドメインと塩基に特異的な水素結合を形成しており,1位のA,G,CはMIDドメインと立体障害あるいは電荷的な反発を起こすことが予想された.これらの構造的な特徴から,piRNAの1位のUに対するSiwiの嗜好性9-12) が説明された.AGOタンパク質と結合したmiRNAと同様に,piRNAの2位~5位のシード領域はA型らせん様の構造をとり塩基を溶媒の側にむけてSiwiと結合していた.したがって,AGOタンパク質の形成するRISCと同様に,Siwiの形成するpiRISCにおいてもシード領域は標的RNAとの塩基対の形成の起点としてはたらくことが示唆された.piRNAの3’末端領域はPAZドメインにより認識され,3’末端の2’-O-メチル基は疎水性のアミノ酸残基が形成するポケットに収容されていた.変異体の解析により,piRNAの5’末端および3’末端の認識はSiwiとの結合に必須であることが明らかにされた.
5.触媒テトラッド
AGOタンパク質のPIWIドメインはRNase Hフォールドをとり,DEDXテトラッド(XはAspあるいはHis)が形成する活性部位をもつ4,6-8).真核生物のAGOタンパク質においては,ガイドRNAとの結合により触媒テトラッドが形成される6-8).一方,高度好熱細菌Thermus thermophilusに由来するAGOタンパク質においては,ガイドDNAと標的RNAあるいは標的DNAとのあいだの塩基対の形成にともないGluが構造変化を起こし触媒テトラッドが完成する4).SiwiのもつPIWIドメインもRNase Hフォールドをとっており触媒テトラッドを形成するアミノ酸残基は保存されていた.さらに,変異体の解析により,Siwiにおいても触媒テトラッドは標的RNAの切断に必須であることが確かめられた.しかし,Siwiの結晶構造において,触媒テトラッドを形成するGluは一定の構造を形成せず活性部位から離れた位置に存在していた.したがって,T. thermophilusのAGOタンパク質と同様に,Siwiの触媒テトラッドは標的RNAとの結合にともない完成すると考えられた.
6.核酸結合チャネル
Siwiは同じくPIWIタンパク質であるBmAgo3とともにピンポンサイクルとよばれるpiRNA経路にかかわる10,11)(図2a).SiwiおよびBmAgo3はトランスポゾンに由来する転写産物を切断したのち,一方の切断産物を互いに受けわたすことによりトランスポゾンの抑制およびpiRNAの増幅を共役する.AGOタンパク質の切断産物が自発的に放出されるのとは対照的に(図2b),Siwiの切断産物は自発的には放出されず,RNAヘリカーゼであるVasaのはたらきによりBmAgo3へと効率的に受けわたされる10,11)(図2).この違いは,AGOタンパク質の切断産物は単なる分解産物にすぎないのに対し,Siwiの切断産物はpiRNAの前駆体RNAとして利用されることを反映すると考えられた.
切断産物の放出機構の違いを理解するため,SiwiあるいはAGOタンパク質のひとつhAgo2とガイドRNAと標的RNAからなる2本鎖RNAをドッキングしたモデルを作製した.ドッキングモデルから,hAgo2と結合した2本鎖RNAはNドメインと立体障害を起こすことが示唆された.したがって,hAgo2が2本鎖RNAを取り込むにはN-PAZローブが外側に開く必要があり,このN-PAZローブの外向きの動きが標的RNAの切断ののち内向きの力の生じる原動力となり,自発的な切断産物の放出を促進する可能性が考えられた.一方,SiwiのNドメインは2本鎖RNAと立体障害を起こさず相互作用することが示唆された.したがって,Siwiは切断産物と安定に結合し,切断産物の効率的な放出にはVasaのRNAヘリカーゼ活性を必要とすると推測された.
おわりに
2007年,筆者らは,当時は未解明だった真核生物のArgonauteタンパク質の構造解析に着手した.いくつかの生物種に由来するAGOタンパク質およびPIWIタンパク質について昆虫細胞を用いて組換えタンパク質の調製を試みたが,高純度の試料を得ることができず結晶化はあきらめた.そのような状況のなか,2012年に真核生物のAGOタンパク質の結晶構造が3つの研究グループにより報告された6-8).3つの研究グループとも,昆虫細胞や大腸菌から調製した組換え体を結晶化しており,PIWIタンパク質の結晶構造が決定されるのも時間の問題だろうと思われた.しかし,これまで,PIWIタンパク質の組換え体の結晶構造が報告されることはなかった.今回,抗Siwi抗体とプロテアーゼによる限定分解を組み合わせることにより内在性のSiwiを精製するというトリッキーな方法がPIWIタンパク質の構造決定のブレークスルーになった.今後は,均一なガイドRNAとの複合体,ガイドRNA-標的RNAからなる2本鎖RNAとの複合体,Vasaなどの相互作用タンパク質との複合体の構造の決定が期待される.さらに,抗PIWI抗体とプロテアーゼを組み合わせた精製法はほかのPIWIタンパク質にも適用が可能と考えられる.今後,異なる生物種に由来するPIWIタンパク質の構造解析により,トランスポゾンの抑制機構の理解が進むことが期待される.
文 献
- Iwasaki, Y. W., Siomi, M. C. & Siomi, H.: PIWI-interacting RNA: its biogenesis and functions. Annu. Rev. Biochem., 84, 405-433 (2015)[PubMed]
- Nishimasu, H., Ishizu, H., Saito, K. et al.: Structure and function of Zucchini endoribonuclease in piRNA biogenesis. Nature, 491, 284-287 (2012)[PubMed] [新着論文レビュー]
- Saito, K., Sakaguchi, Y., Suzuki, T. et al.: Pimet, the Drosophila homolog of HEN1, mediates 2’-O-methylation of Piwi-interacting RNAs at their 3’ ends. Genes Dev., 21, 1603-1608 (2007)[PubMed]
- Sheng, G., Zhao, H., Wang, J. et al.: Structure-based cleavage mechanism of Thermus thermophilus Argonaute DNA guide strand-mediated DNA target cleavage. Proc. Natl. Acad. Sci. USA, 111, 652-657 (2014)[PubMed]
- Schirle, N. T., Sheu-Gruttadauria, J. & MacRae, I. J.: Structural basis for microRNA targeting. Science, 346, 608-613 (2014)[PubMed]
- Schirle, N. T. & MacRae, I. J.: The crystal structure of human Argonaute2. Science, 336, 1037-1040 (2012)[PubMed]
- Nakanishi, K., Weinberg, D. E., Bartel, D. P. et al.: Structure of yeast Argonaute with guide RNA. Nature, 486, 368-374 (2012)[PubMed] [新着論文レビュー]
- Elkayam, E., Kuhn, C. D., Tocilj, A. et al.: The structure of human argonaute-2 in complex with miR-20a. Cell, 150, 100-110 (2012)[PubMed]
- Kawaoka, S., Hayashi, N., Suzuki, Y. et al.: The Bombyx ovary-derived cell line endogenously expresses PIWI/PIWI-interacting RNA complexes. RNA, 15, 1258-1264 (2009)[PubMed]
- Nishida, K. M., Iwasaki, Y. W., Murota, Y. et al.: Respective functions of two distinct Siwi complexes assembled during PIWI-interacting RNA biogenesis in Bombyx germ cells. Cell Rep., 10, 193-203 (2015)[PubMed]
- Xiol, J., Spinelli, P., Laussmann, M. A. et al.: RNA clamping by Vasa assembles a piRNA amplifier complex on transposon transcripts. Cell, 157, 1698-1711 (2014)[PubMed]
- Kawaoka, S., Izumi, N., Katsuma, S. et al.: 3’ end formation of PIWI-interacting RNAs in vitro. Mol. Cell, 43, 1015-1022 (2011)[PubMed] [新着論文レビュー]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2016年 東京大学大学院理学系研究科博士課程 修了.
関心事:人体の正常と異常.
西増 弘志(Hiroshi Nishimasu)
略歴:2007年 東京大学大学院農学生命科学研究科 修了,2013年より東京大学大学院理学系研究科 助教,科学技術振興機構 さきがけ研究者 兼任.
研究テーマ:RNA-guided nucleaseの構造と機能.
関心事:減量.
塩見 美喜子(Mikiko C. Siomi)
東京大学大学院理学系研究科 教授.
研究室URL:http://www-siomilab.biochem.s.u-tokyo.ac.jp
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究室URL:http://www.nurekilab.net/
© 2016 松本直樹・西増弘志・塩見美喜子・濡木 理 Licensed under CC 表示 2.1 日本