ZucchiniはpiRNAの産生に必須のエンドリボヌクレアーゼである
西増弘志・石津大嗣・塩見美喜子・濡木 理
(東京大学大学院理学系研究科 生物化学専攻)
email:西増弘志,石津大嗣,塩見美喜子,濡木 理
DOI: 10.7875/first.author.2012.124
Structure and function of Zucchini endoribonuclease in piRNA biogenesis.
Hiroshi Nishimasu, Hirotsugu Ishizu, Kuniaki Saito, Satoshi Fukuhara, Miharu K. Kamatani, Luc Bonnefond, Naoki Matsumoto, Tomohiro Nishizawa, Keita Nakanaga, Junken Aoki, Ryuichiro Ishitani, Haruhiko Siomi, Mikiko C. Siomi, Osamu Nureki
Nature, 491, 284-287 (2012)
piRNAはPIWIサブファミリータンパク質と結合し生殖細胞のゲノムをトランスポゾンによる損傷から保護する役割を担っている.約30塩基長の成熟型piRNAは,特定のゲノム領域から転写される長い一本鎖RNAがなんらかのリボヌクレアーゼにより切断されることによって産生されると考えられていた.しかし,piRNAの産生にかかわるリボヌクレアーゼの正体は長いあいだ不明であった.今回,筆者らは,ZucchiniがpiRNAの産生に必須なリボヌクレアーゼであることを明らかにした.X線結晶構造解析により,Zucchiniは一本鎖RNAが結合するのに適した形状の酵素活性部位をもつことが明らかになった.生化学的な解析により,Zucchiniは一本鎖RNAを切断する酵素活性をもつことが明らかになった.さらに,細胞生物学的な解析により,Zucchiniのもつリボヌクレアーゼ活性はpiRNAの産生とトランスポゾンの抑制に必須であることが確かめられた.以上の結果から,Zucchiniは動物の生殖細胞においてpiRNAの産生にかかわるエンドリボヌクレアーゼであることが示された.
動物のゲノムにはトランスポゾンとよばれる“動く遺伝子”が存在する.遺伝情報を次世代に正確に受け継ぐ必要のある生殖細胞では,piRNA(PIWI-interacting RNA)とよばれる約30塩基長のsmall RNAが生殖細胞に特異的なArgonauteであるPIWIサブファミリータンパク質と結合し,生殖細胞のゲノムをトランスポゾンによる損傷から保護している1).piRNAは,piRNAクラスターとよばれる特定のゲノム領域から転写される長い一本鎖RNAがなんらかのリボヌクレアーゼにより切断されることによって産生されると考えられていた2-5).また,piRNA経路にはZucchini(Zuc)をふくむ複数のタンパク質が関与している6,7).
zuc遺伝子は,もともと雌不妊の原因遺伝子としてショウジョウバエの遺伝学的なスクリーニングにより同定された8).ショウジョウバエやマウスにおいてZucは生殖細胞に発現しており,ミトコンドリア外膜に局在する6,9-11).zuc遺伝子の変異はpiRNA産生の低下,トランスポゾンの脱抑制,精子形成や卵形成の異常をひき起こす3,5-7,10,12).ZucはN末端にあるミトコンドリア局在シグナル,膜貫通ヘリックス,および,ホスホリパーゼDスーパーファミリーに属する触媒ドメインからなる.ホスホリパーゼDスーパーファミリータンパク質はHKDモチーフを共通にもち,基質のホスホジエステル結合を加水分解する.その基質特異性は多様で,脂質を基質とするホスホリパーゼDや核酸を基質とするNuc 13) などが含まれる(Nucはリパーゼではなくヌクレアーゼであるが,アミノ酸配列にもとづきホスホリパーゼDスーパーファミリーに分類されている).ZucはNucともっとも高い配列同一性(26%)をもつことから,ZucはpiRNAの産生にかかわるリボヌクレアーゼの候補と考えられていた12).一方,マウスのZucホモログであるMmZuc(Pld6やMitoPLDともよばれる)はミトコンドリアに存在する脂質カルジオリピンを加水分解しシグナル分子であるホスファチジン酸を産生することから9),Zucは脂質シグナルを介して間接的にpiRNAの産生にかかわるというモデルも提唱されていた10,11).しかし,Zucのもつヌクレアーゼ活性をていねいに調べた報告はなく,Zucがリボヌクレアーゼとして直接的にpiRNAの産生にかかわる可能性も残されていた.
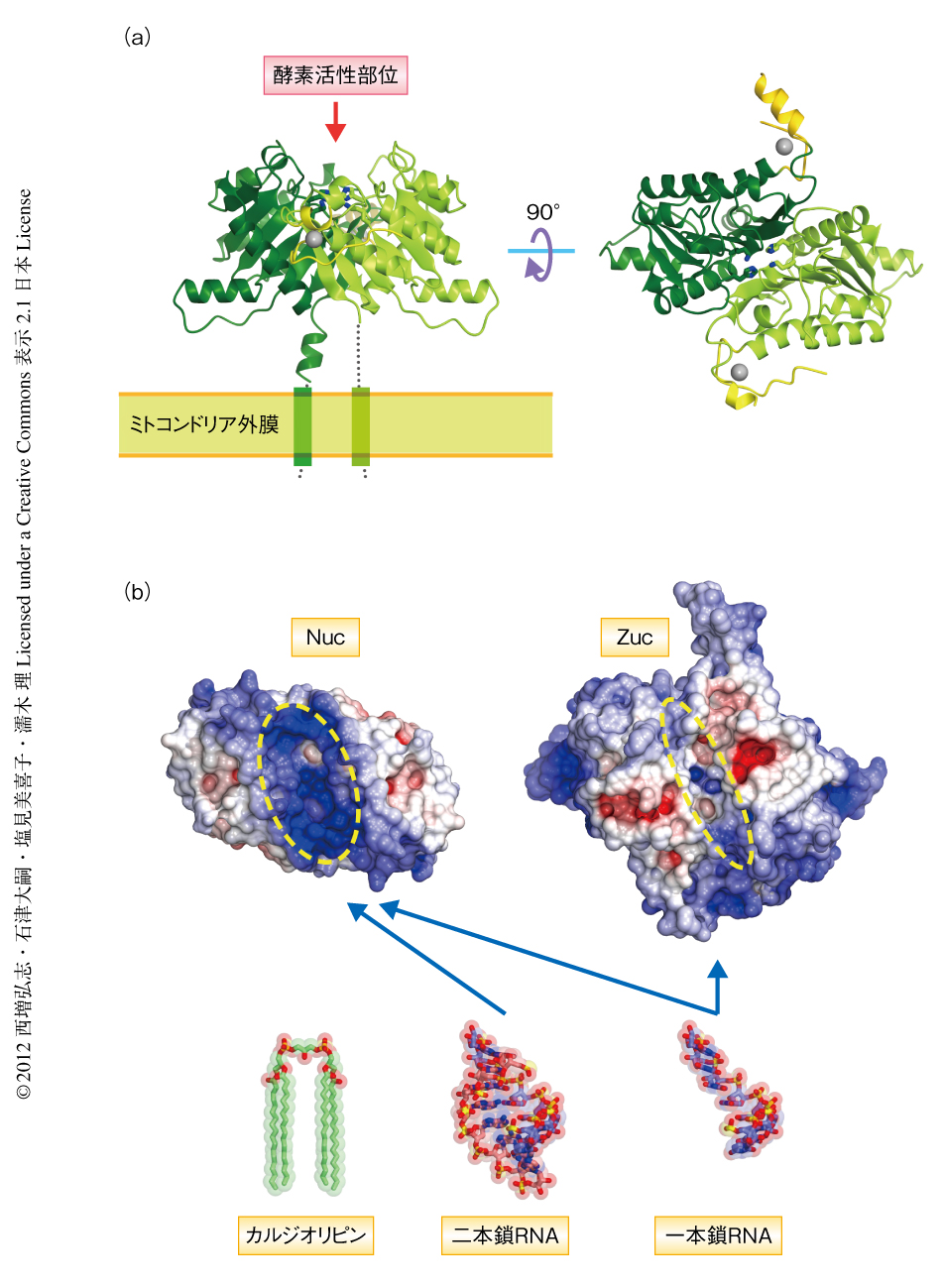
Zucの機能解明の第一歩として,ショウジョウバエに由来するZuc(DmZuc)のX線結晶構造解析を行った.N末端のミトコンドリア局在シグナルと膜貫通ヘリックスを削除したDmZuc(残基41~253)の結晶構造を1.75Å分解能で決定した(図1a).DmZucは触媒ドメイン(残基49~62,残基89~245)とZn2+結合ドメイン(残基63~88)からなり,二量体を形成していた.触媒ドメインは8つのβストランドからなるβシートとその両側をかこむαヘリックスからなり,NucなどのホスホリパーゼDスーパーファミリータンパク質とよく似ていた.HKDモチーフを構成する3つの残基のうち,His169とLys171は酵素活性部位を形成し,Asp176は立体構造の安定化に寄与していた.Cys63,His67,Cys83,Cys88はZn2+と結合しZn2+結合ドメインを形成していた.二次構造予測から,DmZucは既知のホスホリパーゼDスーパーファミリータンパク質とは異なり,触媒ドメインの1番目のβストランドとαヘリックスとのあいだに30残基の一定の構造をとらないディスオーダー領域をもつことが示唆されていたが,この領域がZn2+結合ドメインを形成していたことは予想外の発見だった.Zn2+結合ドメインのほとんどの部分は結晶構造においても一定の構造をとっておらず,この領域は実際にディスオーダー領域であると考えられた.Zn2+と結合すると考えられた残基はほかの動物においても保存されていたことから,Zn2+結合ドメインはZucの構造的な特徴であると考えられた.
酵素活性部位は二量体の界面に存在し,保存された残基(Tyr112,His169,Lys171,Ser204,Asn206,Trp207,Thr208,Asn215,Glu217)により形成されていた.これらの残基はNucにおいてもよく保存されており同様の立体配置をとっている.したがって,ZucはNucと同様の反応機構により基質を加水分解すると考えられた.しかし,DmZucとNucの酵素活性部位の形状には顕著な違いが存在した(図1b).Nucは一本鎖の核酸と二本鎖の核酸の両方を切断し,二本鎖の核酸が結合するのに適した広い溝からなる酵素活性部位をもつ13)(PDB ID:1BYR).一方,DmZucの酵素活性部位はNucに比べ狭く,二本鎖の核酸は結合できないようにみえた.さらに,マウスのZucホモログであるMmZucはカルジオリピンを加水分解することが報告されていたが9),DmZucの酵素活性部位にはカルジオリピンのようなかさ高い脂質分子は結合できないようにみえた.DmZucの酵素活性部位の反対側にはN末端にあるヘリックスが位置し,正に帯電した分子表面が存在していた.この構造的な特徴は,DmZucはミトコンドリア外膜に局在するというモデルとよく一致していた6,9-11).以上の結晶構造解析の結果,Zucはミトコンドリア外膜において一本鎖の核酸を切断するヌクレアーゼであることが示唆された.
Zucがヌクレアーゼであるかどうかを検証するため,結晶化純度にまで精製したDmZucと放射性同位体により標識した合成RNAを用いてリボヌクレアーゼ活性を測定した.その結果,DmZucは一本鎖RNAを切断することがわかった.一方,酵素活性部位の変異体(4種類)はいずれもリボヌクレアーゼ活性を示さなかった.この結果から,酵素活性部位にある保存された残基はリボヌクレアーゼ活性に必須であること,ならびに,リボヌクレアーゼ活性は大腸菌に由来する夾雑タンパク質によるものではないと考えられた.Zn2+結合ドメインを削除したDmZuc変異体のリボヌクレアーゼ活性は著しく低下していたことから,Zn2+結合ドメインはリボヌクレアーゼ活性に重要であることがわかった.一般的なジンクフィンガーと同様に,Zn2+結合ドメインは基質であるRNAとの結合にかかわると考えられた.DmZucは環状の一本鎖RNAを切断したことから,エンドヌクレアーゼであると考えられた.酵素活性部位の形状から予想されたように,DmZucは二本鎖RNAを切断しなかった.さまざまな配列をもつ一本鎖RNAを用いて切断実験を行ったところ,DmZucは厳密な塩基特異性をもたないことが示唆された.DmZucは一本鎖DNAも切断したが,生理的には一本鎖RNAの切断活性が重要だと考えられた.つぎに,DmZucにより切断された一本鎖RNAの5’末端の構造を調べた.5’末端にリン酸基をもつ一本鎖RNAに特異的な末端エキソヌクレアーゼはDmZucによる切断産物を分解したのに対し,これをアルカリホスファターゼにより処理したものは分解しなかった.したがって,DmZucにより切断された一本鎖RNAは5’末端にリン酸基をもつと考えられた.PIWIサブファミリータンパク質に結合した成熟型piRNAは5’末端にリン酸基をもつ2,14).したがって,DmZucはpiRNAの5’末端を規定するリボヌクレアーゼであることが示唆された.最後に,Zucがカルジオリピンを分解するかどうかを検証した.DmZucとカルジオリピンをインキュベーションしたのち反応溶液の脂質を質量分析により定量したところ,カルジオリピンの減少およびそれにともなうホスファチジン酸の増加は観察されなかった.この結果から,DmZucはカルジオリピンに対する分解活性をもたないことが示唆された.さらに,マウスのZucホモログであるMmZuc(残基35~221)もリボヌクレアーゼ活性を示したが,カルジオリピン分解活性は示さなかった.これらの結果は,さきにも述べた,MmZucはカルジオリピン分解酵素であるという過去の報告と一致しなかった9).この理由は不明だが,酵素活性測定に用いたタンパク質の純度の違いによるのかもしれない.以上の生化学的な解析の結果,Zucは一本鎖の核酸に特異的なエンドヌクレアーゼであることが明らかになった.
ショウジョウバエの卵巣の体細胞に由来する培養細胞を用いて,DmZucのリボヌクレアーゼ活性がトランスポゾンの抑制にかかわっているかどうかを検証した.この細胞に発現する内在性のDmZucをRNAiによりノックダウンしたところ,piRNA中間体の蓄積と成熟型piRNAの減少,および,それにともなうトランスポゾンの発現の上昇が観察された.そこで,内在性のDmZucをノックダウンした卵巣の体細胞に由来する培養細胞にRNAiに耐性となる変異をもつ野生型DmZucを発現させ,トランスポゾンの発現量を定量RT-PCR法により定量したところ,トランスポゾンの発現は抑制されることがわかった.一方,野生型DmZucの代わりに酵素活性部位の変異体(8種類)を発現させてもトランスポゾンの発現は抑制されなかった.Zn2+に結合するCys63あるいはHis67をAlaに置換した変異体や,Zn2+結合ドメインを欠損した変異体はトランスポゾンの発現を抑制した.これは,酵素活性部位の変異体はリボヌクレアーゼ活性をまったく示さなかったのに対し,Zn2+結合ドメインを欠損した変異体はわずかにリボヌクレアーゼ活性を保持していたためであると考えられた.酵素活性部位に存在する保存された残基His169,Lys171,Asn206,Asn215のそれぞれをAlaに置換した変異体はリボヌクレアーゼ活性およびトランスポゾン抑制能の両方を示さなかったことから,DmZucのリボヌクレアーゼ活性はトランスポゾンの抑制に必須であると考えられた.さらに,内在性のDmZucをノックダウンした卵巣の体細胞に由来する培養細胞に野生型DmZucを発現させると,piRNA中間体が減少し成熟型piRNAが増加した.一方,酵素活性部位の変異体を発現させてもpiRNAの産生は回復しなかった.したがって,Zucのリボヌクレアーゼ活性はpiRNAの産生にかかわることが示唆された.
今回の結果を総合すると,ZucはpiRNAの産生にかかわるエンドリボヌクレアーゼであると考えられた.ショウジョウバエのもつPIWIサブファミリータンパク質のひとつであるPiwiに結合した成熟型piRNAの1番目の塩基はUにかたよっており2,14),Argonauteの結晶構造からもPiwiはpiRNAの5’末端の塩基を認識していることが示唆されている15).今回の生化学的な解析の結果から,Zucは厳密な塩基特異性をもたずに一本鎖RNAを切断し,5’末端のリン酸基を産生することが示された.したがって,ZucはpiRNA中間体を配列に非依存的に切断することにより5’末端にリン酸基をもつpiRNA断片を産生し,Piwiはそのうち1番目の塩基がUのpiRNA断片を選択的に取り込んでいるという可能性が考えられた.そののち,PiwiからはみだしたpiRNAの3’末端が別のヌクレアーゼ16) によりトリミングされることで,成熟型のpiRNAが産生されると考えられた(図2).
この研究には多くの紆余曲折があった.二次構造予測を行ってディスオーダー領域を削除するのは結晶構造解析の定石のひとつである.そこで,DmZucの二次構造予測を行いさまざまなコンストラクトを検討したところ,膜貫通ヘリックスの直後にあるディスオーダー領域(さきに述べたとおり,この領域はZn2+結合ドメインに対応し,この直前にある1本のβストランドが二量体化に必須だった)までを削除したコンストラクトは大腸菌でよく発現し高純度に精製できることがわかった.また,結晶化スクリーニングを行うなかで,C末端にあるディスオーダー領域が結晶化に影響をあたえていることがわかったので,試行錯誤のすえ,DmZuc(残基89~250)の結晶構造を決定することに成功した.しかし,このDmZuc(残基89~250)はNucと異なり二量体を形成していなかった.あとになって,生化学的な解析からこのDmZuc単量体はリボヌクレアーゼ活性をもたないことが判明し,DmZucの二量体化は酵素活性に必須であることが明らかになった.DmZucとNucの結晶構造を比較したところ,DmZuc(残基89~250)ではNucにおいて二量体化に寄与しているβストランドがひとつ欠けていることがわかった.二次構造予測の結果からも,DmZucは膜貫通ヘリックスとディスオーダー領域とのあいだに1本のβストランドをもつことが予想された.そこで,βストランドをふくむDmZuc(残基41~253)を大腸菌で発現させ,4段階のカラムクロマトグラフィーにより精製した.ゲルろ過の結果からも,このDmZuc(残基41~253)は二量体を形成していることが示唆された.しかしながら,その発現量はきわめて低く,十分に結晶化スクリーニングを行うことができなかった(10リットルの大腸菌培養液から約0.02 mgしか精製できなかった).しかし,幸運なことに,機能解析のため変異体を調製している過程で,Lys171をAlaに置換した変異をもつDmZuc(残基41~253)は大腸菌においてよく発現することを見い出した(この変異体のほかの変異体はどれも発現量が低く,酵素活性測定のためそれぞれ5リットルの大腸菌培養液から精製した).このK171A変異体を用いて結晶化スクリーニングをやり直しその結晶構造を決定したところ,この変異体はNucと同様に二量体を形成していることがわかった.しかし,K171A変異体の酵素活性部位は負に帯電しており核酸が結合しそうにはみえなかった.たった1残基の置換により構造が変化しているとは思っていなかったが,野生型のDmZucも同様の結晶化条件により結晶化することができたので,念のためその結晶構造を決定した.その結果,驚くべきことに,野生型とK171A変異体では酵素活性部位においてループの構造が大きく変化しており,野生型DmZucは正に帯電した酵素活性部位をもつことがわかった.
米国の研究グループによる同様の論文も,Nature誌に同時に掲載された17).この研究グループも生化学的な解析を行い,マウスのZucホモログMmZucは一本鎖の核酸を切断する一方で,カルジオリピンを切断しないことを示していた.さらに,このMmZucの結晶構造を決定し,MmZucのもつ酵素活性部位はNucに比べ狭いことに着目し,Zucは一本鎖の核酸に特異的なリボヌクレアーゼであると結論づけていた.2つの独立した研究が同様の結論にいたったことからも,Zucはリボヌクレアーゼとして直接的にpiRNAの産生にかかわると考えられた.実は,この研究の裏にはこの米国の研究グループとのし烈な競争があった.筆者らが“正しい”結晶構造にたどりつき,さらに,構造解析と同時並行で進んでいた生化学的な解析によりDmZucがリボヌクレアーゼであることを見い出したのは,論文投稿の3週間前のことだった.
略歴:2007年 東京大学大学院農学生命科学研究科 修了,2010年より東京大学大学院理学系研究科 特任助教.
研究テーマ:RNAサイレンシング,疾患にかかわるタンパク質.
抱負:立体構造から新しいコンセプトを創出していきたい.
石津 大嗣(Hirotsugu Ishizu)
略歴:2012年 慶應義塾大学大学院医学研究科 修了,同年より 東京大学大学院理学系研究科 助教.
研究テーマ:RNAサイレンシング.
抱負:piRNAによる遺伝子発現抑制の作用機構を明らかにする.
塩見 美喜子(Mikiko C. Siomi)
東京大学大学院理学系研究科 教授.
研究テーマ:RNAメタボリズム,RNAサイレンシング.
抱負:RNA研究をとおして生命のしくみを分子レベルで理解したい.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究テーマ:遺伝暗号の翻訳,膜輸送,自然炎症.
抱負:細胞の生理機構を原子分解能で解明したい.
研究室URL:http://www.nurekilab.net/
© 2012 西増弘志・石津大嗣・塩見美喜子・濡木 理 Licensed under CC 表示 2.1 日本
(東京大学大学院理学系研究科 生物化学専攻)
email:西増弘志,石津大嗣,塩見美喜子,濡木 理
DOI: 10.7875/first.author.2012.124
Structure and function of Zucchini endoribonuclease in piRNA biogenesis.
Hiroshi Nishimasu, Hirotsugu Ishizu, Kuniaki Saito, Satoshi Fukuhara, Miharu K. Kamatani, Luc Bonnefond, Naoki Matsumoto, Tomohiro Nishizawa, Keita Nakanaga, Junken Aoki, Ryuichiro Ishitani, Haruhiko Siomi, Mikiko C. Siomi, Osamu Nureki
Nature, 491, 284-287 (2012)
要 約
piRNAはPIWIサブファミリータンパク質と結合し生殖細胞のゲノムをトランスポゾンによる損傷から保護する役割を担っている.約30塩基長の成熟型piRNAは,特定のゲノム領域から転写される長い一本鎖RNAがなんらかのリボヌクレアーゼにより切断されることによって産生されると考えられていた.しかし,piRNAの産生にかかわるリボヌクレアーゼの正体は長いあいだ不明であった.今回,筆者らは,ZucchiniがpiRNAの産生に必須なリボヌクレアーゼであることを明らかにした.X線結晶構造解析により,Zucchiniは一本鎖RNAが結合するのに適した形状の酵素活性部位をもつことが明らかになった.生化学的な解析により,Zucchiniは一本鎖RNAを切断する酵素活性をもつことが明らかになった.さらに,細胞生物学的な解析により,Zucchiniのもつリボヌクレアーゼ活性はpiRNAの産生とトランスポゾンの抑制に必須であることが確かめられた.以上の結果から,Zucchiniは動物の生殖細胞においてpiRNAの産生にかかわるエンドリボヌクレアーゼであることが示された.
はじめに
動物のゲノムにはトランスポゾンとよばれる“動く遺伝子”が存在する.遺伝情報を次世代に正確に受け継ぐ必要のある生殖細胞では,piRNA(PIWI-interacting RNA)とよばれる約30塩基長のsmall RNAが生殖細胞に特異的なArgonauteであるPIWIサブファミリータンパク質と結合し,生殖細胞のゲノムをトランスポゾンによる損傷から保護している1).piRNAは,piRNAクラスターとよばれる特定のゲノム領域から転写される長い一本鎖RNAがなんらかのリボヌクレアーゼにより切断されることによって産生されると考えられていた2-5).また,piRNA経路にはZucchini(Zuc)をふくむ複数のタンパク質が関与している6,7).
zuc遺伝子は,もともと雌不妊の原因遺伝子としてショウジョウバエの遺伝学的なスクリーニングにより同定された8).ショウジョウバエやマウスにおいてZucは生殖細胞に発現しており,ミトコンドリア外膜に局在する6,9-11).zuc遺伝子の変異はpiRNA産生の低下,トランスポゾンの脱抑制,精子形成や卵形成の異常をひき起こす3,5-7,10,12).ZucはN末端にあるミトコンドリア局在シグナル,膜貫通ヘリックス,および,ホスホリパーゼDスーパーファミリーに属する触媒ドメインからなる.ホスホリパーゼDスーパーファミリータンパク質はHKDモチーフを共通にもち,基質のホスホジエステル結合を加水分解する.その基質特異性は多様で,脂質を基質とするホスホリパーゼDや核酸を基質とするNuc 13) などが含まれる(Nucはリパーゼではなくヌクレアーゼであるが,アミノ酸配列にもとづきホスホリパーゼDスーパーファミリーに分類されている).ZucはNucともっとも高い配列同一性(26%)をもつことから,ZucはpiRNAの産生にかかわるリボヌクレアーゼの候補と考えられていた12).一方,マウスのZucホモログであるMmZuc(Pld6やMitoPLDともよばれる)はミトコンドリアに存在する脂質カルジオリピンを加水分解しシグナル分子であるホスファチジン酸を産生することから9),Zucは脂質シグナルを介して間接的にpiRNAの産生にかかわるというモデルも提唱されていた10,11).しかし,Zucのもつヌクレアーゼ活性をていねいに調べた報告はなく,Zucがリボヌクレアーゼとして直接的にpiRNAの産生にかかわる可能性も残されていた.
1.ZucのX線結晶構造
Zucの機能解明の第一歩として,ショウジョウバエに由来するZuc(DmZuc)のX線結晶構造解析を行った.N末端のミトコンドリア局在シグナルと膜貫通ヘリックスを削除したDmZuc(残基41~253)の結晶構造を1.75Å分解能で決定した(図1a).DmZucは触媒ドメイン(残基49~62,残基89~245)とZn2+結合ドメイン(残基63~88)からなり,二量体を形成していた.触媒ドメインは8つのβストランドからなるβシートとその両側をかこむαヘリックスからなり,NucなどのホスホリパーゼDスーパーファミリータンパク質とよく似ていた.HKDモチーフを構成する3つの残基のうち,His169とLys171は酵素活性部位を形成し,Asp176は立体構造の安定化に寄与していた.Cys63,His67,Cys83,Cys88はZn2+と結合しZn2+結合ドメインを形成していた.二次構造予測から,DmZucは既知のホスホリパーゼDスーパーファミリータンパク質とは異なり,触媒ドメインの1番目のβストランドとαヘリックスとのあいだに30残基の一定の構造をとらないディスオーダー領域をもつことが示唆されていたが,この領域がZn2+結合ドメインを形成していたことは予想外の発見だった.Zn2+結合ドメインのほとんどの部分は結晶構造においても一定の構造をとっておらず,この領域は実際にディスオーダー領域であると考えられた.Zn2+と結合すると考えられた残基はほかの動物においても保存されていたことから,Zn2+結合ドメインはZucの構造的な特徴であると考えられた.
酵素活性部位は二量体の界面に存在し,保存された残基(Tyr112,His169,Lys171,Ser204,Asn206,Trp207,Thr208,Asn215,Glu217)により形成されていた.これらの残基はNucにおいてもよく保存されており同様の立体配置をとっている.したがって,ZucはNucと同様の反応機構により基質を加水分解すると考えられた.しかし,DmZucとNucの酵素活性部位の形状には顕著な違いが存在した(図1b).Nucは一本鎖の核酸と二本鎖の核酸の両方を切断し,二本鎖の核酸が結合するのに適した広い溝からなる酵素活性部位をもつ13)(PDB ID:1BYR).一方,DmZucの酵素活性部位はNucに比べ狭く,二本鎖の核酸は結合できないようにみえた.さらに,マウスのZucホモログであるMmZucはカルジオリピンを加水分解することが報告されていたが9),DmZucの酵素活性部位にはカルジオリピンのようなかさ高い脂質分子は結合できないようにみえた.DmZucの酵素活性部位の反対側にはN末端にあるヘリックスが位置し,正に帯電した分子表面が存在していた.この構造的な特徴は,DmZucはミトコンドリア外膜に局在するというモデルとよく一致していた6,9-11).以上の結晶構造解析の結果,Zucはミトコンドリア外膜において一本鎖の核酸を切断するヌクレアーゼであることが示唆された.
2.Zucは一本鎖RNAを切断するエンドヌクレアーゼである
Zucがヌクレアーゼであるかどうかを検証するため,結晶化純度にまで精製したDmZucと放射性同位体により標識した合成RNAを用いてリボヌクレアーゼ活性を測定した.その結果,DmZucは一本鎖RNAを切断することがわかった.一方,酵素活性部位の変異体(4種類)はいずれもリボヌクレアーゼ活性を示さなかった.この結果から,酵素活性部位にある保存された残基はリボヌクレアーゼ活性に必須であること,ならびに,リボヌクレアーゼ活性は大腸菌に由来する夾雑タンパク質によるものではないと考えられた.Zn2+結合ドメインを削除したDmZuc変異体のリボヌクレアーゼ活性は著しく低下していたことから,Zn2+結合ドメインはリボヌクレアーゼ活性に重要であることがわかった.一般的なジンクフィンガーと同様に,Zn2+結合ドメインは基質であるRNAとの結合にかかわると考えられた.DmZucは環状の一本鎖RNAを切断したことから,エンドヌクレアーゼであると考えられた.酵素活性部位の形状から予想されたように,DmZucは二本鎖RNAを切断しなかった.さまざまな配列をもつ一本鎖RNAを用いて切断実験を行ったところ,DmZucは厳密な塩基特異性をもたないことが示唆された.DmZucは一本鎖DNAも切断したが,生理的には一本鎖RNAの切断活性が重要だと考えられた.つぎに,DmZucにより切断された一本鎖RNAの5’末端の構造を調べた.5’末端にリン酸基をもつ一本鎖RNAに特異的な末端エキソヌクレアーゼはDmZucによる切断産物を分解したのに対し,これをアルカリホスファターゼにより処理したものは分解しなかった.したがって,DmZucにより切断された一本鎖RNAは5’末端にリン酸基をもつと考えられた.PIWIサブファミリータンパク質に結合した成熟型piRNAは5’末端にリン酸基をもつ2,14).したがって,DmZucはpiRNAの5’末端を規定するリボヌクレアーゼであることが示唆された.最後に,Zucがカルジオリピンを分解するかどうかを検証した.DmZucとカルジオリピンをインキュベーションしたのち反応溶液の脂質を質量分析により定量したところ,カルジオリピンの減少およびそれにともなうホスファチジン酸の増加は観察されなかった.この結果から,DmZucはカルジオリピンに対する分解活性をもたないことが示唆された.さらに,マウスのZucホモログであるMmZuc(残基35~221)もリボヌクレアーゼ活性を示したが,カルジオリピン分解活性は示さなかった.これらの結果は,さきにも述べた,MmZucはカルジオリピン分解酵素であるという過去の報告と一致しなかった9).この理由は不明だが,酵素活性測定に用いたタンパク質の純度の違いによるのかもしれない.以上の生化学的な解析の結果,Zucは一本鎖の核酸に特異的なエンドヌクレアーゼであることが明らかになった.
3.Zucのリボヌクレアーゼ活性はpiRNAの産生とトランスポゾンの抑制にかかわる
ショウジョウバエの卵巣の体細胞に由来する培養細胞を用いて,DmZucのリボヌクレアーゼ活性がトランスポゾンの抑制にかかわっているかどうかを検証した.この細胞に発現する内在性のDmZucをRNAiによりノックダウンしたところ,piRNA中間体の蓄積と成熟型piRNAの減少,および,それにともなうトランスポゾンの発現の上昇が観察された.そこで,内在性のDmZucをノックダウンした卵巣の体細胞に由来する培養細胞にRNAiに耐性となる変異をもつ野生型DmZucを発現させ,トランスポゾンの発現量を定量RT-PCR法により定量したところ,トランスポゾンの発現は抑制されることがわかった.一方,野生型DmZucの代わりに酵素活性部位の変異体(8種類)を発現させてもトランスポゾンの発現は抑制されなかった.Zn2+に結合するCys63あるいはHis67をAlaに置換した変異体や,Zn2+結合ドメインを欠損した変異体はトランスポゾンの発現を抑制した.これは,酵素活性部位の変異体はリボヌクレアーゼ活性をまったく示さなかったのに対し,Zn2+結合ドメインを欠損した変異体はわずかにリボヌクレアーゼ活性を保持していたためであると考えられた.酵素活性部位に存在する保存された残基His169,Lys171,Asn206,Asn215のそれぞれをAlaに置換した変異体はリボヌクレアーゼ活性およびトランスポゾン抑制能の両方を示さなかったことから,DmZucのリボヌクレアーゼ活性はトランスポゾンの抑制に必須であると考えられた.さらに,内在性のDmZucをノックダウンした卵巣の体細胞に由来する培養細胞に野生型DmZucを発現させると,piRNA中間体が減少し成熟型piRNAが増加した.一方,酵素活性部位の変異体を発現させてもpiRNAの産生は回復しなかった.したがって,Zucのリボヌクレアーゼ活性はpiRNAの産生にかかわることが示唆された.
4.piRNAの産生モデル
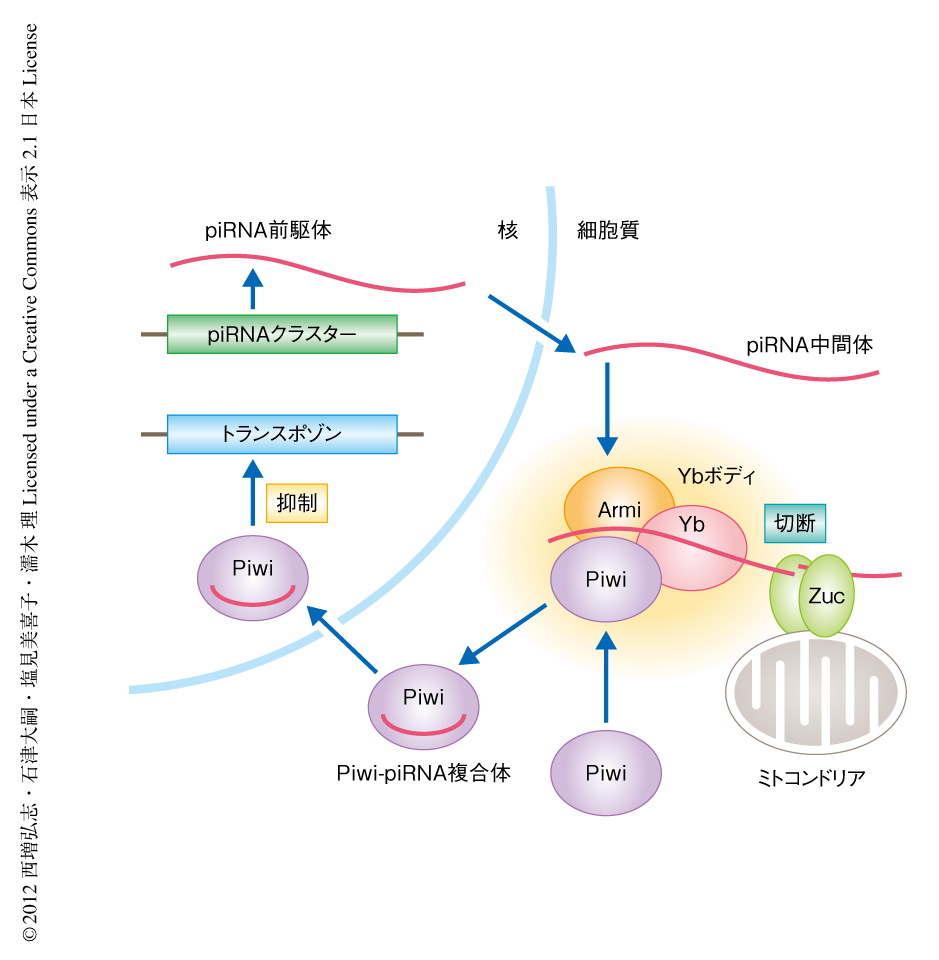
今回の結果を総合すると,ZucはpiRNAの産生にかかわるエンドリボヌクレアーゼであると考えられた.ショウジョウバエのもつPIWIサブファミリータンパク質のひとつであるPiwiに結合した成熟型piRNAの1番目の塩基はUにかたよっており2,14),Argonauteの結晶構造からもPiwiはpiRNAの5’末端の塩基を認識していることが示唆されている15).今回の生化学的な解析の結果から,Zucは厳密な塩基特異性をもたずに一本鎖RNAを切断し,5’末端のリン酸基を産生することが示された.したがって,ZucはpiRNA中間体を配列に非依存的に切断することにより5’末端にリン酸基をもつpiRNA断片を産生し,Piwiはそのうち1番目の塩基がUのpiRNA断片を選択的に取り込んでいるという可能性が考えられた.そののち,PiwiからはみだしたpiRNAの3’末端が別のヌクレアーゼ16) によりトリミングされることで,成熟型のpiRNAが産生されると考えられた(図2).
おわりに
この研究には多くの紆余曲折があった.二次構造予測を行ってディスオーダー領域を削除するのは結晶構造解析の定石のひとつである.そこで,DmZucの二次構造予測を行いさまざまなコンストラクトを検討したところ,膜貫通ヘリックスの直後にあるディスオーダー領域(さきに述べたとおり,この領域はZn2+結合ドメインに対応し,この直前にある1本のβストランドが二量体化に必須だった)までを削除したコンストラクトは大腸菌でよく発現し高純度に精製できることがわかった.また,結晶化スクリーニングを行うなかで,C末端にあるディスオーダー領域が結晶化に影響をあたえていることがわかったので,試行錯誤のすえ,DmZuc(残基89~250)の結晶構造を決定することに成功した.しかし,このDmZuc(残基89~250)はNucと異なり二量体を形成していなかった.あとになって,生化学的な解析からこのDmZuc単量体はリボヌクレアーゼ活性をもたないことが判明し,DmZucの二量体化は酵素活性に必須であることが明らかになった.DmZucとNucの結晶構造を比較したところ,DmZuc(残基89~250)ではNucにおいて二量体化に寄与しているβストランドがひとつ欠けていることがわかった.二次構造予測の結果からも,DmZucは膜貫通ヘリックスとディスオーダー領域とのあいだに1本のβストランドをもつことが予想された.そこで,βストランドをふくむDmZuc(残基41~253)を大腸菌で発現させ,4段階のカラムクロマトグラフィーにより精製した.ゲルろ過の結果からも,このDmZuc(残基41~253)は二量体を形成していることが示唆された.しかしながら,その発現量はきわめて低く,十分に結晶化スクリーニングを行うことができなかった(10リットルの大腸菌培養液から約0.02 mgしか精製できなかった).しかし,幸運なことに,機能解析のため変異体を調製している過程で,Lys171をAlaに置換した変異をもつDmZuc(残基41~253)は大腸菌においてよく発現することを見い出した(この変異体のほかの変異体はどれも発現量が低く,酵素活性測定のためそれぞれ5リットルの大腸菌培養液から精製した).このK171A変異体を用いて結晶化スクリーニングをやり直しその結晶構造を決定したところ,この変異体はNucと同様に二量体を形成していることがわかった.しかし,K171A変異体の酵素活性部位は負に帯電しており核酸が結合しそうにはみえなかった.たった1残基の置換により構造が変化しているとは思っていなかったが,野生型のDmZucも同様の結晶化条件により結晶化することができたので,念のためその結晶構造を決定した.その結果,驚くべきことに,野生型とK171A変異体では酵素活性部位においてループの構造が大きく変化しており,野生型DmZucは正に帯電した酵素活性部位をもつことがわかった.
米国の研究グループによる同様の論文も,Nature誌に同時に掲載された17).この研究グループも生化学的な解析を行い,マウスのZucホモログMmZucは一本鎖の核酸を切断する一方で,カルジオリピンを切断しないことを示していた.さらに,このMmZucの結晶構造を決定し,MmZucのもつ酵素活性部位はNucに比べ狭いことに着目し,Zucは一本鎖の核酸に特異的なリボヌクレアーゼであると結論づけていた.2つの独立した研究が同様の結論にいたったことからも,Zucはリボヌクレアーゼとして直接的にpiRNAの産生にかかわると考えられた.実は,この研究の裏にはこの米国の研究グループとのし烈な競争があった.筆者らが“正しい”結晶構造にたどりつき,さらに,構造解析と同時並行で進んでいた生化学的な解析によりDmZucがリボヌクレアーゼであることを見い出したのは,論文投稿の3週間前のことだった.
文 献
- Siomi, M. C., Sato, K., Pezic, D. et al.: PIWI-interacting small RNAs: the vanguard of genome defence. Nat. Rev. Mol. Cell Biol., 12, 246-258 (2011)[PubMed]
- Brennecke, J., Aravin, A. A., Stark, A. et al.: Discrete small RNA-generating loci as master regulators of transposon activity in Drosophila. Cell, 128, 1089-1103 (2007)[PubMed]
- Malone, C. D., Brennecke, J., Dus, M. et al.: Specialized piRNA pathways act in germline and somatic tissues of the Drosophila ovary. Cell, 137, 522-535 (2009)[PubMed]
- Li, C., Vagin, V. V., Lee, S. et al.: Collapse of germline piRNAs in the absence of Argonaute3 reveals somatic piRNAs in flies. Cell, 137, 509-521 (2009)[PubMed]
- Saito, K., Inagaki, S., Mituyama, T. et al.: A regulatory circuit for piwi by the large Maf gene traffic jam in Drosophila. Nature, 461, 1296-1299 (2009)[PubMed]
- Saito, K., Ishizu, H., Komai, M. et al.: Roles for the Yb body components Armitage and Yb in primary piRNA biogenesis in Drosophila. Genes Dev., 24, 2493-2498 (2010)[PubMed]
- Olivieri, D., Sykora, M. M., Sachidanandam, R. et al.: An in vivo RNAi assay identifies major genetic and cellular requirements for primary piRNA biogenesis in Drosophila. EMBO J., 29, 3301-3317 (2010)[PubMed]
- Schupbach, T. & Wieschaus, E.: Female sterile mutations on the second chromosome of Drosophila melanogaster. II. Mutations blocking oogenesis or altering egg morphology. Genetics, 129, 1119-1136 (1991)[PubMed]
- Choi, S. Y., Huang, P., Jenkins, G. M. et al.: A common lipid links Mfn-mediated mitochondrial fusion and SNARE-regulated exocytosis. Nat. Cell Biol., 8, 1255-1262 (2006)[PubMed]
- Watanabe, T., Chuma, S., Yamamoto, Y. et al.: MITOPLD is a mitochondrial protein essential for nuage formation and piRNA biogenesis in the mouse germline. Dev. Cell, 20, 364-375 (2011)[PubMed] [新着論文レビュー]
- Huang, H., Gao, Q., Peng, X. et al.: piRNA-associated germline nuage formation and spermatogenesis require MitoPLD profusogenic mitochondrial-surface lipid signaling. Dev. Cell, 20, 376-387 (2011)[PubMed]
- Pane, A., Wehr, K. & Schupbach, T.: zucchini and squash encode two putative nucleases required for rasiRNA production in the Drosophila germline. Dev. Cell, 12, 851-862 (2007)[PubMed]
- Stuckey, J. A. & Dixon, J. E.: Crystal structure of a phospholipase D family member. Nat. Struct. Biol., 6, 278-284 (1999)[PubMed]
- Saito, K., Nishida, K. M., Mori, T. et al.: Specific association of Piwi with rasiRNAs derived from retrotransposon and heterochromatic regions in the Drosophila genome. Genes Dev., 20, 2214-2222 (2006)[PubMed]
- Frank, F., Sonenberg, N. & Nagar, B.: Structural basis for 5'-nucleotide base-specific recognition of guide RNA by human AGO2. Nature, 465, 818-822 (2010)[PubMed]
- Kawaoka, S., Izumi, N., Katsuma, S. et al.: 3' end formation of PIWI-interacting RNAs in vitro. Mol. Cell, 43, 1015-1022 (2011)[PubMed] [新着論文レビュー]
- Ipsaro, J. J., Haase, A. D., Knott, S. R. et al.: The structural biochemistry of Zucchini implicates it as a nuclease in piRNA biogenesis. Nature, 491, 279-283 (2012)[PubMed]
著者プロフィール
略歴:2007年 東京大学大学院農学生命科学研究科 修了,2010年より東京大学大学院理学系研究科 特任助教.
研究テーマ:RNAサイレンシング,疾患にかかわるタンパク質.
抱負:立体構造から新しいコンセプトを創出していきたい.
石津 大嗣(Hirotsugu Ishizu)
略歴:2012年 慶應義塾大学大学院医学研究科 修了,同年より 東京大学大学院理学系研究科 助教.
研究テーマ:RNAサイレンシング.
抱負:piRNAによる遺伝子発現抑制の作用機構を明らかにする.
塩見 美喜子(Mikiko C. Siomi)
東京大学大学院理学系研究科 教授.
研究テーマ:RNAメタボリズム,RNAサイレンシング.
抱負:RNA研究をとおして生命のしくみを分子レベルで理解したい.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究テーマ:遺伝暗号の翻訳,膜輸送,自然炎症.
抱負:細胞の生理機構を原子分解能で解明したい.
研究室URL:http://www.nurekilab.net/
© 2012 西増弘志・石津大嗣・塩見美喜子・濡木 理 Licensed under CC 表示 2.1 日本