ガイド鎖と結合した出芽酵母のArgonauteとガイド鎖の結晶構造
中西孝太郎・Dinshaw J. Patel
(米国Memorial Sloan-Kettering Cancer Center,Structural Biology Program)
email:中西孝太郎
DOI: 10.7875/first.author.2012.080
Structure of yeast Argonaute with guide RNA.
Kotaro Nakanishi, David E. Weinberg, David P. Bartel, Dinshaw J. Patel
Nature, 486, 368-374 (2012)
Argonauteはガイド鎖となるRNAとRISCとよばれるRNA-タンパク質複合体を形成し,ガイド鎖に相補的な標的mRNAを認識し切断することで遺伝子発現を制御するRNAi機構におけるエフェクタータンパク質である.これまで,高度好熱細菌Thermus thermophilusのArgonauteの結晶構造が,ガイド鎖DNAとの二者複合体,そして,ガイド鎖DNAおよび標的mRNAとの三者複合体の状態で決定されていたが,真核生物のArgonauteの全長の立体構造は得られてなかった.そのため,真核生物のArgonauteの機構について構造学的な議論をする場合,おもに原核生物であるT. thermophilusのArgonauteの立体構造をもとに議論が展開されてきた.今回,筆者らは,真核生物のArgonauteとしてはじめて,出芽酵母のひとつKluyveromyces polysporusのArgonauteとガイド鎖との複合体の結晶構造を決定した.その結果,真核生物のArgonauteにおいて特有の領域の挿入により,K. polysporusのArgonauteは原核生物のものより長い核酸結合チャネルをもつことが明らかになった.また,すべてのArgonauteにおいて保存されているGlu残基が触媒残基としてはたらいていることを発見し,Glu残基がガイド鎖の取り込みに呼応して既知の3つのAsp残基の近傍へと移動して触媒テトラッドを完成させ,ArgonauteをRNAの切断が可能な状態へと移行させるという機構を提唱した.
RNAi(RNA interference,RNA干渉)は,二本鎖RNAに由来するmiRNA(microRNA)やsiRNA(small interfering RNA)が塩基相補的な配列を含む標的mRNAと結合することにより,そのmRNAが分解される現象である.この真核生物に特有な遺伝子発現の制御機構は,おもにDicerとArgonaute(AGO)が担っている.ArgonauteはDicerから二本鎖RNAを受け取ったのち,一方のRNA鎖を切断し,残ったガイド鎖とRNA-タンパク質複合体であるRISC(RNA-induced silencing complex)を形成する.RISCはガイド鎖と相補的な配列を含む標的mRNAに結合したのち,Argonaute自体がmRNAを切断,もしくは,切断せずmRNAにとどまることにより翻訳を阻害する.
現在まで,原核生物のArgonauteについては,ガイド鎖DNAとの二者複合体,および,ガイド鎖DNAおよび標的RNAとの三者複合体の結晶構造が決定され,その詳細な機構が明らかにされてきた1-3).しかしながら,真核生物のArgonauteはDNAではなくRNAをガイド鎖として用いるため,真核生物におけるArgonauteを介したRNAi機構の解明には真核生物のArgonauteの結晶構造が不可欠であった.また,真核生物のArgonauteには原核生物のArgonauteにはみられない領域が数多く挿入されており分子量が大きい.しかしながら,それらの挿入領域がどのような役割を担っているかについてはまったく明らかにされていなかった.
真核生物のArgonauteとして出芽酵母のひとつKluyveromyces polysporusのArgonauteの結晶構造を決定すべく,その組換えタンパク質を大腸菌において生合成した.7つの精製ステップをへて得られたArgonauteは,タンパク質としての純度はきわめて高かったが夾雑物として核酸を含んでおり,RNaseAを用いてもそれらを除くことはできなかった.しかし,このArgonauteから抽出した核酸はDNaseでは分解されないがRNaseにより分解された.これらの結果から,夾雑していた核酸はRNAであり,そのRNAは溶媒から隔離されるようにArgonauteにより守られていることが示唆された.そこで,このArgonauteに結合しているRNAはガイド鎖に違いないという予想のもと,核酸を含んだままのArgonauteを試料として用い結晶化のスクリーニングを開始した.
K. polysporusのArgonauteの結晶は比較的に容易に得られたが,以下の問題が構造解析に必要な結晶の大きさおよび質の向上を困難にさせた.まず,まったく同じ結晶化緩衝液を用いても同質の結晶が得られる確率がきわめて低かった.その理由として,この試料には長さと塩基配列の異なるRNAが混在し,その混ざり具合は精製ごとに異なっていることがあげられた.つぎに,K. polysporusのArgonauteの結晶はX線を回折しにくい性質をもち,強い回折像を得るためには結晶を大きくする必要があった.しかしながら,このArgonauteの結晶は成長の途中で秋の稲穂のように急に曲がりはじめてしまった.よって,結晶を大きくする目的で結晶化の時間を安易に長くすることはゆるされず,結晶がある程度まで成長しかつ曲がりだすまえを見計らって結晶を収穫する必要があった.また,みた目には真っすぐな結晶も触るとゼリーのように柔らかいものがほとんどであった.
以上の問題をクリアして大きく真っすぐな結晶が得られても,双晶化(twinning)という問題に悩まされた.K. polysporusのArgonauteの立体構造がすでに位相の決定されているほかの分子の構造に似ている場合,分子置換という手法で双晶化の問題は解決できる可能性は高い.そこで,これまでに構造決定されていた原核生物および真核生物のArgonauteの位相情報を用いて分子置換法を試みたが,K. polysporusのArgonauteの位相を決定することはできなかった.結晶化の条件をさらに改良する過程で1,4-ジオキサンが双晶化の抑制に効果的であることがわかり,最終的には双晶化のない結晶を得ることができた.そこで,K. polysporusのArgonauteのセレノメチオニン置換体を調製し,単波長異常散乱法により位相を決定することに成功した(PDB ID:4F1N).最初の結晶が得られてから18カ月のちのことであった.
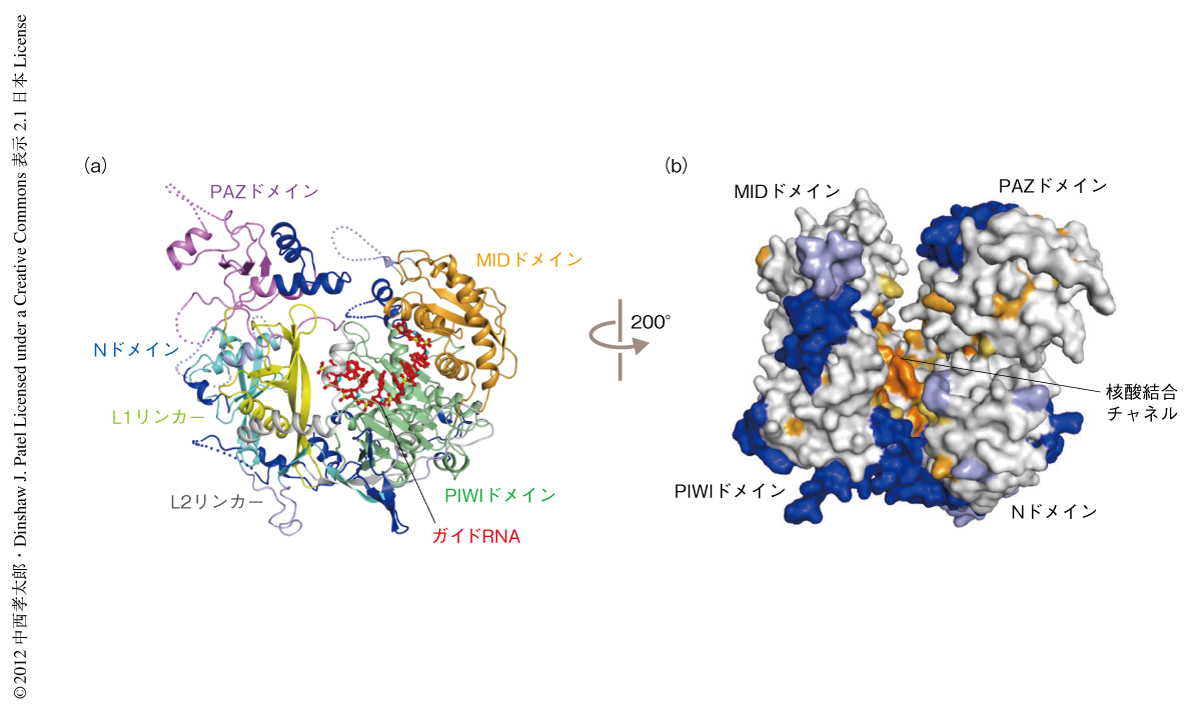
K. polysporusのArgonauteの全体構造は,原核生物のArgonauteの構造と同様に2つのローブから構成されており,そのあいだにガイド鎖の結合する核酸結合チャネルが走っていた(図1).そのチャネルを構成するアミノ酸残基は,原核生物および真核生物のArgonauteにおいてよく保存されており,真核生物に特有の挿入領域は分子の外側でクラスターを形成していた(図1b).このことから,原核生物のArgonauteから真核生物のArgonauteへと分子が進化する過程で,ガイド鎖を用いて標的mRNAを認識し切断するという基本機能を維持しつつ,新たな領域を分子の外側に付加して多機能性を獲得したという経緯がうかがえた.リボソームにおいても同様の進化がみられることは興味深い4).今回,構造比較から原核生物のArgonauteにはみられない19個の挿入部位を同定した.そのうち,11個が真核生物のArgonauteにおいて共通にみられ,残りの8個は真核生物の一部のArgonauteでのみみられた(図1).真核生物のArgonauteはこれらの挿入領域のクラスターを使ってほかのさまざまな分子と相互作用しているものと考えられた.
K. polysporusのArgonauteの結晶構造では,ガイド鎖の5’末端側の1位から8位のヌクレオチドに相当する電子密度が明瞭に確認できた.試料から抽出したガイド鎖を解析した結果,長さおよび塩基配列の異なる5万種類以上のRNAが同様にArgonauteと結合して結晶化していることが明らかになった.また,それらRNAのほとんどがArgonauteの発現に用いたベクターに由来するものであった.ガイド鎖の2’-OH基は,1位,2位,4位,5位,6位,7位でArgonauteによる認識をうけており,それら2’-OH基をデオキシ化したガイド鎖を含む二本鎖RNAはArgonauteにより取り込まれる効率が低下した.
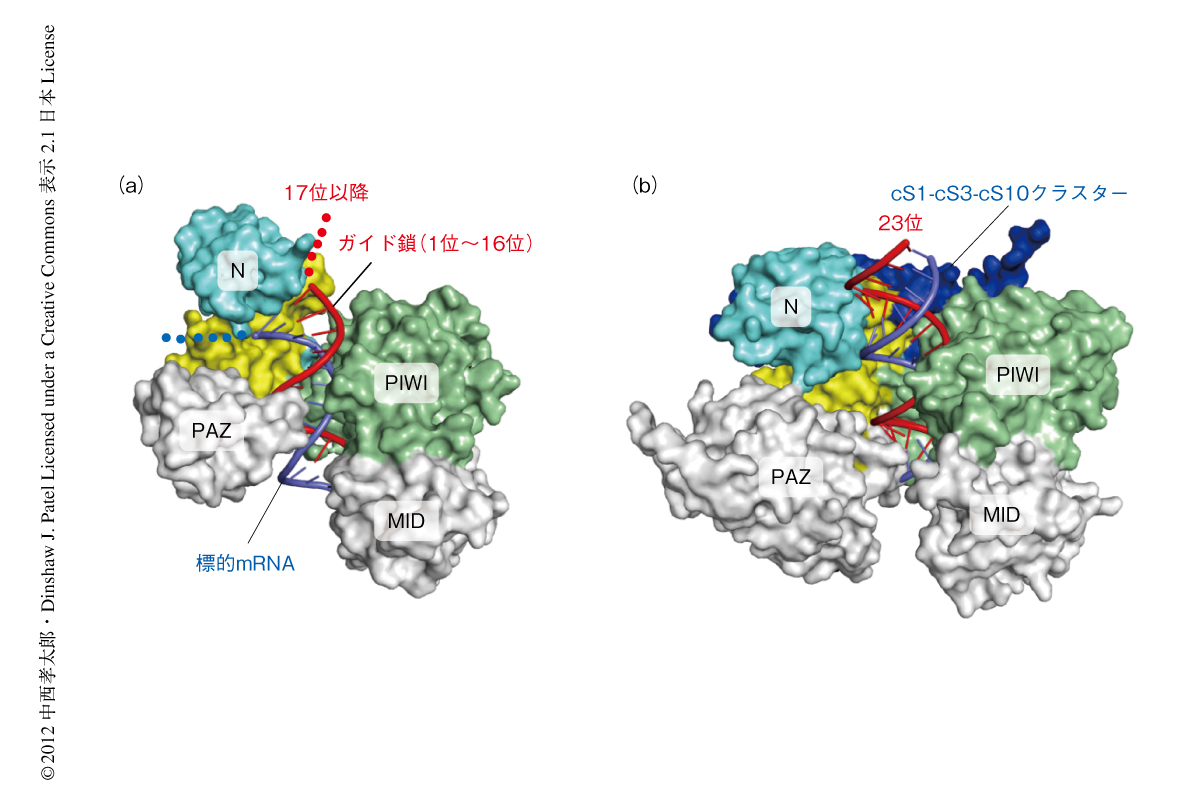
RISCが標的mRNAと結合したのちも両者が安定的に結合しつづけることで,mRNA分解や翻訳抑制にかかわる因子がArgonauteに結合しmiRNAによる遺伝子の発現抑制が起こる.また,速度反応論的な実験から,RISCに含まれるガイド鎖の1位から15位が標的mRNAの認識とその切断に寄与し,16位以降の3’側の配列は標的mRNAとの安定的な結合に寄与することが明らかにされている5).しかしながら,以前に決定された原核生物である高度好熱細菌Thermus thermophilusのArgonauteの結晶構造(PDB ID:3HK2)は,ガイド鎖の1位~16位は標的mRNAと二本鎖を形成するものの,17位以降はNドメインにより標的mRNAとは塩基対を形成できないようすを示していた2)(図2a).このように,原核生物のArgonauteの結晶構造と,真核生物のArgonauteの生化学的な実験によるデータとのあいだには,これまで矛盾が存在していた.今回,決定したK. polysporusのArgonauteの結晶構造では,真核生物のArgonauteに共通にみられる挿入部位cS1,cS3,cS10がPIWIドメインとL2ドメインとのあいだに入り込むことで,Nドメインは核酸結合チャネルの経路から完全に外れていた.その結果,K. polysporusのArgonauteの核酸結合チャネルはT. thermophilusのArgonauteの核酸結合チャネルよりも長く,ガイド鎖はその全長にわたり標的mRNAと塩基対を形成できることが示唆された(図2b).
ガイド鎖の取り込みが真核生物のArgonauteにどのような構造変化をもたらすのかを調べるため,すでに構造の決定されている真核生物Neurospora crassa QDE-2のMIDドメイン-PIWIドメイン単体(PDB ID:2YHA)と,今回,構造決定したガイド鎖と結合したK. polysporusのArgonauteとで,構造比較を行った6).N. crassaではガイド鎖の結合にかかわる4つのループ(ループL1~ループL4)のうち,ループL1がループL2の動きをブロックしていた(図3a).一方,K. polysporusのArgonauteではループL1のゲートが開くことでループL2がArgonauteの触媒残基である3つのAsp残基の近傍へと延びていた(図3a).そして,ループL2に位置するGlu1013が,His977,Arg1045,Glu1060と水素結合ネットワークを形成しその構造を安定化していた(図3b).His977は真核生物のすべてのArgonauteにおいて保存されており,Glu1013,R1045,Glu1060は真核生物および原核生物のすべてのArgonauteにおいて保存されている.また,これらのアミノ酸残基はPIWIファミリータンパク質においても保存されていることから,PIWIファミリータンパク質も同様の構造変化をする可能性が示唆された.
ArgonauteのPIWIドメインはDNA-RNAヘテロ二本鎖を切断することで知られるRNaseHフォールドをもつ.RNaseHフォールドをもつRNaseH1やRNaseH2が“DEDX”(D:Aps,E:Glu,X:AspもしくはHis)という触媒テトラッドをもつのに対し,ArgonauteのPIWIドメインは“DDD”(D:Aps)という触媒トライアッドをもち2番目の触媒残基であるGlu残基を欠失していると考えられてきた7,8).さきに述べたように,K. polysporusのArgonauteはガイド鎖の取り込みに呼応してGlu1013をほかの既知の触媒残基の近傍へと移動させていた(プラグイン).ガイド鎖と結合したK. polysporusのArgonauteの結晶構造をガイド鎖および標的mRNAと結合したT. thermophilusのArgonauteの結晶構造と重ね合わせると,プラグインしたK. polysporusのArgonauteのGlu1013の側鎖は標的mRNAの切断部位であるリン酸基の近傍に位置することがわかった.
この結果から,プラグインしたGlu1013こそが欠失したと考えられてきたArgonauteの触媒テトラッドのGlu残基であろうと考え,その置換体を作製してRNAiによる遺伝子の発現抑制を調べた.Glu1013とともに水素結合ネットワークを形成するHis977,R1045,Glu1060(図3b)のAla置換体のもつRNAi活性は野生型のものに比べ低下していた.しかし,Argonauteの触媒残基としてすでに知られているAsp1046のAla置換体と同じ程度にまでRNAi活性を著しく低下させたのはGlu1013のAla置換体だけであった.これらの結果から,Glu1013こそが欠失したと考えられてきた触媒残基であり,ガイド鎖の取り込みによりArgonauteはDEDX触媒テトラッドを完成させ,RNAの切断を可能な状態へと移行しているという機構を提唱した(図3a).
この研究では,K. polysporusのArgonauteがガイド鎖と結合した状態,つまり,真核生物におけるRISCの結晶構造を決定した.また,N. crassaのMIDドメイン-PIWIドメイン単体との構造比較から,ガイド鎖の取り込みによりGlu残基がほかの3つの触媒残基の近傍へと動き触媒テトラッドを完成させる機構を明らかにした.今回,筆者らのグループと同時期に,2つのグループがそれぞれ,ガイド鎖と結合したヒトのArgonaute2の結晶構造を決定している9,10).K. polysporusのArgonauteの結晶構造でみられた長い核酸結合チャネルはヒトのArgonaute2の構造においても確認できたことから,RISCにおいてガイド鎖がその全長にわたり標的mRNAと塩基対を形成する機構は真核生物のArgonauteに共通な特徴であると考えられた.
今回の結果は,真核生物のArgonauteによるRISCの形成と標的mRNAの認識および切断の機構は,原核生物のArgonauteにおける機構と各ステップにおいて違いがある可能性を示唆し,それらの機構を理解するためには真核生物のArgonauteの立体構造を決定することが重要であることを示したといえよう.その道ははじまったばかりである.
略歴:2007年 東京工業大学大学院生命理工学研究科 修了,同年 同 博士研究員,2008年 米国Memorial Sloan-Kettering Cancer Centerポスドク研究員を経て,2011年より同 リサーチアソシエート.
研究テーマ:非コードRNAによる細胞制御機構の解明.
抱負:立体構造からみえてくる新しい知見をもとに生命現象の謎を解き明かしたい.
Dinshaw J. Patel
米国Memorial Sloan-Kettering Cancer CenterにてLab Head.
研究室URL:http://www.mskcc.org/research/lab/dinshaw-patel
© 2012 中西孝太郎・Dinshaw J. Patel Licensed under CC 表示 2.1 日本
(米国Memorial Sloan-Kettering Cancer Center,Structural Biology Program)
email:中西孝太郎
DOI: 10.7875/first.author.2012.080
Structure of yeast Argonaute with guide RNA.
Kotaro Nakanishi, David E. Weinberg, David P. Bartel, Dinshaw J. Patel
Nature, 486, 368-374 (2012)
要 約
Argonauteはガイド鎖となるRNAとRISCとよばれるRNA-タンパク質複合体を形成し,ガイド鎖に相補的な標的mRNAを認識し切断することで遺伝子発現を制御するRNAi機構におけるエフェクタータンパク質である.これまで,高度好熱細菌Thermus thermophilusのArgonauteの結晶構造が,ガイド鎖DNAとの二者複合体,そして,ガイド鎖DNAおよび標的mRNAとの三者複合体の状態で決定されていたが,真核生物のArgonauteの全長の立体構造は得られてなかった.そのため,真核生物のArgonauteの機構について構造学的な議論をする場合,おもに原核生物であるT. thermophilusのArgonauteの立体構造をもとに議論が展開されてきた.今回,筆者らは,真核生物のArgonauteとしてはじめて,出芽酵母のひとつKluyveromyces polysporusのArgonauteとガイド鎖との複合体の結晶構造を決定した.その結果,真核生物のArgonauteにおいて特有の領域の挿入により,K. polysporusのArgonauteは原核生物のものより長い核酸結合チャネルをもつことが明らかになった.また,すべてのArgonauteにおいて保存されているGlu残基が触媒残基としてはたらいていることを発見し,Glu残基がガイド鎖の取り込みに呼応して既知の3つのAsp残基の近傍へと移動して触媒テトラッドを完成させ,ArgonauteをRNAの切断が可能な状態へと移行させるという機構を提唱した.
はじめに
RNAi(RNA interference,RNA干渉)は,二本鎖RNAに由来するmiRNA(microRNA)やsiRNA(small interfering RNA)が塩基相補的な配列を含む標的mRNAと結合することにより,そのmRNAが分解される現象である.この真核生物に特有な遺伝子発現の制御機構は,おもにDicerとArgonaute(AGO)が担っている.ArgonauteはDicerから二本鎖RNAを受け取ったのち,一方のRNA鎖を切断し,残ったガイド鎖とRNA-タンパク質複合体であるRISC(RNA-induced silencing complex)を形成する.RISCはガイド鎖と相補的な配列を含む標的mRNAに結合したのち,Argonaute自体がmRNAを切断,もしくは,切断せずmRNAにとどまることにより翻訳を阻害する.
現在まで,原核生物のArgonauteについては,ガイド鎖DNAとの二者複合体,および,ガイド鎖DNAおよび標的RNAとの三者複合体の結晶構造が決定され,その詳細な機構が明らかにされてきた1-3).しかしながら,真核生物のArgonauteはDNAではなくRNAをガイド鎖として用いるため,真核生物におけるArgonauteを介したRNAi機構の解明には真核生物のArgonauteの結晶構造が不可欠であった.また,真核生物のArgonauteには原核生物のArgonauteにはみられない領域が数多く挿入されており分子量が大きい.しかしながら,それらの挿入領域がどのような役割を担っているかについてはまったく明らかにされていなかった.
1.精製,結晶化,そして,構造決定
真核生物のArgonauteとして出芽酵母のひとつKluyveromyces polysporusのArgonauteの結晶構造を決定すべく,その組換えタンパク質を大腸菌において生合成した.7つの精製ステップをへて得られたArgonauteは,タンパク質としての純度はきわめて高かったが夾雑物として核酸を含んでおり,RNaseAを用いてもそれらを除くことはできなかった.しかし,このArgonauteから抽出した核酸はDNaseでは分解されないがRNaseにより分解された.これらの結果から,夾雑していた核酸はRNAであり,そのRNAは溶媒から隔離されるようにArgonauteにより守られていることが示唆された.そこで,このArgonauteに結合しているRNAはガイド鎖に違いないという予想のもと,核酸を含んだままのArgonauteを試料として用い結晶化のスクリーニングを開始した.
K. polysporusのArgonauteの結晶は比較的に容易に得られたが,以下の問題が構造解析に必要な結晶の大きさおよび質の向上を困難にさせた.まず,まったく同じ結晶化緩衝液を用いても同質の結晶が得られる確率がきわめて低かった.その理由として,この試料には長さと塩基配列の異なるRNAが混在し,その混ざり具合は精製ごとに異なっていることがあげられた.つぎに,K. polysporusのArgonauteの結晶はX線を回折しにくい性質をもち,強い回折像を得るためには結晶を大きくする必要があった.しかしながら,このArgonauteの結晶は成長の途中で秋の稲穂のように急に曲がりはじめてしまった.よって,結晶を大きくする目的で結晶化の時間を安易に長くすることはゆるされず,結晶がある程度まで成長しかつ曲がりだすまえを見計らって結晶を収穫する必要があった.また,みた目には真っすぐな結晶も触るとゼリーのように柔らかいものがほとんどであった.
以上の問題をクリアして大きく真っすぐな結晶が得られても,双晶化(twinning)という問題に悩まされた.K. polysporusのArgonauteの立体構造がすでに位相の決定されているほかの分子の構造に似ている場合,分子置換という手法で双晶化の問題は解決できる可能性は高い.そこで,これまでに構造決定されていた原核生物および真核生物のArgonauteの位相情報を用いて分子置換法を試みたが,K. polysporusのArgonauteの位相を決定することはできなかった.結晶化の条件をさらに改良する過程で1,4-ジオキサンが双晶化の抑制に効果的であることがわかり,最終的には双晶化のない結晶を得ることができた.そこで,K. polysporusのArgonauteのセレノメチオニン置換体を調製し,単波長異常散乱法により位相を決定することに成功した(PDB ID:4F1N).最初の結晶が得られてから18カ月のちのことであった.
2.K. polysporusのArgonauteの全体構造
K. polysporusのArgonauteの全体構造は,原核生物のArgonauteの構造と同様に2つのローブから構成されており,そのあいだにガイド鎖の結合する核酸結合チャネルが走っていた(図1).そのチャネルを構成するアミノ酸残基は,原核生物および真核生物のArgonauteにおいてよく保存されており,真核生物に特有の挿入領域は分子の外側でクラスターを形成していた(図1b).このことから,原核生物のArgonauteから真核生物のArgonauteへと分子が進化する過程で,ガイド鎖を用いて標的mRNAを認識し切断するという基本機能を維持しつつ,新たな領域を分子の外側に付加して多機能性を獲得したという経緯がうかがえた.リボソームにおいても同様の進化がみられることは興味深い4).今回,構造比較から原核生物のArgonauteにはみられない19個の挿入部位を同定した.そのうち,11個が真核生物のArgonauteにおいて共通にみられ,残りの8個は真核生物の一部のArgonauteでのみみられた(図1).真核生物のArgonauteはこれらの挿入領域のクラスターを使ってほかのさまざまな分子と相互作用しているものと考えられた.
3.ガイド鎖の認識
K. polysporusのArgonauteの結晶構造では,ガイド鎖の5’末端側の1位から8位のヌクレオチドに相当する電子密度が明瞭に確認できた.試料から抽出したガイド鎖を解析した結果,長さおよび塩基配列の異なる5万種類以上のRNAが同様にArgonauteと結合して結晶化していることが明らかになった.また,それらRNAのほとんどがArgonauteの発現に用いたベクターに由来するものであった.ガイド鎖の2’-OH基は,1位,2位,4位,5位,6位,7位でArgonauteによる認識をうけており,それら2’-OH基をデオキシ化したガイド鎖を含む二本鎖RNAはArgonauteにより取り込まれる効率が低下した.
4.真核生物のArgonauteに特有な長い核酸結合チャネル
RISCが標的mRNAと結合したのちも両者が安定的に結合しつづけることで,mRNA分解や翻訳抑制にかかわる因子がArgonauteに結合しmiRNAによる遺伝子の発現抑制が起こる.また,速度反応論的な実験から,RISCに含まれるガイド鎖の1位から15位が標的mRNAの認識とその切断に寄与し,16位以降の3’側の配列は標的mRNAとの安定的な結合に寄与することが明らかにされている5).しかしながら,以前に決定された原核生物である高度好熱細菌Thermus thermophilusのArgonauteの結晶構造(PDB ID:3HK2)は,ガイド鎖の1位~16位は標的mRNAと二本鎖を形成するものの,17位以降はNドメインにより標的mRNAとは塩基対を形成できないようすを示していた2)(図2a).このように,原核生物のArgonauteの結晶構造と,真核生物のArgonauteの生化学的な実験によるデータとのあいだには,これまで矛盾が存在していた.今回,決定したK. polysporusのArgonauteの結晶構造では,真核生物のArgonauteに共通にみられる挿入部位cS1,cS3,cS10がPIWIドメインとL2ドメインとのあいだに入り込むことで,Nドメインは核酸結合チャネルの経路から完全に外れていた.その結果,K. polysporusのArgonauteの核酸結合チャネルはT. thermophilusのArgonauteの核酸結合チャネルよりも長く,ガイド鎖はその全長にわたり標的mRNAと塩基対を形成できることが示唆された(図2b).
5.ガイド鎖の取り込みがひき起こす構造変化
ガイド鎖の取り込みが真核生物のArgonauteにどのような構造変化をもたらすのかを調べるため,すでに構造の決定されている真核生物Neurospora crassa QDE-2のMIDドメイン-PIWIドメイン単体(PDB ID:2YHA)と,今回,構造決定したガイド鎖と結合したK. polysporusのArgonauteとで,構造比較を行った6).N. crassaではガイド鎖の結合にかかわる4つのループ(ループL1~ループL4)のうち,ループL1がループL2の動きをブロックしていた(図3a).一方,K. polysporusのArgonauteではループL1のゲートが開くことでループL2がArgonauteの触媒残基である3つのAsp残基の近傍へと延びていた(図3a).そして,ループL2に位置するGlu1013が,His977,Arg1045,Glu1060と水素結合ネットワークを形成しその構造を安定化していた(図3b).His977は真核生物のすべてのArgonauteにおいて保存されており,Glu1013,R1045,Glu1060は真核生物および原核生物のすべてのArgonauteにおいて保存されている.また,これらのアミノ酸残基はPIWIファミリータンパク質においても保存されていることから,PIWIファミリータンパク質も同様の構造変化をする可能性が示唆された.
ArgonauteのPIWIドメインはDNA-RNAヘテロ二本鎖を切断することで知られるRNaseHフォールドをもつ.RNaseHフォールドをもつRNaseH1やRNaseH2が“DEDX”(D:Aps,E:Glu,X:AspもしくはHis)という触媒テトラッドをもつのに対し,ArgonauteのPIWIドメインは“DDD”(D:Aps)という触媒トライアッドをもち2番目の触媒残基であるGlu残基を欠失していると考えられてきた7,8).さきに述べたように,K. polysporusのArgonauteはガイド鎖の取り込みに呼応してGlu1013をほかの既知の触媒残基の近傍へと移動させていた(プラグイン).ガイド鎖と結合したK. polysporusのArgonauteの結晶構造をガイド鎖および標的mRNAと結合したT. thermophilusのArgonauteの結晶構造と重ね合わせると,プラグインしたK. polysporusのArgonauteのGlu1013の側鎖は標的mRNAの切断部位であるリン酸基の近傍に位置することがわかった.
この結果から,プラグインしたGlu1013こそが欠失したと考えられてきたArgonauteの触媒テトラッドのGlu残基であろうと考え,その置換体を作製してRNAiによる遺伝子の発現抑制を調べた.Glu1013とともに水素結合ネットワークを形成するHis977,R1045,Glu1060(図3b)のAla置換体のもつRNAi活性は野生型のものに比べ低下していた.しかし,Argonauteの触媒残基としてすでに知られているAsp1046のAla置換体と同じ程度にまでRNAi活性を著しく低下させたのはGlu1013のAla置換体だけであった.これらの結果から,Glu1013こそが欠失したと考えられてきた触媒残基であり,ガイド鎖の取り込みによりArgonauteはDEDX触媒テトラッドを完成させ,RNAの切断を可能な状態へと移行しているという機構を提唱した(図3a).
おわりに
この研究では,K. polysporusのArgonauteがガイド鎖と結合した状態,つまり,真核生物におけるRISCの結晶構造を決定した.また,N. crassaのMIDドメイン-PIWIドメイン単体との構造比較から,ガイド鎖の取り込みによりGlu残基がほかの3つの触媒残基の近傍へと動き触媒テトラッドを完成させる機構を明らかにした.今回,筆者らのグループと同時期に,2つのグループがそれぞれ,ガイド鎖と結合したヒトのArgonaute2の結晶構造を決定している9,10).K. polysporusのArgonauteの結晶構造でみられた長い核酸結合チャネルはヒトのArgonaute2の構造においても確認できたことから,RISCにおいてガイド鎖がその全長にわたり標的mRNAと塩基対を形成する機構は真核生物のArgonauteに共通な特徴であると考えられた.
今回の結果は,真核生物のArgonauteによるRISCの形成と標的mRNAの認識および切断の機構は,原核生物のArgonauteにおける機構と各ステップにおいて違いがある可能性を示唆し,それらの機構を理解するためには真核生物のArgonauteの立体構造を決定することが重要であることを示したといえよう.その道ははじまったばかりである.
文 献
- Wang, Y., Juranek, S., Li, H. et al.: Structure of an argonaute silencing complex with a seed-containing guide DNA and target RNA duplex. Nature, 456, 921-926 (2008)[PubMed]
- Wang, Y., Juranek, S., Li, H. et al.: Nucleation, propagation and cleavage of target RNAs in Ago silencing complexes. Nature, 461, 754-761 (2009)[PubMed]
- Wang, Y., Sheng, G., Juranek, S. et al.: Structure of the guide-strand-containing argonaute silencing complex. Nature, 456, 209-213 (2008)[PubMed]
- Ben-Shem, A., Jenner, L., Yusupova, G. et al.: Crystal structure of the eukaryotic ribosome. Science, 330, 1203-1209 (2010)[PubMed]
- Ameres, S. L., Martinez, J. & Schroeder, R.: Molecular basis for target RNA recognition and cleavage by human RISC. Cell, 130, 101-112 (2007)[PubMed]
- Boland, A., Huntzinger, E., Schmidt, S. et al.: Crystal structure of the MID-PIWI lobe of a eukaryotic Argonaute protein. Proc. Natl. Acad. Sci. USA, 108, 10466-10471 (2011)[PubMed]
- Nowotny, M.: Retroviral integrase superfamily: the structural perspective. EMBO Rep., 10, 144-151 (2009)[PubMed]
- Song, J. J., Smith, S. K., Hannon, G. J. et al.: Crystal structure of Argonaute and its implications for RISC slicer activity. Science, 305, 1434-1437 (2004)[PubMed]
- Elkayam, E., Kuhn, C. D., Tocilj, A. et al.: The structure of human Argonaute-2 in complex with miR-20a. Cell, 150, 100-110 (2012)[PubMed]
- Schirle, N. T. & MacRae, I. J.: The crystal structure of human Argonaute2. Science, 336, 1037-1040 (2012)[PubMed]
著者プロフィール
略歴:2007年 東京工業大学大学院生命理工学研究科 修了,同年 同 博士研究員,2008年 米国Memorial Sloan-Kettering Cancer Centerポスドク研究員を経て,2011年より同 リサーチアソシエート.
研究テーマ:非コードRNAによる細胞制御機構の解明.
抱負:立体構造からみえてくる新しい知見をもとに生命現象の謎を解き明かしたい.
Dinshaw J. Patel
米国Memorial Sloan-Kettering Cancer CenterにてLab Head.
研究室URL:http://www.mskcc.org/research/lab/dinshaw-patel
© 2012 中西孝太郎・Dinshaw J. Patel Licensed under CC 表示 2.1 日本