アメーバのもつ5-メチルシトシン酸化酵素TET1の構造
橋本 秀春
(米国Emory大学School of Medicine,Departments of Biochemistry)
email:橋本秀春
DOI: 10.7875/first.author.2014.011
Structure of a Naegleria Tet-like dioxygenase in complex with 5-methylcytosine DNA.
Hideharu Hashimoto, June E. Pais, Xing Zhang, Lana Saleh, Zheng-Qing Fu, Nan Dai, Ivan R. Corrêa, Yu Zheng, Xiaodong Cheng
Nature, 506, 391-395 (2014)
TETはFe2+およびαケトグルタル酸を補酵素として5-メチルシトシンのもつメチル基を酸化する.今回,哺乳類のTETと相同性のあるアメーバのもつTET1についてX線結晶構造解析を行った.アメーバTET1は哺乳類のTETと同様に,Fe2+およびαケトグルタル酸に依存して,5-メチルシトシンのもつメチル基をヒドロキシメチル基,ホルミル基,カルボキシル基へと酸化した.アメーバTET1,5-メチルシトシンを含む基質DNA,Mn2+,αケトグルタル酸のアナログであるN-オキサリルグリシンからなる複合体の共結晶を作製し,2.9Åの解像度でX線結晶構造解析を行うことにより,TETによる5-メチルシトシンの認識の機構を明らかにした.アメーバTET1は副溝の側からDNAを認識し,主溝の側へとDNAを曲げ,基質である5-メチルシトシンをフリップアウトし,活性部位において基質である5-メチルシトシンのWatson-Crickエッジおよびそのメチル基を認識することが明らかになった.さらに,哺乳類のTET1とアメーバTET1とのあいだでは構造および機能において重要なアミノ酸残基が保存されていたことから,このアメーバTET1の構造はTETの普遍的な構造であることが示唆された.
シトシンの5位の炭素原子はメチル化修飾をうけることが知られている.この5-メチルシトシンはヒストンH3の9番目のLysのメチル化と共役して転写の抑制に関与する.5-メチルシトシンに対するDNA脱メチル化酵素については長いあいだ不明であったが,2009年,TET1および5-ヒドロキシメチルシトシンが発見され1),それから5-メチルシトシンのDNA脱メチル化の研究は急速に進んだ.哺乳類はTETファミリーとしてTET1,TET2,TET3の3つをもち,いずれも1800~2100アミノ酸残基からなる.いずれもC末端側に,Fe2+およびαケトグルタル酸を補酵素とする酸化酵素に共通したドメインであるDSBHドメインをもつ1-3).TETはFe2+およびαケトグルタル酸を補酵素として5-メチルシトシンを5-ヒドロキシメチルシトシンへと酸化し,生成物である5-ヒドロキシメチルシトシンもTETによりさらに5-ホルミルシトシン,5-カルボキシルシトシンへと酸化される4).TETが発見されてから5年近くがたち,ようやくTETの構造基盤が明らかにされた.
哺乳類のTETのもつDSBHドメインにはTETファミリーのあいだで相同性の低い300~500アミノ酸残基からなる挿入配列が存在する.これまで,複数の研究グループがTETの結晶化に取り組んできたと思われるが成功していない.そして,この挿入配列が結晶化を困難にしていると考えられている.そこで,筆者らは,DSBHドメインに挿入配列をもたないアメーバ(Naegleria gruberi)のTETを用いて結晶化を試みた.
TETファミリーはTET/JBPスーパーファミリーに属する.アメーバにはこのスーパーファミリーを特徴づけるTET/JBPドメインをもつ8つのcDNAが発見されている5).これらのcDNAを大腸菌において発現させることにより,アメーバTET1およびアメーバTET4が哺乳類のTETと同様に,Fe2+およびαケトグルタル酸に依存して5-メチルシトシンを5-ヒドロキシメチルシトシンへと酸化することを確認した.そこで,このアメーバTET1について解析を進めた.
アメーバTET1はアメーバcDNAに由来する321アミノ酸残基からなる約37 kDaの推定タンパク質である.アメーバTET1をコードするcDNAについて,大腸菌に対しコドンを最適化したものを人工合成し,大腸菌を形質転換した.発現させたタンパク質は超音波による可溶化ののち,Niカラム,イオン交換カラム,ゲルろ過カラムにより精製した.
アメーバTET1はCpG配列がヘミメチル化されたDNAおよび全メチル化されたDNAの両方に対しαケトグルタル酸に依存的なDNA脱メチル化活性をもっていた.酵素活性の測定は,蛍光標識した32塩基対の二本鎖DNA,56塩基対の二本鎖DNA,一本鎖DNA,HeLa細胞のゲノムDNAを用い,pH 6.0において,抗体による5-ヒドロキシメチルシトシン,5-ホルミルシトシン,5-カルボキシルシトシンの検出6),チミンDNAグリコシラーゼを用いた5-ホルミルシトシン,5-カルボキシルシトシンの検出7),および,液体クロマトグラフィー-質量分析計により5-メチルシトシン,5-ヒドロキシメチルシトシン,5-ホルミルシトシン,5-カルボキシルシトシンを定量することにより行った.アメーバTET1は哺乳類のTETと同様に,二本鎖DNAおよび一本鎖DNAに対しともに活性をもち,アメーバTET1は1時間でそれぞれ75%および94%の5-メチルシトシンをカルボキシル化シトシンにまで酸化した.
アメーバTET1と5-メチルシトシンを含む基質DNAとの共結晶化のため,Fe2+の代わりにMn2+を用い,αケトグルタル酸のアナログとしてN-オキサリルグリシンを用いて酵素反応を停止させた.まず,全メチル化CpG配列を含む16塩基対のDNAを用いてアメーバTET1との共結晶を作製し,3.74Åの解像度のデータセットを得た.さらに解像度をあげるため,1塩基対ずつDNAの長さを変えていき,全メチル化CpG配列を含む14塩基対のDNAとの共結晶を作製して2.89Åの解像度のデータを得た.チミジンをブロモデオキシウリジンにより置き換えた14塩基対のDNAとアメーバTET1との共結晶を用い,臭素原子の異常分散により位相を決定した(PDB ID:4LT5).
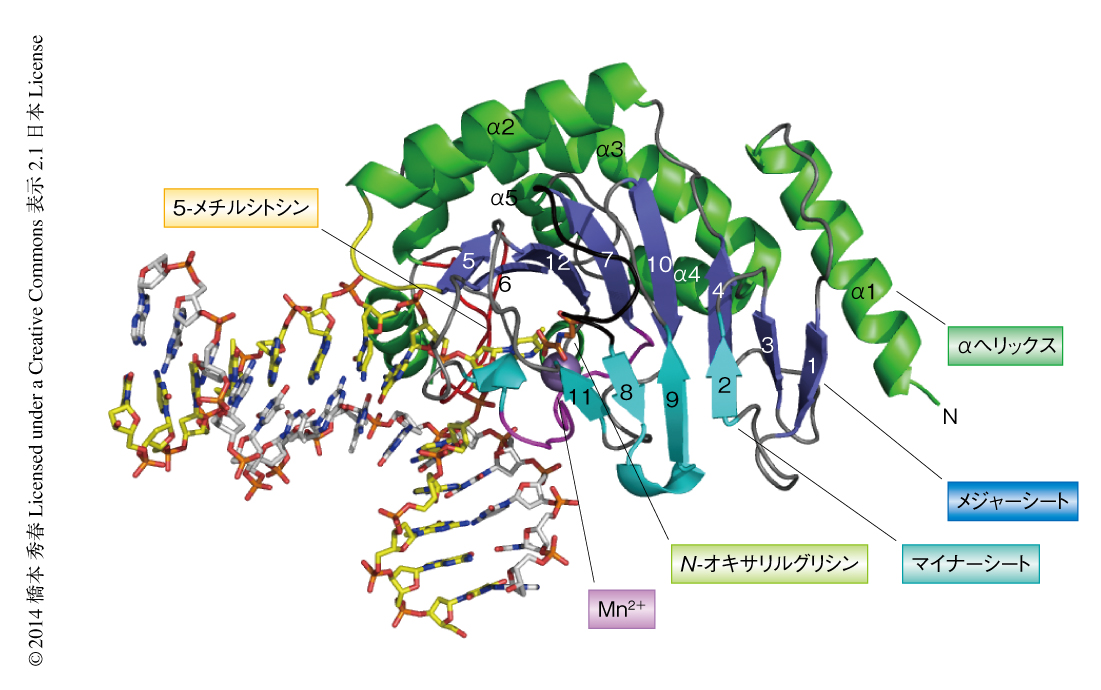
アメーバTET1の全体の構造はDSBHドメインを含み,4つのβストランドからなるマイナーなβシート,8つのβストランドからなるメジャーなβシート,外側の5つのαヘリックス,からなる3層構造をとっていた(図1).アメーバTET1はDNAを65度ほど曲げ,副溝の側からDNAを認識し基質である5-メチルシトシンをフリップアウトしていた.フリップアウトした5-メチルシトシンはPhe295とArg224により安定化されていた.5-メチルシトシンのWatson-CrickエッジはAsn147,His297,Asp234による水素結合により正確に認識されていた.ここから,チミンを認識しないことがわかった.フリップアウトした5-メチルシトシンのメチル基は金属イオンにむかって5.2Å離れた位置に存在した.この距離は,DSBHドメインをもつほかのタンパク質における基質と金属イオンとの距離にほぼ等しかったことから,アメーバTET1もほかのDSBHドメインをもつタンパク質と同じ酸化還元反応機構を利用してシトシン5位のメチル基を酸化していると考えられた1).また,フリップアウトした5-メチルシトシンのあった空間には,アメーバTET1のもつアミノ酸残基の側鎖は挿入されてはいなかった.これは,ほかのタンパク質とは異なっていた.さらに,5-メチルシトシンと対になっていたグアニンはSer148により安定化されていた.全メチル化CpG配列のフリップアウトした3’側のグアニンはGln310により認識されていた.このことから,アメーバTET1はCpA配列にも活性があるものの,CpG配列を好んで酸化することがわかった.また,反対側のDNA鎖の5-メチルシトシンのメチル基は認識されていなかったことから,ヘミメチル化DNAとフルメチル化DNAの両方に活性をもつことがわかった.αケトグルタル酸のアナログであるN-オキサリルグリシンはMn2+とキレートし活性中心の深い部分においてArg224,Tyr242,Arg289により認識されていた.実際の酵素反応ではαケトグルタル酸が用いられ,これはコハク酸へと変換される.つぎの反応のためにはこれを新たなαケトグルタル酸と入れ替える必要があるため,いったんDNAが解離する必要がある.
AlkB,および,そのヒトにおけるホモログであるABHは,DSBHドメインをもち,DNAおよびRNAのWatson-Crickエッジ側のシトシンもしくはチミンのN3位のメチル化,あるいは,アデニンのN1位のメチル化による塩基の損傷を,この塩基をフリップアウトしてFe2+およびαケトグルタル酸に依存してこれを酸化し,3-メチルシトシンを3-ヒドロキシメチルシトシン,3-メチルチミンを3-ヒドロキシルメチルチミン,1-メチルアデニンを1-ヒドロキシメチルアデニンへと変換する酵素である.窒素原子に結合するヒドロキシルメチル基は不安定なため,ホルムアルデヒドとCO2を副産物として脱メチル化する8).そこで,DSBHドメインをもつ2つのタンパク質,5-メチルシトシンを基質とするアメーバTET1と,3-メチルシトシンを基質とするAlkBが,どのようにしてシトシンの異なる位置のメチル基,すなわち,主溝の側の5位のメチル基,あるいは,Watson-Crickエッジ側の3位のメチル基を活性中心に呈示しているかについて注目した.
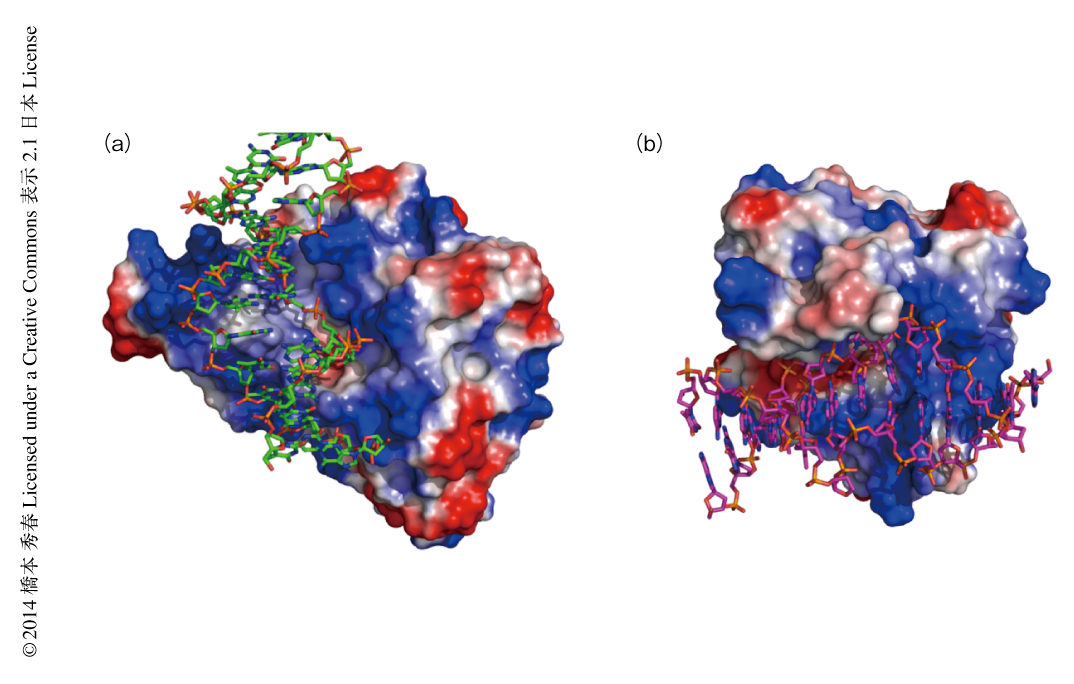
アメーバTET1,5-メチルシトシンを含むDNA,N-オキサリルグリシン,Mn2+からなる複合体の構造(PDB ID:4LT5)と,大腸菌のAlkB,3-メチルシトシンを含むDNA,αケトグルタル酸,Mn2+からなる複合体の構造(PDB ID:3O1M)とを重ね合わせた.アミノ酸の一次配列は異なるもののDSBHドメインの構造は高度に保存されており,2つの構造は金属イオンの位置および補酵素も含め整列させることができた.そして,アメーバTET1およびAlkBともに,β5ストランドおよびβ7ストランドも整列させることができ,両方にそれらからつながるヘアピンループおよび活性ループが存在した.2つのタンパク質のコアドメインが整列し,それぞれの基質DNAはタンパク質の表面の塩基性の領域にそって結合していた.2つの構造を比較したときのもっとも顕著な違いは,アメーバTET1の構造におけるDNAとAlkBの構造におけるDNAが,タンパク質の表面においてほぼ直交したかたちでタンパク質と結合していた点であった(図2).また,フリップアウトした5-メチルシトシンおよび3-メチルシトシンもほぼ直交したかたちで活性中心にむかって位置していた.このように,アメーバTET1およびAlkBにおいて,DNAが直交したかたちでDSBHドメインと結合することにより,塩基がフリップアウトしたのち,主溝の側あるいはWatson-Crickエッジの側に位置するメチル基が活性中心に呈示される位置にきていた.この結合様式が,同じDSBHドメイン構造をもちながらも,シトシンの5位あるいは3位のメチル基を活性中心にむかって呈示させ,アメーバTET1が5位のメチル基を,AlkBが3位のメチル基を特異的に酸化するための分子機構であった.
哺乳類のTET1ではDSBHドメインに約300アミノ酸残基の挿入配列がドメインを分断したかたちで挿入されていた.この挿入配列を除去しても酵素活性が認められたことから,挿入配列は活性には必須でないと考えられた.また,哺乳類のTETにはシステインリッチ領域も挿入されていた.これらの挿入配列を除いたときのアメーバTET1と哺乳類のTET1とのあいだの相同性としては,14%の同一性,39%の類似性があった.このような相同性はDSBHドメインのみならずそれ以外の領域においても認められ,構造および機能において重要なアミノ酸残基は保存されていた.さらに,酵素の機能として,アメーバTET1は哺乳類のTETと同じく,5-メチルシトシンから5-ヒドロキシメチルシトシン,5-ホルミルシトシン,5-カルボキシルシトシンへと,Fe2+およびαケトグルタル酸に依存して酸化したことから,アメーバTET1とDNAとの共結晶構造はTETの普遍的な構造であることが示唆された.
筆者らのグループは,アメーバTET1の結晶構造解析および生化学的な実験を行った.TETを含むTET/JBPスーパーファミリーにおいて,このスーパーファミリーを特徴づけるTET/JBPドメインは進化において散在して存在し5),これらのTETの基本構造は保存されていると考えられる.実際に,筆者らの論文と同じ時期に,ヒトTET2とDNAとの共結晶構造についても発表されている9).この論文では,TET2のDSBHドメインに存在する相同性の低い挿入配列を除去したタンパク質を用いて結晶化に成功している.そして,アメーバTET1とヒトTET2の構造を比較したところ,TETの構造は進化においておおむね保存されていることがわかった.
略歴:2007年 京都大学大学院生命科学研究科 修了,同年 米国Emory大学School of Medicineポストドクトラルフェローを経て,2012年より同 リサーチアソシエート.
研究テーマ:DNAメチル化にかかわるタンパク質の生化学とX線結晶構造解析.
抱負:Enjoy science.
© 2014 橋本 秀春 Licensed under CC 表示 2.1 日本
(米国Emory大学School of Medicine,Departments of Biochemistry)
email:橋本秀春
DOI: 10.7875/first.author.2014.011
Structure of a Naegleria Tet-like dioxygenase in complex with 5-methylcytosine DNA.
Hideharu Hashimoto, June E. Pais, Xing Zhang, Lana Saleh, Zheng-Qing Fu, Nan Dai, Ivan R. Corrêa, Yu Zheng, Xiaodong Cheng
Nature, 506, 391-395 (2014)
要 約
TETはFe2+およびαケトグルタル酸を補酵素として5-メチルシトシンのもつメチル基を酸化する.今回,哺乳類のTETと相同性のあるアメーバのもつTET1についてX線結晶構造解析を行った.アメーバTET1は哺乳類のTETと同様に,Fe2+およびαケトグルタル酸に依存して,5-メチルシトシンのもつメチル基をヒドロキシメチル基,ホルミル基,カルボキシル基へと酸化した.アメーバTET1,5-メチルシトシンを含む基質DNA,Mn2+,αケトグルタル酸のアナログであるN-オキサリルグリシンからなる複合体の共結晶を作製し,2.9Åの解像度でX線結晶構造解析を行うことにより,TETによる5-メチルシトシンの認識の機構を明らかにした.アメーバTET1は副溝の側からDNAを認識し,主溝の側へとDNAを曲げ,基質である5-メチルシトシンをフリップアウトし,活性部位において基質である5-メチルシトシンのWatson-Crickエッジおよびそのメチル基を認識することが明らかになった.さらに,哺乳類のTET1とアメーバTET1とのあいだでは構造および機能において重要なアミノ酸残基が保存されていたことから,このアメーバTET1の構造はTETの普遍的な構造であることが示唆された.
はじめに
シトシンの5位の炭素原子はメチル化修飾をうけることが知られている.この5-メチルシトシンはヒストンH3の9番目のLysのメチル化と共役して転写の抑制に関与する.5-メチルシトシンに対するDNA脱メチル化酵素については長いあいだ不明であったが,2009年,TET1および5-ヒドロキシメチルシトシンが発見され1),それから5-メチルシトシンのDNA脱メチル化の研究は急速に進んだ.哺乳類はTETファミリーとしてTET1,TET2,TET3の3つをもち,いずれも1800~2100アミノ酸残基からなる.いずれもC末端側に,Fe2+およびαケトグルタル酸を補酵素とする酸化酵素に共通したドメインであるDSBHドメインをもつ1-3).TETはFe2+およびαケトグルタル酸を補酵素として5-メチルシトシンを5-ヒドロキシメチルシトシンへと酸化し,生成物である5-ヒドロキシメチルシトシンもTETによりさらに5-ホルミルシトシン,5-カルボキシルシトシンへと酸化される4).TETが発見されてから5年近くがたち,ようやくTETの構造基盤が明らかにされた.
1.アメーバTET1の発現および精製
哺乳類のTETのもつDSBHドメインにはTETファミリーのあいだで相同性の低い300~500アミノ酸残基からなる挿入配列が存在する.これまで,複数の研究グループがTETの結晶化に取り組んできたと思われるが成功していない.そして,この挿入配列が結晶化を困難にしていると考えられている.そこで,筆者らは,DSBHドメインに挿入配列をもたないアメーバ(Naegleria gruberi)のTETを用いて結晶化を試みた.
TETファミリーはTET/JBPスーパーファミリーに属する.アメーバにはこのスーパーファミリーを特徴づけるTET/JBPドメインをもつ8つのcDNAが発見されている5).これらのcDNAを大腸菌において発現させることにより,アメーバTET1およびアメーバTET4が哺乳類のTETと同様に,Fe2+およびαケトグルタル酸に依存して5-メチルシトシンを5-ヒドロキシメチルシトシンへと酸化することを確認した.そこで,このアメーバTET1について解析を進めた.
アメーバTET1はアメーバcDNAに由来する321アミノ酸残基からなる約37 kDaの推定タンパク質である.アメーバTET1をコードするcDNAについて,大腸菌に対しコドンを最適化したものを人工合成し,大腸菌を形質転換した.発現させたタンパク質は超音波による可溶化ののち,Niカラム,イオン交換カラム,ゲルろ過カラムにより精製した.
2.アメーバTET1の生化学的な特性
アメーバTET1はCpG配列がヘミメチル化されたDNAおよび全メチル化されたDNAの両方に対しαケトグルタル酸に依存的なDNA脱メチル化活性をもっていた.酵素活性の測定は,蛍光標識した32塩基対の二本鎖DNA,56塩基対の二本鎖DNA,一本鎖DNA,HeLa細胞のゲノムDNAを用い,pH 6.0において,抗体による5-ヒドロキシメチルシトシン,5-ホルミルシトシン,5-カルボキシルシトシンの検出6),チミンDNAグリコシラーゼを用いた5-ホルミルシトシン,5-カルボキシルシトシンの検出7),および,液体クロマトグラフィー-質量分析計により5-メチルシトシン,5-ヒドロキシメチルシトシン,5-ホルミルシトシン,5-カルボキシルシトシンを定量することにより行った.アメーバTET1は哺乳類のTETと同様に,二本鎖DNAおよび一本鎖DNAに対しともに活性をもち,アメーバTET1は1時間でそれぞれ75%および94%の5-メチルシトシンをカルボキシル化シトシンにまで酸化した.
3.アメーバTET1と基質DNAとの共結晶の作製と位相の決定
アメーバTET1と5-メチルシトシンを含む基質DNAとの共結晶化のため,Fe2+の代わりにMn2+を用い,αケトグルタル酸のアナログとしてN-オキサリルグリシンを用いて酵素反応を停止させた.まず,全メチル化CpG配列を含む16塩基対のDNAを用いてアメーバTET1との共結晶を作製し,3.74Åの解像度のデータセットを得た.さらに解像度をあげるため,1塩基対ずつDNAの長さを変えていき,全メチル化CpG配列を含む14塩基対のDNAとの共結晶を作製して2.89Åの解像度のデータを得た.チミジンをブロモデオキシウリジンにより置き換えた14塩基対のDNAとアメーバTET1との共結晶を用い,臭素原子の異常分散により位相を決定した(PDB ID:4LT5).
4.アメーバTET1の結晶構造と5-メチルシトシンの認識の機構
アメーバTET1の全体の構造はDSBHドメインを含み,4つのβストランドからなるマイナーなβシート,8つのβストランドからなるメジャーなβシート,外側の5つのαヘリックス,からなる3層構造をとっていた(図1).アメーバTET1はDNAを65度ほど曲げ,副溝の側からDNAを認識し基質である5-メチルシトシンをフリップアウトしていた.フリップアウトした5-メチルシトシンはPhe295とArg224により安定化されていた.5-メチルシトシンのWatson-CrickエッジはAsn147,His297,Asp234による水素結合により正確に認識されていた.ここから,チミンを認識しないことがわかった.フリップアウトした5-メチルシトシンのメチル基は金属イオンにむかって5.2Å離れた位置に存在した.この距離は,DSBHドメインをもつほかのタンパク質における基質と金属イオンとの距離にほぼ等しかったことから,アメーバTET1もほかのDSBHドメインをもつタンパク質と同じ酸化還元反応機構を利用してシトシン5位のメチル基を酸化していると考えられた1).また,フリップアウトした5-メチルシトシンのあった空間には,アメーバTET1のもつアミノ酸残基の側鎖は挿入されてはいなかった.これは,ほかのタンパク質とは異なっていた.さらに,5-メチルシトシンと対になっていたグアニンはSer148により安定化されていた.全メチル化CpG配列のフリップアウトした3’側のグアニンはGln310により認識されていた.このことから,アメーバTET1はCpA配列にも活性があるものの,CpG配列を好んで酸化することがわかった.また,反対側のDNA鎖の5-メチルシトシンのメチル基は認識されていなかったことから,ヘミメチル化DNAとフルメチル化DNAの両方に活性をもつことがわかった.αケトグルタル酸のアナログであるN-オキサリルグリシンはMn2+とキレートし活性中心の深い部分においてArg224,Tyr242,Arg289により認識されていた.実際の酵素反応ではαケトグルタル酸が用いられ,これはコハク酸へと変換される.つぎの反応のためにはこれを新たなαケトグルタル酸と入れ替える必要があるため,いったんDNAが解離する必要がある.
5.3-メチルシトシン酸化酵素AlkBとの比較
AlkB,および,そのヒトにおけるホモログであるABHは,DSBHドメインをもち,DNAおよびRNAのWatson-Crickエッジ側のシトシンもしくはチミンのN3位のメチル化,あるいは,アデニンのN1位のメチル化による塩基の損傷を,この塩基をフリップアウトしてFe2+およびαケトグルタル酸に依存してこれを酸化し,3-メチルシトシンを3-ヒドロキシメチルシトシン,3-メチルチミンを3-ヒドロキシルメチルチミン,1-メチルアデニンを1-ヒドロキシメチルアデニンへと変換する酵素である.窒素原子に結合するヒドロキシルメチル基は不安定なため,ホルムアルデヒドとCO2を副産物として脱メチル化する8).そこで,DSBHドメインをもつ2つのタンパク質,5-メチルシトシンを基質とするアメーバTET1と,3-メチルシトシンを基質とするAlkBが,どのようにしてシトシンの異なる位置のメチル基,すなわち,主溝の側の5位のメチル基,あるいは,Watson-Crickエッジ側の3位のメチル基を活性中心に呈示しているかについて注目した.
アメーバTET1,5-メチルシトシンを含むDNA,N-オキサリルグリシン,Mn2+からなる複合体の構造(PDB ID:4LT5)と,大腸菌のAlkB,3-メチルシトシンを含むDNA,αケトグルタル酸,Mn2+からなる複合体の構造(PDB ID:3O1M)とを重ね合わせた.アミノ酸の一次配列は異なるもののDSBHドメインの構造は高度に保存されており,2つの構造は金属イオンの位置および補酵素も含め整列させることができた.そして,アメーバTET1およびAlkBともに,β5ストランドおよびβ7ストランドも整列させることができ,両方にそれらからつながるヘアピンループおよび活性ループが存在した.2つのタンパク質のコアドメインが整列し,それぞれの基質DNAはタンパク質の表面の塩基性の領域にそって結合していた.2つの構造を比較したときのもっとも顕著な違いは,アメーバTET1の構造におけるDNAとAlkBの構造におけるDNAが,タンパク質の表面においてほぼ直交したかたちでタンパク質と結合していた点であった(図2).また,フリップアウトした5-メチルシトシンおよび3-メチルシトシンもほぼ直交したかたちで活性中心にむかって位置していた.このように,アメーバTET1およびAlkBにおいて,DNAが直交したかたちでDSBHドメインと結合することにより,塩基がフリップアウトしたのち,主溝の側あるいはWatson-Crickエッジの側に位置するメチル基が活性中心に呈示される位置にきていた.この結合様式が,同じDSBHドメイン構造をもちながらも,シトシンの5位あるいは3位のメチル基を活性中心にむかって呈示させ,アメーバTET1が5位のメチル基を,AlkBが3位のメチル基を特異的に酸化するための分子機構であった.
6.哺乳類のTET1との比較
哺乳類のTET1ではDSBHドメインに約300アミノ酸残基の挿入配列がドメインを分断したかたちで挿入されていた.この挿入配列を除去しても酵素活性が認められたことから,挿入配列は活性には必須でないと考えられた.また,哺乳類のTETにはシステインリッチ領域も挿入されていた.これらの挿入配列を除いたときのアメーバTET1と哺乳類のTET1とのあいだの相同性としては,14%の同一性,39%の類似性があった.このような相同性はDSBHドメインのみならずそれ以外の領域においても認められ,構造および機能において重要なアミノ酸残基は保存されていた.さらに,酵素の機能として,アメーバTET1は哺乳類のTETと同じく,5-メチルシトシンから5-ヒドロキシメチルシトシン,5-ホルミルシトシン,5-カルボキシルシトシンへと,Fe2+およびαケトグルタル酸に依存して酸化したことから,アメーバTET1とDNAとの共結晶構造はTETの普遍的な構造であることが示唆された.
おわりに
筆者らのグループは,アメーバTET1の結晶構造解析および生化学的な実験を行った.TETを含むTET/JBPスーパーファミリーにおいて,このスーパーファミリーを特徴づけるTET/JBPドメインは進化において散在して存在し5),これらのTETの基本構造は保存されていると考えられる.実際に,筆者らの論文と同じ時期に,ヒトTET2とDNAとの共結晶構造についても発表されている9).この論文では,TET2のDSBHドメインに存在する相同性の低い挿入配列を除去したタンパク質を用いて結晶化に成功している.そして,アメーバTET1とヒトTET2の構造を比較したところ,TETの構造は進化においておおむね保存されていることがわかった.
文 献
- Tahiliani, M., Kian, P. -K., Yinghua, S. et al.: Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science, 324, 930-935 (2009)[PubMed]
- Ito, S., D'Alessio, A. C., Taranova, O. V. et al.: Role of Tet proteins in 5mC to 5hmC conversion, ES-cell self-renewal and inner cell mass specification. Nature, 466, 1129-1133 (2010)[PubMed] [新着論文レビュー]
- Aik, W., McDonough, M. A., Thalhammer, A. et al.: Role of the jelly-roll fold in substrate binding by 2-oxoglutarate oxygenases. Curr. Opin. Struct. Biol., 22, 691-700 (2012)[PubMed]
- Ito, S., Li, S., Qing D. et al.: Tet proteins can convert 5-methylcytosine to 5-formylcytosine and 5-carboxylcytosine. Science, 333, 1300-1303 (2011)[PubMed]
- Iyer, L. M., Zhang, D., Burroughs, A. M. et al.: Computational identification of novel biochemical systems involved in oxidation, glycosylation and other complex modifications of bases in DNA. Nucleic Acids Res., 41, 7635-7655 (2013)[PubMed]
- Inoue, A., Shen, L., Dai, Q. et al.: Generation and replication-dependent dilution of 5fC and 5caC during mouse preimplantation development. Cell Res., 21, 1670-1676 (2011)[PubMed]
- Hashimoto, H., Hong, S., Bhagwat, A. S. et al.: Excision of 5-hydroxymethyluracil and 5-carboxylcytosine by the thymine DNA glycosylase domain: its structural basis and implications for active DNA demethylation. Nucleic Acids Res., 40, 10203-10214 (2012)[PubMed]
- Yi, C., Jia, G., Hou, G. et al.: Iron-catalysed oxidation intermediates captured in a DNA repair dioxygenase. Nature, 468, 330-333 (2010)[PubMed]
- Hu, L., Li, Z., Cheng, J. et al.: Crystal structure of TET2-DNA complex: insight into TET-mediated 5mC oxidation. Cell, 155, 1545-1555 (2013)[PubMed]
著者プロフィール
略歴:2007年 京都大学大学院生命科学研究科 修了,同年 米国Emory大学School of Medicineポストドクトラルフェローを経て,2012年より同 リサーチアソシエート.
研究テーマ:DNAメチル化にかかわるタンパク質の生化学とX線結晶構造解析.
抱負:Enjoy science.
© 2014 橋本 秀春 Licensed under CC 表示 2.1 日本