哺乳類におけるRNAポリメラーゼIIのC末端ドメインのリン酸化を介する転写と共役したスプライシングの機構
野島 孝之
(英国Oxford大学Sir William Dunn School of Pathology)
email:野島孝之
DOI: 10.7875/first.author.2018.111
RNA polymerase II phosphorylated on CTD serine 5 interacts with the spliceosome during co-transcriptional splicing.
Takayuki Nojima, Kenny Rebelo, Tomás Gomes, Ana Rita Grosso, Nicholas J. Proudfoot, Maria Carmo-Fonseca
Molecular Cell, 72, 369-379.e4 (2018)
哺乳類においてゲノムDNAに存在するタンパク質の情報はエキソンにコードされており,イントロンにより分断されている.遺伝子がその機能を発現する過程において,mRNA前駆体からスプライシングによりイントロンを除去する必要がある.スプライシングはRNAポリメラーゼIIによる転写の際に起こると考えられているが,その機構についてはいまだ不明な点が多い.この研究において,筆者らは,哺乳類NET-seq法および新たに開発された哺乳類NET-MS法を用いて,C末端ドメインが特定のリン酸化をうけたRNAポリメラーゼIIと活性型のスプライソソームとの特異的な相互作用を明らかにした.さらに,スプライシング中間体やRNAポリメラーゼII複合体に含まれるスプライシングをうけた新生RNAの解析から,哺乳類の細胞におけるエキソン認識スプライシングモデルが支持された.
哺乳類において細胞の核に存在するゲノムDNAには約2万のタンパク質をコードする遺伝子が存在し,RNAポリメラーゼIIによりmRNA前駆体に転写される.多くの場合,mRNA前駆体は複数のイントロンを含む.RNAポリメラーゼIIによる転写ののち,イントロンはスプライシングにより除去され,エキソンどうしが連結したmRNAへと成熟する.スプライシングは,5つのU snRNAおよび200以上のタンパク質から構成されるスプライソソームにおいて起こる1).スプライシングはひとつのステップではなく複数のステップからなる複雑な反応である.エキソンとイントロンの境界には進化的に保存された配列が存在し,これらの配列はスプライシングに必要不可欠である.まず,分岐部位の2’-OH基が5’側スプライス部位のリン酸基と求核置換反応を起こす.この段階で上流のエキソンは切断される.下流のエキソンは投げ縄状のイントロンとともにイントロン-エキソン構造を形成する.これらはスプライシング中間体とよばれ,活性型のスプライソソームに保持される.そののち,切断された上流のエキソンの5’側スプライス部位の3’-OH基が3’側スプライス部位のリン酸基と求核置換反応を起こすことにより,投げ縄状のイントロンは遊離し,上流のエキソンと下流のエキソンとが連結されスプライシングは完了する.このように複雑なスプライシングは,RNAポリメラーゼIIによる転写とすることにより正確かつ効率よく起こると考えられている2).
哺乳類のRNAポリメラーゼIIは,その最大サブユニットのC末端ドメインに進化的に保存された尻尾のような構造,すなわち,Tyr-Ser-Pro-Thr-Ser-Pro-Serが52回くり返した配列をもち,転写の際にはダイナミックにリン酸化され,そのリン酸化の変化によりスプライシングの精密な制御が実現されると考えられている2,3).以前に,筆者らは,C末端ドメインのリン酸化に依存的な新生RNAのプロファイルについて報告し,とくに,5番目のSerのリン酸化が転写の際のスプライシング中間体の保持およびエキソンにおけるRNAポリメラーゼIIの一時停止にかかわることを明らかにした4,5).(文献4) は 新着論文レビュー でも掲載,文献5) は 新着論文レビュー でも掲載).しかしながら,転写の際にRNAポリメラーゼIIと相互作用するタンパク質や,転写とスプライシングのキネティクスなど,時空間的な制御において不明な点が多く残されていた.
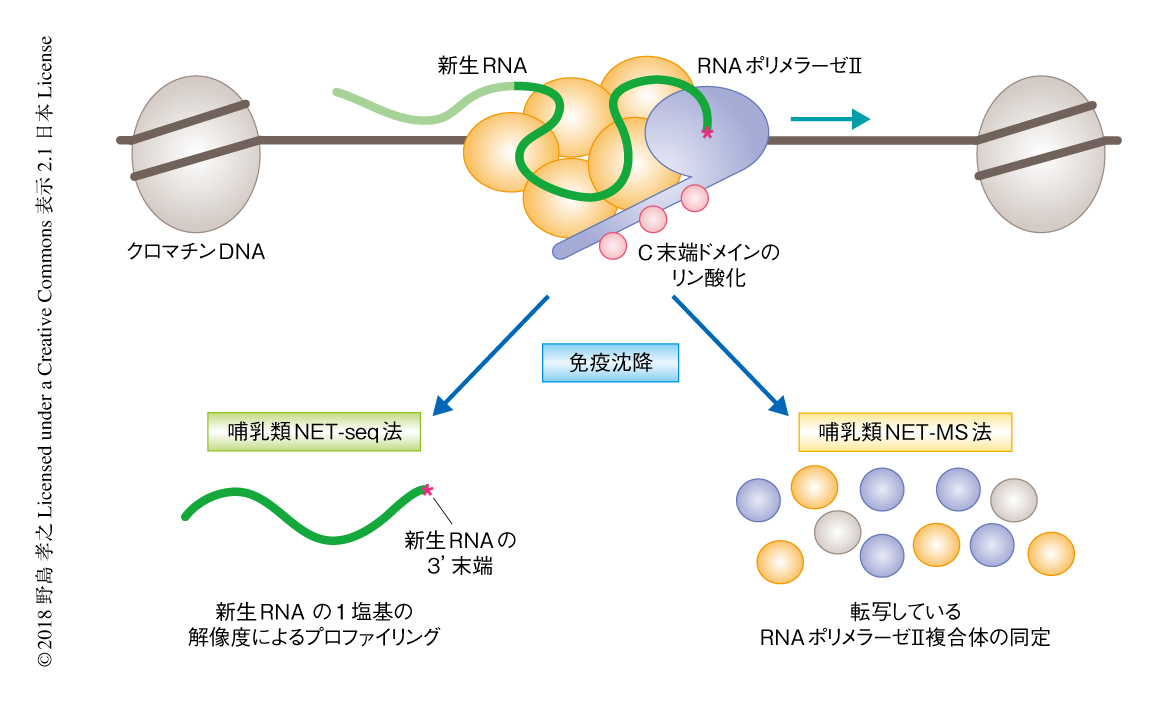
この研究において,筆者らは,RNAポリメラーゼIIと相互作用する複合体を網羅的に解析するため,哺乳類NET-seq法を派生させて,質量分析法を用いた哺乳類NET-MS法を開発した(図1).さらには,C末端ドメインのリン酸化がどのようにRNAポリメラーゼII複合体やスプライシングを制御するのかを解析した.
哺乳類NET-seq法は,クロマチン画分からRNAポリメラーゼIIにより転写された新生RNAの3’末端を1塩基の解像度で解析する手法である4).哺乳類NET-seq法により検出される新生RNAの3’末端は,大きく2種類に分類される.ひとつはRNAポリメラーゼIIの活性中心に存在する新生RNAの3’末端であり,もうひとつはRNAポリメラーゼIIと相互作用する複合体に含まれる新生RNAの3’末端である.以前に,筆者らは,これら2種類の新生RNAの3’末端を界面活性剤の一種であるEmpigenの処理により分離した5).また,さらなる解析により,RNAポリメラーゼIIと相互作用する複合体に含まれる新生RNAの3’末端のひとつとして,スプライシング中間体の5’側スプライス部位が同定された4).今回,スプライシング中間体を保持するRNAポリメラーゼII複合体を解析するため,哺乳類NET-seq法を派生させ質量分析を用いた哺乳類NET-MS法により,C末端ドメインが修飾されたRNAポリメラーゼIIと相互作用するタンパク質を網羅的に解析した.C末端ドメインの5番目のSerのリン酸化,2番目のSerのリン酸化,4番目のThrのリン酸化について,バイオインフォマティクス的な解析により,RNAポリメラーゼIIのサブユニット,ヒストン,クロマチンリモデリングタンパク質,スプライシングタンパク質,転写伸長因子,DNA修復タンパク質など,200以上のタンパク質がC末端ドメインが修飾されたRNAポリメラーゼIIと相互作用することが示された.なかでも,スプライシングタンパク質,とくに,活性型のスプライソソームに多く含まれるU2 snRNA結合タンパク質,U5 snRNA結合タンパク質,PRPF8,PRPF19などが,C末端ドメインの5番目のSerがリン酸化されたRNAポリメラーゼIIと相互作用するタンパク質として特異的に検出された.
クロマチン画分に含まれるRNAポリメラーゼII複合体をグリセロール勾配法により分画した.その結果,U1 snRNA結合タンパク質はRNAポリメラーゼIIの主要な画分に特異的に検出されたが,U5 snRNA結合タンパク質は高グリセロール画分に特異的に検出された.この画分には,C末端ドメインの5番目のSerがリン酸化されたRNAポリメラーゼIIも比較的多く検出された.さらに,哺乳類NET-seq法において,U5 snRNAおよびスプライシング中間体の5’側スプライス部位のシグナルは高グリセロール画分に特異的であることも示された.これらの結果から,C末端ドメインの5番目のSerがリン酸化されたRNAポリメラーゼIIは,クロマチンにおいて活性型のスプライソソームと複合体を形成することが示された.
哺乳類NET-seq法においては,マイクロコッカルヌクレアーゼを利用してクロマチンDNAおよび新生RNAを切断する.しかしながら,ある程度の長さをもつ新生RNAは,RNAポリメラーゼII複合体により保護される.保護される新生RNAの多くは長さ20~60塩基であるが,それより長い新生RNAも存在する.そこで,20~60塩基および60~150塩基の2つの画分を精製し哺乳類NET-seq法により解析した.その結果,RNAポリメラーゼII複合体はおもに,27塩基の短鎖を保護する領域と60塩基の長鎖を保護する領域をもつことが明らかにされた.短鎖を保護する領域は小さなRNAポリメラーゼII複合体に,長鎖を保護する領域は大きなRNAポリメラーゼII複合体にあると考えられた.長鎖を保護する領域には約70倍ものスプライシングをうけた新生RNAが検出された.この新生RNAのフットプリントは,ほかの研究グループにより報告されたスプライソソームに含まれるRNAのフットプリントと同様であり6),スプライシングの後期において新生RNAは巨大なスプライソソームにより保護されることが考えられた.
長鎖の新生RNAの画分に哺乳類NET-seq法を適用したところ,スプライシングをうけた新生RNAが多く検出された.そこで,この新生RNAがRNAポリメラーゼIIの活性中心に存在するのか,あるいは,スプライソソームなどRNAポリメラーゼIIと相互作用する複合体に含まれるのかを調べるため,RNAポリメラーゼIIとRNAポリメラーゼIIと相互作用する複合体とを分離するEmpigenによる処理を施した.その結果,Empigenの処理はエキソンやイントロンにおける哺乳類NET-seq法のシグナルには影響しなかったが,スプライシングをうけたRNAにおいて哺乳類NET-seq法のシグナルは大部分が消失した.この結果から,スプライシングののちの新生RNAはRNAポリメラーゼIIの活性中心に存在するのではなく,RNAポリメラーゼIIと相互作用する複合体に含まれることが示された.さらに,RNAポリメラーゼIIの活性中心に存在するスプライシングをうけた新生RNAは長鎖の新生RNAの画分には検出されなかったことから,スプライシングを完了させるためRNAポリメラーゼIIはさらに下流へと転写を進める必要があることが考えられた.哺乳類においてエキソンの平均の長さは130塩基であることから,スプライシングの完了はエキソンにおいては起こっておらず,下流のイントロンのどこかで起こると推察された.この結果から,スプライシングをうけるエキソンの上流のイントロンの3’側スプライス部位と,下流のイントロンの5’側スプライス部位の両者がエキソンの認識を強化するとする,エキソン認識スプライシングモデルが支持された7).近年,ほかの研究グループにより報告された出芽酵母におけるスプライシングのモデルとは異なっており8),スプライシングの制御機構の進化を考えるうえで興味深い.
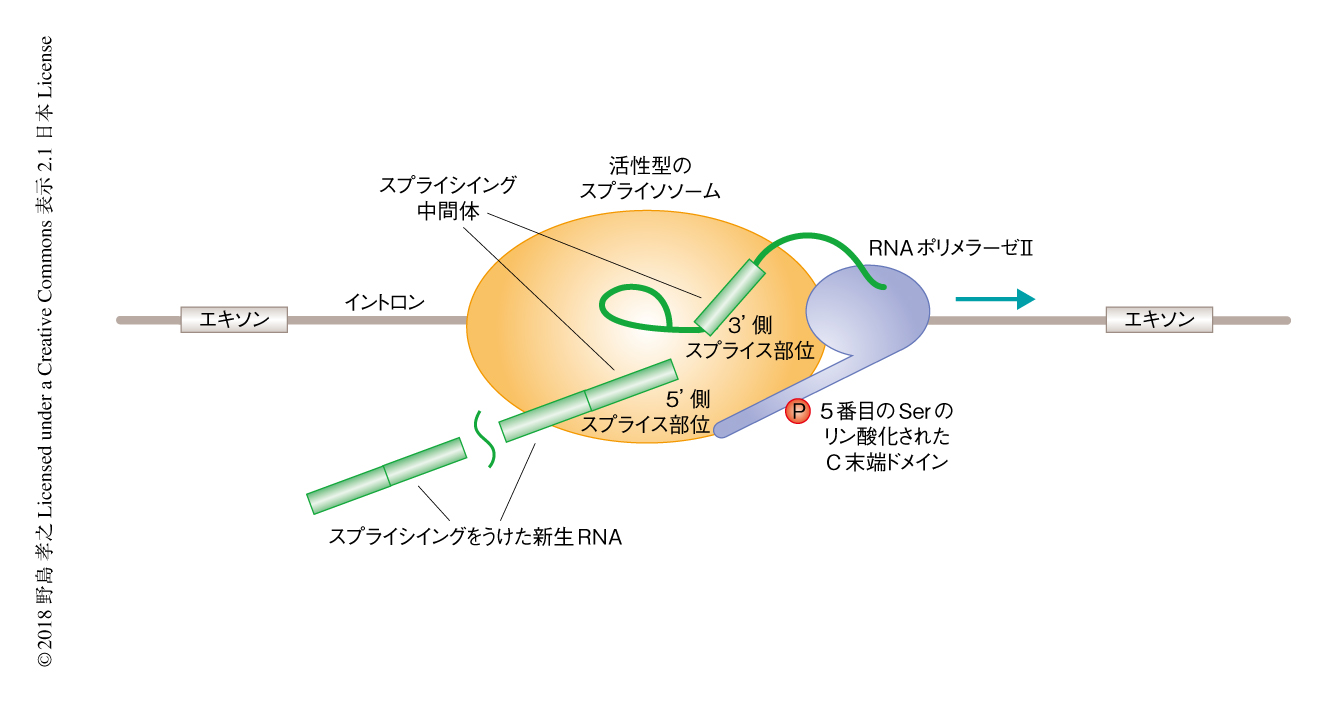
スプライシングをうけた新生RNAはどこに存在するのだろうか.スプライシングをうけた新生RNAにおいて哺乳類NET-seq法のシグナルを解析した結果,スプライシングをうけた新生RNAの3’末端はスプライシング中間体の5’側スプライス部位と一致した.この結果から,下流のイントロンを除去するためにRNAポリメラーゼIIが活性型のスプライソソームおよびスプライシング中間体を保持するときには,上流のイントロンを除去するためのスプライシングは完了していることが示された(図2).つまり,スプライシングは上流のイントロンから下流のイントロンへと連続的に起こりうることが示された.この現象は,スプライシングの速度が遅いと考えられているU12 snRNAに依存的なマイナーなイントロンにおいても同様であり9),また,ヒトとマウスに共通する機構であった.
この研究においては,エキソン認識スプライシングモデルを支持する結果が得られたが,シークエンサーのリード長などの技術的な制限から,スプライシングが完了したときRNAポリメラーゼIIはどこにいるのか,位置あるいは配列に特異的なRNAポリメラーゼIIの一時停止があるのかなど,いまだ不明な点が多い.これらは今後の重要な課題であり,1分子のRNAの解析が可能なシークエンサーの利用を検討している.これにより,スプライシングのより正確なキネティクスが明らかにされると思われる.とくに,RNAポリメラーゼIIの一時停止がどのように選択的スプライシングを制御するのかは興味深い.
この研究において,筆者らは,新たに開発した哺乳類NET-MS法を用いて,C末端ドメインの5番目のSerがリン酸化されたRNAポリメラーゼIIと活性型のスプライソソームとの特異的な相互作用を明らかにした.原理的には,内在性のRNAポリメラーゼIIとスプライソソームとの複合体をクライオ電子顕微鏡により解析することも可能である.このアプローチは,転写とスプライシングが共役する機構を構造の面から理解するのに役だつだろう.
哺乳類NET-MS法は哺乳類において転写を理解するうえで非常に強力な手法である.筆者らは,RNAポリメラーゼIIと相互作用するタンパク質として,スプライシングタンパク質だけでなく,クロマチンリモデリングタンパク質やDNA修復タンパク質など興味深いタンパク質を数多く検出している.さらに,免疫沈降に用いる抗体を変えることにより,C末端ドメインにおけるほかの残基のリン酸化,アセチル化,メチル化,ユビキチン化など,種々の修飾に特異的な相互作用も検出することが可能である.また,化合物の処理や紫外線の照射など外的な刺激のもとでのRNAポリメラーゼIIによる転写の制御を理解するのにも役だつ.哺乳類NET-seq法と哺乳類NET-MS法とを組み合わせることにより,新生RNAおよびRNAポリメラーゼII複合体を同時にプロファイリングすることが可能になり,哺乳類における転写のより深い理解が得られると考えている.
略歴:2006年 東京医科歯科大学大学院生命情報教育部博士課程 修了,同年 同 特任助教,2010年 英国Oxford大学 博士研究員を経て,2014年より同 シニアリサーチフェロー.
研究テーマ:RNAポリメラーゼIIによる転写およびスプライシングの制御機構.ノンコーディングRNAの転写とゲノムの安定性.
抱負:ノンコーディング領域によるゲノムの制御について理解し,がん細胞の増殖を抑制したい.
© 2018 野島 孝之 Licensed under CC 表示 2.1 日本
(英国Oxford大学Sir William Dunn School of Pathology)
email:野島孝之
DOI: 10.7875/first.author.2018.111
RNA polymerase II phosphorylated on CTD serine 5 interacts with the spliceosome during co-transcriptional splicing.
Takayuki Nojima, Kenny Rebelo, Tomás Gomes, Ana Rita Grosso, Nicholas J. Proudfoot, Maria Carmo-Fonseca
Molecular Cell, 72, 369-379.e4 (2018)
要 約
哺乳類においてゲノムDNAに存在するタンパク質の情報はエキソンにコードされており,イントロンにより分断されている.遺伝子がその機能を発現する過程において,mRNA前駆体からスプライシングによりイントロンを除去する必要がある.スプライシングはRNAポリメラーゼIIによる転写の際に起こると考えられているが,その機構についてはいまだ不明な点が多い.この研究において,筆者らは,哺乳類NET-seq法および新たに開発された哺乳類NET-MS法を用いて,C末端ドメインが特定のリン酸化をうけたRNAポリメラーゼIIと活性型のスプライソソームとの特異的な相互作用を明らかにした.さらに,スプライシング中間体やRNAポリメラーゼII複合体に含まれるスプライシングをうけた新生RNAの解析から,哺乳類の細胞におけるエキソン認識スプライシングモデルが支持された.
はじめに
哺乳類において細胞の核に存在するゲノムDNAには約2万のタンパク質をコードする遺伝子が存在し,RNAポリメラーゼIIによりmRNA前駆体に転写される.多くの場合,mRNA前駆体は複数のイントロンを含む.RNAポリメラーゼIIによる転写ののち,イントロンはスプライシングにより除去され,エキソンどうしが連結したmRNAへと成熟する.スプライシングは,5つのU snRNAおよび200以上のタンパク質から構成されるスプライソソームにおいて起こる1).スプライシングはひとつのステップではなく複数のステップからなる複雑な反応である.エキソンとイントロンの境界には進化的に保存された配列が存在し,これらの配列はスプライシングに必要不可欠である.まず,分岐部位の2’-OH基が5’側スプライス部位のリン酸基と求核置換反応を起こす.この段階で上流のエキソンは切断される.下流のエキソンは投げ縄状のイントロンとともにイントロン-エキソン構造を形成する.これらはスプライシング中間体とよばれ,活性型のスプライソソームに保持される.そののち,切断された上流のエキソンの5’側スプライス部位の3’-OH基が3’側スプライス部位のリン酸基と求核置換反応を起こすことにより,投げ縄状のイントロンは遊離し,上流のエキソンと下流のエキソンとが連結されスプライシングは完了する.このように複雑なスプライシングは,RNAポリメラーゼIIによる転写とすることにより正確かつ効率よく起こると考えられている2).
哺乳類のRNAポリメラーゼIIは,その最大サブユニットのC末端ドメインに進化的に保存された尻尾のような構造,すなわち,Tyr-Ser-Pro-Thr-Ser-Pro-Serが52回くり返した配列をもち,転写の際にはダイナミックにリン酸化され,そのリン酸化の変化によりスプライシングの精密な制御が実現されると考えられている2,3).以前に,筆者らは,C末端ドメインのリン酸化に依存的な新生RNAのプロファイルについて報告し,とくに,5番目のSerのリン酸化が転写の際のスプライシング中間体の保持およびエキソンにおけるRNAポリメラーゼIIの一時停止にかかわることを明らかにした4,5).(文献4) は 新着論文レビュー でも掲載,文献5) は 新着論文レビュー でも掲載).しかしながら,転写の際にRNAポリメラーゼIIと相互作用するタンパク質や,転写とスプライシングのキネティクスなど,時空間的な制御において不明な点が多く残されていた.
この研究において,筆者らは,RNAポリメラーゼIIと相互作用する複合体を網羅的に解析するため,哺乳類NET-seq法を派生させて,質量分析法を用いた哺乳類NET-MS法を開発した(図1).さらには,C末端ドメインのリン酸化がどのようにRNAポリメラーゼII複合体やスプライシングを制御するのかを解析した.
1.活性型のスプライソソームとC末端ドメインの5番目のSerがリン酸化されたRNAポリメラーゼIIとの特異的な相互作用
哺乳類NET-seq法は,クロマチン画分からRNAポリメラーゼIIにより転写された新生RNAの3’末端を1塩基の解像度で解析する手法である4).哺乳類NET-seq法により検出される新生RNAの3’末端は,大きく2種類に分類される.ひとつはRNAポリメラーゼIIの活性中心に存在する新生RNAの3’末端であり,もうひとつはRNAポリメラーゼIIと相互作用する複合体に含まれる新生RNAの3’末端である.以前に,筆者らは,これら2種類の新生RNAの3’末端を界面活性剤の一種であるEmpigenの処理により分離した5).また,さらなる解析により,RNAポリメラーゼIIと相互作用する複合体に含まれる新生RNAの3’末端のひとつとして,スプライシング中間体の5’側スプライス部位が同定された4).今回,スプライシング中間体を保持するRNAポリメラーゼII複合体を解析するため,哺乳類NET-seq法を派生させ質量分析を用いた哺乳類NET-MS法により,C末端ドメインが修飾されたRNAポリメラーゼIIと相互作用するタンパク質を網羅的に解析した.C末端ドメインの5番目のSerのリン酸化,2番目のSerのリン酸化,4番目のThrのリン酸化について,バイオインフォマティクス的な解析により,RNAポリメラーゼIIのサブユニット,ヒストン,クロマチンリモデリングタンパク質,スプライシングタンパク質,転写伸長因子,DNA修復タンパク質など,200以上のタンパク質がC末端ドメインが修飾されたRNAポリメラーゼIIと相互作用することが示された.なかでも,スプライシングタンパク質,とくに,活性型のスプライソソームに多く含まれるU2 snRNA結合タンパク質,U5 snRNA結合タンパク質,PRPF8,PRPF19などが,C末端ドメインの5番目のSerがリン酸化されたRNAポリメラーゼIIと相互作用するタンパク質として特異的に検出された.
クロマチン画分に含まれるRNAポリメラーゼII複合体をグリセロール勾配法により分画した.その結果,U1 snRNA結合タンパク質はRNAポリメラーゼIIの主要な画分に特異的に検出されたが,U5 snRNA結合タンパク質は高グリセロール画分に特異的に検出された.この画分には,C末端ドメインの5番目のSerがリン酸化されたRNAポリメラーゼIIも比較的多く検出された.さらに,哺乳類NET-seq法において,U5 snRNAおよびスプライシング中間体の5’側スプライス部位のシグナルは高グリセロール画分に特異的であることも示された.これらの結果から,C末端ドメインの5番目のSerがリン酸化されたRNAポリメラーゼIIは,クロマチンにおいて活性型のスプライソソームと複合体を形成することが示された.
2.新生RNAのフットプリント解析
哺乳類NET-seq法においては,マイクロコッカルヌクレアーゼを利用してクロマチンDNAおよび新生RNAを切断する.しかしながら,ある程度の長さをもつ新生RNAは,RNAポリメラーゼII複合体により保護される.保護される新生RNAの多くは長さ20~60塩基であるが,それより長い新生RNAも存在する.そこで,20~60塩基および60~150塩基の2つの画分を精製し哺乳類NET-seq法により解析した.その結果,RNAポリメラーゼII複合体はおもに,27塩基の短鎖を保護する領域と60塩基の長鎖を保護する領域をもつことが明らかにされた.短鎖を保護する領域は小さなRNAポリメラーゼII複合体に,長鎖を保護する領域は大きなRNAポリメラーゼII複合体にあると考えられた.長鎖を保護する領域には約70倍ものスプライシングをうけた新生RNAが検出された.この新生RNAのフットプリントは,ほかの研究グループにより報告されたスプライソソームに含まれるRNAのフットプリントと同様であり6),スプライシングの後期において新生RNAは巨大なスプライソソームにより保護されることが考えられた.
3.哺乳類の細胞におけるエキソン認識スプライシングモデル
長鎖の新生RNAの画分に哺乳類NET-seq法を適用したところ,スプライシングをうけた新生RNAが多く検出された.そこで,この新生RNAがRNAポリメラーゼIIの活性中心に存在するのか,あるいは,スプライソソームなどRNAポリメラーゼIIと相互作用する複合体に含まれるのかを調べるため,RNAポリメラーゼIIとRNAポリメラーゼIIと相互作用する複合体とを分離するEmpigenによる処理を施した.その結果,Empigenの処理はエキソンやイントロンにおける哺乳類NET-seq法のシグナルには影響しなかったが,スプライシングをうけたRNAにおいて哺乳類NET-seq法のシグナルは大部分が消失した.この結果から,スプライシングののちの新生RNAはRNAポリメラーゼIIの活性中心に存在するのではなく,RNAポリメラーゼIIと相互作用する複合体に含まれることが示された.さらに,RNAポリメラーゼIIの活性中心に存在するスプライシングをうけた新生RNAは長鎖の新生RNAの画分には検出されなかったことから,スプライシングを完了させるためRNAポリメラーゼIIはさらに下流へと転写を進める必要があることが考えられた.哺乳類においてエキソンの平均の長さは130塩基であることから,スプライシングの完了はエキソンにおいては起こっておらず,下流のイントロンのどこかで起こると推察された.この結果から,スプライシングをうけるエキソンの上流のイントロンの3’側スプライス部位と,下流のイントロンの5’側スプライス部位の両者がエキソンの認識を強化するとする,エキソン認識スプライシングモデルが支持された7).近年,ほかの研究グループにより報告された出芽酵母におけるスプライシングのモデルとは異なっており8),スプライシングの制御機構の進化を考えるうえで興味深い.
4.転写の際に起こる連続的なスプライシング
スプライシングをうけた新生RNAはどこに存在するのだろうか.スプライシングをうけた新生RNAにおいて哺乳類NET-seq法のシグナルを解析した結果,スプライシングをうけた新生RNAの3’末端はスプライシング中間体の5’側スプライス部位と一致した.この結果から,下流のイントロンを除去するためにRNAポリメラーゼIIが活性型のスプライソソームおよびスプライシング中間体を保持するときには,上流のイントロンを除去するためのスプライシングは完了していることが示された(図2).つまり,スプライシングは上流のイントロンから下流のイントロンへと連続的に起こりうることが示された.この現象は,スプライシングの速度が遅いと考えられているU12 snRNAに依存的なマイナーなイントロンにおいても同様であり9),また,ヒトとマウスに共通する機構であった.
おわりに
この研究においては,エキソン認識スプライシングモデルを支持する結果が得られたが,シークエンサーのリード長などの技術的な制限から,スプライシングが完了したときRNAポリメラーゼIIはどこにいるのか,位置あるいは配列に特異的なRNAポリメラーゼIIの一時停止があるのかなど,いまだ不明な点が多い.これらは今後の重要な課題であり,1分子のRNAの解析が可能なシークエンサーの利用を検討している.これにより,スプライシングのより正確なキネティクスが明らかにされると思われる.とくに,RNAポリメラーゼIIの一時停止がどのように選択的スプライシングを制御するのかは興味深い.
この研究において,筆者らは,新たに開発した哺乳類NET-MS法を用いて,C末端ドメインの5番目のSerがリン酸化されたRNAポリメラーゼIIと活性型のスプライソソームとの特異的な相互作用を明らかにした.原理的には,内在性のRNAポリメラーゼIIとスプライソソームとの複合体をクライオ電子顕微鏡により解析することも可能である.このアプローチは,転写とスプライシングが共役する機構を構造の面から理解するのに役だつだろう.
哺乳類NET-MS法は哺乳類において転写を理解するうえで非常に強力な手法である.筆者らは,RNAポリメラーゼIIと相互作用するタンパク質として,スプライシングタンパク質だけでなく,クロマチンリモデリングタンパク質やDNA修復タンパク質など興味深いタンパク質を数多く検出している.さらに,免疫沈降に用いる抗体を変えることにより,C末端ドメインにおけるほかの残基のリン酸化,アセチル化,メチル化,ユビキチン化など,種々の修飾に特異的な相互作用も検出することが可能である.また,化合物の処理や紫外線の照射など外的な刺激のもとでのRNAポリメラーゼIIによる転写の制御を理解するのにも役だつ.哺乳類NET-seq法と哺乳類NET-MS法とを組み合わせることにより,新生RNAおよびRNAポリメラーゼII複合体を同時にプロファイリングすることが可能になり,哺乳類における転写のより深い理解が得られると考えている.
文 献
- Papasaikas, P. & Valcarcel, J.: The spliceosome: the ultimate RNA chaperone and sculptor. Trends Biochem. Sci., 41, 33-45 (2016)[PubMed]
- Zaborowska, J., Egloff, S. & Murphy, S.: The pol II CTD: new twists in the tail. Nat. Struct. Mol. Biol., 23, 771-777 (2016)[PubMed]
- Heidemann, M., Hintermair, C., Voss, K. et al.: Dynamic phosphorylation patterns of RNA polymerase II CTD during transcription. Biochim. Biophys. Acta, 1829, 55-62 (2013)[PubMed]
- Nojima, T., Gomes, T., Grosso, A. R. et al.: Mammalian NET-Seq reveals genome-wide nascent transcription coupled to RNA processing. Cell, 161, 526-540 (2015)[PubMed] [新着論文レビュー]
- Schlackow, M., Nojima, T., Gomes, T. et al.: Distinctive patterns of transcription and RNA processing for human lincRNAs. Mol. Cell, 65, 25-38 (2017)[PubMed] [新着論文レビュー]
- Chen, W., Moore, J., Ozadam, H. et al.: Transcriptome-wide interrogation of the functional intronome by spliceosome profiling. Cell, 173, 1031-1044.e13 (2018)[PubMed]
- Keren, H., Lev-Maor, G. & Ast, G.: Alternative splicing and evolution: diversification, exon definition and function. Nat. Rev. Genet., 11, 345-355 (2010)[PubMed]
- Oesterreich, F. C., Herzel, L., Straube, K. et al.: Splicing of nascent RNA coincides with intron exit from RNA polymerase II. Cell, 165, 372-381 (2016)[PubMed]
- Turunen, J. J., Niemela, E. H., Verma, B. et al.: The significant other: splicing by the minor spliceosome. Wiley Interdiscip. Rev. RNA, 4, 61-76 (2013)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2006年 東京医科歯科大学大学院生命情報教育部博士課程 修了,同年 同 特任助教,2010年 英国Oxford大学 博士研究員を経て,2014年より同 シニアリサーチフェロー.
研究テーマ:RNAポリメラーゼIIによる転写およびスプライシングの制御機構.ノンコーディングRNAの転写とゲノムの安定性.
抱負:ノンコーディング領域によるゲノムの制御について理解し,がん細胞の増殖を抑制したい.
© 2018 野島 孝之 Licensed under CC 表示 2.1 日本