哺乳類NET-seq法:RNAプロセシングと共役した新生転写反応の網羅的な解析
野島 孝之
(英国Oxford大学Sir William Dunn School of Pathology)
email:野島孝之
DOI: 10.7875/first.author.2015.058
Mammalian NET-seq reveals genome-wide nascent transcription coupled to RNA processing.
Takayuki Nojima, Tomás Gomes, Ana Rita Fialho Grosso, Hiroshi Kimura, Michael J. Dye, Somdutta Dhir, Maria Carmo-Fonseca, Nicholas J. Proudfoot
Cell, 161, 526-540 (2015)
RNAポリメラーゼIIによる転写はダイナミックな反応である.今回,筆者らは,哺乳類細胞において1塩基の解像度でRNAポリメラーゼIIによる転写をとらえることのできる手法を開発した.この哺乳類NET-seq法は,RNAポリメラーゼIIの転写活性部位をゲノムにおいて正確にマッピングすることができ,さらに,スプライシング中間体やマイクロRNA前駆体などRNAプロセシング反応の中間産物を検出することができる.この手法により,RNAポリメラーゼIIのC末端ドメインにおける5番目のセリン残基のリン酸化が,転写と共役したスプライシングの制御にかかわることが明らかにされた.また,転写終結因子がポリAシグナルの下流においてRNAポリメラーゼIIの速度を低下させ転写の終結を促進していることが示唆された.さらに,RNAポリメラーゼIIのC末端ドメインにおける2番目のセリン残基のリン酸化に特異的に,転写終結因子が遺伝子プロモーターの近傍において非生産的な転写産物の代謝にかかわることが明らかにされた.以上のように,哺乳類NET-seq法はRNAポリメラーゼIIのC末端ドメインのリン酸化状態に特異的な新生RNAの超高解像度での解析を可能にし,哺乳類においてRNAポリメラーゼIIによる複雑な転写反応を理解する強力なツールになると期待される.
RNAポリメラーゼIIにより合成された新生RNAは,転写反応の過程において修飾され成熟した機能性mRNAになる1).まず,新生RNAの5’末端にキャップ構造が付加され,そののち,スプライシングによりイントロンが除去される.スプライシングはU snRNPによるスプライス部位の認識からはじまる.上流のエキソンは切断され,3’末端にはOH基が生じる.切断された上流のエキソンは下流のエキソンと結合し転写複合体に保持され,イントロンはスプライソソームから放出される.また,マイクロRNA前駆体の多くはイントロンに存在し,転写と共役した新生RNAの切断により産生される.さらに,最下流ではポリA化複合体によりポリAシグナルが認識され,その下流において新生RNAは切断され,そののち,ポリA鎖が付加される.ポリA鎖の付加は転写の終結に不可欠であると考えられている.これら多くの知見は試験官内反応系を中心に得られており,転写と共役したRNAプロセシングを細胞内に近い条件において詳細に解析した例はまだない.
RNAポリメラーゼIIによる転写反応がダイナミックであるのには理由がある.RNAポリメラーゼIIのC末端ドメインは特徴的な7アミノ酸残基を52回くり返し高度にリン酸化されている.転写反応においてそのリン酸化状態の組合せは非常に複雑なものになり,その複雑さがRNAプロセシングにかかわるタンパク質の部位特異的な結合に重要であると考えられている2).このC末端ドメインのリン酸化状態によるRNAポリメラーゼIIの転写制御は“CTDコード”といわれ,一般的に広く受け入れられてはいるが技術的な問題によりいまだ不明な点が多い.
RNAポリメラーゼIIによる転写反応と共役したRNAプロセンシングを詳細に解析するためには,合成された直後の新生RNAを解析する技術が必要になる.新生RNAを解析する代表的な技術としてはGRO-seq法およびPRO-seq法があげられる3,4).これらの方法では,修飾塩基を取り込ませることにより新生RNAを標識し,修飾塩基に特異的な抗体によりこれを精製する.また,クロマチン画分に含まれる転写産物をキャップ構造に特異的な抗体により精製しその3’末端を解析する3’NT法もある5).これらは,新生RNA,とくに,RNAポリメラーゼIIがゲノムのどこで一時停止するかを解析するのに有効な方法であるが,RNAポリメラーゼIIのC末端ドメインのリン酸化状態や転写と共役したRNAプロセシングを解析することは不可能である.
では,どのようにしたらRNAポリメラーゼIIのC末端ドメインのリン酸化状態に特異的に新生RNAを解析できるだろうか.RNAポリメラーゼII転写複合体に含まれる新生RNAを解析する方法としてNET-seq(native elongating transcript sequencing)法が報告されている6).この方法は酵母を用いて開発され大腸菌にも応用されている7).NET-seq法では,Flagタグを付加したRNAポリメラーゼIIの遺伝子をゲノムに挿入し,Flag抗体により免疫沈降したRNAポリメラーゼII転写複合体に含まれる新生RNAの3’末端,つまり,RNAポリメラーゼIIの転写活性部位を解析する.Flagタグによる単離ではC末端ドメインのリン酸化状態はみわけられないが,RNAポリメラーゼII転写複合体を免疫沈降する際にC末端ドメインのリン酸化状態を特異的に認識する抗体を用いることにより理論的には区別が可能になる.しかし,これまでNET-seq法を哺乳類細胞において応用した例はなく,C末端ドメインのリン酸化状態と新生RNAとを同時に調べた例はなかった.筆者らは,低濃度の尿素により処理したクロマチン画分に活性をもつRNAポリメラーゼIIが濃縮されることを見い出し,C末端ドメインのリン酸化状態を特異的に認識する抗体によりRNAポリメラーゼII複合体に含まれる新生RNAを単離することに成功し,この方法を哺乳類NET-seq法と名づけた.
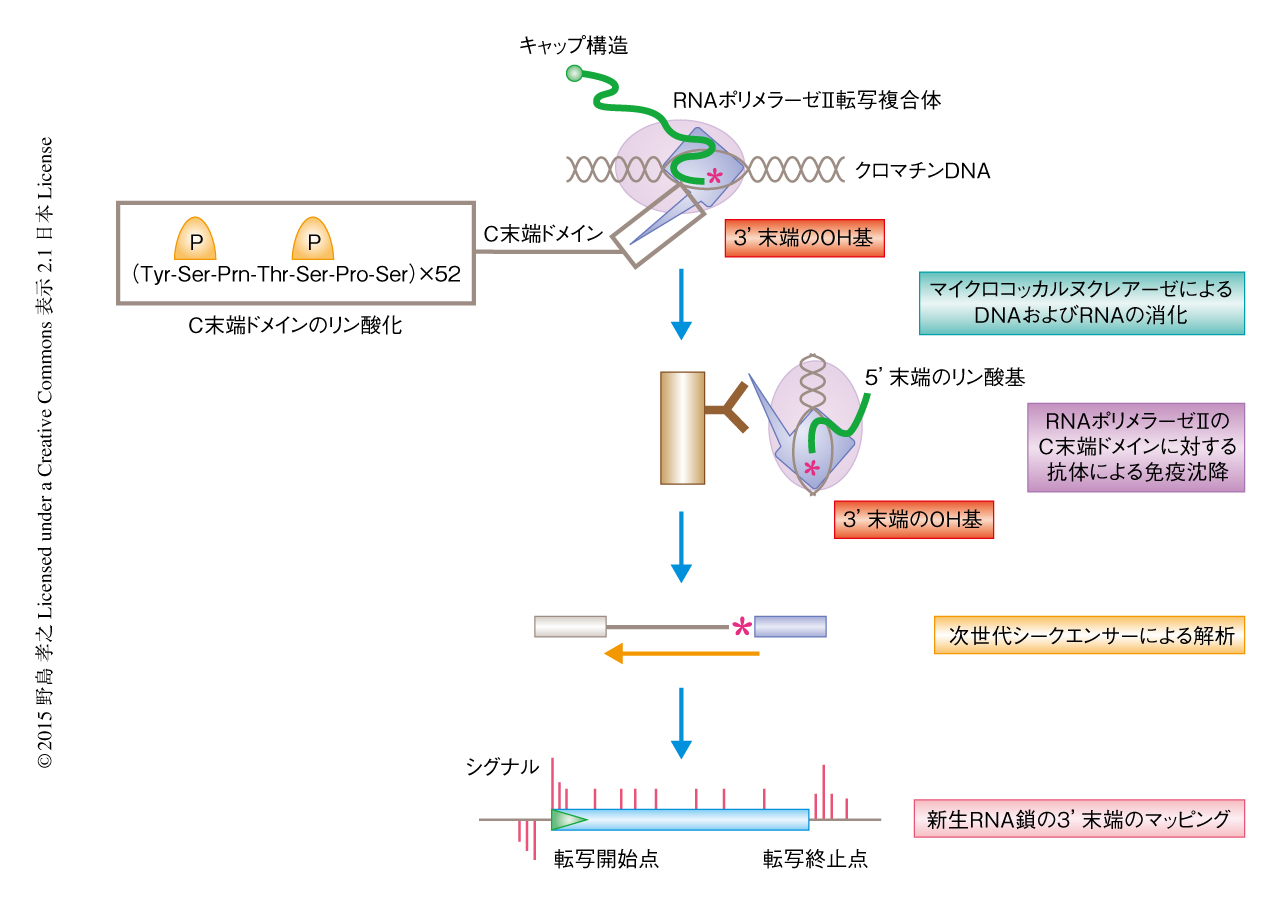
哺乳類NET-seq法の原理は至極単純である(図1).哺乳類細胞のクロマチン画分をマイクロコッカルヌクレアーゼにより可溶化したのち,抗RNAポリメラーゼII抗体により免疫沈降し,得られたRNAポリメラーゼII転写複合体に含まれる新生RNAの断片を次世代シークエンサーにより解析した.さらに,得られたリード配列の5’末端をバイオインフォマティクス解析によりヒトのゲノムにマッピングし,RNAポリメラーゼII転写複合体に含まれる新生RNAの3’末端の位置情報,つまり,RNAポリメラーゼIIの転写活性部位を1塩基の解像度で明らかにした.哺乳類NET-seq法の最大の利点は,免疫沈降の際に異なる抗RNAポリメラーゼII抗体を使用できることである.リン酸化されていないC末端ドメインを認識する抗RNAポリメラーゼII抗体を用いた場合,新生RNAは転写開始点の近傍に多く検出された.一方,2番目のセリン残基がリン酸化されたC末端ドメインを特異的に認識する抗RNAポリメラーゼII抗体を用いた場合,新生RNAは転写終結点の下流に多く検出された.これらの結果から,哺乳類NET-seq法はC末端ドメインのリン酸化状態に特異的な新生RNAのプロファイルを1塩基のレベルで網羅的に解析できることを示した.
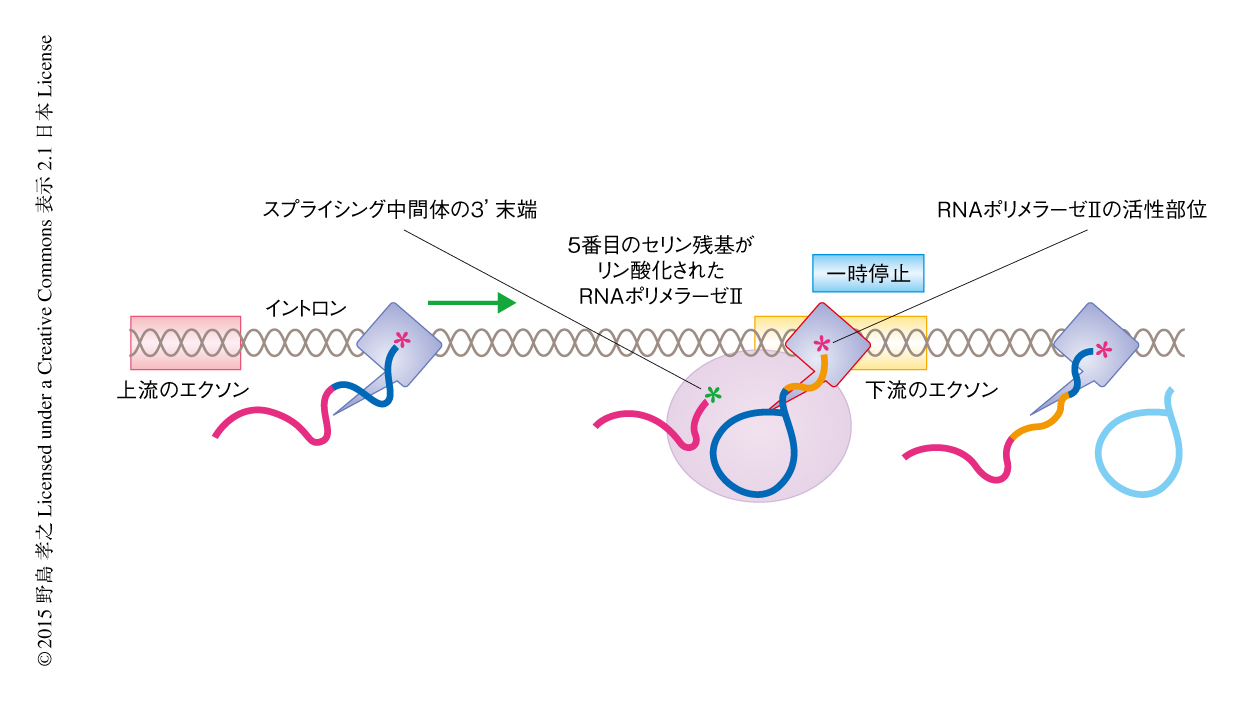
哺乳類NET-seq法により,5番目のセリン残基がリン酸化されたC末端ドメインに特異的な抗RNAポリメラーゼII抗体を用いることによりエキソンの3’末端に1塩基のピークが検出された.このピークは選択されていないエキソンには検出されず,さらに,スプラシシングの阻害剤の存在により減弱したことから,スプライシングに依存的であると結論づけられた.スプライシング中間体の上流のエキソンはスプライソソームにより切断され3’末端にOH基を生じることから,このエキソンの3’末端が検出されたことが示唆された.この結果は,哺乳類NET-seq法はRNAポリメラーゼIIの転写活性部位だけでなく,スプライソソームなどRNAポリメラーゼII転写複合体において起こる新生RNAの切断にともなう3’末端のOH基も検出できることを示した(図2).哺乳類NET-seq法により,イントロンよりエキソンにおいてより多くのシグナルが検出され,このシグナルは5番目のセリン残基がリン酸化されたC末端ドメインに特異的であった.これらの知見から,転写反応においてRNAポリメラーゼIIはイントロンとエキソンの境界で減速し,そのことが活性化したエキソンの認識に重要であることが示唆された.また,5番目のセリン残基がリン酸化されたC末端ドメインはエキソンにおけるRNAポリメラーゼIIの速度の制御およびスプライソソームとの相互作用に密接にかかわることが考えられた.
哺乳類においてマイクロRNA前駆体の多くはイントロンに存在し,2本鎖RNA鎖に特異的なヌクレアーゼ複合体であるDrosha/DGCR8マイクロプロセッサーにより転写反応の過程において切断される8).この新生RNAの切断により上流のRNA鎖およびマイクロRNA前駆体の3’末端にOH基が生じる.哺乳類NET-seq法により,転写反応において生じるマイクロRNA前駆体の切断において1塩基のピークが検出された.マイクロRNAとして,has-mir-27bやhas-mir-21においては前駆体の5’側および3’側の部位にピークが検出されたが,has-mir-26bやhas-mir-181a-1においては前駆体の5’側の部位にのみピークが検出された.これらの結果は,RNAポリメラーゼII転写複合体からのマイクロRNA前駆体の放出に関して少なくとも2種類のモードが存在することを示唆した.5’側の部位のピークは切断された上流のRNA鎖の3’末端のOH基を示し,3’側の部位のピークはマイクロRNA前駆体の3’末端のOH基を示すと考えられた.5’側および3’側の部位にピークが検出されたマイクロRNA前駆体は切断ののちRNAポリメラーゼII複合体にとどまりやすいが,5’側の部位にのみピークが検出されるマイクロRNA前駆体はただちに放出されると考えられた.重要なことに,これらマイクロRNA前駆体とRNAポリメラーゼII複合体との相互作用はリン酸化されていないC末端ドメインを認識する抗RNAポリメラーゼII抗体では検出されなかったことから,C末端ドメインのリン酸化状態が重要であることが示された.
RNAポリメラーゼIIによる転写の終結はポリAシグナルに大きく依存することが知られており,mRNA前駆体はポリAシグナルから10~30塩基ほど下流において切断されポリA鎖が付加される9).この新生RNAの切断はポリA化複合体の構成タンパク質であるCPSF73によるもので,CstF64はこの切断を促進することが知られている.哺乳類NET-seq法により,シグナルは転写終結領域に有意に蓄積していたことから,転写終結領域におけるRNAポリメラーゼII転写反応に対するポリA化複合体の影響について調べた.ポリA化複合体の種々の構成タンパク質をsiRNA法によりノックダウンした結果,転写の終結に大きな破綻がみられた.さらに,哺乳類NET-seq法により,このノックダウンのもとでは転写終結領域においてシグナルが減少していたことから,転写の終結の破綻はRNAポリメラーゼIIのC末端ドメインのリン酸化には依存していないことが示唆された.これらの結果から,ポリA化複合体は転写終結部位においてRNAポリメラーゼIIを一時停止させることにより,新生RNAの切断およびポリA鎖の付加のための時間をあたえ,転写終結を促進するというモデルが示唆された.
哺乳類NET-seq法により,転写開始点の近傍のセンス鎖およびアンチセンス鎖にもっとも大きな新生RNAのピークが検出された.この結果は,ほかの方法による新生RNAの検出とも一致しており,遺伝子プロモーターの近傍におけるRNAポリメラーゼIIの一時停止を示した.この一時停止は新生RNAの品質管理にかかわると考えられており,遺伝子の発現制御における重要なステップである10).ポリA化複合体による新生RNAの切断にともなう転写終結の機構は,遺伝子プロモーターの近傍におけるRNAポリメラーゼIIの一時停止と関連があるのではないかと考え,ポリA化複合体の構成タンパク質をノックダウンした.その結果,哺乳類NET-seq法により,CPSF73あるいはCstF64/64tauのノックダウンは転写開始点の近傍のセンス鎖およびアンチセンス鎖において有意にシグナルを増加させた.興味深いことに,このシグナルの増加は2番目のセリン残基がリン酸化されたC末端ドメインに特異的であった.また,転写の終結にかかわる5’-3’エクソヌクレアーゼXrn2をノックダウンも同様の結果を示した.さらに,CLIP(crosslinking and immunoprecipitation,架橋および免疫沈降法)法により,転写開始点の近傍におけるポリA化複合体の構成タンパク質の新生RNAに対する直接的な結合能が示された.以上の知見から,ポリA化複合体を含む転写終結複合体が転写開始点の近傍における非生産的な転写産物の代謝にかかわることが強く示唆された.
哺乳類NET-seq法は新生RNAのプロファイルおよびRNAポリメラーゼIIのC末端ドメインのリン酸化状態を同時かつ高解像度で解析できる唯一の方法である.5番目のセリン残基がリン酸化されたC末端ドメインを認識する抗RNAポリメラーゼII抗体がスプライシング中間体を選択的に単離できることを示したように,特定のC末端ドメイン修飾抗体を用いることにより特定の新生RNAを濃縮でき,転写反応における新たな現象を発見できる可能性がある.また,哺乳類NET-seq法は簡便であり,あらゆる細胞に応用が可能である.今後,医薬品の候補となる化合物の転写反応への影響を調べたり,細胞が分化するときの遺伝子発現の変化を観察したりすることなどに用いることで,いままでみすごされていた不安定なRNAが同定されるかもしれない.CTDコードがおりなす新生RNAワールドへの扉は,たったいま開かれたばかりである.近い将来,転写反応における多くの現象が哺乳類NET-seq法により明らかになる日を楽しみにしている.
略歴:2006年 東京医科歯科大学大学院生命情報教育部博士課程 修了,同年 同 特任助教,2010年 英国Oxford大学 博士研究員を経て,2014年より同 シニアリサーチフェロー.
研究テーマ:哺乳類細胞におけるRNAポリメラーゼIIによる新生転写反応とCTDコード.
抱負:哺乳類のゲノムにうもれている新生RNAを発掘し,その発現制御の機構および生理学的な機能を明らかにしたい.
© 2015 野島 孝之 Licensed under CC 表示 2.1 日本
(英国Oxford大学Sir William Dunn School of Pathology)
email:野島孝之
DOI: 10.7875/first.author.2015.058
Mammalian NET-seq reveals genome-wide nascent transcription coupled to RNA processing.
Takayuki Nojima, Tomás Gomes, Ana Rita Fialho Grosso, Hiroshi Kimura, Michael J. Dye, Somdutta Dhir, Maria Carmo-Fonseca, Nicholas J. Proudfoot
Cell, 161, 526-540 (2015)
要 約
RNAポリメラーゼIIによる転写はダイナミックな反応である.今回,筆者らは,哺乳類細胞において1塩基の解像度でRNAポリメラーゼIIによる転写をとらえることのできる手法を開発した.この哺乳類NET-seq法は,RNAポリメラーゼIIの転写活性部位をゲノムにおいて正確にマッピングすることができ,さらに,スプライシング中間体やマイクロRNA前駆体などRNAプロセシング反応の中間産物を検出することができる.この手法により,RNAポリメラーゼIIのC末端ドメインにおける5番目のセリン残基のリン酸化が,転写と共役したスプライシングの制御にかかわることが明らかにされた.また,転写終結因子がポリAシグナルの下流においてRNAポリメラーゼIIの速度を低下させ転写の終結を促進していることが示唆された.さらに,RNAポリメラーゼIIのC末端ドメインにおける2番目のセリン残基のリン酸化に特異的に,転写終結因子が遺伝子プロモーターの近傍において非生産的な転写産物の代謝にかかわることが明らかにされた.以上のように,哺乳類NET-seq法はRNAポリメラーゼIIのC末端ドメインのリン酸化状態に特異的な新生RNAの超高解像度での解析を可能にし,哺乳類においてRNAポリメラーゼIIによる複雑な転写反応を理解する強力なツールになると期待される.
はじめに
RNAポリメラーゼIIにより合成された新生RNAは,転写反応の過程において修飾され成熟した機能性mRNAになる1).まず,新生RNAの5’末端にキャップ構造が付加され,そののち,スプライシングによりイントロンが除去される.スプライシングはU snRNPによるスプライス部位の認識からはじまる.上流のエキソンは切断され,3’末端にはOH基が生じる.切断された上流のエキソンは下流のエキソンと結合し転写複合体に保持され,イントロンはスプライソソームから放出される.また,マイクロRNA前駆体の多くはイントロンに存在し,転写と共役した新生RNAの切断により産生される.さらに,最下流ではポリA化複合体によりポリAシグナルが認識され,その下流において新生RNAは切断され,そののち,ポリA鎖が付加される.ポリA鎖の付加は転写の終結に不可欠であると考えられている.これら多くの知見は試験官内反応系を中心に得られており,転写と共役したRNAプロセシングを細胞内に近い条件において詳細に解析した例はまだない.
RNAポリメラーゼIIによる転写反応がダイナミックであるのには理由がある.RNAポリメラーゼIIのC末端ドメインは特徴的な7アミノ酸残基を52回くり返し高度にリン酸化されている.転写反応においてそのリン酸化状態の組合せは非常に複雑なものになり,その複雑さがRNAプロセシングにかかわるタンパク質の部位特異的な結合に重要であると考えられている2).このC末端ドメインのリン酸化状態によるRNAポリメラーゼIIの転写制御は“CTDコード”といわれ,一般的に広く受け入れられてはいるが技術的な問題によりいまだ不明な点が多い.
RNAポリメラーゼIIによる転写反応と共役したRNAプロセンシングを詳細に解析するためには,合成された直後の新生RNAを解析する技術が必要になる.新生RNAを解析する代表的な技術としてはGRO-seq法およびPRO-seq法があげられる3,4).これらの方法では,修飾塩基を取り込ませることにより新生RNAを標識し,修飾塩基に特異的な抗体によりこれを精製する.また,クロマチン画分に含まれる転写産物をキャップ構造に特異的な抗体により精製しその3’末端を解析する3’NT法もある5).これらは,新生RNA,とくに,RNAポリメラーゼIIがゲノムのどこで一時停止するかを解析するのに有効な方法であるが,RNAポリメラーゼIIのC末端ドメインのリン酸化状態や転写と共役したRNAプロセシングを解析することは不可能である.
では,どのようにしたらRNAポリメラーゼIIのC末端ドメインのリン酸化状態に特異的に新生RNAを解析できるだろうか.RNAポリメラーゼII転写複合体に含まれる新生RNAを解析する方法としてNET-seq(native elongating transcript sequencing)法が報告されている6).この方法は酵母を用いて開発され大腸菌にも応用されている7).NET-seq法では,Flagタグを付加したRNAポリメラーゼIIの遺伝子をゲノムに挿入し,Flag抗体により免疫沈降したRNAポリメラーゼII転写複合体に含まれる新生RNAの3’末端,つまり,RNAポリメラーゼIIの転写活性部位を解析する.Flagタグによる単離ではC末端ドメインのリン酸化状態はみわけられないが,RNAポリメラーゼII転写複合体を免疫沈降する際にC末端ドメインのリン酸化状態を特異的に認識する抗体を用いることにより理論的には区別が可能になる.しかし,これまでNET-seq法を哺乳類細胞において応用した例はなく,C末端ドメインのリン酸化状態と新生RNAとを同時に調べた例はなかった.筆者らは,低濃度の尿素により処理したクロマチン画分に活性をもつRNAポリメラーゼIIが濃縮されることを見い出し,C末端ドメインのリン酸化状態を特異的に認識する抗体によりRNAポリメラーゼII複合体に含まれる新生RNAを単離することに成功し,この方法を哺乳類NET-seq法と名づけた.
1.哺乳類NET-seq法の原理
哺乳類NET-seq法の原理は至極単純である(図1).哺乳類細胞のクロマチン画分をマイクロコッカルヌクレアーゼにより可溶化したのち,抗RNAポリメラーゼII抗体により免疫沈降し,得られたRNAポリメラーゼII転写複合体に含まれる新生RNAの断片を次世代シークエンサーにより解析した.さらに,得られたリード配列の5’末端をバイオインフォマティクス解析によりヒトのゲノムにマッピングし,RNAポリメラーゼII転写複合体に含まれる新生RNAの3’末端の位置情報,つまり,RNAポリメラーゼIIの転写活性部位を1塩基の解像度で明らかにした.哺乳類NET-seq法の最大の利点は,免疫沈降の際に異なる抗RNAポリメラーゼII抗体を使用できることである.リン酸化されていないC末端ドメインを認識する抗RNAポリメラーゼII抗体を用いた場合,新生RNAは転写開始点の近傍に多く検出された.一方,2番目のセリン残基がリン酸化されたC末端ドメインを特異的に認識する抗RNAポリメラーゼII抗体を用いた場合,新生RNAは転写終結点の下流に多く検出された.これらの結果から,哺乳類NET-seq法はC末端ドメインのリン酸化状態に特異的な新生RNAのプロファイルを1塩基のレベルで網羅的に解析できることを示した.
2.転写と共役したスプライシングの機構
哺乳類NET-seq法により,5番目のセリン残基がリン酸化されたC末端ドメインに特異的な抗RNAポリメラーゼII抗体を用いることによりエキソンの3’末端に1塩基のピークが検出された.このピークは選択されていないエキソンには検出されず,さらに,スプラシシングの阻害剤の存在により減弱したことから,スプライシングに依存的であると結論づけられた.スプライシング中間体の上流のエキソンはスプライソソームにより切断され3’末端にOH基を生じることから,このエキソンの3’末端が検出されたことが示唆された.この結果は,哺乳類NET-seq法はRNAポリメラーゼIIの転写活性部位だけでなく,スプライソソームなどRNAポリメラーゼII転写複合体において起こる新生RNAの切断にともなう3’末端のOH基も検出できることを示した(図2).哺乳類NET-seq法により,イントロンよりエキソンにおいてより多くのシグナルが検出され,このシグナルは5番目のセリン残基がリン酸化されたC末端ドメインに特異的であった.これらの知見から,転写反応においてRNAポリメラーゼIIはイントロンとエキソンの境界で減速し,そのことが活性化したエキソンの認識に重要であることが示唆された.また,5番目のセリン残基がリン酸化されたC末端ドメインはエキソンにおけるRNAポリメラーゼIIの速度の制御およびスプライソソームとの相互作用に密接にかかわることが考えられた.
3.転写と共役したマイクロRNA前駆体の産生のキネティクス
哺乳類においてマイクロRNA前駆体の多くはイントロンに存在し,2本鎖RNA鎖に特異的なヌクレアーゼ複合体であるDrosha/DGCR8マイクロプロセッサーにより転写反応の過程において切断される8).この新生RNAの切断により上流のRNA鎖およびマイクロRNA前駆体の3’末端にOH基が生じる.哺乳類NET-seq法により,転写反応において生じるマイクロRNA前駆体の切断において1塩基のピークが検出された.マイクロRNAとして,has-mir-27bやhas-mir-21においては前駆体の5’側および3’側の部位にピークが検出されたが,has-mir-26bやhas-mir-181a-1においては前駆体の5’側の部位にのみピークが検出された.これらの結果は,RNAポリメラーゼII転写複合体からのマイクロRNA前駆体の放出に関して少なくとも2種類のモードが存在することを示唆した.5’側の部位のピークは切断された上流のRNA鎖の3’末端のOH基を示し,3’側の部位のピークはマイクロRNA前駆体の3’末端のOH基を示すと考えられた.5’側および3’側の部位にピークが検出されたマイクロRNA前駆体は切断ののちRNAポリメラーゼII複合体にとどまりやすいが,5’側の部位にのみピークが検出されるマイクロRNA前駆体はただちに放出されると考えられた.重要なことに,これらマイクロRNA前駆体とRNAポリメラーゼII複合体との相互作用はリン酸化されていないC末端ドメインを認識する抗RNAポリメラーゼII抗体では検出されなかったことから,C末端ドメインのリン酸化状態が重要であることが示された.
4.ポリA化複合体による転写の終結とRNAポリメラーゼIIの一時停止
RNAポリメラーゼIIによる転写の終結はポリAシグナルに大きく依存することが知られており,mRNA前駆体はポリAシグナルから10~30塩基ほど下流において切断されポリA鎖が付加される9).この新生RNAの切断はポリA化複合体の構成タンパク質であるCPSF73によるもので,CstF64はこの切断を促進することが知られている.哺乳類NET-seq法により,シグナルは転写終結領域に有意に蓄積していたことから,転写終結領域におけるRNAポリメラーゼII転写反応に対するポリA化複合体の影響について調べた.ポリA化複合体の種々の構成タンパク質をsiRNA法によりノックダウンした結果,転写の終結に大きな破綻がみられた.さらに,哺乳類NET-seq法により,このノックダウンのもとでは転写終結領域においてシグナルが減少していたことから,転写の終結の破綻はRNAポリメラーゼIIのC末端ドメインのリン酸化には依存していないことが示唆された.これらの結果から,ポリA化複合体は転写終結部位においてRNAポリメラーゼIIを一時停止させることにより,新生RNAの切断およびポリA鎖の付加のための時間をあたえ,転写終結を促進するというモデルが示唆された.
5.ポリA化複合体の介在する転写の未成熟な終結
哺乳類NET-seq法により,転写開始点の近傍のセンス鎖およびアンチセンス鎖にもっとも大きな新生RNAのピークが検出された.この結果は,ほかの方法による新生RNAの検出とも一致しており,遺伝子プロモーターの近傍におけるRNAポリメラーゼIIの一時停止を示した.この一時停止は新生RNAの品質管理にかかわると考えられており,遺伝子の発現制御における重要なステップである10).ポリA化複合体による新生RNAの切断にともなう転写終結の機構は,遺伝子プロモーターの近傍におけるRNAポリメラーゼIIの一時停止と関連があるのではないかと考え,ポリA化複合体の構成タンパク質をノックダウンした.その結果,哺乳類NET-seq法により,CPSF73あるいはCstF64/64tauのノックダウンは転写開始点の近傍のセンス鎖およびアンチセンス鎖において有意にシグナルを増加させた.興味深いことに,このシグナルの増加は2番目のセリン残基がリン酸化されたC末端ドメインに特異的であった.また,転写の終結にかかわる5’-3’エクソヌクレアーゼXrn2をノックダウンも同様の結果を示した.さらに,CLIP(crosslinking and immunoprecipitation,架橋および免疫沈降法)法により,転写開始点の近傍におけるポリA化複合体の構成タンパク質の新生RNAに対する直接的な結合能が示された.以上の知見から,ポリA化複合体を含む転写終結複合体が転写開始点の近傍における非生産的な転写産物の代謝にかかわることが強く示唆された.
おわりに
哺乳類NET-seq法は新生RNAのプロファイルおよびRNAポリメラーゼIIのC末端ドメインのリン酸化状態を同時かつ高解像度で解析できる唯一の方法である.5番目のセリン残基がリン酸化されたC末端ドメインを認識する抗RNAポリメラーゼII抗体がスプライシング中間体を選択的に単離できることを示したように,特定のC末端ドメイン修飾抗体を用いることにより特定の新生RNAを濃縮でき,転写反応における新たな現象を発見できる可能性がある.また,哺乳類NET-seq法は簡便であり,あらゆる細胞に応用が可能である.今後,医薬品の候補となる化合物の転写反応への影響を調べたり,細胞が分化するときの遺伝子発現の変化を観察したりすることなどに用いることで,いままでみすごされていた不安定なRNAが同定されるかもしれない.CTDコードがおりなす新生RNAワールドへの扉は,たったいま開かれたばかりである.近い将来,転写反応における多くの現象が哺乳類NET-seq法により明らかになる日を楽しみにしている.
文 献
- Moore, M. J. & Proudfoot, N. J.: Pre-mRNA processing reaches back to transcription and ahead to translation. Cell, 136, 688-700 (2009)[PubMed]
- Heidemann, M., Hintermair, C., Voss, K. et al.: Dynamic phosphorylation patterns of RNA polymerase II CTD during transcription. Biochim. Biophys. Acta, 1829, 55-62 (2013)[PubMed]
- Core, L. J. & Lis, J. T.: Transcription regulation through promoter-proximal pausing of RNA polymerase II. Science, 319, 1791-1792 (2008)[PubMed]
- Kwak, H., Fuda, N. J., Core, L. J. et al.: Precise maps of RNA polymerase reveal how promoters direct initiation and pausing. Science, 339, 950-953 (2013)[PubMed]
- Weber, C. M., Ramachandran, S. & Henikoff, S.: Nucleosomes are context-specific, H2A.Z-modulated barriers to RNA polymerase. Mol. Cell, 53, 819-830 (2014)[PubMed]
- Churchman, L. S. & Weissman, J. S.: Nascent transcript sequencing visualizes transcription at nucleotide resolution. Nature, 469, 368-373 (2011)[PubMed]
- Larson, M. H., Mooney, R. A., Peters, J. M. et al.: A pause sequence enriched at translation start sites drives transcription dynamics in vivo. Science, 344, 1042-1047 (2014)[PubMed]
- Morlando, M., Ballarino, M., Gromak, N. et al.: Primary microRNA transcripts are processed co-transcriptionally. Nat. Struct. Mol. Biol., 15, 902-909 (2008)[PubMed]
- Proudfoot, N. J.: Ending the message: poly(A) signals then and now. Genes Dev., 25, 1770-1782 (2011)[PubMed]
- Kwak, H. & Lis, J. T.: Control of transcriptional elongation. Annu. Rev. Genet., 47, 483-508 (2013)[PubMed]
著者プロフィール
略歴:2006年 東京医科歯科大学大学院生命情報教育部博士課程 修了,同年 同 特任助教,2010年 英国Oxford大学 博士研究員を経て,2014年より同 シニアリサーチフェロー.
研究テーマ:哺乳類細胞におけるRNAポリメラーゼIIによる新生転写反応とCTDコード.
抱負:哺乳類のゲノムにうもれている新生RNAを発掘し,その発現制御の機構および生理学的な機能を明らかにしたい.
© 2015 野島 孝之 Licensed under CC 表示 2.1 日本