クロマチンにおける転写の構造基盤
鯨井智也1・江原晴彦2・関根俊一2・胡桃坂仁志1
(1東京大学定量生命科学研究所 クロマチン構造機能研究分野,2理化学研究所生命機能科学研究センター 転写制御構造生物学研究チーム)
email:関根俊一,胡桃坂仁志
DOI: 10.7875/first.author.2018.104
Structural basis of the nucleosome transition during RNA polymerase II passage.
Tomoya Kujirai, Haruhiko Ehara, Yuka Fujino, Mikako Shirouzu, Shun-ichi Sekine, Hitoshi Kurumizaka
Science, 362, 595-598 (2018)
真核生物においてゲノムDNAはヌクレオソームを基本単位とするクロマチンを形成し核に収納される.生命活動の根幹である遺伝子の発現は,RNAポリメラーゼIIによりDNAからRNAが転写される反応からはじまる.クロマチンにおいてRNAポリメラーゼIIが転写反応を行うためには,ヌクレオソームにおいてヒストンに強固に巻きついたDNAの配列を読み取る必要がある.しかし,RNAポリメラーゼIIによるヌクレオソームDNAの読み取りの機構については不明であった.この研究において,筆者らは,試験管内においてヌクレオソームDNAからの転写反応を再現し,クライオ電子顕微鏡を用いた単粒子解析により転写反応中のRNAポリメラーゼII-ヌクレオソーム複合体の立体構造を明らかにした.一連の構造から,RNAポリメラーゼIIがヌクレオソームからDNAを段階的にはがしながら転写反応を行うようすが明らかにされた.ヌクレオソームDNAの中心の付近にて停止した複合体には外来のDNAがトランスに結合していた.このDNAがトランスに結合した複合体は,転写反応におけるヒストンの転移の中間体の構造である“テンプレートルーピング”構造を示唆した.これらの知見は,ゲノムのエピジェネティックな制御の中心であるクロマチンにおけるRNAポリメラーゼIIによる転写の構造基盤を示すものである.
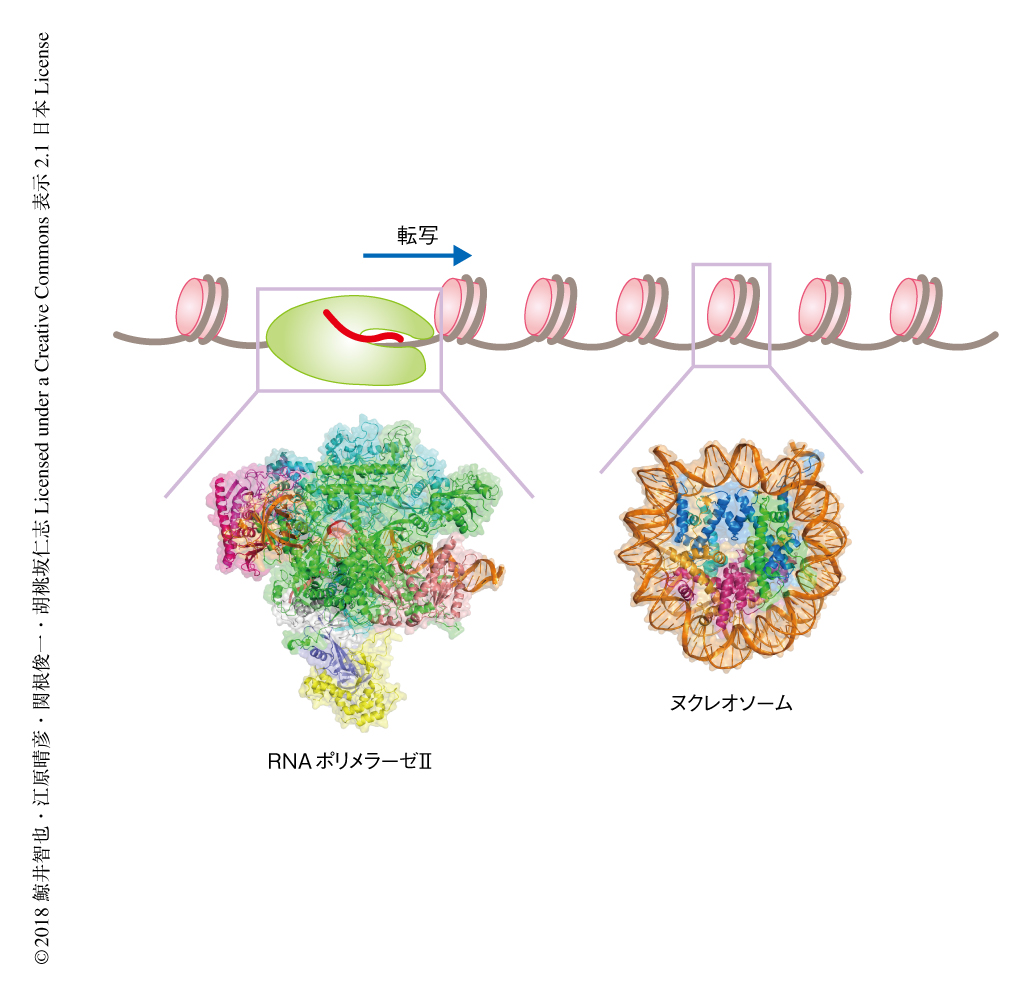
真核生物においてゲノムDNAはさまざまなタンパク質が結合したクロマチンを形成し核に収納される.クロマチンの最小単位はヌクレオソームであり,ヌクレオソームは4種類のヒストンタンパク質,ヒストンH2A,ヒストンH2B,ヒストンH3,ヒストンH4をそれぞれ2分子ずつ含むヒストン八量体に約150 bpのDNAが巻きついた円盤状の構造体である1).クロマチンの基本構造はヌクレオソームが数珠状につらなった超分子ポリマーである.転写,DNA修復,複製などのDNAの機能はこのクロマチンにおいて実現される(図1,PDB ID:5XON,5X7X).遺伝子の発現を担うDNAからRNAが転写される反応はRNAポリメラーゼIIにより触媒される.RNAポリメラーゼIIによる転写の機構については,これまで,非常に多くの構造生物的な研究が進められてきたが,それらはすべてクロマチンではない,はだかのDNAにおける構造を解析したものであった.ヌクレオソームにおいてDNAはヒストン八量体に強固に巻きついているため,ヌクレオソームの構造は転写にとり大きな障害になる2,3).そのため,クロマチンにおける転写,とくに,RNAポリメラーゼIIがヒストンに巻きついたDNAを読み取る機構は,ヌクレオソームが発見されて以来の未解決の重要な課題であった.先行研究において,生体においてRNAポリメラーゼIIはヌクレオソームにて段階的に停止しながら転写反応を行うことが示されたが4),その詳細な機構は不明であった.
この研究において,筆者らは,ヌクレオソームDNAからRNAポリメラーゼIIにより転写反応を行わせ,得られた複数の複合体を混合物として調製した.そして,クライオ電子顕微鏡を用いた単粒子解析により,ヌクレオソームにおいて段階的に停止した一連のRNAポリメラーゼII-ヌクレオソーム複合体の立体構造を明らかにした.
先行研究において,生体においてRNAポリメラーゼIIがヌクレオソームDNAから転写をする際には,ヌクレオソームDNAの中心の付近まではさまざまな位置で一時停止しつつ伸長反応を行う一方,中心の付近でヒストンが脱落し,それ以降は停止することなく伸長反応を行うと考えられていた4).そこで,RNAポリメラーゼIIによりヌクレオソームDNAから転写反応を行わせるための鋳型DNAのデザインとして,ヌクレオソームの脱落をふせぐためヌクレオソームDNAの中心の手前の位置でRNAポリメラーゼIIが停止するようDNAの配列を設計した.具体的には,中心の手前までをチミンを含まない配列とし,反応溶液のATPを3’-dATPに置換することにより転写がヌクレオソームDNAの中心の手前で停止するようにした.さらに,構造解析にむけ,ヌクレオソームの位置を正確に固定するためのDNAの配列として,ヌクレオソームに強固に巻きつくWidom601配列を用いた.このようにデザインした鋳型DNAと,組換えタンパク質として精製したヒストンH2A,ヒストンH2B,ヒストンH3,ヒストンH4を用いてヌクレオソームを再構成した.ヌクレオソームのリンカー領域にバブルDNA構造を模倣したミスマッチ2本鎖DNAを連結し,RNAポリメラーゼIIによる転写の開始点とした.精製した酵母Komagataella pastorisのRNAポリメラーゼIIおよび転写伸長因子TFIISを,バブルDNA構造をとる領域に相補的なプライマーRNA,GTP,CTP,UTP,3’-dATPを含む条件において,バブルDNA構造をもつヌクレオソームに添加することにより転写反応を行わせた.そののち,GraFix法により反応液を分画および架橋し,伸長したRNAを含むポリメラーゼII-ヌクレオソーム複合体を安定化させて精製した.精製されたRNAポリメラーゼII-ヌクレオソーム複合体に含まれるRNAを解析した結果,RNAポリメラーゼIIは,ヌクレオソームの入り口の手前の位置,20 bp侵入した位置,50 bp侵入した位置,60 bp侵入した位置において,それぞれ,停止していることがわかった.これらの停止位置に対応するRNAポリメラーゼII-ヌクレオソーム複合体を,停止位置のヌクレオソームの超らせんの位置(super helical location)を参照して,それぞれ,SHL(-6) 複合体,SHL(-5) 複合体,SHL(-2) 複合体,SHL(-1) 複合体と名づけた.
これまで頻繁に用いられてきたX線結晶構造解析法は,結晶の作製のため試料の溶液に含まれる粒子が均一である必要があった.そこで,近年,急速に発展しているクライオ電子顕微鏡による単粒子解析を試みた.単粒子解析においては,さまざまな粒子を含む不均一な試料でも,溶液に含まれる粒子を分類することにより立体構造解析が可能である.単粒子解析の結果,ヌクレオソームのSHL(-6),SHL(-5),SHL(-2),SHL(-1) の位置においてRNAポリメラーゼIIが停止した,合計で7つのRNAポリメラーゼII-ヌクレオソーム複合体の立体構造が決定された(PDB ID:6A5O ,6A5P ,6A5R ,6A5T ,6A5L ,6A5U ,6A5V ).これら一連の構造は,RNAポリメラーゼIIがヌクレオソームDNAから転写反応をしている最中のスナップショットを表わしており,RNAポリメラーゼIIはヌクレオソームからDNAを段階的にはがすことによりDNAを読み取っていくことが明らかにされた.一連の構造において,RNAポリメラーゼIIは活性型の構造を形成しており,転写反応のおのおのの段階において大きな変化はなかった.
RNAポリメラーゼIIはヌクレオソームにおいてヒストンとDNAとが強固に相互作用している部位にて停止していた.SHL(-5) 複合体はヒストンH2A-ヒストンH2B二量体とDNAとの相互作用部位において,SHL(-6) 複合体,SHL(-2) 複合体,SHL(-1) 複合体はヒストンH3-ヒストンH4二量体とDNAとの相互作用部位において停止していた.RNAポリメラーゼIIはヌクレオソームにて複数の異なる位置で停止していたが,いずれの停止位置においても,RNAポリメラーゼIIに対するヌクレオソームの角度は類似していた.そして,どの停止位置においても,RNAポリメラーゼIIのサブユニットのひとつであるRpb1のclamp headドメインが,ヌクレオソームからはがされていないDNAにそうよう結合していた.ヒストンとDNAとの強固な相互作用にくわえ,RNAポリメラーゼIIとヌクレオソームDNAとの相互作用が,RNAポリメラーゼIIがヌクレオソームにて停止する機構において重要であると考えられた.
RNAポリメラーゼIIにより転写反応が行われるのにともない,ヌクレオソームからDNAがはがされていく.SHL(-6) 複合体は,ヌクレオソームからDNAがはがされるまえの,RNAポリメラーゼIIがヌクレオソームに衝突した状態の複合体であった.また,SHL(-5) 複合体はヌクレオソームからDNAが20 bpほどはがされた構造であった.これらの位置の近傍にはヒストンH3のN末端領域が位置していた.ヒストンH3のN末端領域はアセチル化をうけることが知られており,このアセチル化はヌクレオソームの末端においてRNAポリメラーゼIIがDNAをはがすことを助けている可能性が考えられた.SHL(-2) 複合体においてDNAは50 bpほどはがされ,SHL(-1) 複合体においてDNAは60 bpほどはがされていた.SHL(-2) 複合体およびSHL(-1) 複合体において,ヒストンH2A-ヒストンH2B二量体はヌクレオソームに存在した.このことは,先行研究において報告されていた,ヌクレオソームからDNAが40 bpほどはがされた場合,ヒストンH2A-ヒストンH2B二量体は脱落するという知見に反していた5).SHL(-2) 複合体の構造において,ヒストンH2A-ヒストンH2B二量体と,RNAポリメラーゼIIのサブユニットのひとつであるRpb2のlobeドメインとが直接に相互作用していることが判明し,この相互作用により,ヌクレオソームにおいてヒストンH2A-ヒストンH2B二量体は安定化されると考えられた.
SHL(-1) 複合体においてRNAポリメラーゼIIの停止に寄与すると考えられたヒストンH3のLys64は,転写開始点の周辺においてアセチル化されることによりDNAとの相互作用を弱めると考えられている6).そのため,SHL(-1) はヒストンの修飾による転写のエピジェネティックな制御に重要な位置であると考えられた.
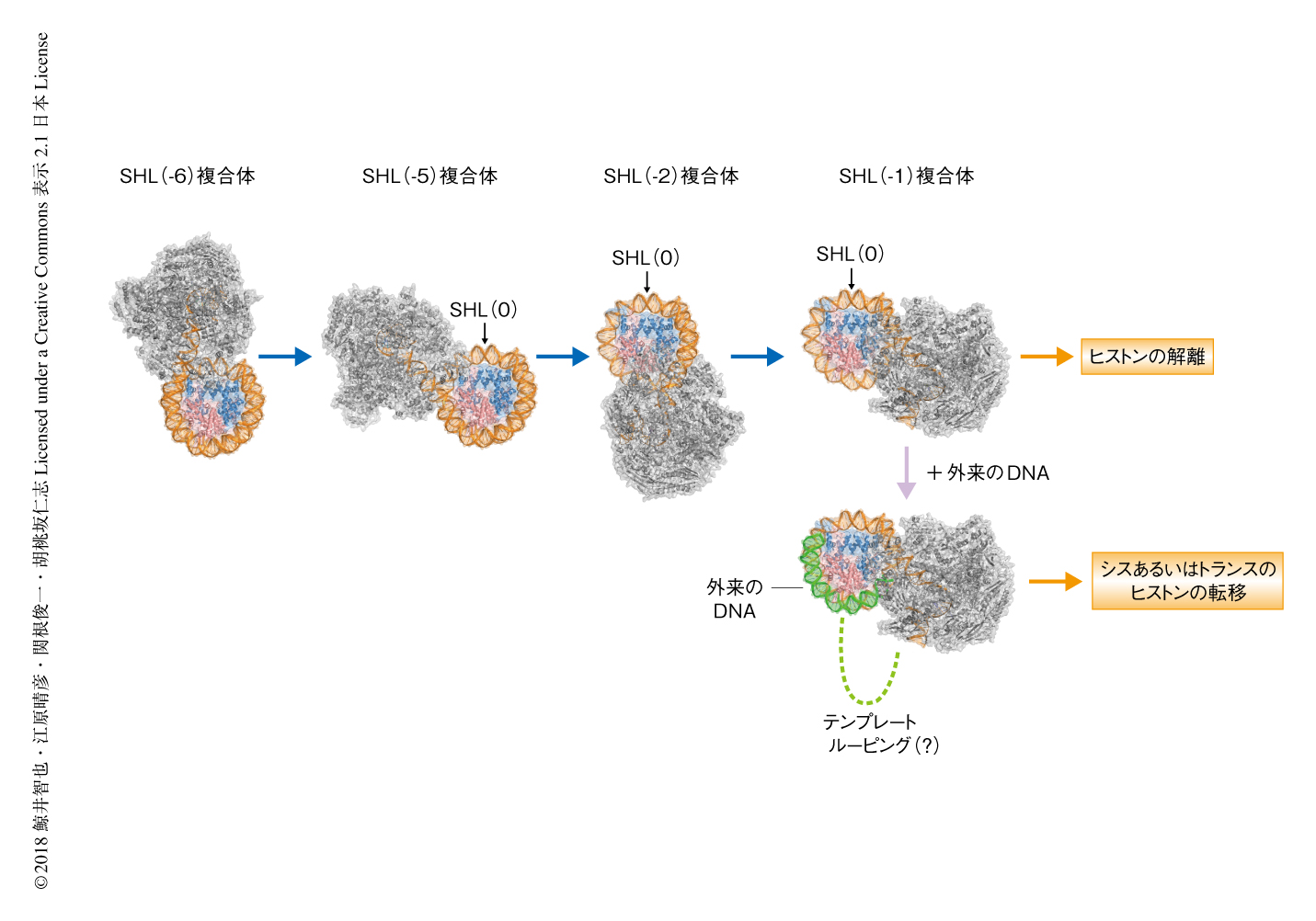
SHL(-1) 複合体においては,DNAがはがされ露出したヒストンH2A-ヒストンH2B二量体に試料の溶液に存在したほかのDNA,すなわち,外来のDNAが結合している画分が存在した.この外来のDNAが結合する現象は架橋していない試料の構造解析においても観察されたため,架橋によるアーティファクトではなかった.電子顕微鏡による写真から,外来のDNAは溶液に存在したほかのヌクレオソームのリンカー領域のDNAであると考えられた.この現象から,露出したヒストンH2A-ヒストンH2B二量体はほかのDNAと結合してヌクレオソーム様の構造体を再形成する傾向のあることが示されたことと同時に,ヒストンがシスまたはトランスに転移する際の中間体の構造を模している可能性が示唆された.先行研究において,RNAポリメラーゼIIによりヌクレオソームDNAから転写反応が行われる際に,ヒストンがRNAポリメラーゼIIの下流側から上流側へと転移する現象が知られている.その際,RNAポリメラーゼIIの通過した下流側のDNAが,上流側に存在するヌクレオソームと結合することにより,テンプレートルーピング構造を形成することが報告されている7,8).この研究において観察された外来のDNAが結合した複合体は,RNAポリメラーゼIIの上流側のDNAが下流側のヌクレオソームのヒストンH2A-ヒストンH2B複合体と結合したテンプレートルーピング構造を模倣したものなのかもしれない.
外来のDNAを含むSHL(-1) 複合体についてさらなる構造解析を進めた結果,SHL(-1) 複合体から1 bp伸長したSHL(-1)+1複合体,および,SHL(-1) 複合体とSHL(-1)+1複合体の中間の状態と考えられるtilted SHL(-1) 複合体の構造も明らかにされた.SHL(-1) 複合体に対し,SHL(-1)+1複合体においてヌクレオソームに対するRNAポリメラーゼIIの角度は36度ほど回転しており,tilted SHL(-1) 複合体はその中間の構造であった.これらの構造は,RNAポリメラーゼIIがヌクレオソームDNAを1 bpだけ進んだ際のスナップショット構造を示しているのだろう.
この研究において,筆者らは,RNAポリメラーゼIIによる転写反応にともなうヌクレオソームの構造の一連の変化を明らかにし,RNAポリメラーゼがヌクレオソームからDNAを段階的にはがしながら転写反応を行うことをはじめて明らかにした.まず,RNAポリメラーゼIIはヌクレオソームと衝突し,そののち,ヌクレオソームから20 bp,50 bp,60 bpと段階的にDNAがはがされる.SHL(-1) 複合体においてはヒストンH2A-ヒストンH2B二量体が露出し外来のDNAが結合できる状態になる.転写反応中に外来のDNAが結合した場合にはシスあるいはトランスにヒストンの転移が起こり,外来のDNAが結合しない場合にはヒストンの解離が起こるのかもしれない(図2).
正常なクロマチンの維持は細胞にとりきわめて重要であり,その破綻はがんをはじめとするさまざまな疾患の原因になりうるほか,細胞の老化などにも深くかかわることが知られている.細胞においてRNAポリメラーゼに対しさまざまな転写伸長因子が結合することによりクロマチンから効率的な転写が起こると考えられている9,10)(文献9) は 新着論文レビュー でも掲載).また,ヌクレオソームリモデリングタンパク質,ヒストンシャペロン,ヒストン修飾酵素などが協調的にはたらくことにより,ヒストンの修飾やリモデリングによるクロマチンの機能的な構造の変化が実現されている.この研究において明らかにされた一連の構造および実験の手法は,これら転写反応と同時に起こる高次のクロマチンの構造の変化を理解していくための基盤を提供するだろう.
構造解析に用いた反応液にはRNAポリメラーゼIIがヌクレオソームのさまざまな地点で停止した複数の種類の複合体が含まれていたが,計算機による画像処理により立体構造が同時に解明された.明らかにさまざまな粒子を含む不均一な混合液を用いて7種類もの立体構造が同時に明らかにされたことは世界的にもほとんど例のないことから,クライオ電子顕微鏡のさらなる可能性が提示されたと考えている.
この論文がScience誌に掲載されてから数日後,別の研究グループにより,RNAポリメラーゼII-ヌクレオソーム複合体のクライオ電子顕微鏡による構造がプレプリントリポジトリであるBioRxivに発表された(DOI:10.1101/437574).これは,この研究分野に対する関心の高さや将来性を示すものである.今後の発展を期待したい.
略歴:2018年 早稲田大学大学院先進理工学研究科 修了,同年より東京大学定量生命科学研究所 助教.
研究テーマ:クロマチンの構造と遺伝子発現の制御.
関心事:つぎの研究.
江原 晴彦(Haruhiko Ehara)
理化学研究所生命機能科学研究センター 研究員.
関根 俊一(Shun-ichi Sekine)
理化学研究所生命機能科学研究センター チームリーダー.
研究室URL:https://www.bdr.riken.jp/jp/research/labs/sekine-s/index.html
胡桃坂 仁志(Hitoshi Kurumizaka)
東京大学定量生命科学研究所 教授.
研究室URL:http://www.iam.u-tokyo.ac.jp/kurumizakalab/
© 2018 鯨井智也・江原晴彦・関根俊一・胡桃坂仁志 Licensed under CC 表示 2.1 日本
(1東京大学定量生命科学研究所 クロマチン構造機能研究分野,2理化学研究所生命機能科学研究センター 転写制御構造生物学研究チーム)
email:関根俊一,胡桃坂仁志
DOI: 10.7875/first.author.2018.104
Structural basis of the nucleosome transition during RNA polymerase II passage.
Tomoya Kujirai, Haruhiko Ehara, Yuka Fujino, Mikako Shirouzu, Shun-ichi Sekine, Hitoshi Kurumizaka
Science, 362, 595-598 (2018)
この論文に出現する遺伝子・タンパク質のUniprot ID
RNA polymerase II, ヌクレオソーム, RNAポリメラーゼII, ヒストン, ヒストンH2A, ヒストンH2B, ヒストンH3, ヒストンH4(P62805), TFIIS, Rpb1(P24928), Rpb2(P30876), RNAポリメラーゼ, ヌクレオソームリモデリングタンパク質, ヒストンシャペロン, ヒストン修飾酵素
要 約
真核生物においてゲノムDNAはヌクレオソームを基本単位とするクロマチンを形成し核に収納される.生命活動の根幹である遺伝子の発現は,RNAポリメラーゼIIによりDNAからRNAが転写される反応からはじまる.クロマチンにおいてRNAポリメラーゼIIが転写反応を行うためには,ヌクレオソームにおいてヒストンに強固に巻きついたDNAの配列を読み取る必要がある.しかし,RNAポリメラーゼIIによるヌクレオソームDNAの読み取りの機構については不明であった.この研究において,筆者らは,試験管内においてヌクレオソームDNAからの転写反応を再現し,クライオ電子顕微鏡を用いた単粒子解析により転写反応中のRNAポリメラーゼII-ヌクレオソーム複合体の立体構造を明らかにした.一連の構造から,RNAポリメラーゼIIがヌクレオソームからDNAを段階的にはがしながら転写反応を行うようすが明らかにされた.ヌクレオソームDNAの中心の付近にて停止した複合体には外来のDNAがトランスに結合していた.このDNAがトランスに結合した複合体は,転写反応におけるヒストンの転移の中間体の構造である“テンプレートルーピング”構造を示唆した.これらの知見は,ゲノムのエピジェネティックな制御の中心であるクロマチンにおけるRNAポリメラーゼIIによる転写の構造基盤を示すものである.
はじめに
真核生物においてゲノムDNAはさまざまなタンパク質が結合したクロマチンを形成し核に収納される.クロマチンの最小単位はヌクレオソームであり,ヌクレオソームは4種類のヒストンタンパク質,ヒストンH2A,ヒストンH2B,ヒストンH3,ヒストンH4をそれぞれ2分子ずつ含むヒストン八量体に約150 bpのDNAが巻きついた円盤状の構造体である1).クロマチンの基本構造はヌクレオソームが数珠状につらなった超分子ポリマーである.転写,DNA修復,複製などのDNAの機能はこのクロマチンにおいて実現される(図1,PDB ID:5XON,5X7X).遺伝子の発現を担うDNAからRNAが転写される反応はRNAポリメラーゼIIにより触媒される.RNAポリメラーゼIIによる転写の機構については,これまで,非常に多くの構造生物的な研究が進められてきたが,それらはすべてクロマチンではない,はだかのDNAにおける構造を解析したものであった.ヌクレオソームにおいてDNAはヒストン八量体に強固に巻きついているため,ヌクレオソームの構造は転写にとり大きな障害になる2,3).そのため,クロマチンにおける転写,とくに,RNAポリメラーゼIIがヒストンに巻きついたDNAを読み取る機構は,ヌクレオソームが発見されて以来の未解決の重要な課題であった.先行研究において,生体においてRNAポリメラーゼIIはヌクレオソームにて段階的に停止しながら転写反応を行うことが示されたが4),その詳細な機構は不明であった.
この研究において,筆者らは,ヌクレオソームDNAからRNAポリメラーゼIIにより転写反応を行わせ,得られた複数の複合体を混合物として調製した.そして,クライオ電子顕微鏡を用いた単粒子解析により,ヌクレオソームにおいて段階的に停止した一連のRNAポリメラーゼII-ヌクレオソーム複合体の立体構造を明らかにした.
1.転写反応中のRNAポリメラーゼIIとヌクレオソームとの複合体の調製
先行研究において,生体においてRNAポリメラーゼIIがヌクレオソームDNAから転写をする際には,ヌクレオソームDNAの中心の付近まではさまざまな位置で一時停止しつつ伸長反応を行う一方,中心の付近でヒストンが脱落し,それ以降は停止することなく伸長反応を行うと考えられていた4).そこで,RNAポリメラーゼIIによりヌクレオソームDNAから転写反応を行わせるための鋳型DNAのデザインとして,ヌクレオソームの脱落をふせぐためヌクレオソームDNAの中心の手前の位置でRNAポリメラーゼIIが停止するようDNAの配列を設計した.具体的には,中心の手前までをチミンを含まない配列とし,反応溶液のATPを3’-dATPに置換することにより転写がヌクレオソームDNAの中心の手前で停止するようにした.さらに,構造解析にむけ,ヌクレオソームの位置を正確に固定するためのDNAの配列として,ヌクレオソームに強固に巻きつくWidom601配列を用いた.このようにデザインした鋳型DNAと,組換えタンパク質として精製したヒストンH2A,ヒストンH2B,ヒストンH3,ヒストンH4を用いてヌクレオソームを再構成した.ヌクレオソームのリンカー領域にバブルDNA構造を模倣したミスマッチ2本鎖DNAを連結し,RNAポリメラーゼIIによる転写の開始点とした.精製した酵母Komagataella pastorisのRNAポリメラーゼIIおよび転写伸長因子TFIISを,バブルDNA構造をとる領域に相補的なプライマーRNA,GTP,CTP,UTP,3’-dATPを含む条件において,バブルDNA構造をもつヌクレオソームに添加することにより転写反応を行わせた.そののち,GraFix法により反応液を分画および架橋し,伸長したRNAを含むポリメラーゼII-ヌクレオソーム複合体を安定化させて精製した.精製されたRNAポリメラーゼII-ヌクレオソーム複合体に含まれるRNAを解析した結果,RNAポリメラーゼIIは,ヌクレオソームの入り口の手前の位置,20 bp侵入した位置,50 bp侵入した位置,60 bp侵入した位置において,それぞれ,停止していることがわかった.これらの停止位置に対応するRNAポリメラーゼII-ヌクレオソーム複合体を,停止位置のヌクレオソームの超らせんの位置(super helical location)を参照して,それぞれ,SHL(-6) 複合体,SHL(-5) 複合体,SHL(-2) 複合体,SHL(-1) 複合体と名づけた.
2.RNAポリメラーゼII-ヌクレオソーム複合体の立体構造
これまで頻繁に用いられてきたX線結晶構造解析法は,結晶の作製のため試料の溶液に含まれる粒子が均一である必要があった.そこで,近年,急速に発展しているクライオ電子顕微鏡による単粒子解析を試みた.単粒子解析においては,さまざまな粒子を含む不均一な試料でも,溶液に含まれる粒子を分類することにより立体構造解析が可能である.単粒子解析の結果,ヌクレオソームのSHL(-6),SHL(-5),SHL(-2),SHL(-1) の位置においてRNAポリメラーゼIIが停止した,合計で7つのRNAポリメラーゼII-ヌクレオソーム複合体の立体構造が決定された(PDB ID:6A5O ,6A5P ,6A5R ,6A5T ,6A5L ,6A5U ,6A5V ).これら一連の構造は,RNAポリメラーゼIIがヌクレオソームDNAから転写反応をしている最中のスナップショットを表わしており,RNAポリメラーゼIIはヌクレオソームからDNAを段階的にはがすことによりDNAを読み取っていくことが明らかにされた.一連の構造において,RNAポリメラーゼIIは活性型の構造を形成しており,転写反応のおのおのの段階において大きな変化はなかった.
RNAポリメラーゼIIはヌクレオソームにおいてヒストンとDNAとが強固に相互作用している部位にて停止していた.SHL(-5) 複合体はヒストンH2A-ヒストンH2B二量体とDNAとの相互作用部位において,SHL(-6) 複合体,SHL(-2) 複合体,SHL(-1) 複合体はヒストンH3-ヒストンH4二量体とDNAとの相互作用部位において停止していた.RNAポリメラーゼIIはヌクレオソームにて複数の異なる位置で停止していたが,いずれの停止位置においても,RNAポリメラーゼIIに対するヌクレオソームの角度は類似していた.そして,どの停止位置においても,RNAポリメラーゼIIのサブユニットのひとつであるRpb1のclamp headドメインが,ヌクレオソームからはがされていないDNAにそうよう結合していた.ヒストンとDNAとの強固な相互作用にくわえ,RNAポリメラーゼIIとヌクレオソームDNAとの相互作用が,RNAポリメラーゼIIがヌクレオソームにて停止する機構において重要であると考えられた.
3.RNAポリメラーゼIIの転写反応にともなうヌクレオソームの構造の変化
RNAポリメラーゼIIにより転写反応が行われるのにともない,ヌクレオソームからDNAがはがされていく.SHL(-6) 複合体は,ヌクレオソームからDNAがはがされるまえの,RNAポリメラーゼIIがヌクレオソームに衝突した状態の複合体であった.また,SHL(-5) 複合体はヌクレオソームからDNAが20 bpほどはがされた構造であった.これらの位置の近傍にはヒストンH3のN末端領域が位置していた.ヒストンH3のN末端領域はアセチル化をうけることが知られており,このアセチル化はヌクレオソームの末端においてRNAポリメラーゼIIがDNAをはがすことを助けている可能性が考えられた.SHL(-2) 複合体においてDNAは50 bpほどはがされ,SHL(-1) 複合体においてDNAは60 bpほどはがされていた.SHL(-2) 複合体およびSHL(-1) 複合体において,ヒストンH2A-ヒストンH2B二量体はヌクレオソームに存在した.このことは,先行研究において報告されていた,ヌクレオソームからDNAが40 bpほどはがされた場合,ヒストンH2A-ヒストンH2B二量体は脱落するという知見に反していた5).SHL(-2) 複合体の構造において,ヒストンH2A-ヒストンH2B二量体と,RNAポリメラーゼIIのサブユニットのひとつであるRpb2のlobeドメインとが直接に相互作用していることが判明し,この相互作用により,ヌクレオソームにおいてヒストンH2A-ヒストンH2B二量体は安定化されると考えられた.
SHL(-1) 複合体においてRNAポリメラーゼIIの停止に寄与すると考えられたヒストンH3のLys64は,転写開始点の周辺においてアセチル化されることによりDNAとの相互作用を弱めると考えられている6).そのため,SHL(-1) はヒストンの修飾による転写のエピジェネティックな制御に重要な位置であると考えられた.
4.SHL(-1) 複合体において外来のDNAがヒストンH2A-ヒストンH2B二量体と結合する
SHL(-1) 複合体においては,DNAがはがされ露出したヒストンH2A-ヒストンH2B二量体に試料の溶液に存在したほかのDNA,すなわち,外来のDNAが結合している画分が存在した.この外来のDNAが結合する現象は架橋していない試料の構造解析においても観察されたため,架橋によるアーティファクトではなかった.電子顕微鏡による写真から,外来のDNAは溶液に存在したほかのヌクレオソームのリンカー領域のDNAであると考えられた.この現象から,露出したヒストンH2A-ヒストンH2B二量体はほかのDNAと結合してヌクレオソーム様の構造体を再形成する傾向のあることが示されたことと同時に,ヒストンがシスまたはトランスに転移する際の中間体の構造を模している可能性が示唆された.先行研究において,RNAポリメラーゼIIによりヌクレオソームDNAから転写反応が行われる際に,ヒストンがRNAポリメラーゼIIの下流側から上流側へと転移する現象が知られている.その際,RNAポリメラーゼIIの通過した下流側のDNAが,上流側に存在するヌクレオソームと結合することにより,テンプレートルーピング構造を形成することが報告されている7,8).この研究において観察された外来のDNAが結合した複合体は,RNAポリメラーゼIIの上流側のDNAが下流側のヌクレオソームのヒストンH2A-ヒストンH2B複合体と結合したテンプレートルーピング構造を模倣したものなのかもしれない.
外来のDNAを含むSHL(-1) 複合体についてさらなる構造解析を進めた結果,SHL(-1) 複合体から1 bp伸長したSHL(-1)+1複合体,および,SHL(-1) 複合体とSHL(-1)+1複合体の中間の状態と考えられるtilted SHL(-1) 複合体の構造も明らかにされた.SHL(-1) 複合体に対し,SHL(-1)+1複合体においてヌクレオソームに対するRNAポリメラーゼIIの角度は36度ほど回転しており,tilted SHL(-1) 複合体はその中間の構造であった.これらの構造は,RNAポリメラーゼIIがヌクレオソームDNAを1 bpだけ進んだ際のスナップショット構造を示しているのだろう.
おわりに
この研究において,筆者らは,RNAポリメラーゼIIによる転写反応にともなうヌクレオソームの構造の一連の変化を明らかにし,RNAポリメラーゼがヌクレオソームからDNAを段階的にはがしながら転写反応を行うことをはじめて明らかにした.まず,RNAポリメラーゼIIはヌクレオソームと衝突し,そののち,ヌクレオソームから20 bp,50 bp,60 bpと段階的にDNAがはがされる.SHL(-1) 複合体においてはヒストンH2A-ヒストンH2B二量体が露出し外来のDNAが結合できる状態になる.転写反応中に外来のDNAが結合した場合にはシスあるいはトランスにヒストンの転移が起こり,外来のDNAが結合しない場合にはヒストンの解離が起こるのかもしれない(図2).
正常なクロマチンの維持は細胞にとりきわめて重要であり,その破綻はがんをはじめとするさまざまな疾患の原因になりうるほか,細胞の老化などにも深くかかわることが知られている.細胞においてRNAポリメラーゼに対しさまざまな転写伸長因子が結合することによりクロマチンから効率的な転写が起こると考えられている9,10)(文献9) は 新着論文レビュー でも掲載).また,ヌクレオソームリモデリングタンパク質,ヒストンシャペロン,ヒストン修飾酵素などが協調的にはたらくことにより,ヒストンの修飾やリモデリングによるクロマチンの機能的な構造の変化が実現されている.この研究において明らかにされた一連の構造および実験の手法は,これら転写反応と同時に起こる高次のクロマチンの構造の変化を理解していくための基盤を提供するだろう.
構造解析に用いた反応液にはRNAポリメラーゼIIがヌクレオソームのさまざまな地点で停止した複数の種類の複合体が含まれていたが,計算機による画像処理により立体構造が同時に解明された.明らかにさまざまな粒子を含む不均一な混合液を用いて7種類もの立体構造が同時に明らかにされたことは世界的にもほとんど例のないことから,クライオ電子顕微鏡のさらなる可能性が提示されたと考えている.
この論文がScience誌に掲載されてから数日後,別の研究グループにより,RNAポリメラーゼII-ヌクレオソーム複合体のクライオ電子顕微鏡による構造がプレプリントリポジトリであるBioRxivに発表された(DOI:10.1101/437574).これは,この研究分野に対する関心の高さや将来性を示すものである.今後の発展を期待したい.
文 献
- Luger, K., Mader, A. W., Richmond, R. K. et al.: Crystal structure of the nucleosome core particle at 2.8Å resolution. Nature, 389, 251-260 (1997)[PubMed]
- Teves, S. S., Weber, C. M. & Henikoff, S.: Transcribing through the nucleosome. Trends Biochem. Sci., 39, 577-586 (2014)[PubMed]
- Lai, W. K. M. & Pugh, B. F.: Understanding nucleosome dynamics and their links to gene expression and DNA replication. Nat. Rev. Mol. Cell Biol., 18, 548-562 (2017)[PubMed]
- Weber. C. M., Ramachandran, S. & Henikoff, S.: Nucleosomes are context-specific, H2A.Z-modulated barriers to RNA polymerase. Mol. Cell, 53, 819-830 (2014)[PubMed]
- Arimura, Y., Tachiwana, H., Oda, T. et al.: Structural analysis of the hexasome, lacking one histone H2A/H2B dimer from the conventional nucleosome. Biochemistry, 51, 3302-3309 (2012)[PubMed]
- Di Cerbo, V., Mohn, F., Ryan, D. P. et al.: Acetylation of histone H3 at lysine 64 regulates nucleosome dynamics and facilitates transcription. Elife, 3, e01632 (2014)[PubMed]
- Studitsky, V. M., Clark, D. J. & Felsenfeld, G.: A histone octamer can step around a transcribing polymerase without leaving the template. Cell, 76, 371-382 (1994)[PubMed]
- Hodges, C., Bintu, L., Lubkowska, L. et al.: Nucleosomal fluctuations govern the transcription dynamics of RNA polymerase II. Science, 325, 626-628 (2009)[PubMed]
- Ehara, H., Yokoyama, T., Shigematsu, H. et al.: Structure of the complete elongation complex of RNA polymerase II with basal factors. Science, 357, 921-924 (2017)[PubMed] [新着論文レビュー]
- Vos, S. M., Farnung, L., Boehning, M. et al.: Structure of activated transcription complex Pol II-DSIF-PAF-SPT6. Nature, 560, 607-612 (2018)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2018年 早稲田大学大学院先進理工学研究科 修了,同年より東京大学定量生命科学研究所 助教.
研究テーマ:クロマチンの構造と遺伝子発現の制御.
関心事:つぎの研究.
江原 晴彦(Haruhiko Ehara)
理化学研究所生命機能科学研究センター 研究員.
関根 俊一(Shun-ichi Sekine)
理化学研究所生命機能科学研究センター チームリーダー.
研究室URL:https://www.bdr.riken.jp/jp/research/labs/sekine-s/index.html
胡桃坂 仁志(Hitoshi Kurumizaka)
東京大学定量生命科学研究所 教授.
研究室URL:http://www.iam.u-tokyo.ac.jp/kurumizakalab/
© 2018 鯨井智也・江原晴彦・関根俊一・胡桃坂仁志 Licensed under CC 表示 2.1 日本