基本的な転写伸長因子の結合したRNAポリメラーゼII転写伸長複合体の構造
江原晴彦・関根俊一
(理化学研究所ライフサイエンス技術基盤研究センター 超分子構造解析研究チーム)
email:関根俊一
DOI: 10.7875/first.author.2017.086
Structure of the complete elongation complex of RNA polymerase II with basal factors.
Haruhiko Ehara, Takeshi Yokoyama, Hideki Shigematsu, Shigeyuki Yokoyama, Mikako Shirouzu, Shun-ichi Sekine
Science, 357, 921-924 (2017)
真核生物においてmRNAの転写を担うRNAポリメラーゼIIは遺伝子の発現において中核的な役割をはたす.転写の伸長の際のRNAポリメラーゼIIにはDNAやRNAだけでなくさまざまな転写伸長因子が結合するが,それらすべてを含む高次複合体の分子構造は不明であった.筆者らは,X線結晶構造解析法とクライオ電子顕微鏡による単粒子解析法という2つの手法を組み合わせることにより,RNAポリメラーゼIIに高度に保存された基本的な転写伸長因子であるSpt4-Spt5複合体,Elf1,TFIISの結合した転写伸長複合体の構造を解明し,それら転写伸長因子の役割や転写開始複合体との大きな違いについて明らかにした.
RNAポリメラーゼIIによる転写は高度な制御をうけており,転写の開始,伸長,終結といった複数の段階から構成される.また,それぞれの段階に特有の機能を実現するため,さまざまに異なる転写因子がRNAポリメラーゼIIと結合し,転写の段階ごとに固有の高次複合体を形成することが知られている.たとえば,転写の開始においては,基本転写因子とよばれる一群の転写因子がRNAポリメラーゼIIと結合することにより巨大な転写開始複合体が形成される.最近のクライオ電子顕微鏡を用いた研究などにより,転写の開始にかかわる高次複合体の構造が明らかにされつつある1-3).一方,転写の伸長のときに形成される高次複合体の構造についてはこれまでほとんど知見がなかった.転写伸長複合体と結合する転写伸長因子としては多くの種類が知られているが,なかでも,Spt4-Spt5複合体,Elf1,TFIISとよばれる3種類の転写伸長因子は真核生物をこえてホモログの存在が知られており,転写の伸長においてもっとも基本的な機能に関与すると予想された.
Spt4-Spt5複合体は約100 kDaの大きなタンパク質複合体である.Spt5は古細菌だけでなく細菌まで保存された唯一の転写因子であり,転写の伸長へのかかわりが報告されてきた.たとえば,Spt4-Spt5複合体には転写の持続性を高めるはたらきのあるほか,転写の制御への関与や,転写に共役した高次機能とのかかわりが知られている4).Spt5はマルチドメインタンパク質であり,酵母においてはNGNドメイン,5つのKOWドメイン,CTRとよばれるリピート配列から構成される.
Elf1は約10 kDaの小さなタンパク質であり,亜鉛結合モチーフをもつ.Spt4-Spt5複合体やTFIISなど,転写の伸長にかかわるタンパク質と合成致死性を示すことが知られているほか5),クロマチン-免疫沈降法を用いた解析により転写の伸長の際のRNAポリメラーゼIIとの結合が示唆されている6).また,真核生物のみならず一部の古細菌にも存在するなど高い保存性をもち7),転写の伸長において基本的な機能にかかわると考えられるが具体的な知見には乏しい.
TFIISは古細菌および真核生物に保存されており,転写の伸長が途中で停止した際にRNAポリメラーゼIIのもつRNA切断活性を促進して転写を再開することが知られている.これまで,RNAポリメラーゼIIとTFIISとの複合体の構造はいくつか報告されており,2次チャネルとよばれる活性部位に連なる穴の近辺に結合することが知られている8,9).
この研究においては,Spt4-Spt5複合体,Elf1,TFIISの結合したRNAポリメラーゼII転写伸長複合体の構造を解析し,細胞において実際にはたらく状態に近い,転写の伸長に特化したRNAポリメラーゼII転写伸長複合体の構造をはじめて明らかにした.
RNAポリメラーゼIIについては,ゲノムに精製用のタグを組み込んだメタノール資化性酵母を用い,内在性の複合体を精製して用いた10,11).Spt4-Spt5複合体,Elf1,TFIISについては,いずれも大腸菌を用いた系において発現させ精製した.それぞれの転写伸長因子について,構造の解析や機能の解析に用いるため一部のドメインのみを含む断片や変異体を調製した.RNAポリメラーゼIIにくわえ,長さのさまざまに異なる転写の伸長を模擬した核酸や,それぞれの転写伸長因子を含むもの,含まないもの,さらには,断片や変異体など,多くの組合せについて結晶化をスクリーニングした.その結果,Spt5のKOW5ドメインおよびElf1を含むRNAポリメラーゼII転写伸長複合体について良好な結晶が得られ,X線結晶構造解析法により3.0Åの分解能で構造を決定した(PDB ID:5XOG).
Spt4-Spt5複合体のNGNドメインからKOW5ドメインまでを含む断片およびTFIISの不活性な変異体を含むRNAポリメラーゼII転写伸長複合体について,クライオ電子顕微鏡による単粒子解析法により構造を決定した.核酸としては,結晶化に用いたものと比べ上流に長い配列をもつDNAを用いた.精製の手順の最適化,クロスリンカーの利用,界面活性剤の添加などのくふうをつうじて観察するタンパク質の溶液からなる氷の薄膜の性質を改善したのち,3000枚をこえる画像を撮影した.約68万個の粒子を用いた画像解析により3.8Åの分解能をもつマップが得られた(PDB ID:5XON).しかし,転写伸長因子が結合すると思われる3箇所に大きな構造多型が存在したため,それぞれの部位に着目して粒子の画像を分類した.その結果,上流のDNA,Spt4,Spt5のNGNドメインおよびKOW1ドメインがよく分離したマップが4.3Åの分解能で,StalkとよばれるRNAポリメラーゼIIから飛び出た領域とSpt5のKOW4ドメインが分離したマップが4.7Åの分解能で,TFIISがよく分離したマップが3.9Åの分解能で,それぞれ得られた.
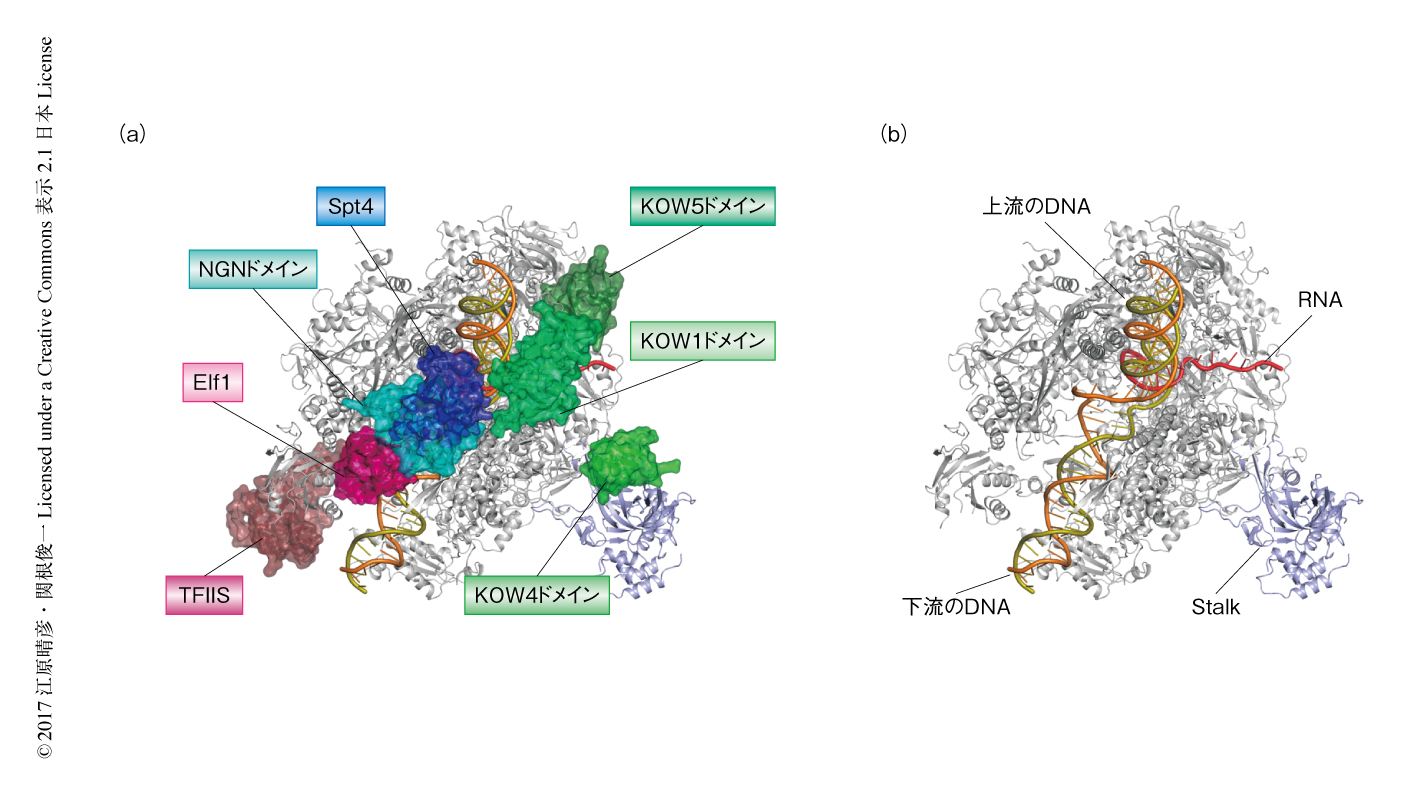
X線結晶構造解析法により得られた構造とクライオ電子顕微鏡による単粒子解析法から得られた複数のマップとを組み合わせることにより,Spt4-Spt5複合体,Elf1,TFIISを含むRNAポリメラーゼII転写伸長複合体の構造モデルを作製した(図1).この完全なRNAポリメラーゼII転写伸長複合体において,おのおのの転写伸長因子はRNAポリメラーゼIIの表面の広い範囲に分布していた.Elf1は下流のDNAの近傍に結合すること,Spt4とSpt5のNGNドメインおよびKOW1ドメインは上流のDNAをとりかこむように位置すること,Spt5のKOW4ドメインはRNAポリメラーゼIIのStalkに結合すること,Spt5のKOW5ドメインはRNAの出口の付近に形成されたくぼみに結合することが明らかにされた.Spt4-Spt5複合体およびElf1は核酸の溶媒への露出部をおおうように結合しており,転写の伸長の際にDNAおよびRNAの結合を安定化させることが示唆された.
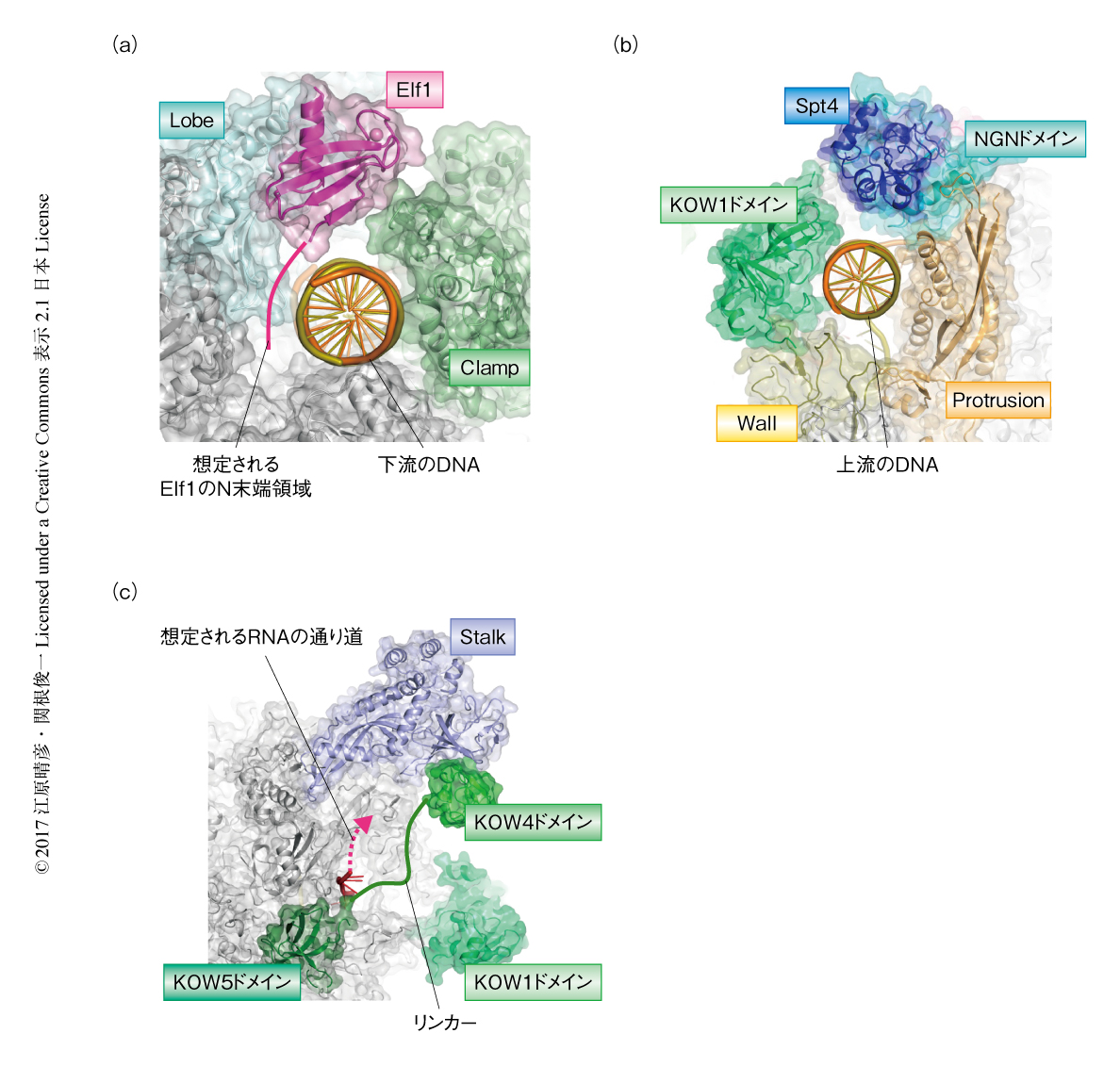
Elf1はRNAポリメラーゼIIのLobeとよばれる領域とClampとよばれる領域とのあいだにはさまれるように位置し,下流の2本鎖DNAが結合する溝の上部の隙間をうめていた.Elf1の結合により完全に閉じた“DNA導入トンネル”が形成され(図2a),下流のDNAの安定な保持に寄与すると考えられた.さらに,Elf1には塩基性残基に富んだN末端領域が存在し,この領域が下流のDNAと直接に相互作用することが機能面においても重要であることが示唆された.
Spt5のNGNドメインはRNAポリメラーゼIIのClampとProtrusionとよばれる領域とのあいだにはさまるように位置していた.また,Spt5のKOW1ドメインはNGNドメインのとなりに位置し,RNAポリメラーゼIIのClampおよびWallとよばれる領域に接していた.Spt4はSpt5のNGNドメインと強く結合しており,RNAポリメラーゼIIからもっとも遠い位置に位置していた.Spt4,Spt5のNGNドメインおよびKOW1ドメインは上流の2本鎖DNAをとりかこむようなかたちで配置しており,RNAポリメラーゼIIの一部の領域とともに完全に閉じた“DNA排出トンネル”を形成していた(図2b).2本鎖DNAのRNAポリメラーゼIIに近い部分や転写バブルの非鋳型鎖DNAは,Spt5のNGNドメインあるいはKOW1ドメインと直接に相互作用しており,これらのドメインが,転写バブルの1本鎖DNAがよりあわさり2本鎖DNAにもどるのを促進することが示唆された.一方で,DNA排出トンネルのRNAポリメラーゼIIから遠い部位については,複数の塩基性残基が存在するものの,2本鎖DNAからはやや距離がありDNAを比較的ゆるくとりかこむかたちになっていた.結果として,DNA排出トンネルはジョウゴのような形状をとり,2本鎖DNAの根元をしっかりと固定する一方で,RNAポリメラーゼIIから遠い部位に関してはある程度のゆらぎを許容していることが明らかにされた.
Spt5について,KOW4ドメインはRNAポリメラーゼIIのStalkと結合していた.また,KOW5ドメインはRNAの出口の近傍のくぼみにはまり込んでいた.さらに,KOW4ドメインとKOW5ドメインとのあいだのリンカーに相当すると考えられる弱い密度が観測された.RNAポリメラーゼIIのStalk,Spt5のKOW4ドメインおよびKOW5ドメインはRNAの出口をとりかこむように存在し,RNAポリメラーゼIIからでてくるRNAを正しく導くための“RNA排出トンネル”を構成することが示唆された(図2c).また,KOW4ドメインおよびKOW5ドメインにくわえて,近傍にはKOW1ドメイン,および,密度は観測されなかったもののKOW2ドメインおよびKOW3ドメインが存在すると考えられ,これらSpt5のKOWドメインが共同してRNAポリメラーゼIIから排出されるDNAとRNAとを物理的に隔離することが明らかにされた.さらに,RNAの出口の近傍には新しく合成されたRNAを修飾したりするさまざまな因子が結合することが知られており,Spt5により補強および拡張されたRNA排出トンネルは新しく合成されたRNAを別の酵素へとうけわたすための土台として重要なはたらきをすることが示唆された.
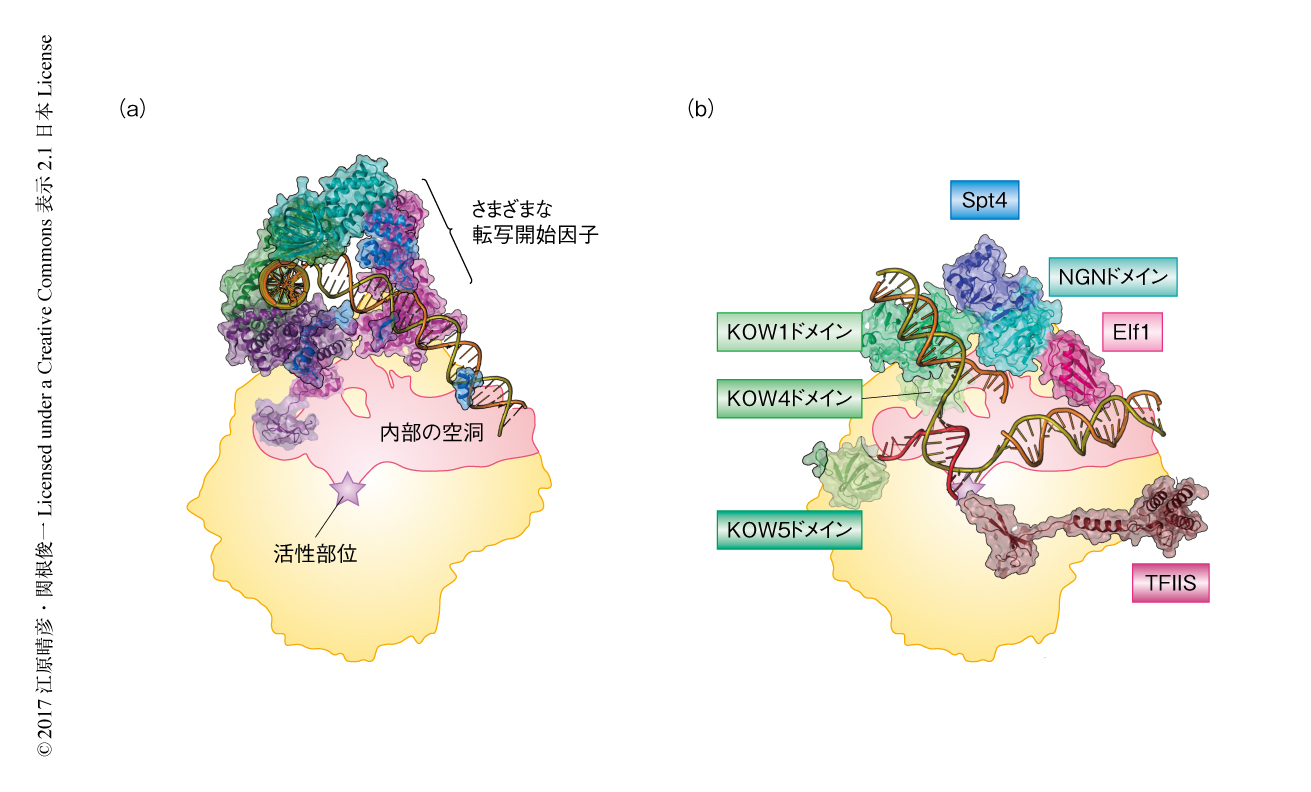
今回,構造の明らかにされたRNAポリメラーゼII転写伸長複合体は,以前に構造が報告されていたRNAポリメラーゼII転写開始前複合体2)(図3a,PDB ID:5FZ5)とは構造が大きく異なっていた.転写開始前複合体においては,DNAは基本転写因子によりRNAポリメラーゼIIの外側に結合し,それら基本転写因子の協調的なはたらきにより2本鎖DNAの一部がほどけDNAはRNAポリメラーゼIIの内部の空洞に取り込まれると考えられている.一方,転写伸長複合体(図3b)においては,DNAおよびRNAはRNAポリメラーゼIIの内部に完全に取り込まれた状態にあり,転写伸長因子がそれらの周囲をとりかこみ完全に独立したトンネルを形成することにより安定かつ継続した転写の伸長を可能すると考えられた.また,核酸の通り道をより規定されたものとすることが,ほかの因子への核酸のうけわたしなどをつうじた転写と共役したさまざまな高次機能の実現に重要であると考えられた.
さらに,基本転写因子どうし,および,転写伸長因子どうしは互いに協調的にRNAポリメラーゼII複合体と結合する一方,基本転写因子と転写伸長因子は構造において相互に排他的な結合様式をもっていた.たとえば,Elf1,Spt4,Spt5のNGNドメインは,転写開始前複合体のDNAと衝突してしまうため転写の開始の際には結合できないと考えられた.同様に,転写の伸長の際のSpt4やSpt5のKOW4ドメインは,転写の開始の際のTFIIFやTFIIEといった基本転写因子と重複した位置に結合しており,それらの結合は相互に排他的であると考えられた.このような特徴は,RNAポリメラーゼIIという同じ複合体を核にしているにもかかわらず,転写開始複合体と転写伸長複合体とが大きく異なる構造および役割をもつことを示すとともに,転写開始複合体が転写伸長複合体に転換される過程を理解するうえでも重要である.
転写はきわめて複雑な過程であり,転写の開始あるいは伸長を含む多くのステージにおいてRNAポリメラーゼはさまざまに異なる高次複合体を形成する.この研究においては,そのなかのひとつ,RNAポリメラーゼII転写伸長複合体の構造を明らかにすることにより,転写の持続的な伸長を可能とするしくみや,転写の開始から伸長への移行の機構の一端が明らかにされた.一方,転写の伸長の際のRNAポリメラーゼIIには,この研究において解析されたもっとも基本的な3種類の転写伸長因子にくわえ,きわめて多様な因子が相互作用する.それらは,たとえば転写と共役したDNAの修復といった高次機能に必須であるが,この研究は,それら高次機能の足場となる複合体の構造を明らかにしたという点でも重要である.これらの高次機能は,その分子機構それ自体が興味深いことにくわえ,疾患とのかかわりも深い.いまだ多くの技術的な困難はあろうが,すべての因子を含む超巨大複合体の構造が原子レベルで明らかにされる日を期待したい.
略歴:理化学研究所ライフサイエンス技術基盤研究センター 研究員.
関根 俊一(Shun-ichi Sekine)
理化学研究所ライフサイエンス技術基盤研究センター チームリーダー.
研究室URL:http://www.clst.riken.jp/ja/science/labs/ssb/struc/smsb/
© 2017 江原晴彦・関根俊一 Licensed under CC 表示 2.1 日本
(理化学研究所ライフサイエンス技術基盤研究センター 超分子構造解析研究チーム)
email:関根俊一
DOI: 10.7875/first.author.2017.086
Structure of the complete elongation complex of RNA polymerase II with basal factors.
Haruhiko Ehara, Takeshi Yokoyama, Hideki Shigematsu, Shigeyuki Yokoyama, Mikako Shirouzu, Shun-ichi Sekine
Science, 357, 921-924 (2017)
要 約
真核生物においてmRNAの転写を担うRNAポリメラーゼIIは遺伝子の発現において中核的な役割をはたす.転写の伸長の際のRNAポリメラーゼIIにはDNAやRNAだけでなくさまざまな転写伸長因子が結合するが,それらすべてを含む高次複合体の分子構造は不明であった.筆者らは,X線結晶構造解析法とクライオ電子顕微鏡による単粒子解析法という2つの手法を組み合わせることにより,RNAポリメラーゼIIに高度に保存された基本的な転写伸長因子であるSpt4-Spt5複合体,Elf1,TFIISの結合した転写伸長複合体の構造を解明し,それら転写伸長因子の役割や転写開始複合体との大きな違いについて明らかにした.
はじめに
RNAポリメラーゼIIによる転写は高度な制御をうけており,転写の開始,伸長,終結といった複数の段階から構成される.また,それぞれの段階に特有の機能を実現するため,さまざまに異なる転写因子がRNAポリメラーゼIIと結合し,転写の段階ごとに固有の高次複合体を形成することが知られている.たとえば,転写の開始においては,基本転写因子とよばれる一群の転写因子がRNAポリメラーゼIIと結合することにより巨大な転写開始複合体が形成される.最近のクライオ電子顕微鏡を用いた研究などにより,転写の開始にかかわる高次複合体の構造が明らかにされつつある1-3).一方,転写の伸長のときに形成される高次複合体の構造についてはこれまでほとんど知見がなかった.転写伸長複合体と結合する転写伸長因子としては多くの種類が知られているが,なかでも,Spt4-Spt5複合体,Elf1,TFIISとよばれる3種類の転写伸長因子は真核生物をこえてホモログの存在が知られており,転写の伸長においてもっとも基本的な機能に関与すると予想された.
Spt4-Spt5複合体は約100 kDaの大きなタンパク質複合体である.Spt5は古細菌だけでなく細菌まで保存された唯一の転写因子であり,転写の伸長へのかかわりが報告されてきた.たとえば,Spt4-Spt5複合体には転写の持続性を高めるはたらきのあるほか,転写の制御への関与や,転写に共役した高次機能とのかかわりが知られている4).Spt5はマルチドメインタンパク質であり,酵母においてはNGNドメイン,5つのKOWドメイン,CTRとよばれるリピート配列から構成される.
Elf1は約10 kDaの小さなタンパク質であり,亜鉛結合モチーフをもつ.Spt4-Spt5複合体やTFIISなど,転写の伸長にかかわるタンパク質と合成致死性を示すことが知られているほか5),クロマチン-免疫沈降法を用いた解析により転写の伸長の際のRNAポリメラーゼIIとの結合が示唆されている6).また,真核生物のみならず一部の古細菌にも存在するなど高い保存性をもち7),転写の伸長において基本的な機能にかかわると考えられるが具体的な知見には乏しい.
TFIISは古細菌および真核生物に保存されており,転写の伸長が途中で停止した際にRNAポリメラーゼIIのもつRNA切断活性を促進して転写を再開することが知られている.これまで,RNAポリメラーゼIIとTFIISとの複合体の構造はいくつか報告されており,2次チャネルとよばれる活性部位に連なる穴の近辺に結合することが知られている8,9).
この研究においては,Spt4-Spt5複合体,Elf1,TFIISの結合したRNAポリメラーゼII転写伸長複合体の構造を解析し,細胞において実際にはたらく状態に近い,転写の伸長に特化したRNAポリメラーゼII転写伸長複合体の構造をはじめて明らかにした.
1.RNAポリメラーゼII転写伸長複合体の再構成および構造の決定
RNAポリメラーゼIIについては,ゲノムに精製用のタグを組み込んだメタノール資化性酵母を用い,内在性の複合体を精製して用いた10,11).Spt4-Spt5複合体,Elf1,TFIISについては,いずれも大腸菌を用いた系において発現させ精製した.それぞれの転写伸長因子について,構造の解析や機能の解析に用いるため一部のドメインのみを含む断片や変異体を調製した.RNAポリメラーゼIIにくわえ,長さのさまざまに異なる転写の伸長を模擬した核酸や,それぞれの転写伸長因子を含むもの,含まないもの,さらには,断片や変異体など,多くの組合せについて結晶化をスクリーニングした.その結果,Spt5のKOW5ドメインおよびElf1を含むRNAポリメラーゼII転写伸長複合体について良好な結晶が得られ,X線結晶構造解析法により3.0Åの分解能で構造を決定した(PDB ID:5XOG).
Spt4-Spt5複合体のNGNドメインからKOW5ドメインまでを含む断片およびTFIISの不活性な変異体を含むRNAポリメラーゼII転写伸長複合体について,クライオ電子顕微鏡による単粒子解析法により構造を決定した.核酸としては,結晶化に用いたものと比べ上流に長い配列をもつDNAを用いた.精製の手順の最適化,クロスリンカーの利用,界面活性剤の添加などのくふうをつうじて観察するタンパク質の溶液からなる氷の薄膜の性質を改善したのち,3000枚をこえる画像を撮影した.約68万個の粒子を用いた画像解析により3.8Åの分解能をもつマップが得られた(PDB ID:5XON).しかし,転写伸長因子が結合すると思われる3箇所に大きな構造多型が存在したため,それぞれの部位に着目して粒子の画像を分類した.その結果,上流のDNA,Spt4,Spt5のNGNドメインおよびKOW1ドメインがよく分離したマップが4.3Åの分解能で,StalkとよばれるRNAポリメラーゼIIから飛び出た領域とSpt5のKOW4ドメインが分離したマップが4.7Åの分解能で,TFIISがよく分離したマップが3.9Åの分解能で,それぞれ得られた.
X線結晶構造解析法により得られた構造とクライオ電子顕微鏡による単粒子解析法から得られた複数のマップとを組み合わせることにより,Spt4-Spt5複合体,Elf1,TFIISを含むRNAポリメラーゼII転写伸長複合体の構造モデルを作製した(図1).この完全なRNAポリメラーゼII転写伸長複合体において,おのおのの転写伸長因子はRNAポリメラーゼIIの表面の広い範囲に分布していた.Elf1は下流のDNAの近傍に結合すること,Spt4とSpt5のNGNドメインおよびKOW1ドメインは上流のDNAをとりかこむように位置すること,Spt5のKOW4ドメインはRNAポリメラーゼIIのStalkに結合すること,Spt5のKOW5ドメインはRNAの出口の付近に形成されたくぼみに結合することが明らかにされた.Spt4-Spt5複合体およびElf1は核酸の溶媒への露出部をおおうように結合しており,転写の伸長の際にDNAおよびRNAの結合を安定化させることが示唆された.
2.おのおのの転写伸長因子の結合および転写の伸長の際の核酸とのかかわり
Elf1はRNAポリメラーゼIIのLobeとよばれる領域とClampとよばれる領域とのあいだにはさまれるように位置し,下流の2本鎖DNAが結合する溝の上部の隙間をうめていた.Elf1の結合により完全に閉じた“DNA導入トンネル”が形成され(図2a),下流のDNAの安定な保持に寄与すると考えられた.さらに,Elf1には塩基性残基に富んだN末端領域が存在し,この領域が下流のDNAと直接に相互作用することが機能面においても重要であることが示唆された.
Spt5のNGNドメインはRNAポリメラーゼIIのClampとProtrusionとよばれる領域とのあいだにはさまるように位置していた.また,Spt5のKOW1ドメインはNGNドメインのとなりに位置し,RNAポリメラーゼIIのClampおよびWallとよばれる領域に接していた.Spt4はSpt5のNGNドメインと強く結合しており,RNAポリメラーゼIIからもっとも遠い位置に位置していた.Spt4,Spt5のNGNドメインおよびKOW1ドメインは上流の2本鎖DNAをとりかこむようなかたちで配置しており,RNAポリメラーゼIIの一部の領域とともに完全に閉じた“DNA排出トンネル”を形成していた(図2b).2本鎖DNAのRNAポリメラーゼIIに近い部分や転写バブルの非鋳型鎖DNAは,Spt5のNGNドメインあるいはKOW1ドメインと直接に相互作用しており,これらのドメインが,転写バブルの1本鎖DNAがよりあわさり2本鎖DNAにもどるのを促進することが示唆された.一方で,DNA排出トンネルのRNAポリメラーゼIIから遠い部位については,複数の塩基性残基が存在するものの,2本鎖DNAからはやや距離がありDNAを比較的ゆるくとりかこむかたちになっていた.結果として,DNA排出トンネルはジョウゴのような形状をとり,2本鎖DNAの根元をしっかりと固定する一方で,RNAポリメラーゼIIから遠い部位に関してはある程度のゆらぎを許容していることが明らかにされた.
Spt5について,KOW4ドメインはRNAポリメラーゼIIのStalkと結合していた.また,KOW5ドメインはRNAの出口の近傍のくぼみにはまり込んでいた.さらに,KOW4ドメインとKOW5ドメインとのあいだのリンカーに相当すると考えられる弱い密度が観測された.RNAポリメラーゼIIのStalk,Spt5のKOW4ドメインおよびKOW5ドメインはRNAの出口をとりかこむように存在し,RNAポリメラーゼIIからでてくるRNAを正しく導くための“RNA排出トンネル”を構成することが示唆された(図2c).また,KOW4ドメインおよびKOW5ドメインにくわえて,近傍にはKOW1ドメイン,および,密度は観測されなかったもののKOW2ドメインおよびKOW3ドメインが存在すると考えられ,これらSpt5のKOWドメインが共同してRNAポリメラーゼIIから排出されるDNAとRNAとを物理的に隔離することが明らかにされた.さらに,RNAの出口の近傍には新しく合成されたRNAを修飾したりするさまざまな因子が結合することが知られており,Spt5により補強および拡張されたRNA排出トンネルは新しく合成されたRNAを別の酵素へとうけわたすための土台として重要なはたらきをすることが示唆された.
3.転写伸長複合体と転写開始前複合体との構造の比較
今回,構造の明らかにされたRNAポリメラーゼII転写伸長複合体は,以前に構造が報告されていたRNAポリメラーゼII転写開始前複合体2)(図3a,PDB ID:5FZ5)とは構造が大きく異なっていた.転写開始前複合体においては,DNAは基本転写因子によりRNAポリメラーゼIIの外側に結合し,それら基本転写因子の協調的なはたらきにより2本鎖DNAの一部がほどけDNAはRNAポリメラーゼIIの内部の空洞に取り込まれると考えられている.一方,転写伸長複合体(図3b)においては,DNAおよびRNAはRNAポリメラーゼIIの内部に完全に取り込まれた状態にあり,転写伸長因子がそれらの周囲をとりかこみ完全に独立したトンネルを形成することにより安定かつ継続した転写の伸長を可能すると考えられた.また,核酸の通り道をより規定されたものとすることが,ほかの因子への核酸のうけわたしなどをつうじた転写と共役したさまざまな高次機能の実現に重要であると考えられた.
さらに,基本転写因子どうし,および,転写伸長因子どうしは互いに協調的にRNAポリメラーゼII複合体と結合する一方,基本転写因子と転写伸長因子は構造において相互に排他的な結合様式をもっていた.たとえば,Elf1,Spt4,Spt5のNGNドメインは,転写開始前複合体のDNAと衝突してしまうため転写の開始の際には結合できないと考えられた.同様に,転写の伸長の際のSpt4やSpt5のKOW4ドメインは,転写の開始の際のTFIIFやTFIIEといった基本転写因子と重複した位置に結合しており,それらの結合は相互に排他的であると考えられた.このような特徴は,RNAポリメラーゼIIという同じ複合体を核にしているにもかかわらず,転写開始複合体と転写伸長複合体とが大きく異なる構造および役割をもつことを示すとともに,転写開始複合体が転写伸長複合体に転換される過程を理解するうえでも重要である.
おわりに
転写はきわめて複雑な過程であり,転写の開始あるいは伸長を含む多くのステージにおいてRNAポリメラーゼはさまざまに異なる高次複合体を形成する.この研究においては,そのなかのひとつ,RNAポリメラーゼII転写伸長複合体の構造を明らかにすることにより,転写の持続的な伸長を可能とするしくみや,転写の開始から伸長への移行の機構の一端が明らかにされた.一方,転写の伸長の際のRNAポリメラーゼIIには,この研究において解析されたもっとも基本的な3種類の転写伸長因子にくわえ,きわめて多様な因子が相互作用する.それらは,たとえば転写と共役したDNAの修復といった高次機能に必須であるが,この研究は,それら高次機能の足場となる複合体の構造を明らかにしたという点でも重要である.これらの高次機能は,その分子機構それ自体が興味深いことにくわえ,疾患とのかかわりも深い.いまだ多くの技術的な困難はあろうが,すべての因子を含む超巨大複合体の構造が原子レベルで明らかにされる日を期待したい.
文 献
- He, Y., Yan, C., Fang, J. et al.: Near-atomic resolution visualization of human transcription promoter opening. Nature, 533, 359-365 (2016)[PubMed]
- Plaschka, C., Hantsche, M., Dienemann, C. et al.: Transcription initiation complex structures elucidate DNA opening. Nature, 533, 353-358 (2016)[PubMed]
- Murakami, K., Tsai, K. L., Kalisman, N. et al.: Structure of an RNA polymerase II preinitiation complex. Proc. Natl. Acad. Sci. USA, 112, 13543-13548 (2015)[PubMed]
- Hartzog, G. A. & Fu, J.: The Spt4-Spt5 complex: a multi-faceted regulator of transcription elongation. Biochim. Biophys. Acta, 1829, 105-115 (2013)[PubMed]
- Prather, D., Krogan, N. J., Emili, A. et al.: Identification and characterization of Elf1, a conserved transcription elongation factor in Saccharomyces cerevisiae. Mol. Cell. Biol., 25, 10122-10135 (2005)[PubMed]
- Mayer, A., Lidschreiber, M., Siebert, M. et al.: Uniform transitions of the general RNA polymerase II transcription complex. Nat. Struct. Mol. Biol., 17, 1272-1278 (2010)[PubMed]
- Daniels, J. P., Kelly, S., Wickstead, B. et al.: Identification of a crenarchaeal orthologue of Elf1: implications for chromatin and transcription in Archaea. Biol. Direct., 4, 24 (2009)[PubMed]
- Kettenberger, H., Armache, K. -J. & Cramer, P.: Architecture of the RNA polymerase II-TFIIS complex and implications for mRNA cleavage. Cell, 114, 347-357 (2003)[PubMed]
- Wang, D., Bushnell, D. A., Huang, X. et al.: Structural basis of transcription: backtracked RNA polymerase II at 3.4 angstrom resolution. Science, 324, 1203-1206 (2009)[PubMed]
- Higo, T., Suka, N., Ehara, H. et al.: Development of a hexahistidine-3× FLAG-tandem affinity purification method for endogenous protein complexes in Pichia pastoris. J. Struct. Funct. Genomics., 15, 191-199 (2014)[PubMed]
- Ehara, H., Umehara, T., Sekine, S. et al.: Crystal structure of RNA polymerase II from Komagataella pastoris. Biochem. Biophys. Res. Commun., 487, 230-235 (2017)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:理化学研究所ライフサイエンス技術基盤研究センター 研究員.
関根 俊一(Shun-ichi Sekine)
理化学研究所ライフサイエンス技術基盤研究センター チームリーダー.
研究室URL:http://www.clst.riken.jp/ja/science/labs/ssb/struc/smsb/
© 2017 江原晴彦・関根俊一 Licensed under CC 表示 2.1 日本