CRISPR-Cpf1によるPAMの寛容な認識機構
山野 峻・西増弘志・濡木 理
(東京大学大学院理学系研究科 生物科学専攻生物化学講座構造生命科学研究室)
email:山野 峻,西増弘志,濡木 理
DOI: 10.7875/first.author.2017.078
Structural basis for the canonical and non-canonical PAM recognition by CRISPR-Cpf1.
Takashi Yamano, Bernd Zetsche, Ryuichiro Ishitani, Feng Zhang, Hiroshi Nishimasu, Osamu Nureki
Molecular Cell, 67, 633-645.e3 (2017)
V型CRISPR-Cas系に関与するRNA依存性DNAヌクレアーゼCpf1は,ガイドRNAと相補な2本鎖DNAを特異的に切断するはたらきをもち,ゲノム編集ツールとして利用されている.標的となるDNAのターゲティングには,ガイドRNAとの配列相補性にくわえ,標的となる配列の近傍に存在するPAMとよばれる特定の配列が必要である.ゲノム編集に利用されているCpf1はTTTV(VはA,C,G)という配列をPAMとして認識するが,最近の研究により,Cをふくむ配列もPAMとして認識されることが明らかにされてきた.しかし,Cpf1が複数の配列をPAMとして認識する分子機構は不明であった.今回,筆者らは,PAMとして4種類の異なる配列をふくむCpf1-ガイドRNA-標的DNA複合体の結晶構造を決定し,Cpf1によるPAMの寛容な認識機構について明らかにした.
原核生物はCRISPR(clustered regularly interspaced short palindromic repeat)-Cas(CRISPR-associated protein)とよばれる獲得免疫機構をもつ.CRISPR-Cas系においては,Casタンパク質がcrRNA(CRISPR RNA)とエフェクター複合体を形成し,crRNAと相補な外来性の核酸を認識し切断する.CRISPR-Cas系はクラス1とクラス2に分類され,クラス2のCRISPR-Cas系においては単一のCasがエフェクター分子としてはたらく.クラス2のCRISPR-Cas系はII型,V型,VI型に分類され,V型CRISPR-Cas系においてはCpf1(Cas12aともよばれる)がエフェクター分子としてはたらく1).細菌Lachnospiraceae bacterium ND2006や細菌Acidaminoncoccus sp. BV3L6に由来するCpf1は真核生物の細胞においてもDNAの切断活性を示すことから,ゲノム編集ツールとして用いられている2-5).II型CRISPR-Cas系に関与するCas9もゲノム編集ツールとして広く利用されているが,Cpf1はCas9とは異なる特徴をもつ.両者とも,標的となるDNAのターゲティングには,ガイドRNAと標的となる配列とのあいだの相補性にくわえ,標的となる配列の近傍にPAM(protospacer adjacent motif)とよばれる特定の配列が存在することが必須であるが,Cas9はGにとむPAMを認識する一方,Cpf1はTにとむPAMを認識する.L. bacterium ND2006およびAcidaminoncoccus sp. BV3L6に由来するCpf1はTTTV(VはA,C,G)という配列をPAMとして認識する.さらに,最近の研究により,Cpf1はTTTVにくわえCをふくむいくつかの配列もPAMとして認識することが明らかにされた4).これまでに,結晶構造から,Cpf1の全体構造,crRNAによる認識機構,標的となるDNAの切断機構,PAMとしてTTTVを認識する機構について明らかにされてきた6-10)(文献8) は新着論文レビュー でも掲載).しかし,Cpf1によるPAMの寛容な認識機構については不明であった.
哺乳類の細胞においてCpf1はCをふくむ配列をPAMとして認識することが報告されていたが4),in vitroにおけるPAMに対する嗜好性については詳細には調べられていなかった.そこで,PAMとしてさまざまな配列をふくむプラスミドを用いて,in vitroにおけるCpf1の切断活性を測定した.その結果,Cpf1はPAMとしてTTTVをふくむプラスミドをもっとも効率的に切断することが確認された.また,Cを1つふくむTTCA,TCTA,CTTAもPAMとして認識された.一方,Cを2つ以上ふくむTCCA,CCTA,CCCAはほとんど認識されなかった.これらの結果から,先行研究の結果と一致して,Cpf1はTTTVおよびCを1つふくむTTCV,TCTV,CTTVをPAMとして認識することが確かめられた.
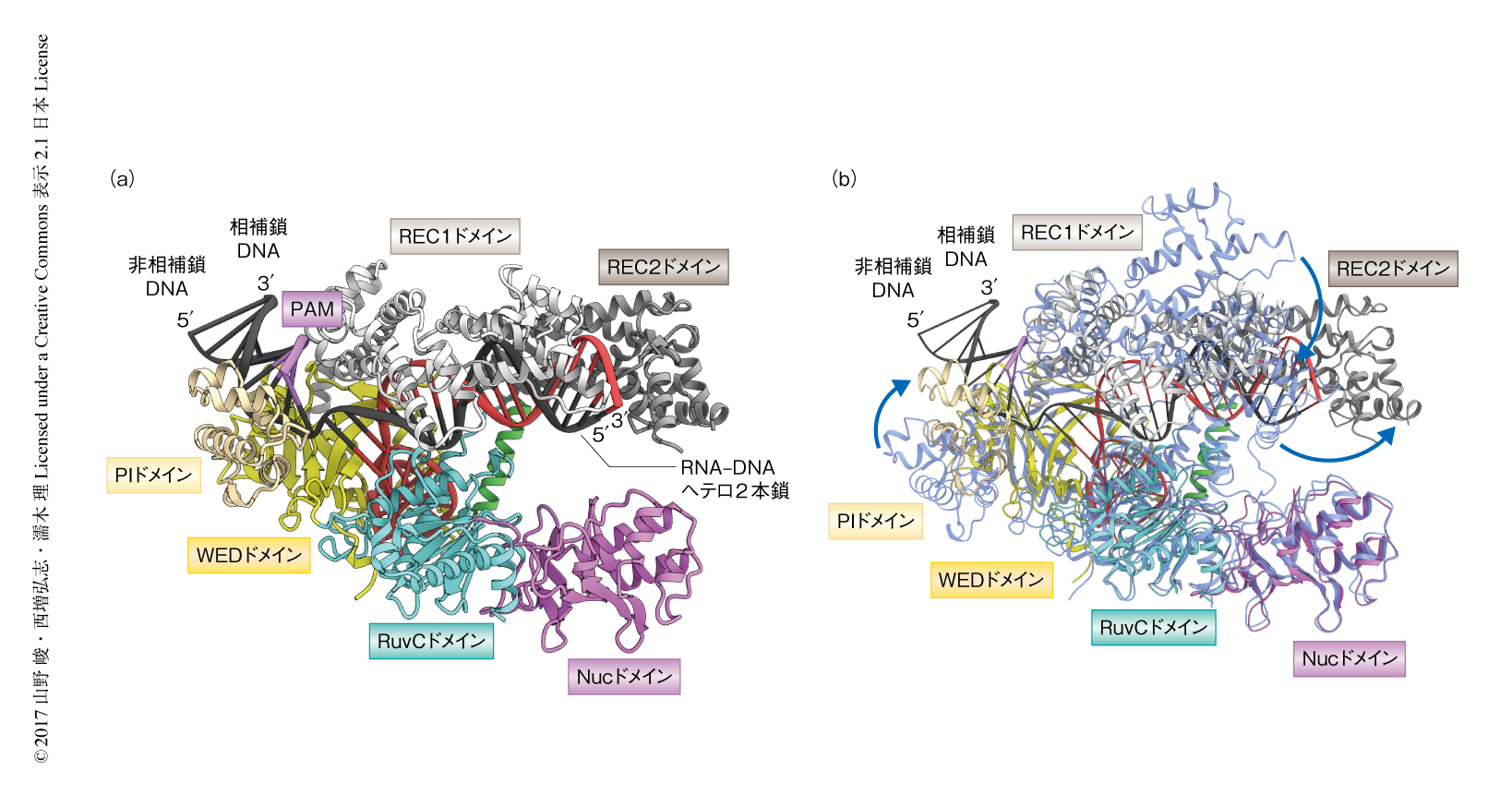
L. bacterium ND2006に由来するCpf1(1~1228残基),crRNA(40塩基),相補鎖DNA(29塩基),PAMとしてTTTAをふくむ非相補鎖DNA(9塩基)からなる複合体の結晶構造を2.5Å分解能で決定した(PDB ID:5XUS,図1a).Cpf1は,REC1ドメインとREC2ドメインから構成されるRECローブ,および,WEDドメイン,PIドメイン,RuvCドメイン,Nucドメインから構成されるNUCローブからなるバイローブ構造をとっていた.crRNAは5’ハンドルとガイド配列から構成されていた.5’ハンドルはシュードノット構造を形成し,WEDドメインおよびRuvCドメインにより認識されていた.ガイド配列は相補鎖DNAと20塩基対のヘテロ2本鎖を形成し,RECローブに収容されていた.非相補鎖DNAは相補鎖DNAとPAM 2本鎖を形成し,REC1ドメイン,WEDドメイン,PIドメインにより形成されるPAM結合チャネルにより認識されていた.ほかの細菌に由来するCpf1との構造の比較から7-10),Cpf1ファミリーは保存された立体構造をもつことが明らかにされた.さらに,Cpf1-crRNA複合体(PDB ID:5ID6)との比較から6),標的となるDNAの結合にともないRECローブおよびPIドメインが構造変化を起こすことが明らかにされた(図1b).
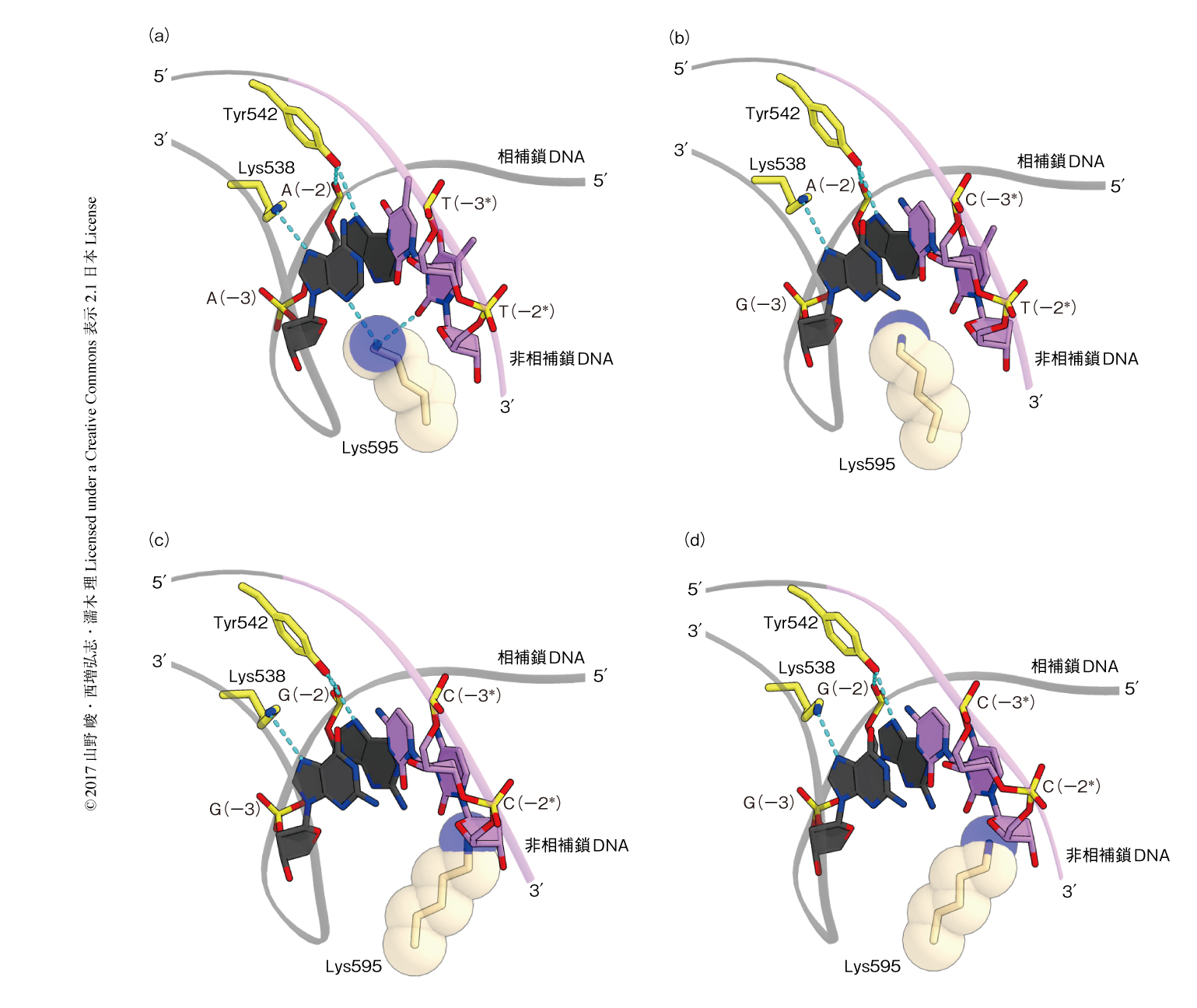
Cpf1-crRNA-標的DNA複合体において,TTTAをふくむPAM 2本鎖はB型DNAに比べ狭い副溝をもつゆがんだ二重らせん構造をとり,PIドメインのLys595により副溝の側から認識されていた(図2a).さらに,PAM 2本鎖はWEDドメインのLys538およびTyr542により主溝の側から認識されていた.このような,PAM 2本鎖の形状および配列の両方を認識する機構は,ほかの細菌に由来するCpf1と共通していた8).
Cpf1によるPAMの寛容な認識機構について明らかにするため,PAMとしてTCTA(PDB ID:5XUT),TCCA(PDB ID:5XUU),CCCA(PDB ID:5XUZ)をふくむ3つの複合体の結晶構造を決定した.これらの構造の比較から,PAM 2本鎖にふくまれるGC塩基対が増えるにしたがい二重らせん構造のゆがみが減り,PAM 2本鎖がB型DNAに近づくことが明らかにされた.それにともないPIドメインは外側へ移動し,構造の異なるPAM 2本鎖を収容していた.PAMとしてTTTA,TCTA,TCCA,CCCAをふくむ4つの複合体の構造において,WEDドメインのLys538およびTyr542は同様にPAM 2本鎖と相互作用していた一方,PIドメインのLys595は異なる位置に存在していた.TTTAをふくむ複合体とは異なり,TCTAをふくむ複合体においては,Lys595はPAM 2本鎖の副溝に挿入されていたが,PAMとは水素結合を形成していなかった(図2b).一方,TCCAをふくむ複合体およびCCCAをふくむ複合体においては,Lys595はPAM 2本鎖の副溝に挿入されていなかった(図2cおよび図2d).以上の結果から,Cpf1のPIドメインは構造変化を起こし異なる配列をもつPAM 2本鎖と相互作用すること,および,この相互作用の強さがPAMの認識に重要であることが明らかにされた.
Cas9とCpf1との構造の比較から,両者のあいだのPAMの認識機構の違いが明らかにされた.Cas9においては,PAM 2本鎖はPIドメインの溝に結合し,おもに主溝の側から塩基に特異的に認識される11).一方,Cpf1においては,PAM 2本鎖は3つのドメインから形成されるPAM結合チャネルに収容され,その形状および配列の両方が認識される.Cpf1のPIドメインは構造的に柔軟であるため,PAM結合チャネルは異なる配列をふくむPAM 2本鎖を認識することができる.したがって,Cpf1はCas9とは異なり,複数の配列をPAMとして認識できると考えられた.
Cpf1はCas9よりも標的となるDNAに対する特異性が高く,オフターゲット配列の切断の少ないことが報告されている2,3).今回の研究により,TTTVだけでなくCをふくむ配列もPAMとして認識することがはっきりしたことから,Cpf1をゲノム編集に利用する際はこれらのPAMも考慮して標的となる配列を選択するのがよいと考えられる.近年,構造情報と分子進化的な手法を組み合わせてCpf1の改変体が創出されている12,13)(文献12) は新着論文レビュー でも掲載).今回,得られた知見をもとに,TTTVのみをPAMとして認識するCpf1の改変体を創出することができれば,より特異性の高いゲノム編集ツールとなる可能性が考えられる.
略歴:東京大学大学院理学系研究科博士課程 在学中.
研究テーマ:CRISPR-Cas機構の構造解析.
抱負:倒れない.
西増 弘志(Hiroshi Nishimasu)
略歴:2007年 東京大学大学院農学生命科学研究科 修了,2013年より東京大学大学院理学系研究科 助教.
研究テーマ:CRISPR-Casの構造および機能.
関心事:減量.つぎの10年,何をするか.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究室URL:http://www.nurekilab.net/
© 2017 山野 峻・西増弘志・濡木 理 Licensed under CC 表示 2.1 日本
(東京大学大学院理学系研究科 生物科学専攻生物化学講座構造生命科学研究室)
email:山野 峻,西増弘志,濡木 理
DOI: 10.7875/first.author.2017.078
Structural basis for the canonical and non-canonical PAM recognition by CRISPR-Cpf1.
Takashi Yamano, Bernd Zetsche, Ryuichiro Ishitani, Feng Zhang, Hiroshi Nishimasu, Osamu Nureki
Molecular Cell, 67, 633-645.e3 (2017)
要 約
V型CRISPR-Cas系に関与するRNA依存性DNAヌクレアーゼCpf1は,ガイドRNAと相補な2本鎖DNAを特異的に切断するはたらきをもち,ゲノム編集ツールとして利用されている.標的となるDNAのターゲティングには,ガイドRNAとの配列相補性にくわえ,標的となる配列の近傍に存在するPAMとよばれる特定の配列が必要である.ゲノム編集に利用されているCpf1はTTTV(VはA,C,G)という配列をPAMとして認識するが,最近の研究により,Cをふくむ配列もPAMとして認識されることが明らかにされてきた.しかし,Cpf1が複数の配列をPAMとして認識する分子機構は不明であった.今回,筆者らは,PAMとして4種類の異なる配列をふくむCpf1-ガイドRNA-標的DNA複合体の結晶構造を決定し,Cpf1によるPAMの寛容な認識機構について明らかにした.
はじめに
原核生物はCRISPR(clustered regularly interspaced short palindromic repeat)-Cas(CRISPR-associated protein)とよばれる獲得免疫機構をもつ.CRISPR-Cas系においては,Casタンパク質がcrRNA(CRISPR RNA)とエフェクター複合体を形成し,crRNAと相補な外来性の核酸を認識し切断する.CRISPR-Cas系はクラス1とクラス2に分類され,クラス2のCRISPR-Cas系においては単一のCasがエフェクター分子としてはたらく.クラス2のCRISPR-Cas系はII型,V型,VI型に分類され,V型CRISPR-Cas系においてはCpf1(Cas12aともよばれる)がエフェクター分子としてはたらく1).細菌Lachnospiraceae bacterium ND2006や細菌Acidaminoncoccus sp. BV3L6に由来するCpf1は真核生物の細胞においてもDNAの切断活性を示すことから,ゲノム編集ツールとして用いられている2-5).II型CRISPR-Cas系に関与するCas9もゲノム編集ツールとして広く利用されているが,Cpf1はCas9とは異なる特徴をもつ.両者とも,標的となるDNAのターゲティングには,ガイドRNAと標的となる配列とのあいだの相補性にくわえ,標的となる配列の近傍にPAM(protospacer adjacent motif)とよばれる特定の配列が存在することが必須であるが,Cas9はGにとむPAMを認識する一方,Cpf1はTにとむPAMを認識する.L. bacterium ND2006およびAcidaminoncoccus sp. BV3L6に由来するCpf1はTTTV(VはA,C,G)という配列をPAMとして認識する.さらに,最近の研究により,Cpf1はTTTVにくわえCをふくむいくつかの配列もPAMとして認識することが明らかにされた4).これまでに,結晶構造から,Cpf1の全体構造,crRNAによる認識機構,標的となるDNAの切断機構,PAMとしてTTTVを認識する機構について明らかにされてきた6-10)(文献8) は新着論文レビュー でも掲載).しかし,Cpf1によるPAMの寛容な認識機構については不明であった.
1.Cpf1のPAMに対する嗜好性
哺乳類の細胞においてCpf1はCをふくむ配列をPAMとして認識することが報告されていたが4),in vitroにおけるPAMに対する嗜好性については詳細には調べられていなかった.そこで,PAMとしてさまざまな配列をふくむプラスミドを用いて,in vitroにおけるCpf1の切断活性を測定した.その結果,Cpf1はPAMとしてTTTVをふくむプラスミドをもっとも効率的に切断することが確認された.また,Cを1つふくむTTCA,TCTA,CTTAもPAMとして認識された.一方,Cを2つ以上ふくむTCCA,CCTA,CCCAはほとんど認識されなかった.これらの結果から,先行研究の結果と一致して,Cpf1はTTTVおよびCを1つふくむTTCV,TCTV,CTTVをPAMとして認識することが確かめられた.
2.Cpf1-crRNA-標的DNA複合体の結晶構造
L. bacterium ND2006に由来するCpf1(1~1228残基),crRNA(40塩基),相補鎖DNA(29塩基),PAMとしてTTTAをふくむ非相補鎖DNA(9塩基)からなる複合体の結晶構造を2.5Å分解能で決定した(PDB ID:5XUS,図1a).Cpf1は,REC1ドメインとREC2ドメインから構成されるRECローブ,および,WEDドメイン,PIドメイン,RuvCドメイン,Nucドメインから構成されるNUCローブからなるバイローブ構造をとっていた.crRNAは5’ハンドルとガイド配列から構成されていた.5’ハンドルはシュードノット構造を形成し,WEDドメインおよびRuvCドメインにより認識されていた.ガイド配列は相補鎖DNAと20塩基対のヘテロ2本鎖を形成し,RECローブに収容されていた.非相補鎖DNAは相補鎖DNAとPAM 2本鎖を形成し,REC1ドメイン,WEDドメイン,PIドメインにより形成されるPAM結合チャネルにより認識されていた.ほかの細菌に由来するCpf1との構造の比較から7-10),Cpf1ファミリーは保存された立体構造をもつことが明らかにされた.さらに,Cpf1-crRNA複合体(PDB ID:5ID6)との比較から6),標的となるDNAの結合にともないRECローブおよびPIドメインが構造変化を起こすことが明らかにされた(図1b).
3.PAMとしてTTTVを認識する機構
Cpf1-crRNA-標的DNA複合体において,TTTAをふくむPAM 2本鎖はB型DNAに比べ狭い副溝をもつゆがんだ二重らせん構造をとり,PIドメインのLys595により副溝の側から認識されていた(図2a).さらに,PAM 2本鎖はWEDドメインのLys538およびTyr542により主溝の側から認識されていた.このような,PAM 2本鎖の形状および配列の両方を認識する機構は,ほかの細菌に由来するCpf1と共通していた8).
4.CをふくむPAMの認識機構
Cpf1によるPAMの寛容な認識機構について明らかにするため,PAMとしてTCTA(PDB ID:5XUT),TCCA(PDB ID:5XUU),CCCA(PDB ID:5XUZ)をふくむ3つの複合体の結晶構造を決定した.これらの構造の比較から,PAM 2本鎖にふくまれるGC塩基対が増えるにしたがい二重らせん構造のゆがみが減り,PAM 2本鎖がB型DNAに近づくことが明らかにされた.それにともないPIドメインは外側へ移動し,構造の異なるPAM 2本鎖を収容していた.PAMとしてTTTA,TCTA,TCCA,CCCAをふくむ4つの複合体の構造において,WEDドメインのLys538およびTyr542は同様にPAM 2本鎖と相互作用していた一方,PIドメインのLys595は異なる位置に存在していた.TTTAをふくむ複合体とは異なり,TCTAをふくむ複合体においては,Lys595はPAM 2本鎖の副溝に挿入されていたが,PAMとは水素結合を形成していなかった(図2b).一方,TCCAをふくむ複合体およびCCCAをふくむ複合体においては,Lys595はPAM 2本鎖の副溝に挿入されていなかった(図2cおよび図2d).以上の結果から,Cpf1のPIドメインは構造変化を起こし異なる配列をもつPAM 2本鎖と相互作用すること,および,この相互作用の強さがPAMの認識に重要であることが明らかにされた.
5.Cas9とCpf1におけるPAMの認識機構の違い
Cas9とCpf1との構造の比較から,両者のあいだのPAMの認識機構の違いが明らかにされた.Cas9においては,PAM 2本鎖はPIドメインの溝に結合し,おもに主溝の側から塩基に特異的に認識される11).一方,Cpf1においては,PAM 2本鎖は3つのドメインから形成されるPAM結合チャネルに収容され,その形状および配列の両方が認識される.Cpf1のPIドメインは構造的に柔軟であるため,PAM結合チャネルは異なる配列をふくむPAM 2本鎖を認識することができる.したがって,Cpf1はCas9とは異なり,複数の配列をPAMとして認識できると考えられた.
おわりに
Cpf1はCas9よりも標的となるDNAに対する特異性が高く,オフターゲット配列の切断の少ないことが報告されている2,3).今回の研究により,TTTVだけでなくCをふくむ配列もPAMとして認識することがはっきりしたことから,Cpf1をゲノム編集に利用する際はこれらのPAMも考慮して標的となる配列を選択するのがよいと考えられる.近年,構造情報と分子進化的な手法を組み合わせてCpf1の改変体が創出されている12,13)(文献12) は新着論文レビュー でも掲載).今回,得られた知見をもとに,TTTVのみをPAMとして認識するCpf1の改変体を創出することができれば,より特異性の高いゲノム編集ツールとなる可能性が考えられる.
文 献
- Zetsche, B., Gootenberg, J. S., Abudayyeh, O. O. et al.: Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 163, 759-771 (2015)[PubMed]
- Kim, D., Kim, J., Hur, J. K. et al.: Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nat. Biotechnol., 34, 863-868 (2016)[PubMed]
- Kleinstiver, B. P., Tsai, S. Q., Prew, M. S. et al.: Genome-wide specificities of CRISPR-Cas Cpf1 nucleases in human cells. Nat. Biotechnol., 34, 869-874 (2016)[PubMed]
- Kim, H. K., Song, M., Lee, J. et al.: In vivo high-throughput profiling of CRISPR-Cpf1 activity. Nat. Methods, 14, 153-159 (2017)[PubMed]
- Zetsche, B., Heidenreich, M., Mohanraju, P. et al.: Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array. Nat. Biotechnol., 35, 31-34 (2017)[PubMed]
- Dong, D., Ren, K., Qiu, X. et al.: The crystal structure of Cpf1 in complex with CRISPR RNA. Nature, 532, 522-526 (2016)[PubMed]
- Gao, P., Yang, H., Rajashankar, K. R. et al.: Type V CRISPR-Cas Cpf1 endonuclease employs a unique mechanism for crRNA-mediated target DNA recognition. Cell Res., 26, 901-913 (2016)[PubMed]
- Yamano, T., Nishimasu, H., Zetsche, B. et al.: Crystal structure of Cpf1 in complex with guide RNA and target DNA. Cell, 165, 949-962 (2016)[PubMed] [新着論文レビュー]
- Swarts, D., van der Oost, J. & Jinek, M.: Structural basis for guide RNA processing and seed-dependent DNA targeting by CRISPR-Cas12a. Mol. Cell, 66, 221-233 (2017)[PubMed]
- Stella, S., Alcon, P. & Montoya, G.: Structure of the Cpf1 endonuclease R-loop complex after target DNA cleavage. Nature, 546, 559-563 (2017)[PubMed]
- Anders, C., Niewoehner, O., Duerst, A. et al.: Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature, 513, 569-573 (2014)[PubMed]
- Nishimasu, H., Yamano, T., Gao, L. et al.: Structural basis for the altered PAM recognition by engineered CRISPR-Cpf1. Mol. Cell, 67, 139-147 (2017)[PubMed] [新着論文レビュー]
- Gao, L., Cox, D. B. T., Yan, W. X. et al.: Engineered Cpf1 variants with altered PAM specificities. Nat. Biotechnol., 35, 789-792 (2017)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:東京大学大学院理学系研究科博士課程 在学中.
研究テーマ:CRISPR-Cas機構の構造解析.
抱負:倒れない.
西増 弘志(Hiroshi Nishimasu)
略歴:2007年 東京大学大学院農学生命科学研究科 修了,2013年より東京大学大学院理学系研究科 助教.
研究テーマ:CRISPR-Casの構造および機能.
関心事:減量.つぎの10年,何をするか.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究室URL:http://www.nurekilab.net/
© 2017 山野 峻・西増弘志・濡木 理 Licensed under CC 表示 2.1 日本