V型CRISPR-Cas系にかかわるCpf1の結晶構造
山野 峻・西増弘志・濡木 理
(東京大学大学院理学系研究科 生物科学専攻生物化学講座構造生命科学研究室)
email:山野 峻,西増弘志,濡木 理
DOI: 10.7875/first.author.2016.048
Crystal structure of Cpf1 in complex with guide RNA and target DNA.
Takashi Yamano, Hiroshi Nishimasu, Bernd Zetsche, Hisato Hirano, Ian M. Slaymaker, Yinqing Li, Iana Fedorova, Takanori Nakane, Kira S. Makarova, Eugene V. Koonin, Ryuichiro Ishitani, Feng Zhang, Osamu Nureki
Cell, 165, 949-962 (2016)
II型CRISPR-Cas系に関与するRNA依存性DNAエンドヌクレアーゼCas9は,ガイドRNAと相補的な2本鎖DNAを特異的に切断する機能をもち,ゲノム編集ツールとして広く利用されている.最近,V型CRISPR-Cas系にかかわるRNA依存性DNAエンドヌクレアーゼCpf1が発見され,新たなゲノム編集ツールとして注目されている.Cas9とは異なり,Cpf1は標的となる2本鎖DNAを切断し突出末端を形成するが,その分子機構は不明であった.今回,筆者らは,Cpf1,ガイドRNA,標的となるDNAからなる複合体の結晶構造を決定し,その作動機構を解明した.さらに,Cpf1とCas9との構造の比較から,それらの作動機構における共通性および多様性が明らかにされた.
原核生物はCRISPR-Cas系(CRISPR:clustered regularly interspaced short palindromic repeat,Cas:CRISPR-associated protein)とよばれる獲得免疫機構をもつ.CRISPR-Cas系においては,CasがcrRNA(CRISPR RNA)とエフェクター複合体を形成し,crRNAと相補的な塩基配列をもつ外来の核酸を認識し分解する.CRISPR-Cas系はエフェクター複合体の構造にもとづき2つのクラスに分類される.クラス1のCRISPR-Cas系には複数のCasからなるマルチサブユニット複合体が関与する.一方,クラス2のCRISPR-Cas系には単一のCasが関与する.クラス1はI型,III型,IV型に分類され,クラス2はII型,V型,VI型に分類される.II型のCRISPR-Cas系においてはCas9がcrRNAおよびtracrRNA(trans-activating crRNA)とエフェクター複合体を形成し,標的となる2本鎖DNAを切断する1).Cas9による2本鎖DNAの切断には標的となる配列の近傍にPAM(protospacer adjacent motif)とよばれる特定の塩基配列が存在することが必須である.したがって,PAMによる制限はあるものの,ガイドとなるRNAの配列を変更することにより任意の配列をもつ2本鎖DNAを特異的に認識することができるため,Cas9はゲノム編集技術をはじめとするさまざまな新規の技術に応用されている2).
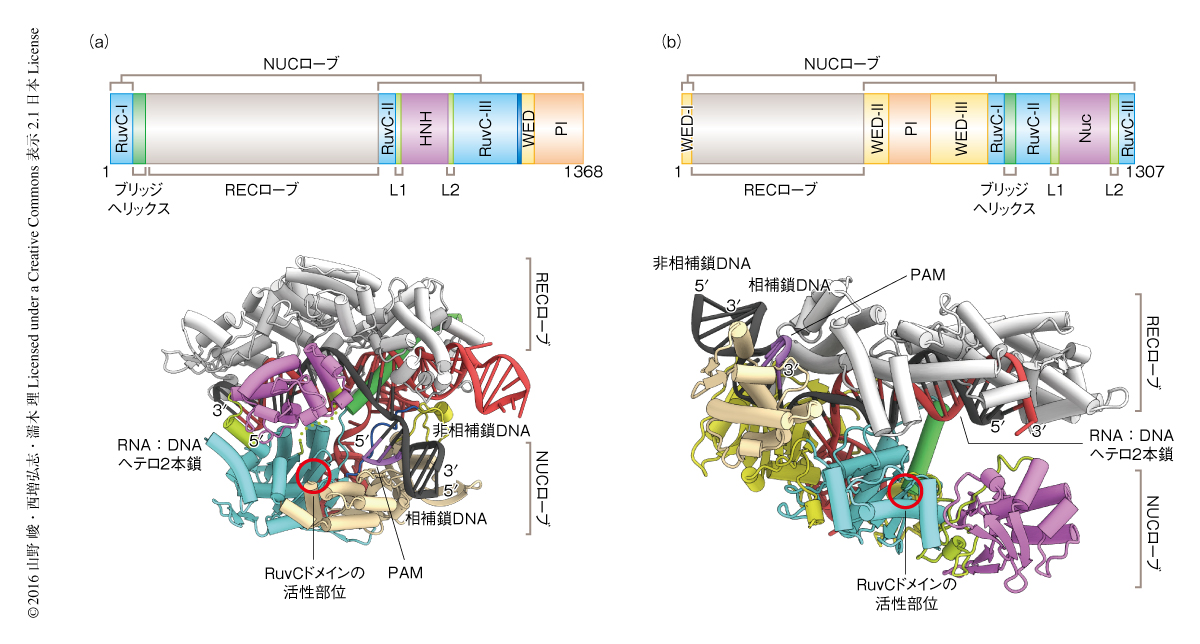
細菌Streptococcus pyogenesに由来するCas9の結晶構造(PDB ID:4UN3,図1a)から,Cas9の作動機構が明らかにされてきた.これまでに,単体のCas9 3),Cas9-ガイドRNA複合体4),Cas9-ガイドRNA-DNA複合体5-7)(文献6) は新着論文レビュー でも掲載))の結晶構造が決定され,1)Cas9は2つのローブからなる構造をもつこと,2)ガイドRNAの結合によりCas9は大きく構造を変化させること,3)ガイドRNAと相補鎖DNAはヘテロ2本鎖を形成しCas9の2つのローブのあいだに収容されること,4)Cas9のもつ2つのヌクレアーゼドメインであるRuvCドメインおよびHNHドメインはそれぞれ非相補鎖DNAおよび相補鎖DNAを切断するのに適した位置に存在すること,5)非相補鎖DNAに存在する5’-NGG-3’というPAMはPIドメインにより認識されること,などが明らかにされた.さらに,細菌Staphylococcus aureusに由来する小型のCas9などCas9オルソログの結晶構造から,CRISPR-Cas9系の多様性が解明されてきた8)(新着論文レビュー でも掲載).
2015年,V型のCRISPR-Cas系にかかわるエフェクタータンパク質としてCpf1が発見された9).Cpf1はCas9と同様にガイドRNAと協働して2本鎖DNAを切断するが,Cas9とは以下のような異なる特徴をもつ.1)Cpf1はcrRNAのみをガイドRNAとして利用しtracrRNAを必要としない.2)Cas9はGリッチなPAMを認識するが,Cpf1はTリッチなPAMを認識する.3)Cas9は標的となる2本鎖DNAをPAMの近傍において切断し平滑末端を形成するが,Cpf1はPAMから離れた位置において2本鎖DNAを切断し突出末端を形成する.4)Cas9はヌクレアーゼドメインとしてRuvCドメインおよびHNHドメインをもつが,Cpf1はHNHドメインをもたずRuvCドメインのみをもつ.RuvCドメインを除き,Cpf1は既知のタンパク質とアミノ酸配列の相同性をもたないため,そのDNA切断機構は不明であった.
Cpf1の作動機構を理解するため,細菌Acidaminococcus sp.に由来するCpf1,crRNA,PAMとして5’-TTTA-3’を含むDNAからなる複合体の結晶構造を2.8Å分解能で決定した(PDB ID:5B43,図1b).Cpf1はRECローブおよびNUCローブと名づけた2つのローブからなり,この2つのローブはブリッジヘリックスと名づけた特徴的な長いαヘリックスにより連結されていた.RECローブはREC1ドメインおよびREC2ドメインから構成されていた.NUCローブはRuvCドメインおよび3つのドメインから構成され,このうち2つのドメインは,Cas9のWEDドメインおよびPIドメインと構造は異なるが類似の役割をもっていたため,それぞれ,WEDドメインおよびPIドメインと名づけた.残りの1つのドメインは,のちに述べるように,DNAの切断にかかわることが明らかにされたため,Nucドメインと名づけた.Nucドメインは2つのリンカー領域L1およびL2によりRuvCドメインと連結されていた.RuvCドメインを除く5つのドメインは新規な構造をもっていた.
crRNAは5’ハンドルと名づけた5’末端領域とガイド配列から構成されていた(図1b).ガイド配列(G1~C24)は標的となる2本鎖DNAの相補鎖DNA(dC1~dG24)と20塩基対のRNA:DNAヘテロ2本鎖を形成し,RECローブとNUCローブとのあいだに収容され,塩基配列に非特異的に認識されていた.相補鎖DNA(dG-10~dT-1)は非相補鎖DNA(dC-10*~dA-1*)とPAMを含む2本鎖DNAを形成していた.塩基配列から5’ハンドルは単純なステムループ構造をもつと予想されていたが,結晶構造においてより複雑なシュードノット構造をもち,WEDドメインおよびRuvCドメインにより認識されていた(図1b).
PAMを含む2本鎖DNAはATリッチな2本鎖DNAに特徴的なゆがんだ二重らせん構造をとり,WEDドメイン,REC1ドメイン,PIドメインにより認識されていた(図1b).4文字目のdT-1:dA-1*塩基対はCpf1と塩基に特異的な相互作用を形成していなかった.一方,dT-2*,dA-3,dA-4はCpf1の保存されたLys607により副溝の側から認識されていた.Lys607をAlaに置換したCpf1変異体は活性をほとんど示さなかったことから,PAMの認識におけるLys607の重要性が確かめられた.これらの結果から,Cpf1はPAMを含む2本鎖DNAの塩基配列およびゆがんだ二重らせん構造の両方を認識していることが明らかにされた.
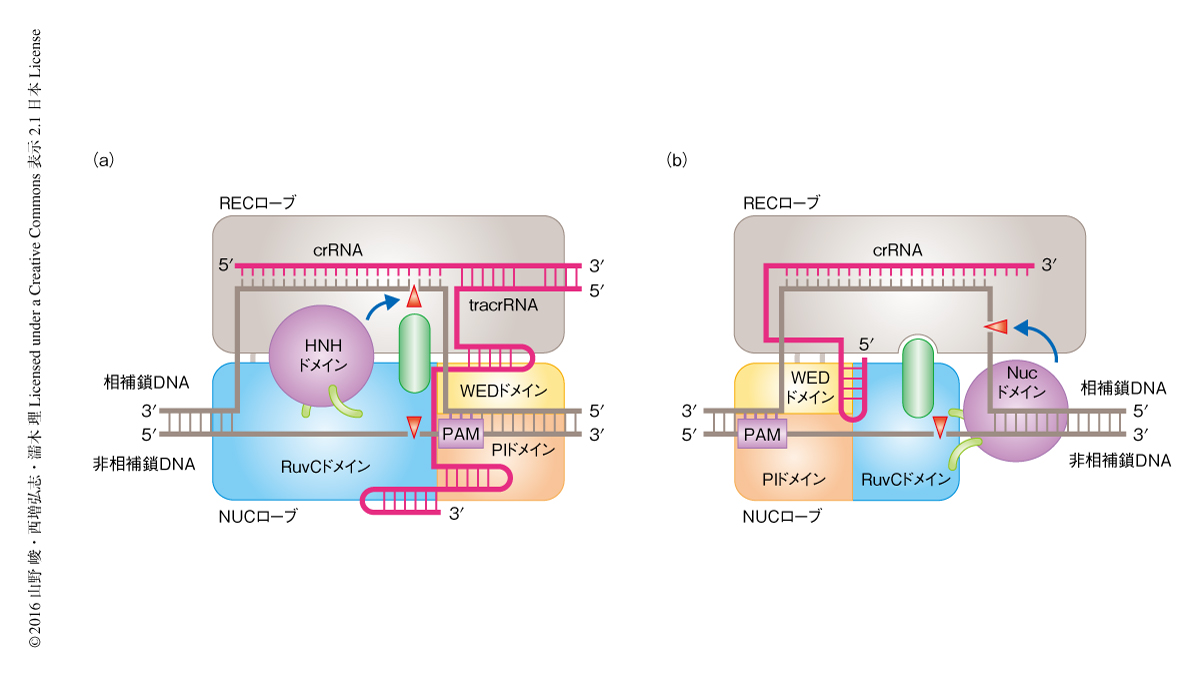
RuvCドメインはRNase Hフォールドをもち,生物種のあいだで保存された触媒残基Asp908,Glu993,Asp1263が活性部位を形成していた.Cpf1のアミノ酸配列からは第2のヌクレアーゼドメインの存在は不明だったが,結晶構造から,RuvCドメインの近傍に存在するNucドメインが同定された(図1b).Nucドメインは新規な構造をもち既知のヌクレアーゼとは類似性をもたなかったが,相補鎖DNAの切断に適した位置に存在していた.そこで,NucドメインがDNAの切断にかかわるかどうかを調べるため,生物種のあいだで保存された表面の残基をAlaに置換した複数のCpf1変異体を作製し,DNA切断活性を測定した.その結果,NucドメインにあるArg1226の変異体は非相補鎖DNAを切断する一方,相補鎖DNAは切断しないことが明らかにされた.したがって,Nucドメインは相補鎖DNAの切断にかかわることが示唆された.一方,RuvCドメインの触媒残基の変異体は相補鎖DNAと非相補鎖DNAのどちらも切断しなかった.これらの結果から,Nucドメインが相補鎖DNAを切断したのち,RuvCドメインが非相補鎖DNAを切断すると考えられた(図2).
Cas9とCpf1はRuvCドメインを除きアミノ酸配列の相同性をもたないにもかかわらず,ともに2つのローブからなる全体構造をとっていた(図1).一方,顕著な違いも存在した.まず,Cas9とCpf1のPAMの認識の機構は大きく異なっていた.Cas9はPAMを含む2本鎖DNAの主溝の側から塩基配列に特異的にPAMを認識する.たとえば,S. pyogenesに由来するCas9においては,5’-NGG-3’というPAMのGGはArg1333およびArg1335により水素結合を介し認識されていた5).一方,Cpf1は塩基配列に特異的な認識にくわえて,ゆがんだ二重らせん構造を認識していた.さらに,ヌクレアーゼドメインに関しても大きな違いが存在した.Cas9とは異なり,Cpf1はRuvCドメインおよびNucドメインをもち,これらは標的となる2本鎖DNAをPAMから離れた位置で切断し突出末端を形成するように配置されていた.以上の構造の比較から,Cas9とCpf1とのあいだの機能の収斂が明らかにされた(図2).
今回の研究により,CRISPRに関連する新規のヌクレアーゼCpf1の結晶構造が明らかにされた.構造の情報にもとづき機能の向上したCas9改変体が数多く開発されていることから,今後,Cpf1の機能の改変により新たなゲノム編集ツールが開発されるのはまちがいないだろう.
筆者らは,2016年2月,Cpf1の構造決定に成功し論文を書きはじめた.ちょうどそのころ,“Cpf1”をWebで検索したところ,中国の研究グループによりCpf1-crRNA複合体の構造がPDBに登録されていることが判明した.これは,近いうちに論文が発表されることを暗示した.急転直下,逆境に追い込まれることになったが,“あきらめたらそこで試合終了だよ”という言葉を胸に論文の執筆を急いだ.その結果,なんとか同じ週に論文を発表することができた.中国の研究グループは異なる生物種に由来するCpf1とcrRNAとの二者複合体の構造を決定していた10).また,電子顕微鏡を用いた解析により,crRNAの結合にともないCpf1は大きく構造を変化させることが明らかにされていた.このCpf1-crRNA複合体とCpf1-crRNA-DNA複合体とを比較することにより,標的となるDNAの結合による構造の変化が明らかにされた.今回,新規のNucドメインがDNAの切断にかかわることが示唆されたが,その詳細な分子機構は不明である.今後の構造機能解析により,Cpf1によるDNA切断機構についての全容の解明が期待される.
略歴:東京大学大学院理学系研究科博士課程 在学中.
研究テーマ:CRISPR-Cas系における構造解析.
抱負:倒れたら立ち上がれ.
西増 弘志(Hiroshi Nishimasu)
略歴:2007年 東京大学大学院農学生命科学研究科 修了,2013年より東京大学大学院理学系研究科 助教,科学技術振興機構 さきがけ研究者 兼任.
研究テーマ:RNAサイレンシング,CRISPR-Cas9.
抱負:日々感謝の気持ちを忘れず,いまやるべきことを見極め,新しいことに挑戦していきたい.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究室URL:http://www.nurekilab.net/
© 2016 山野 峻・西増弘志・濡木 理 Licensed under CC 表示 2.1 日本
(東京大学大学院理学系研究科 生物科学専攻生物化学講座構造生命科学研究室)
email:山野 峻,西増弘志,濡木 理
DOI: 10.7875/first.author.2016.048
Crystal structure of Cpf1 in complex with guide RNA and target DNA.
Takashi Yamano, Hiroshi Nishimasu, Bernd Zetsche, Hisato Hirano, Ian M. Slaymaker, Yinqing Li, Iana Fedorova, Takanori Nakane, Kira S. Makarova, Eugene V. Koonin, Ryuichiro Ishitani, Feng Zhang, Osamu Nureki
Cell, 165, 949-962 (2016)
要 約
II型CRISPR-Cas系に関与するRNA依存性DNAエンドヌクレアーゼCas9は,ガイドRNAと相補的な2本鎖DNAを特異的に切断する機能をもち,ゲノム編集ツールとして広く利用されている.最近,V型CRISPR-Cas系にかかわるRNA依存性DNAエンドヌクレアーゼCpf1が発見され,新たなゲノム編集ツールとして注目されている.Cas9とは異なり,Cpf1は標的となる2本鎖DNAを切断し突出末端を形成するが,その分子機構は不明であった.今回,筆者らは,Cpf1,ガイドRNA,標的となるDNAからなる複合体の結晶構造を決定し,その作動機構を解明した.さらに,Cpf1とCas9との構造の比較から,それらの作動機構における共通性および多様性が明らかにされた.
はじめに
原核生物はCRISPR-Cas系(CRISPR:clustered regularly interspaced short palindromic repeat,Cas:CRISPR-associated protein)とよばれる獲得免疫機構をもつ.CRISPR-Cas系においては,CasがcrRNA(CRISPR RNA)とエフェクター複合体を形成し,crRNAと相補的な塩基配列をもつ外来の核酸を認識し分解する.CRISPR-Cas系はエフェクター複合体の構造にもとづき2つのクラスに分類される.クラス1のCRISPR-Cas系には複数のCasからなるマルチサブユニット複合体が関与する.一方,クラス2のCRISPR-Cas系には単一のCasが関与する.クラス1はI型,III型,IV型に分類され,クラス2はII型,V型,VI型に分類される.II型のCRISPR-Cas系においてはCas9がcrRNAおよびtracrRNA(trans-activating crRNA)とエフェクター複合体を形成し,標的となる2本鎖DNAを切断する1).Cas9による2本鎖DNAの切断には標的となる配列の近傍にPAM(protospacer adjacent motif)とよばれる特定の塩基配列が存在することが必須である.したがって,PAMによる制限はあるものの,ガイドとなるRNAの配列を変更することにより任意の配列をもつ2本鎖DNAを特異的に認識することができるため,Cas9はゲノム編集技術をはじめとするさまざまな新規の技術に応用されている2).
細菌Streptococcus pyogenesに由来するCas9の結晶構造(PDB ID:4UN3,図1a)から,Cas9の作動機構が明らかにされてきた.これまでに,単体のCas9 3),Cas9-ガイドRNA複合体4),Cas9-ガイドRNA-DNA複合体5-7)(文献6) は新着論文レビュー でも掲載))の結晶構造が決定され,1)Cas9は2つのローブからなる構造をもつこと,2)ガイドRNAの結合によりCas9は大きく構造を変化させること,3)ガイドRNAと相補鎖DNAはヘテロ2本鎖を形成しCas9の2つのローブのあいだに収容されること,4)Cas9のもつ2つのヌクレアーゼドメインであるRuvCドメインおよびHNHドメインはそれぞれ非相補鎖DNAおよび相補鎖DNAを切断するのに適した位置に存在すること,5)非相補鎖DNAに存在する5’-NGG-3’というPAMはPIドメインにより認識されること,などが明らかにされた.さらに,細菌Staphylococcus aureusに由来する小型のCas9などCas9オルソログの結晶構造から,CRISPR-Cas9系の多様性が解明されてきた8)(新着論文レビュー でも掲載).
2015年,V型のCRISPR-Cas系にかかわるエフェクタータンパク質としてCpf1が発見された9).Cpf1はCas9と同様にガイドRNAと協働して2本鎖DNAを切断するが,Cas9とは以下のような異なる特徴をもつ.1)Cpf1はcrRNAのみをガイドRNAとして利用しtracrRNAを必要としない.2)Cas9はGリッチなPAMを認識するが,Cpf1はTリッチなPAMを認識する.3)Cas9は標的となる2本鎖DNAをPAMの近傍において切断し平滑末端を形成するが,Cpf1はPAMから離れた位置において2本鎖DNAを切断し突出末端を形成する.4)Cas9はヌクレアーゼドメインとしてRuvCドメインおよびHNHドメインをもつが,Cpf1はHNHドメインをもたずRuvCドメインのみをもつ.RuvCドメインを除き,Cpf1は既知のタンパク質とアミノ酸配列の相同性をもたないため,そのDNA切断機構は不明であった.
1.Cpf1-crRNA-DNA複合体の全体構造
Cpf1の作動機構を理解するため,細菌Acidaminococcus sp.に由来するCpf1,crRNA,PAMとして5’-TTTA-3’を含むDNAからなる複合体の結晶構造を2.8Å分解能で決定した(PDB ID:5B43,図1b).Cpf1はRECローブおよびNUCローブと名づけた2つのローブからなり,この2つのローブはブリッジヘリックスと名づけた特徴的な長いαヘリックスにより連結されていた.RECローブはREC1ドメインおよびREC2ドメインから構成されていた.NUCローブはRuvCドメインおよび3つのドメインから構成され,このうち2つのドメインは,Cas9のWEDドメインおよびPIドメインと構造は異なるが類似の役割をもっていたため,それぞれ,WEDドメインおよびPIドメインと名づけた.残りの1つのドメインは,のちに述べるように,DNAの切断にかかわることが明らかにされたため,Nucドメインと名づけた.Nucドメインは2つのリンカー領域L1およびL2によりRuvCドメインと連結されていた.RuvCドメインを除く5つのドメインは新規な構造をもっていた.
crRNAは5’ハンドルと名づけた5’末端領域とガイド配列から構成されていた(図1b).ガイド配列(G1~C24)は標的となる2本鎖DNAの相補鎖DNA(dC1~dG24)と20塩基対のRNA:DNAヘテロ2本鎖を形成し,RECローブとNUCローブとのあいだに収容され,塩基配列に非特異的に認識されていた.相補鎖DNA(dG-10~dT-1)は非相補鎖DNA(dC-10*~dA-1*)とPAMを含む2本鎖DNAを形成していた.塩基配列から5’ハンドルは単純なステムループ構造をもつと予想されていたが,結晶構造においてより複雑なシュードノット構造をもち,WEDドメインおよびRuvCドメインにより認識されていた(図1b).
2.Cpf1によるPAMの認識の機構
PAMを含む2本鎖DNAはATリッチな2本鎖DNAに特徴的なゆがんだ二重らせん構造をとり,WEDドメイン,REC1ドメイン,PIドメインにより認識されていた(図1b).4文字目のdT-1:dA-1*塩基対はCpf1と塩基に特異的な相互作用を形成していなかった.一方,dT-2*,dA-3,dA-4はCpf1の保存されたLys607により副溝の側から認識されていた.Lys607をAlaに置換したCpf1変異体は活性をほとんど示さなかったことから,PAMの認識におけるLys607の重要性が確かめられた.これらの結果から,Cpf1はPAMを含む2本鎖DNAの塩基配列およびゆがんだ二重らせん構造の両方を認識していることが明らかにされた.
3.Cpf1の2つのヌクレアーゼドメイン
RuvCドメインはRNase Hフォールドをもち,生物種のあいだで保存された触媒残基Asp908,Glu993,Asp1263が活性部位を形成していた.Cpf1のアミノ酸配列からは第2のヌクレアーゼドメインの存在は不明だったが,結晶構造から,RuvCドメインの近傍に存在するNucドメインが同定された(図1b).Nucドメインは新規な構造をもち既知のヌクレアーゼとは類似性をもたなかったが,相補鎖DNAの切断に適した位置に存在していた.そこで,NucドメインがDNAの切断にかかわるかどうかを調べるため,生物種のあいだで保存された表面の残基をAlaに置換した複数のCpf1変異体を作製し,DNA切断活性を測定した.その結果,NucドメインにあるArg1226の変異体は非相補鎖DNAを切断する一方,相補鎖DNAは切断しないことが明らかにされた.したがって,Nucドメインは相補鎖DNAの切断にかかわることが示唆された.一方,RuvCドメインの触媒残基の変異体は相補鎖DNAと非相補鎖DNAのどちらも切断しなかった.これらの結果から,Nucドメインが相補鎖DNAを切断したのち,RuvCドメインが非相補鎖DNAを切断すると考えられた(図2).
4.Cas9とCpf1との比較
Cas9とCpf1はRuvCドメインを除きアミノ酸配列の相同性をもたないにもかかわらず,ともに2つのローブからなる全体構造をとっていた(図1).一方,顕著な違いも存在した.まず,Cas9とCpf1のPAMの認識の機構は大きく異なっていた.Cas9はPAMを含む2本鎖DNAの主溝の側から塩基配列に特異的にPAMを認識する.たとえば,S. pyogenesに由来するCas9においては,5’-NGG-3’というPAMのGGはArg1333およびArg1335により水素結合を介し認識されていた5).一方,Cpf1は塩基配列に特異的な認識にくわえて,ゆがんだ二重らせん構造を認識していた.さらに,ヌクレアーゼドメインに関しても大きな違いが存在した.Cas9とは異なり,Cpf1はRuvCドメインおよびNucドメインをもち,これらは標的となる2本鎖DNAをPAMから離れた位置で切断し突出末端を形成するように配置されていた.以上の構造の比較から,Cas9とCpf1とのあいだの機能の収斂が明らかにされた(図2).
おわりに
今回の研究により,CRISPRに関連する新規のヌクレアーゼCpf1の結晶構造が明らかにされた.構造の情報にもとづき機能の向上したCas9改変体が数多く開発されていることから,今後,Cpf1の機能の改変により新たなゲノム編集ツールが開発されるのはまちがいないだろう.
筆者らは,2016年2月,Cpf1の構造決定に成功し論文を書きはじめた.ちょうどそのころ,“Cpf1”をWebで検索したところ,中国の研究グループによりCpf1-crRNA複合体の構造がPDBに登録されていることが判明した.これは,近いうちに論文が発表されることを暗示した.急転直下,逆境に追い込まれることになったが,“あきらめたらそこで試合終了だよ”という言葉を胸に論文の執筆を急いだ.その結果,なんとか同じ週に論文を発表することができた.中国の研究グループは異なる生物種に由来するCpf1とcrRNAとの二者複合体の構造を決定していた10).また,電子顕微鏡を用いた解析により,crRNAの結合にともないCpf1は大きく構造を変化させることが明らかにされていた.このCpf1-crRNA複合体とCpf1-crRNA-DNA複合体とを比較することにより,標的となるDNAの結合による構造の変化が明らかにされた.今回,新規のNucドメインがDNAの切断にかかわることが示唆されたが,その詳細な分子機構は不明である.今後の構造機能解析により,Cpf1によるDNA切断機構についての全容の解明が期待される.
文 献
- Jinek, M., Chylinski, K., Fonfara, I. et al.: A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 337, 816-821 (2012)[PubMed]
- Cong, L., Ran, F. A., Cox, D. et al.: Multiplex genome engineering using CRISPR/Cas systems. Science, 339, 819-823 (2013)[PubMed]
- Jinek, M., Jiang, F., Taylor, D. W. et al.: Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. Science, 343, 1247997 (2014)[PubMed]
- Jiang, F., Zhou, K., Ma, L. et al.: A Cas9-guide RNA complex preorganized for target DNA recognition. Science, 348, 1477-1481 (2015)[PubMed]
- Anders, C., Niewoehner, O., Duerst, A. et al.: Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature, 513, 569-573 (2014)[PubMed]
- Nishimasu, H., Ran, F. A., Hsu, P. D. et al.: Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell, 156, 935-949 (2014)[PubMed] [新着論文レビュー]
- Jiang, F., Taylor, D. W., Chen, J. S. et al.: Structure of a CRISPR-Cas9 R-loop complex primed for DNA cleavage. Science, 351, 867-871 (2016)[PubMed]
- Nishimasu, H., Cong, L., Yan, W. X. et al.: Crystal structure of Staphylococcus aureus Cas9. Cell, 162, 1113-1126 (2015)[PubMed] [新着論文レビュー]
- Zetsche, B., Gootenberg, J. S., Abudayyeh, O. O. et al.: Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 163, 759-771 (2015)[PubMed]
- Dong, D., Ren, K., Qiu, X. et al.: The crystal structure of Cpf1 in complex with CRISPR RNA. Nature, 532, 522-526 (2016)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:東京大学大学院理学系研究科博士課程 在学中.
研究テーマ:CRISPR-Cas系における構造解析.
抱負:倒れたら立ち上がれ.
西増 弘志(Hiroshi Nishimasu)
略歴:2007年 東京大学大学院農学生命科学研究科 修了,2013年より東京大学大学院理学系研究科 助教,科学技術振興機構 さきがけ研究者 兼任.
研究テーマ:RNAサイレンシング,CRISPR-Cas9.
抱負:日々感謝の気持ちを忘れず,いまやるべきことを見極め,新しいことに挑戦していきたい.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究室URL:http://www.nurekilab.net/
© 2016 山野 峻・西増弘志・濡木 理 Licensed under CC 表示 2.1 日本