Staphylococcus aureusに由来する小型のCas9の結晶構造
西増弘志・濡木 理
(東京大学大学院理学系研究科 生物科学専攻生物化学講座構造生命科学研究室)
email:西増弘志,濡木 理
DOI: 10.7875/first.author.2015.104
Crystal structure of Staphylococcus aureus Cas9.
Hiroshi Nishimasu, Le Cong, Winston X. Yan, F. Ann Ran, Bernd Zetsche, Yinqing Li, Arisa Kurabayashi, Ryuichiro Ishitani, Feng Zhang, Osamu Nureki
Cell, 162, 1113-1126 (2015)
原核生物のCRISPR-Cas獲得免疫機構にかかわるRNA依存性DNAヌクレアーゼとして発見されたCas9は,ゲノム編集をはじめとするさまざまな新規の技術に応用され生命科学に革命を起こしている.現在,ゲノム編集にはStreptococcus pyogenesに由来するCas9が広く利用されているが,最近の研究により,小型でゲノム編集の効率の高いStaphylococcus aureusに由来するCas9が発見された.今回,筆者らは,S. aureusに由来するCas9,ガイド鎖RNA,標的DNAからなる複合体の結晶構造を決定し,そのRNA依存性のDNA切断機構を解明した.S. aureusに由来するCas9とS. pyogenesに由来するCas9との構造の比較から,CRISPR-Cas9系における構造の保存性および多様性が明らかにされた.さらに,構造の情報をもとにS. aureusに由来するCas9を改変し,新規の転写活性化系および誘導型ヌクレアーゼの創出にも成功した.今回の結果により,CRISPR-Cas9系を利用したゲノム編集のさらなる効率化および高度化が期待される.
Cas9は2012年,原核生物のもつII型CRISPR(clustered regularly interspaced short palindromic repeat)-Cas(CRISPR-associated protein)獲得免疫機構にかかわるRNA依存性DNAエンドヌクレアーゼとして発見された1).Cas9は2つのヌクレアーゼドメイン(RuvCドメインおよびHNHドメイン)をもち,crRNA(CRISPR RNA)およびtracrRNA(trans-activating crRNA)とよばれる2種類の非コードRNAと複合体を形成し,crRNAのもつガイド配列(20~22塩基)と相補的な2本鎖DNAを認識しこれを切断する.標的2本鎖DNAのうち,crRNAと相補的なDNA鎖(相補鎖DNA)はHNHドメインにより切断され,もう一方のDNA鎖(非相補鎖DNA)はRuvCドメインにより切断される.Cas9による標的2本鎖DNAの認識には,PAM(protospacer adjacent motif)とよばれる特定の数塩基が標的となる配列の近傍に存在することが必要である.crRNAとtracrRNAを人工的に連結したsgRNA(single-guide RNA)もガイド鎖RNAとして機能する1).任意のガイド配列をもつsgRNAとCas9を細胞に共発現させることにより,ゲノムDNAに存在する標的配列を特異的に切断できることから,Cas9は簡便かつ迅速なゲノム編集ツールとして瞬く間に普及した2).さらに,sgRNAに依存的にゲノムの任意の場所にターゲッティングできるというCas9の性質を利用した新規の技術も続々と報告されている3).これまで,Streptococcus pyogenesに由来するCas9(SpCas9)の構造に関する研究により,Cas9によるRNA依存性のDNA切断機構が明らかにされてきている.SpCas9単体4),SpCas9-sgRNA複合体5),SpCas9-sgRNA-標的DNA複合体6,7)(文献6) は,新着論文レビュー でも掲載)の結晶構造から,1)SpCas9はREC(recognition)ローブとNUC(nuclease)ローブからなること,2)sgRNAのガイド配列は標的DNAとRNA:DNAヘテロ2本鎖を形成し2つのローブのあいだに収容されること,3)非相補鎖DNAのPAMはPIドメインにより認識されること,などが明らかにされている.
現在,ゲノム編集ツールとしてはSpCas9が広く利用されているが,分子量が大きくウイルスベクターへの導入の効率が低いなどの問題点が残されていた.この問題の解決策として,最近,Staphylococcus aureusに由来する小型のCas9(SaCas9)が報告された8).SaCas9(1053残基)はSpCas9(1368残基)に比べ分子量が小さく,配列同一性も低い(17%).SpCas9はPAMとして5’-NGG-3’を認識する一方,SaCas9は5’-NNGRRT-3’(Rはプリン塩基,AまたはG)を認識する.さらに,SaCas9はSpCas9とは異なるガイド鎖RNAと協働し標的DNAを切断する.しかし,SaCas9によるDNA切断機構は不明であった.
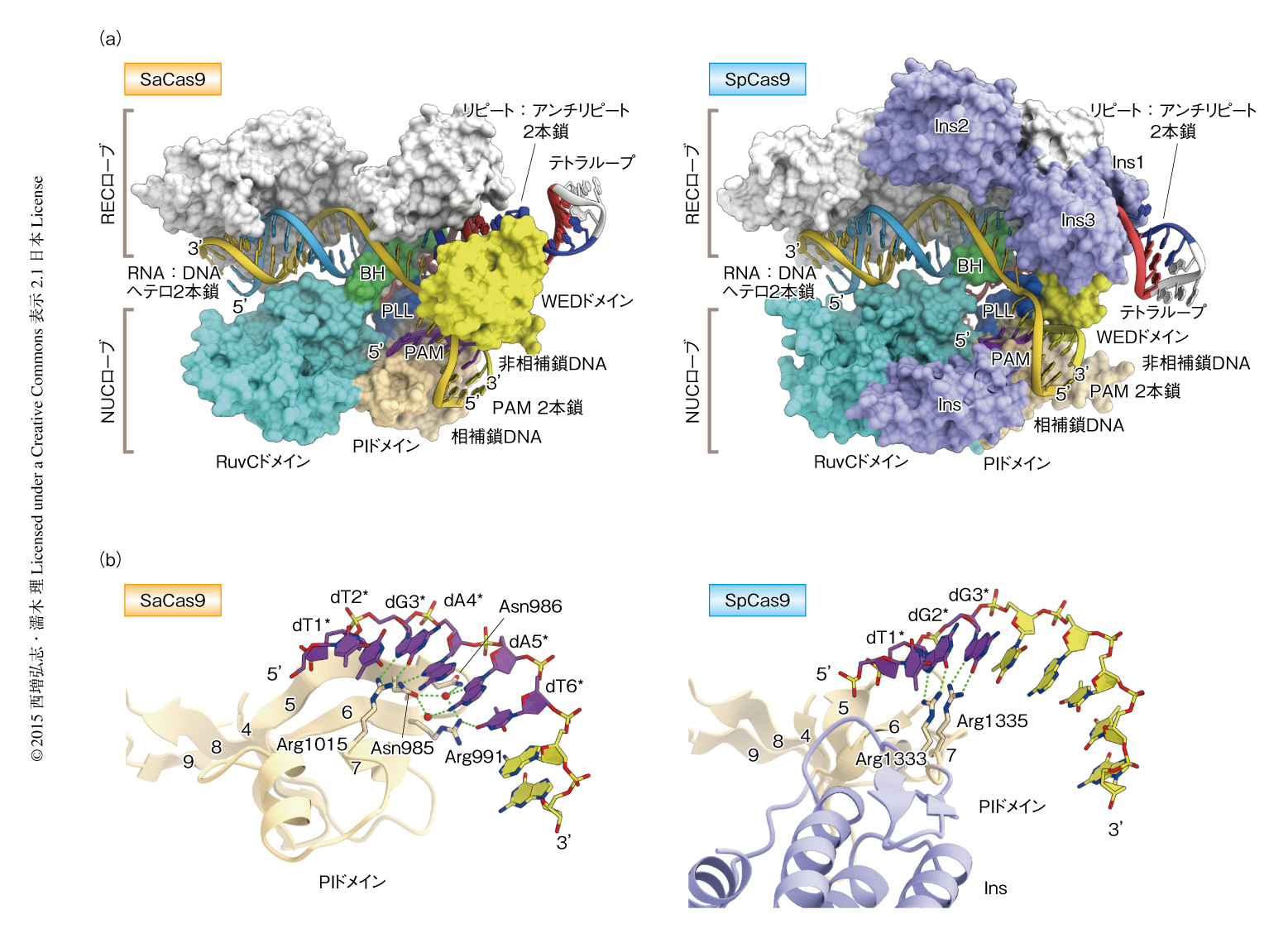
SaCas9(1~1053残基),sgRNA(73塩基),相補鎖DNA(28塩基),PAM(5’-TTGAAT-3’あるいは5’-TTGGGT-3’)を含む非相補鎖DNA(8塩基)からなる四者複合体の結晶構造を2.6Å分解能(PDB ID:5CZZ)および2.7Å分解能(PDB ID:5AXW)でそれぞれ決定した(図1).SpCas9と同様に,SaCas9はRECローブとNUCローブからなり,2つのローブはArgに富むαヘリックス(ブリッジヘリックス)とリンカーループにより連結されていた.NUCローブはRuvCドメイン,HNHドメイン,WEDドメイン,PIドメインから構成されていた.HNHドメインはRuvCドメインと2つのリンカー領域(L1とL2)によりつながっていた.RuvCドメインとPIドメインはPLループによりつながっていた.HNHドメインはRuvCドメインと相互作用し,DNAの切断部位からは離れていた.したがって,今回の結晶構造は不活性型をとらえたものであると考えられた.
sgRNAはガイド配列,リピート:アンチリピート2本鎖,ステムループ1から構成されていた(図1).ガイド配列(G1~C20)は相補鎖DNA(dG1~dC20)とRNA:DNAヘテロ2本鎖を形成し,相補鎖DNA(dC(-8)~dA(-1))は非相補鎖DNA(dT1*~dG8*)とPAM 2本鎖を形成していた.塩基配列からsgRNAの3’側の領域は2つのステムループ(ステムループ1とステムループ2)を形成することが予想されたが,良質の結晶を得るためにはステムループ2の削除が必要だった.SpCas9との構造の比較から,今回,決定した結晶構造には含まれていないステムループ2は,RuvCドメインとPIドメインのあいだの溝に結合すると考えられた.
一連の結晶構造解析により,sgRNAの結合によりSpCas9は大きな構造変化を起こすことが明らかにされていた4-7).SpCas9単体は閉じた不活性型の構造をとる一方,sgRNAとの結合により開いた構造をとり,RECローブとNUCローブのあいだにRNA:DNAヘテロ2本鎖が収容される4-7)(図2a).SpCas9-sgRNA-標的DNA複合体と同様に,SaCas9-sgRNA-標的DNA複合体は開いた構造をとり,RNA:DNAヘテロ2本鎖は2つのローブのあいだに収容されていた(図2a).したがって,sgRNAとの結合による構造変化はSaCas9とSpCas9において保存されていると考えられた.
SaCas9とSpCas9の構造の比較からドメインの境界をより正確に決定することが可能になった結果,HNHドメインとRuvCドメインは2つのリンカー領域(L1とL2)により連結されていることがわかった.SaCas9とSpCas9のヌクレアーゼドメイン(RuvCドメインおよびHNHドメイン)の構造は類似していたことから,2つのCas9においてDNA切断機構は保存されていると考えられた.生化学的な解析からPAM 2本鎖の結合により標的DNAの切断が促進されることが報告されているが9),PAM 2本鎖を含むSpCas9およびSaCas9の結晶構造において,HNHドメインは標的DNAから離れた位置に存在していた.したがって,Cas9の活性化の機構は不明であり,その解明にはさらなる研究が必要である.
SaCas9において,RNA:DNAヘテロ2本鎖は2つのローブのあいだに結合し,おもにRECドメインおよびRuvCドメインにより認識されていた(図2a).SaCas9とSpCas9においてDNA切断活性にはRNA:DNAヘテロ2本鎖のうちシード領域(PAMの近傍の約8塩基)における塩基対の形成が重要である1,8).SaCas9の結晶構造において,SpCas9と同様に5-7),sgRNAのシード領域は標的DNAと塩基対を形成するのに適したかたちをとりブリッジヘリックスにより認識されていた.一方,RNA:DNAヘテロ2本鎖のうちPAMから離れた領域はRECローブのC末端側の領域により認識されていた.SaCas9とSpCas9においてこの領域は類似の構造をもち,塩基配列に非依存的にRNA:DNAヘテロ2本鎖を認識していた.したがって,SaCas9とSpCas9において,RNA:DNAヘテロ2本鎖の認識機構は保存されていることが明らかにされた.
SpCas9において,PLループ(Glu1108とSer1109)は相補鎖DNAの+1リン酸基(dA(-1) とdT1のあいだのリン酸基)と相互作用し,標的DNAの巻きもどしを促進する役割をもつ7).結晶構造から,配列相同性は低いにもかかわらず,SaCas9はSpCas9と同様のPLループをもつことが明らかにされた(図2a).SaCas9においても,PLループ(Asp786とThr787)は相補鎖DNAの+1リン酸基と相互作用し,標的DNAの巻きもどしに貢献していた.実際,Thr787のAlaへの置換によりDNA切断活性は低下した.したがって,SaCas9とSpCas9において標的DNAの巻きもどしの機構は保存されていることが明らかにされた.
異なる生物種のCRISPR-Cas9系においてリピート:アンチリピート2本鎖の塩基配列は異なり,Cas9のオルソログはそれぞれのガイド鎖RNAを特異的に認識する8)(ガイド鎖RNAの直交性).これらの報告と一致して,SaCas9とSpCas9はそれぞれ構造の異なるRECドメインおよびWEDドメインをもち,それぞれのsgRNAのリピート:アンチリピート2本鎖を特異的に認識していることが明らかにされた(図2a).SaCas9とSpCas9の構造の比較から,両者のRECローブの構造は類似性をもつ一方,SpCas9のRECローブには4つの挿入領域Ins1~Ins4が存在していた(図2a).Ins1およびIns3はリピート:アンチリピート2本鎖を認識し,Ins4はステムループ1を認識していた.Ins2は独立したドメインとして存在し核酸とは相互作用していなかった(図2a).SpCas9のIns2がDNA切断活性に必須ではないという結果と一致して6),SaCas9はIns2に対応する領域(約130残基)をもっていなかった(図2a).この違いはSaCas9の小型化に貢献していた.SpCas9のWEDドメインは短いループ構造をもつ一方,SaCas9のWEDドメインは新規のフォールドをもっていた(図2a).SpCas9のWEDドメインは2次構造をもたないため,以前にSpCas9の結晶構造が解かれた時点では独立したドメインとして認識されておらず,PIドメインの一部と考えられていた6).WEDドメインはリピート:アンチリピート2本鎖とPAM 2本鎖のあいだに“wedge”(くさび)のように入り込んでいることから命名された.Cas9のオルソログにおいてWEDドメインに対応する領域は多様な配列をもつことから,Cas9のオルソログのWEDドメインは多様な構造をもち,それぞれのリピート:アンチリピート2本鎖を特異的に認識していると考えられた.以上をまとめると,Cas9とガイド鎖RNAとのあいだの直交性は,おもにRECドメインおよびWEDドメインとリピート:アンチリピート2本鎖とのあいだの立体構造の相補性により規定されていることが明らかにされた.
結晶構造からSaCas9によるPAM(5’-NNGRRT-3’)の認識機構が明らかにされた.PAMとして5’-TTGAAT-3’を含む複合体および5’-TTGGGT-3’を含む複合体において,PAM 2本鎖はWEDドメインとPIドメインのあいだに結合し(図2a),非相補鎖DNAに存在するPAMはPIドメインにより“解読”されていた(図2b).dT1*とdT2*の塩基はSaCas9と相互作用していなかった(図2b).一方,PAMの3文字目がGであることと一致して,dG3*はArg1015と2本の水素結合を形成していた(図2b).PAMとして5’-TTGAAT-3’を含む複合体において,dA4*のN7はAsn985と水素結合し,dA5*のN7はAsn985,Asn986,Arg991と水分子を介して水素結合していた(図2b).一方,PAMとして5’-TTGGGT-3’を含む複合体において,dG4*のN7はAsn985と水素結合し,dG5*のN7はAsn985,Asn986,Arg991と水分子を介して水素結合していた.したがって,SaCas9はプリン塩基に共通するN7を認識することにより,PAMの4文字目および5文字目のプリン塩基を“解読”していることが明らかにされた.さらに,PAMの6文字目のTへの嗜好性と一致して,dT6*のO4はArg991と水素結合していた(図2b).PAMを認識する残基の変異によりSaCas9によるDNA切断活性は低下したことから,これらの残基の重要性が確認された.
SaCas9とSpCas9の構造の比較から,PIドメインのアミノ酸配列は保存されていないにもかかわらず,9つのβストランドβ1~β9を含む共通のコアフォールドの存在が明らかにされた.SpCas9のPIドメインにはSaCas9に存在しない挿入領域(約110残基)が存在し,この違いはSaCas9の小型化に貢献していた.SaCas9とSpCas9において,PAMはβ5-β7領域により“解読”されていた.SaCas9においてはArg1015がPAM(5’-NNGRRT-3’)の3文字目のGを認識していた(図2b).同様に,SpCas9においてはArg1335(SaCas9のArg1015に対応する)がPAM(5’-NGG-3’)の3文字目のGを認識していた7)(図2b).一方,SpCas9においてPAMの2文字目のGを認識しているArg1333は,SaCas9においてはPro1013に置換されていた(図2b).さらに,SpCas9のβ5-β6領域はSaCas9に比べて短いため,SpCas9はSaCas9のAsn985,Asn986,Arg991に対応する残基をもっていなかった(図2b).これらのPIドメインの構造の違いから,SaCas9とSpCas9のPAMに対する特異性の違いが説明された.
これまで,SpCas9の構造の情報にもとづき,転写活性化系3) や誘導型ヌクレアーゼ10) などCRISPR-Cas9系を利用した新たなツールが開発されてきた.SpCas9にもとづく転写活性化系を参考にして,不活性型のSaCas9,改変型sgRNA,転写活性化因子MS2-p65-HSF1を哺乳類細胞に共発現させることにより,標的となる遺伝子を活性化することに成功した.さらに,構造の情報にもとづき,SaCas9を2分割しFKBPとFRBをそれぞれ融合させることにより,誘導型のSaCas9を開発することにも成功した.
今回,SaCas9-sgRNA-標的DNA複合体の結晶構造が明らかにされたことから,SpCas9-sgRNA-標的DNA複合体との構造の比較がはじめて可能になった.この結果,SaCas9とSpCas9のあいだのアミノ酸配列の保存性は低いにもかかわらず,基本的な作動原理は高度に保存されていることが明らかにされた.一方,SaCas9とSpCas9は構造の異なるWEDドメインおよびPIドメインをもち,ガイド鎖RNAおよびPAMを特異的に認識していることも明らかにされた.ガイド鎖RNAおよびPAMの多様性と一致して,Cas9のオルソログにおいてWEDドメインおよびPIドメインのアミノ酸配列は保存性が低い.今後,Cas9のオルソログの構造解析により,ガイド鎖RNAおよびPAMの認識機構の多様性が解明され,CRISPR-Cas9系の作動機構の理解の深まることが期待される.また,SaCas9にもとづく転写活性化系の開発にも成功した.S. aureusとS. pyogenesに由来するCRISPR-Cas9系を利用したツールは直交性をもつため,これらを組み合せることにより複数の標的遺伝子を同時に編集,活性化,抑制できる可能性が広がった.さらに,SaCas9の構造の情報はより有用なCas9の合理的な設計の基盤としても期待される.
この研究は米国Massachusetts Institute of TechnologyのFeng Zhang博士のグループとの共同研究として行った.若きスーパースターFeng Zhangとともにしたこの1年間は,これまでの人生で2番目にエキサイティングな1年だった.
略歴:2007年 東京大学大学院農学生命科学研究科 修了,2013年より東京大学大学院理学系研究科 助教,科学技術振興機構 さきがけ研究者 兼任.
研究テーマ:RNAサイレンシング,CRISPR-Cas9.
抱負:研究がしたい.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究室URL:http://www.nurekilab.net/
© 2015 西増弘志・濡木 理 Licensed under CC 表示 2.1 日本
(東京大学大学院理学系研究科 生物科学専攻生物化学講座構造生命科学研究室)
email:西増弘志,濡木 理
DOI: 10.7875/first.author.2015.104
Crystal structure of Staphylococcus aureus Cas9.
Hiroshi Nishimasu, Le Cong, Winston X. Yan, F. Ann Ran, Bernd Zetsche, Yinqing Li, Arisa Kurabayashi, Ryuichiro Ishitani, Feng Zhang, Osamu Nureki
Cell, 162, 1113-1126 (2015)
要 約
原核生物のCRISPR-Cas獲得免疫機構にかかわるRNA依存性DNAヌクレアーゼとして発見されたCas9は,ゲノム編集をはじめとするさまざまな新規の技術に応用され生命科学に革命を起こしている.現在,ゲノム編集にはStreptococcus pyogenesに由来するCas9が広く利用されているが,最近の研究により,小型でゲノム編集の効率の高いStaphylococcus aureusに由来するCas9が発見された.今回,筆者らは,S. aureusに由来するCas9,ガイド鎖RNA,標的DNAからなる複合体の結晶構造を決定し,そのRNA依存性のDNA切断機構を解明した.S. aureusに由来するCas9とS. pyogenesに由来するCas9との構造の比較から,CRISPR-Cas9系における構造の保存性および多様性が明らかにされた.さらに,構造の情報をもとにS. aureusに由来するCas9を改変し,新規の転写活性化系および誘導型ヌクレアーゼの創出にも成功した.今回の結果により,CRISPR-Cas9系を利用したゲノム編集のさらなる効率化および高度化が期待される.
はじめに
Cas9は2012年,原核生物のもつII型CRISPR(clustered regularly interspaced short palindromic repeat)-Cas(CRISPR-associated protein)獲得免疫機構にかかわるRNA依存性DNAエンドヌクレアーゼとして発見された1).Cas9は2つのヌクレアーゼドメイン(RuvCドメインおよびHNHドメイン)をもち,crRNA(CRISPR RNA)およびtracrRNA(trans-activating crRNA)とよばれる2種類の非コードRNAと複合体を形成し,crRNAのもつガイド配列(20~22塩基)と相補的な2本鎖DNAを認識しこれを切断する.標的2本鎖DNAのうち,crRNAと相補的なDNA鎖(相補鎖DNA)はHNHドメインにより切断され,もう一方のDNA鎖(非相補鎖DNA)はRuvCドメインにより切断される.Cas9による標的2本鎖DNAの認識には,PAM(protospacer adjacent motif)とよばれる特定の数塩基が標的となる配列の近傍に存在することが必要である.crRNAとtracrRNAを人工的に連結したsgRNA(single-guide RNA)もガイド鎖RNAとして機能する1).任意のガイド配列をもつsgRNAとCas9を細胞に共発現させることにより,ゲノムDNAに存在する標的配列を特異的に切断できることから,Cas9は簡便かつ迅速なゲノム編集ツールとして瞬く間に普及した2).さらに,sgRNAに依存的にゲノムの任意の場所にターゲッティングできるというCas9の性質を利用した新規の技術も続々と報告されている3).これまで,Streptococcus pyogenesに由来するCas9(SpCas9)の構造に関する研究により,Cas9によるRNA依存性のDNA切断機構が明らかにされてきている.SpCas9単体4),SpCas9-sgRNA複合体5),SpCas9-sgRNA-標的DNA複合体6,7)(文献6) は,新着論文レビュー でも掲載)の結晶構造から,1)SpCas9はREC(recognition)ローブとNUC(nuclease)ローブからなること,2)sgRNAのガイド配列は標的DNAとRNA:DNAヘテロ2本鎖を形成し2つのローブのあいだに収容されること,3)非相補鎖DNAのPAMはPIドメインにより認識されること,などが明らかにされている.
現在,ゲノム編集ツールとしてはSpCas9が広く利用されているが,分子量が大きくウイルスベクターへの導入の効率が低いなどの問題点が残されていた.この問題の解決策として,最近,Staphylococcus aureusに由来する小型のCas9(SaCas9)が報告された8).SaCas9(1053残基)はSpCas9(1368残基)に比べ分子量が小さく,配列同一性も低い(17%).SpCas9はPAMとして5’-NGG-3’を認識する一方,SaCas9は5’-NNGRRT-3’(Rはプリン塩基,AまたはG)を認識する.さらに,SaCas9はSpCas9とは異なるガイド鎖RNAと協働し標的DNAを切断する.しかし,SaCas9によるDNA切断機構は不明であった.
1.SaCas9-sgRNA-標的DNA複合体の全体構造
SaCas9(1~1053残基),sgRNA(73塩基),相補鎖DNA(28塩基),PAM(5’-TTGAAT-3’あるいは5’-TTGGGT-3’)を含む非相補鎖DNA(8塩基)からなる四者複合体の結晶構造を2.6Å分解能(PDB ID:5CZZ)および2.7Å分解能(PDB ID:5AXW)でそれぞれ決定した(図1).SpCas9と同様に,SaCas9はRECローブとNUCローブからなり,2つのローブはArgに富むαヘリックス(ブリッジヘリックス)とリンカーループにより連結されていた.NUCローブはRuvCドメイン,HNHドメイン,WEDドメイン,PIドメインから構成されていた.HNHドメインはRuvCドメインと2つのリンカー領域(L1とL2)によりつながっていた.RuvCドメインとPIドメインはPLループによりつながっていた.HNHドメインはRuvCドメインと相互作用し,DNAの切断部位からは離れていた.したがって,今回の結晶構造は不活性型をとらえたものであると考えられた.
sgRNAはガイド配列,リピート:アンチリピート2本鎖,ステムループ1から構成されていた(図1).ガイド配列(G1~C20)は相補鎖DNA(dG1~dC20)とRNA:DNAヘテロ2本鎖を形成し,相補鎖DNA(dC(-8)~dA(-1))は非相補鎖DNA(dT1*~dG8*)とPAM 2本鎖を形成していた.塩基配列からsgRNAの3’側の領域は2つのステムループ(ステムループ1とステムループ2)を形成することが予想されたが,良質の結晶を得るためにはステムループ2の削除が必要だった.SpCas9との構造の比較から,今回,決定した結晶構造には含まれていないステムループ2は,RuvCドメインとPIドメインのあいだの溝に結合すると考えられた.
2.SaCas9とSpCas9との比較
一連の結晶構造解析により,sgRNAの結合によりSpCas9は大きな構造変化を起こすことが明らかにされていた4-7).SpCas9単体は閉じた不活性型の構造をとる一方,sgRNAとの結合により開いた構造をとり,RECローブとNUCローブのあいだにRNA:DNAヘテロ2本鎖が収容される4-7)(図2a).SpCas9-sgRNA-標的DNA複合体と同様に,SaCas9-sgRNA-標的DNA複合体は開いた構造をとり,RNA:DNAヘテロ2本鎖は2つのローブのあいだに収容されていた(図2a).したがって,sgRNAとの結合による構造変化はSaCas9とSpCas9において保存されていると考えられた.
SaCas9とSpCas9の構造の比較からドメインの境界をより正確に決定することが可能になった結果,HNHドメインとRuvCドメインは2つのリンカー領域(L1とL2)により連結されていることがわかった.SaCas9とSpCas9のヌクレアーゼドメイン(RuvCドメインおよびHNHドメイン)の構造は類似していたことから,2つのCas9においてDNA切断機構は保存されていると考えられた.生化学的な解析からPAM 2本鎖の結合により標的DNAの切断が促進されることが報告されているが9),PAM 2本鎖を含むSpCas9およびSaCas9の結晶構造において,HNHドメインは標的DNAから離れた位置に存在していた.したがって,Cas9の活性化の機構は不明であり,その解明にはさらなる研究が必要である.
3.RNA:DNAヘテロ2本鎖の認識機構
SaCas9において,RNA:DNAヘテロ2本鎖は2つのローブのあいだに結合し,おもにRECドメインおよびRuvCドメインにより認識されていた(図2a).SaCas9とSpCas9においてDNA切断活性にはRNA:DNAヘテロ2本鎖のうちシード領域(PAMの近傍の約8塩基)における塩基対の形成が重要である1,8).SaCas9の結晶構造において,SpCas9と同様に5-7),sgRNAのシード領域は標的DNAと塩基対を形成するのに適したかたちをとりブリッジヘリックスにより認識されていた.一方,RNA:DNAヘテロ2本鎖のうちPAMから離れた領域はRECローブのC末端側の領域により認識されていた.SaCas9とSpCas9においてこの領域は類似の構造をもち,塩基配列に非依存的にRNA:DNAヘテロ2本鎖を認識していた.したがって,SaCas9とSpCas9において,RNA:DNAヘテロ2本鎖の認識機構は保存されていることが明らかにされた.
SpCas9において,PLループ(Glu1108とSer1109)は相補鎖DNAの+1リン酸基(dA(-1) とdT1のあいだのリン酸基)と相互作用し,標的DNAの巻きもどしを促進する役割をもつ7).結晶構造から,配列相同性は低いにもかかわらず,SaCas9はSpCas9と同様のPLループをもつことが明らかにされた(図2a).SaCas9においても,PLループ(Asp786とThr787)は相補鎖DNAの+1リン酸基と相互作用し,標的DNAの巻きもどしに貢献していた.実際,Thr787のAlaへの置換によりDNA切断活性は低下した.したがって,SaCas9とSpCas9において標的DNAの巻きもどしの機構は保存されていることが明らかにされた.
4.sgRNAの認識機構
異なる生物種のCRISPR-Cas9系においてリピート:アンチリピート2本鎖の塩基配列は異なり,Cas9のオルソログはそれぞれのガイド鎖RNAを特異的に認識する8)(ガイド鎖RNAの直交性).これらの報告と一致して,SaCas9とSpCas9はそれぞれ構造の異なるRECドメインおよびWEDドメインをもち,それぞれのsgRNAのリピート:アンチリピート2本鎖を特異的に認識していることが明らかにされた(図2a).SaCas9とSpCas9の構造の比較から,両者のRECローブの構造は類似性をもつ一方,SpCas9のRECローブには4つの挿入領域Ins1~Ins4が存在していた(図2a).Ins1およびIns3はリピート:アンチリピート2本鎖を認識し,Ins4はステムループ1を認識していた.Ins2は独立したドメインとして存在し核酸とは相互作用していなかった(図2a).SpCas9のIns2がDNA切断活性に必須ではないという結果と一致して6),SaCas9はIns2に対応する領域(約130残基)をもっていなかった(図2a).この違いはSaCas9の小型化に貢献していた.SpCas9のWEDドメインは短いループ構造をもつ一方,SaCas9のWEDドメインは新規のフォールドをもっていた(図2a).SpCas9のWEDドメインは2次構造をもたないため,以前にSpCas9の結晶構造が解かれた時点では独立したドメインとして認識されておらず,PIドメインの一部と考えられていた6).WEDドメインはリピート:アンチリピート2本鎖とPAM 2本鎖のあいだに“wedge”(くさび)のように入り込んでいることから命名された.Cas9のオルソログにおいてWEDドメインに対応する領域は多様な配列をもつことから,Cas9のオルソログのWEDドメインは多様な構造をもち,それぞれのリピート:アンチリピート2本鎖を特異的に認識していると考えられた.以上をまとめると,Cas9とガイド鎖RNAとのあいだの直交性は,おもにRECドメインおよびWEDドメインとリピート:アンチリピート2本鎖とのあいだの立体構造の相補性により規定されていることが明らかにされた.
5.PAMの認識機構
結晶構造からSaCas9によるPAM(5’-NNGRRT-3’)の認識機構が明らかにされた.PAMとして5’-TTGAAT-3’を含む複合体および5’-TTGGGT-3’を含む複合体において,PAM 2本鎖はWEDドメインとPIドメインのあいだに結合し(図2a),非相補鎖DNAに存在するPAMはPIドメインにより“解読”されていた(図2b).dT1*とdT2*の塩基はSaCas9と相互作用していなかった(図2b).一方,PAMの3文字目がGであることと一致して,dG3*はArg1015と2本の水素結合を形成していた(図2b).PAMとして5’-TTGAAT-3’を含む複合体において,dA4*のN7はAsn985と水素結合し,dA5*のN7はAsn985,Asn986,Arg991と水分子を介して水素結合していた(図2b).一方,PAMとして5’-TTGGGT-3’を含む複合体において,dG4*のN7はAsn985と水素結合し,dG5*のN7はAsn985,Asn986,Arg991と水分子を介して水素結合していた.したがって,SaCas9はプリン塩基に共通するN7を認識することにより,PAMの4文字目および5文字目のプリン塩基を“解読”していることが明らかにされた.さらに,PAMの6文字目のTへの嗜好性と一致して,dT6*のO4はArg991と水素結合していた(図2b).PAMを認識する残基の変異によりSaCas9によるDNA切断活性は低下したことから,これらの残基の重要性が確認された.
SaCas9とSpCas9の構造の比較から,PIドメインのアミノ酸配列は保存されていないにもかかわらず,9つのβストランドβ1~β9を含む共通のコアフォールドの存在が明らかにされた.SpCas9のPIドメインにはSaCas9に存在しない挿入領域(約110残基)が存在し,この違いはSaCas9の小型化に貢献していた.SaCas9とSpCas9において,PAMはβ5-β7領域により“解読”されていた.SaCas9においてはArg1015がPAM(5’-NNGRRT-3’)の3文字目のGを認識していた(図2b).同様に,SpCas9においてはArg1335(SaCas9のArg1015に対応する)がPAM(5’-NGG-3’)の3文字目のGを認識していた7)(図2b).一方,SpCas9においてPAMの2文字目のGを認識しているArg1333は,SaCas9においてはPro1013に置換されていた(図2b).さらに,SpCas9のβ5-β6領域はSaCas9に比べて短いため,SpCas9はSaCas9のAsn985,Asn986,Arg991に対応する残基をもっていなかった(図2b).これらのPIドメインの構造の違いから,SaCas9とSpCas9のPAMに対する特異性の違いが説明された.
6.立体構造にもとづくSaCas9の改変
これまで,SpCas9の構造の情報にもとづき,転写活性化系3) や誘導型ヌクレアーゼ10) などCRISPR-Cas9系を利用した新たなツールが開発されてきた.SpCas9にもとづく転写活性化系を参考にして,不活性型のSaCas9,改変型sgRNA,転写活性化因子MS2-p65-HSF1を哺乳類細胞に共発現させることにより,標的となる遺伝子を活性化することに成功した.さらに,構造の情報にもとづき,SaCas9を2分割しFKBPとFRBをそれぞれ融合させることにより,誘導型のSaCas9を開発することにも成功した.
おわりに
今回,SaCas9-sgRNA-標的DNA複合体の結晶構造が明らかにされたことから,SpCas9-sgRNA-標的DNA複合体との構造の比較がはじめて可能になった.この結果,SaCas9とSpCas9のあいだのアミノ酸配列の保存性は低いにもかかわらず,基本的な作動原理は高度に保存されていることが明らかにされた.一方,SaCas9とSpCas9は構造の異なるWEDドメインおよびPIドメインをもち,ガイド鎖RNAおよびPAMを特異的に認識していることも明らかにされた.ガイド鎖RNAおよびPAMの多様性と一致して,Cas9のオルソログにおいてWEDドメインおよびPIドメインのアミノ酸配列は保存性が低い.今後,Cas9のオルソログの構造解析により,ガイド鎖RNAおよびPAMの認識機構の多様性が解明され,CRISPR-Cas9系の作動機構の理解の深まることが期待される.また,SaCas9にもとづく転写活性化系の開発にも成功した.S. aureusとS. pyogenesに由来するCRISPR-Cas9系を利用したツールは直交性をもつため,これらを組み合せることにより複数の標的遺伝子を同時に編集,活性化,抑制できる可能性が広がった.さらに,SaCas9の構造の情報はより有用なCas9の合理的な設計の基盤としても期待される.
この研究は米国Massachusetts Institute of TechnologyのFeng Zhang博士のグループとの共同研究として行った.若きスーパースターFeng Zhangとともにしたこの1年間は,これまでの人生で2番目にエキサイティングな1年だった.
文 献
- Jinek, M., Chylinski, K., Fonfara, I. et al.: A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 337, 816-821 (2012)[PubMed]
- Cong, L., Ran, F. A., Cox, D. et al.: Multiplex genome engineering using CRISPR/Cas systems. Science, 339, 819-823 (2013)[PubMed]
- Konermann, S., Brigham, M. D., Trevino, A. E. et al.: Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature, 517, 583-588 (2015)[PubMed]
- Jinek, M., Jiang, F., Taylor, D. W. et al.: Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. Science, 343, 1247997 (2014)[PubMed]
- Jiang, F., Zhou, K., Ma, L. et al.: A Cas9-guide RNA complex preorganized for target DNA recognition. Science, 348, 1477-1481 (2015)[PubMed]
- Nishimasu, H., Ran, F. A., Hsu, P. D. et al.: Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell, 156, 935-949 (2014)[PubMed] [新着論文レビュー]
- Anders, C., Niewoehner, O., Duerst, A. et al.: Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature, 513, 569-573 (2014)[PubMed]
- Ran, F. A., Cong, L., Yan, W. X. et al.: In vivo genome editing using Staphylococcus aureus Cas9. Nature 520, 186-191 (2015)[PubMed]
- Sternberg, S. H., Redding, S., Jinek, M. et al.: DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature, 507, 62-67 (2014)[PubMed]
- Zetsche, B., Volz, S. E. & Zhang, F.: A split-Cas9 architecture for inducible genome editing and transcription modulation. Nat. Biotechnol., 33, 139-142 (2015)[PubMed]
著者プロフィール
略歴:2007年 東京大学大学院農学生命科学研究科 修了,2013年より東京大学大学院理学系研究科 助教,科学技術振興機構 さきがけ研究者 兼任.
研究テーマ:RNAサイレンシング,CRISPR-Cas9.
抱負:研究がしたい.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究室URL:http://www.nurekilab.net/
© 2015 西増弘志・濡木 理 Licensed under CC 表示 2.1 日本