Cpf1改変体によるPAMの認識機構
西増弘志・山野 峻・濡木 理
(東京大学大学院理学系研究科 生物科学専攻生物化学講座構造生命科学研究室)
email:西増弘志,山野 峻,濡木 理
DOI: 10.7875/first.author.2017.063
Structural basis for the altered PAM recognition by engineered CRISPR-Cpf1.
Hiroshi Nishimasu, Takashi Yamano, Linyi Gao, Feng Zhang, Ryuichiro Ishitani, Osamu Nureki
Molecular Cell, 67, 139-147 (2017)
RNA依存性のDNAヌクレアーゼCpf1はCRISPR RNA(crRNA)と複合体を形成し,crRNAのガイド配列と相補的な2本鎖DNAを切断する.crRNAのガイド配列は自由に変更できるためCpf1は効率的なゲノム編集ツールとして利用されている.しかし,Cpf1による2本鎖DNAの認識には,crRNAと標的となるDNA配列とのあいだの相補性にくわえ,その近傍にPAMとよばれる特定の塩基配列が必要なため,標的となるゲノム領域には制限が存在する.ごく最近,標的となるゲノム領域の拡張を目的として,異なるDNA配列をPAMとして認識する2種類のCpf1改変体が開発された.しかし,これらCpf1改変体によるPAMの認識機構は不明であった.今回,筆者らは,これら2種類のCpf1改変体の結晶構造を決定し,PAMの認識機構を明らかにすることに成功した.
Cpf1はCas12aともよばれ,原核生物のもつCRISPR-Cas獲得免疫機構にかかわるRNA依存性のDNAヌクレアーゼであり,CRISPR RNA(crRNA)と複合体を形成し,crRNAと相補的な2本鎖DNAを認識し切断する1).Cpf1が標的となる2本鎖DNAを認識するためには,標的となるDNA配列の近傍にPAM(protospacer adjacent motif)とよばれる特定の塩基配列が必要であり,Cpf1はPAMとしてTTTVという配列(VはA,C,G)を認識する.細菌Acidaminococcus sp. BV3L6や細菌Lachnospiraceae bacterium ND2006に由来するCpf1は真核生物においてもDNA切断活性を示すため,Cpf1はゲノム編集技術に応用されている1-5).
結晶構造解析によりCpf1によるDNA切断機構が明らかにされてきた6-8)(文献8) は 新着論文レビュー でも掲載).Cpf1はWEDドメイン,REC1ドメイン,REC2ドメイン,PIドメイン,RuvCドメイン,Nucドメインの6つのドメインから構成され,RECローブとNUCローブからなる構造をとる.crRNAのガイド配列と相補鎖DNAとはヘテロ2本鎖を形成し2つのローブのあいだに収容される一方,crRNAの5’側の領域はシュードノット構造をとりWEDドメイン-RuvCドメインにより認識される.PAMを含む非相補鎖DNAと相補鎖DNAは2本鎖(PAM 2本鎖DNA)を形成し,WEDドメイン-REC1ドメイン-PIドメインからなるPAM結合チャネルにより配列および構造が特異的に認識される.
Cpf1によるDNAの認識にはPAMが必要であるため,ゲノム編集において標的とすることのできるゲノム領域には制限が存在する.そこで,標的となるゲノム領域の拡張のため,構造情報と分子進化的な手法とを組み合わせることにより,Acidaminococcus sp. BV3L6に由来する2種類のCpf1改変体が開発された9).RVR改変体はSer542がArg,Lys548がVal,Asn552がArgという3つの置換をもち,TTTVにくわえTATVをPAMとして認識する一方,RR改変体はSer542がArg,Lys607がArgという2つの置換をもち,TYCV(YはTあるいはC)をPAMとして認識する.しかし,これらのCpf1改変体によるPAMの認識機構は不明であった.とくに,野生型のCpf1においてLys607はPAMの認識に必須であったため,RR改変体においてArg607がどのようにPAMの認識に関与するかは謎であった.
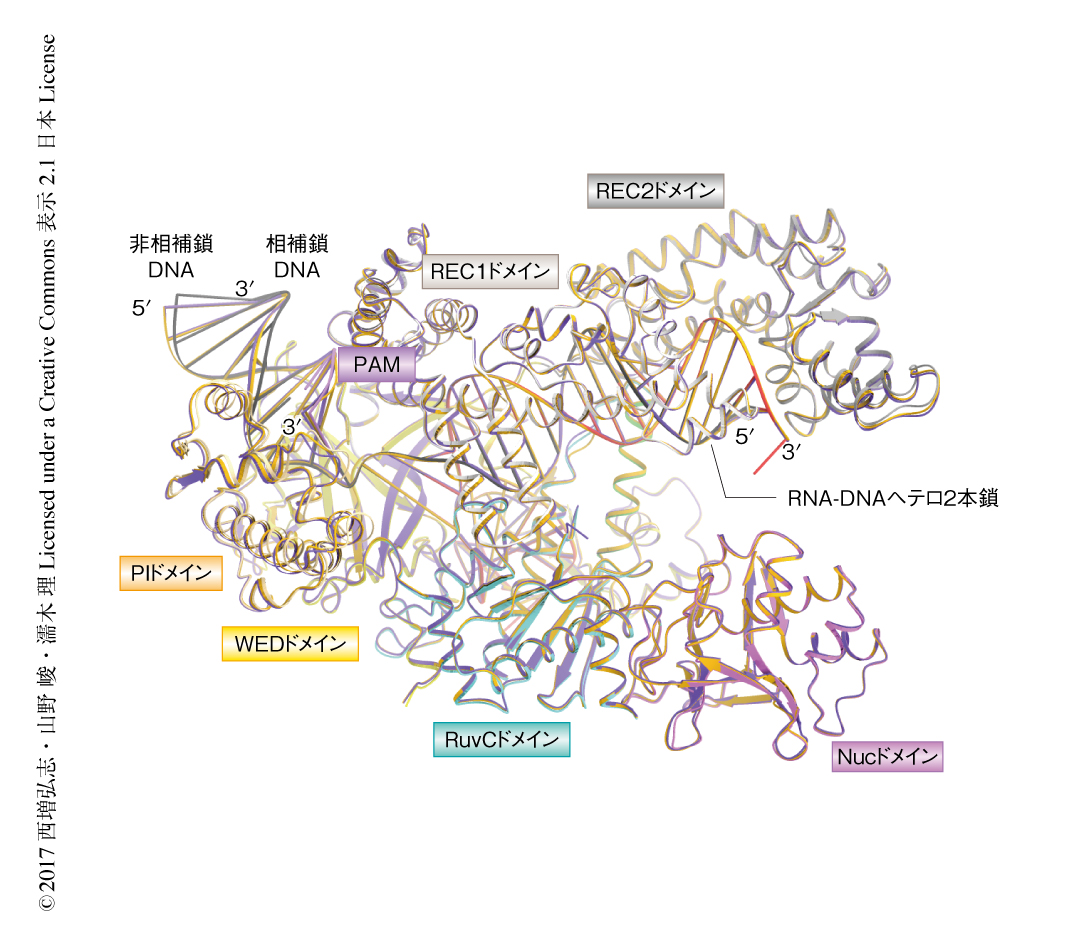
RVR改変体およびRR改変体によるPAMの認識機構を解明するため,RVR改変体-crRNA-標的DNA(PAMとしてTATAをもつ)複合体(PDB ID:5XH6),および,RR改変体-crRNA-標的DNA(PAMとしてTCCAをもつ)複合体(PDB ID:5XH7)について,結晶構造を2.0Å分解能で決定した.2種類のCpf1改変体は野生型のCpf1 8) と同様の全体構造をとり,crRNAおよび標的DNAと結合していた(図1).野生型Cpf1-crRNA-DNA複合体の構造と同様に,PAM 2本鎖DNAはPAM結合チャネルに結合していた.
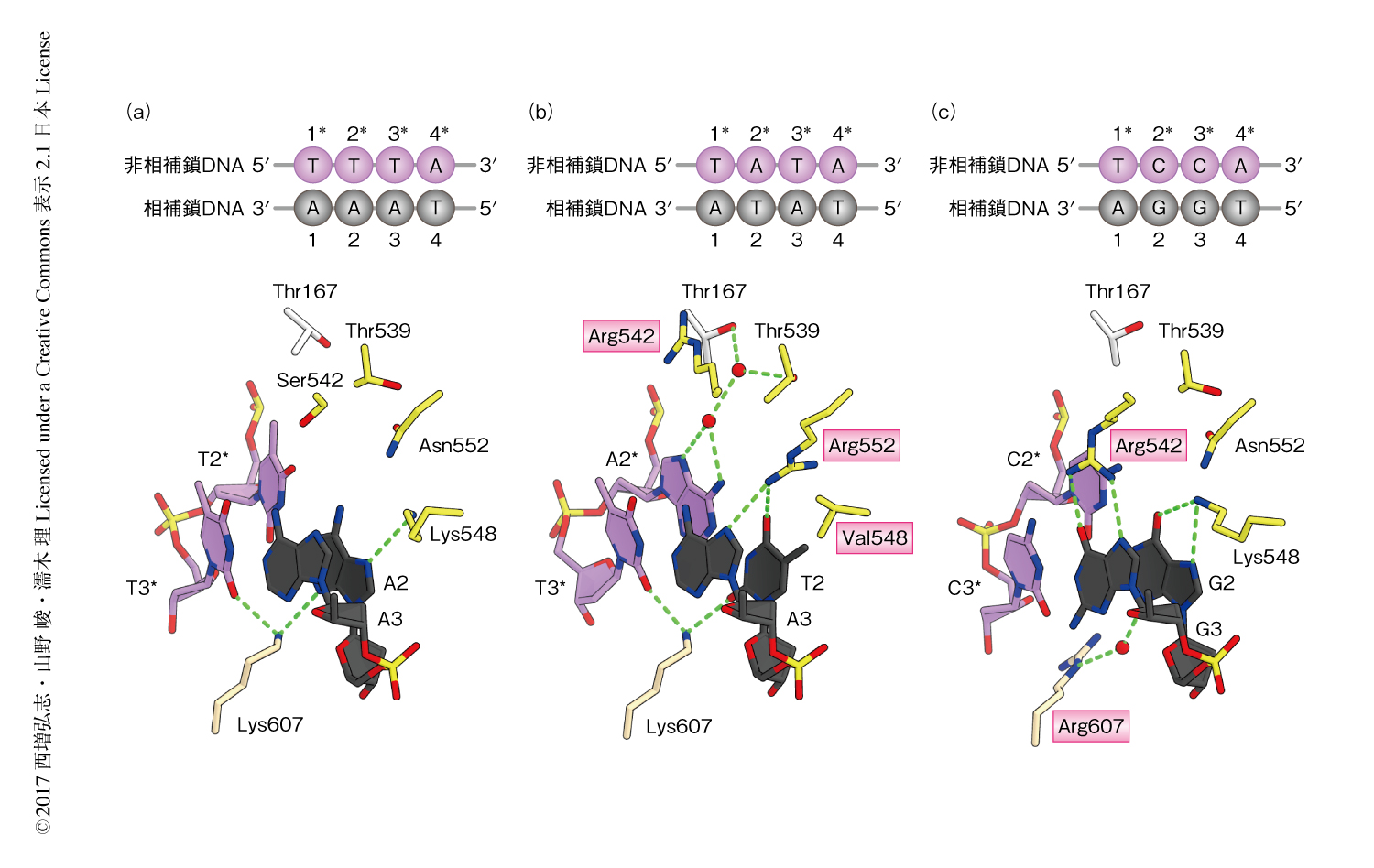
野生型のCpf1がPAMとしてTTTVを認識する一方,RVR改変体はPAMとしてTATVを認識する.すなわち,PAMの2文字目の特異性が異なる.この違いと一致して,野生型とRVR改変体はPAMの2番目の塩基対と異なる相互作用を形成していた.野生型において,非相補鎖のT2*は認識されておらず,相補鎖のA2がLys548およびLys607と水素結合を形成していた8)(図2).一方,RVR改変体においては,非相補鎖のA2*はThr167およびThr539と水分子を介して水素結合していた(図2).さらに,相補鎖のT2はArg552(野生型ではAsn),Val548(野生型ではLys),Lys607により密に認識されていた(図2).Arg542(野生型ではSer)はPAM 2本鎖DNAとは相互作用せず,REC1ドメインと相互作用しPAM結合チャネルの構造を安定化することによりPAMの認識に貢献していた.これらの結果から,RVR改変体がPAMとしてTATVを認識する機構が明らかにされた.
RR改変体の構造において,C2*は認識されておらず,G2がLys548およびArg607(野生型ではLys)と水素結合を形成していた(図2).A2はG2と同様の相互作用が可能であることから,RR改変体がPAMの2文字目としてYを好むことが説明された.C3*は認識されていない一方,G3はArg542(野生型ではSer)により水素結合を介して認識されていた(図2).したがって,野生型とは異なり,RR改変体はPAMの3文字目としてCを好むことが説明された.Arg542(野生型ではSer)はRVR改変体とRR改変体において異なる役割をはたしていた.また,野生型におけるLys607とは異なり,RR改変体においてArg607はPAM 2本鎖DNAの副溝と疎水性の相互作用を形成していた.これらの結果から,RR改変体がPAMとしてTYCVを認識する機構が明らかにされた.
広くゲノム編集に利用されているCas9に関しても異なるPAMを認識するCas9改変体が開発されており10),結晶構造からPAMの認識機構が明らかにされている11)(新着論文レビュー でも掲載).今回の結果とあわせ,構造情報と分子進化的な手法とを組み合わせたアプローチがCRISPR-Casヌクレアーゼの機能の改変において有効であること実証された.したがって,今後も,さまざまな機能をもつCas9およびCpf1の改変体が開発されると予想される.また,機能の異なるCRISPR-Casヌクレアーゼの発見もあいついでおり,これらを改変した新規のツールの開発も期待される.
略歴:2007年 東京大学大学院農学生命科学研究科 修了,2013年より東京大学大学院理学系研究科 助教.
研究テーマ:CRISPR-Cas.
関心事:“次”なにをやるか.
山野 峻(Takashi Yamano)
略歴:東京大学大学院理学系研究科博士課程 在学中.
研究テーマ:CRISPR-Cas機構の構造解析.
抱負:倒れるときは前のめり.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究室URL:http://www.nurekilab.net/
© 2017 西増弘志・山野 峻・濡木 理 Licensed under CC 表示 2.1 日本
(東京大学大学院理学系研究科 生物科学専攻生物化学講座構造生命科学研究室)
email:西増弘志,山野 峻,濡木 理
DOI: 10.7875/first.author.2017.063
Structural basis for the altered PAM recognition by engineered CRISPR-Cpf1.
Hiroshi Nishimasu, Takashi Yamano, Linyi Gao, Feng Zhang, Ryuichiro Ishitani, Osamu Nureki
Molecular Cell, 67, 139-147 (2017)
要 約
RNA依存性のDNAヌクレアーゼCpf1はCRISPR RNA(crRNA)と複合体を形成し,crRNAのガイド配列と相補的な2本鎖DNAを切断する.crRNAのガイド配列は自由に変更できるためCpf1は効率的なゲノム編集ツールとして利用されている.しかし,Cpf1による2本鎖DNAの認識には,crRNAと標的となるDNA配列とのあいだの相補性にくわえ,その近傍にPAMとよばれる特定の塩基配列が必要なため,標的となるゲノム領域には制限が存在する.ごく最近,標的となるゲノム領域の拡張を目的として,異なるDNA配列をPAMとして認識する2種類のCpf1改変体が開発された.しかし,これらCpf1改変体によるPAMの認識機構は不明であった.今回,筆者らは,これら2種類のCpf1改変体の結晶構造を決定し,PAMの認識機構を明らかにすることに成功した.
はじめに
Cpf1はCas12aともよばれ,原核生物のもつCRISPR-Cas獲得免疫機構にかかわるRNA依存性のDNAヌクレアーゼであり,CRISPR RNA(crRNA)と複合体を形成し,crRNAと相補的な2本鎖DNAを認識し切断する1).Cpf1が標的となる2本鎖DNAを認識するためには,標的となるDNA配列の近傍にPAM(protospacer adjacent motif)とよばれる特定の塩基配列が必要であり,Cpf1はPAMとしてTTTVという配列(VはA,C,G)を認識する.細菌Acidaminococcus sp. BV3L6や細菌Lachnospiraceae bacterium ND2006に由来するCpf1は真核生物においてもDNA切断活性を示すため,Cpf1はゲノム編集技術に応用されている1-5).
結晶構造解析によりCpf1によるDNA切断機構が明らかにされてきた6-8)(文献8) は 新着論文レビュー でも掲載).Cpf1はWEDドメイン,REC1ドメイン,REC2ドメイン,PIドメイン,RuvCドメイン,Nucドメインの6つのドメインから構成され,RECローブとNUCローブからなる構造をとる.crRNAのガイド配列と相補鎖DNAとはヘテロ2本鎖を形成し2つのローブのあいだに収容される一方,crRNAの5’側の領域はシュードノット構造をとりWEDドメイン-RuvCドメインにより認識される.PAMを含む非相補鎖DNAと相補鎖DNAは2本鎖(PAM 2本鎖DNA)を形成し,WEDドメイン-REC1ドメイン-PIドメインからなるPAM結合チャネルにより配列および構造が特異的に認識される.
Cpf1によるDNAの認識にはPAMが必要であるため,ゲノム編集において標的とすることのできるゲノム領域には制限が存在する.そこで,標的となるゲノム領域の拡張のため,構造情報と分子進化的な手法とを組み合わせることにより,Acidaminococcus sp. BV3L6に由来する2種類のCpf1改変体が開発された9).RVR改変体はSer542がArg,Lys548がVal,Asn552がArgという3つの置換をもち,TTTVにくわえTATVをPAMとして認識する一方,RR改変体はSer542がArg,Lys607がArgという2つの置換をもち,TYCV(YはTあるいはC)をPAMとして認識する.しかし,これらのCpf1改変体によるPAMの認識機構は不明であった.とくに,野生型のCpf1においてLys607はPAMの認識に必須であったため,RR改変体においてArg607がどのようにPAMの認識に関与するかは謎であった.
1.Cpf1改変体の結晶構造
RVR改変体およびRR改変体によるPAMの認識機構を解明するため,RVR改変体-crRNA-標的DNA(PAMとしてTATAをもつ)複合体(PDB ID:5XH6),および,RR改変体-crRNA-標的DNA(PAMとしてTCCAをもつ)複合体(PDB ID:5XH7)について,結晶構造を2.0Å分解能で決定した.2種類のCpf1改変体は野生型のCpf1 8) と同様の全体構造をとり,crRNAおよび標的DNAと結合していた(図1).野生型Cpf1-crRNA-DNA複合体の構造と同様に,PAM 2本鎖DNAはPAM結合チャネルに結合していた.
2.RVR改変体によるPAMの認識機構
野生型のCpf1がPAMとしてTTTVを認識する一方,RVR改変体はPAMとしてTATVを認識する.すなわち,PAMの2文字目の特異性が異なる.この違いと一致して,野生型とRVR改変体はPAMの2番目の塩基対と異なる相互作用を形成していた.野生型において,非相補鎖のT2*は認識されておらず,相補鎖のA2がLys548およびLys607と水素結合を形成していた8)(図2).一方,RVR改変体においては,非相補鎖のA2*はThr167およびThr539と水分子を介して水素結合していた(図2).さらに,相補鎖のT2はArg552(野生型ではAsn),Val548(野生型ではLys),Lys607により密に認識されていた(図2).Arg542(野生型ではSer)はPAM 2本鎖DNAとは相互作用せず,REC1ドメインと相互作用しPAM結合チャネルの構造を安定化することによりPAMの認識に貢献していた.これらの結果から,RVR改変体がPAMとしてTATVを認識する機構が明らかにされた.
3.RR改変体によるPAMの認識機構
RR改変体の構造において,C2*は認識されておらず,G2がLys548およびArg607(野生型ではLys)と水素結合を形成していた(図2).A2はG2と同様の相互作用が可能であることから,RR改変体がPAMの2文字目としてYを好むことが説明された.C3*は認識されていない一方,G3はArg542(野生型ではSer)により水素結合を介して認識されていた(図2).したがって,野生型とは異なり,RR改変体はPAMの3文字目としてCを好むことが説明された.Arg542(野生型ではSer)はRVR改変体とRR改変体において異なる役割をはたしていた.また,野生型におけるLys607とは異なり,RR改変体においてArg607はPAM 2本鎖DNAの副溝と疎水性の相互作用を形成していた.これらの結果から,RR改変体がPAMとしてTYCVを認識する機構が明らかにされた.
おわりに
広くゲノム編集に利用されているCas9に関しても異なるPAMを認識するCas9改変体が開発されており10),結晶構造からPAMの認識機構が明らかにされている11)(新着論文レビュー でも掲載).今回の結果とあわせ,構造情報と分子進化的な手法とを組み合わせたアプローチがCRISPR-Casヌクレアーゼの機能の改変において有効であること実証された.したがって,今後も,さまざまな機能をもつCas9およびCpf1の改変体が開発されると予想される.また,機能の異なるCRISPR-Casヌクレアーゼの発見もあいついでおり,これらを改変した新規のツールの開発も期待される.
文 献
- Zetsche, B., Gootenberg, J. S., Abudayyeh, O. O. et al.: Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 163, 759-771 (2015)[PubMed]
- Kim, D., Kim, J., Hur, J. K. et al.: Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nat. Biotechnol., 34, 863-868 (2016)[PubMed]
- Kleinstiver, B. P., Tsai, S. Q., Prew, M. S. et al.: Genome-wide specificities of CRISPR-Cas Cpf1 nucleases in human cells. Nat. Biotechnol., 34, 869-874 (2016)[PubMed]
- Kim, H. K., Song, M., Lee, J. et al.: In vivo high-throughput profiling of CRISPR-Cpf1 activity. Nat. Methods, 14, 153-159 (2017)[PubMed]
- Zetsche, B., Heidenreich, M., Mohanraju, P. et al.: Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array. Nat. Biotechnol., 35, 31-34 (2017)[PubMed]
- Dong, D., Ren, K., Qiu, X. et al.: The crystal structure of Cpf1 in complex with CRISPR RNA. Nature, 532, 522-526 (2016)[PubMed]
- Gao, P., Yang, H., Rajashankar, K. R. et al.: Type V CRISPR-Cas Cpf1 endonuclease employs a unique mechanism for crRNA-mediated target DNA recognition. Cell Res., 26, 901-913 (2016)[PubMed]
- Yamano, T., Nishimasu, H., Zetsche, B. et al.: Crystal structure of Cpf1 in complex with guide RNA and target DNA. Cell, 165, 949-962 (2016)[PubMed] [新着論文レビュー]
- Gao, L., Cox, D. B. T., Yan, W. X. et al.: Engineered Cpf1 variants with altered PAM specificities. Nat. Biotechnol., 35, 789-792 (2017)[PubMed]
- Kleinstiver, B. P., Prew, M. S., Tsai, S. Q. et al.: Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature, 523, 481-485 (2015)[PubMed]
- Hirano, S., Nishimasu, H., Ishitani, R. et al.: Structural basis for the altered PAM specificities of engineered CRISPR-Cas9. Mol. Cell, 61, 886-894 (2016)[PubMed] [新着論文レビュー]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2007年 東京大学大学院農学生命科学研究科 修了,2013年より東京大学大学院理学系研究科 助教.
研究テーマ:CRISPR-Cas.
関心事:“次”なにをやるか.
山野 峻(Takashi Yamano)
略歴:東京大学大学院理学系研究科博士課程 在学中.
研究テーマ:CRISPR-Cas機構の構造解析.
抱負:倒れるときは前のめり.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究室URL:http://www.nurekilab.net/
© 2017 西増弘志・山野 峻・濡木 理 Licensed under CC 表示 2.1 日本