Mediatorの結晶構造から明らかにされた転写開始機構
野澤 佳世
(ドイツMax Planck Institute for Biophysical Chemistry,Department of Molecular Biology)
email:野澤佳世
DOI: 10.7875/first.author.2017.046
Core Mediator structure at 3.4Å extends model of transcription initiation complex.
Kayo Nozawa, Thomas R. Schneider, Patrick Cramer
Nature, 545, 248-251 (2017)
Mediatorは転写開始領域から遠く離れたアクチベーターと転写マシーナリーとを直接につなぐことによりほとんどすべての遺伝子プロモーターに活性化シグナルを伝達する.この研究においては,X線結晶構造解析法により,生存に必要不可欠なサブユニットすべてを含む分裂酵母のコアMediatorの立体構造を原子分解能レベルで解明した.コアMediatorは,13のサブモジュールから構成され,なかでも,Med14はユビキチン結合酵素に似たユニークなコンフォメーションをとっていた.分裂酵母のコアMediatorの結晶構造を出芽酵母の転写開始前複合体のクライオ電子顕微鏡像にあてはめることにより,MediatorとRNAポリメラーゼIIおよび基本転写因子TFIIHとのあいだに新たな結合面が可視化され,転写制御機構の一端が明らかにされた.
生物の部品であるタンパク質の情報はDNAに書き込まれた遺伝子に保存され,RNAポリメラーゼIIのはたらきによりその設計図となるmRNAへと書き出される.mRNAや遺伝子発現のオンオフを切り替える小分子RNAの合成は,RNAポリメラーゼIIが基本転写因子であるTFIIA,TFIIB,TFIID,TFIIE,TFIIF,TFIIHのセットと協同しながら,開始,伸長,終結の転写サイクルを制御することにより可能となる.しかし,RNAポリメラーゼIIや基本転写因子だけでは細胞の分化や分裂,生育状態に応じた転写制御はできない.Mediatorは転写開始領域から数kbpも離れたアクチベーター(高等生物におけるエンハンサー)と転写マシーナリーとを直接につなぐことにより,真核生物のほとんどすべての遺伝子プロモーターに対し活性化シグナルを伝達する1).出芽酵母において,Mediatorは分子量が約1,400,000,25のサブユニットから構成される超分子複合体であり,ヘッドモジュール,ミドルモジュール,テイルモジュール,キナーゼモジュールからなるその構造のほとんどは,菌類,植物,動物において高度に保存されている.テイルモジュールおよびキナーゼモジュールはアクチベーターとの相互作用や転写の抑制と関係して高度な発現制御を担うにもかかわらず,その構成サブユニットは出芽酵母の生存には必須でない.一方,ヘッドモジュールあるいはミドルモジュールの変異はmRNAの合成不全をひき起こすことから,ヘッドモジュールおよびミドルモジュールのセットはコアMediatorとよばれている2).
近年の生化学的および構造生物学的なアプローチにより,コアMediatorがRNAポリメラーゼIIおよび基本転写因子と直接に作用し,転写開始前複合体(pre-initiation complex)を遺伝子プロモーターへとリクルートする機構が明らかにされてきた3).さらに,転写開始前複合体のアセンブリーが完了すると,コアMediatorはTFIIHによるRNAポリメラーゼIIのC末端ドメインのリン酸化を促進し,RNAポリメラーゼIIの遺伝子プロモーターからの遊離および伸長状態への変換をもたらす4).RNAポリメラーゼIIのC末端ドメインはTrp-Ser-Pro-Thr-Ser-Pro-Serというアミノ酸残基のくり返し構造(出芽酵母では26回,ヒトでは52回のくり返し)から構成され,リン酸化されたC末端ドメインは転写サイクルのあいだ種々の転写因子の足場として機能する.
これまで,多くの研究室がMediatorの構造解析および機能解析に取り組んできたが,その巨大さ複雑さゆえに研究は難航しており,Mediatorがどのようにして綿密に転写を制御するかは長いあいだ不明であった.
転写開始前複合体の解析が困難だった背景には試料の調製法があった.ほとんどの場合は,ゲノムに存在する目的とするタンパク質をコードする遺伝子に直接にタグを導入し数百リットルというスケールで培養した出芽酵母から内因性のタンパク質を精製していたため,試料に多量のコンタミネーションやプロテアーゼが含まれてしまうのであった.この問題を克服するため,Mediatorを構成するサブユニットそれぞれの長さが出芽酵母より短い分裂酵母に由来するMediatorを選択し,大腸菌においてコアMediatorの組換え発現系を確立した.具体的には,ヘッドモジュールおよびミドルモジュールを構成する14のサブユニット,Med19,Med14を3つの大腸菌ベクターにポリシストロニックに組み込んだ共発現系を構築し,150種類以上のコンストラクトを作製した.条件を検討した結果,Med1の存在下において複合体の収量が増大することを見い出し,4リットルの培養液からmg単位の高純度のタンパク質の獲得に成功した.この発現トライアルをつうじ,単体では非常に不安定なMed14は,N末端側でミドルモジュール,C末端側でヘッドモジュールと結合してコアMediatorの全体をつなぎとめる,要のような役割をはたすことが明らかにされた.
しかし,精製タンパク質のスクリーニングにより得られた結晶は初期の段階では8Åまでの分解能の回折像しか得られず,構造がまったく未知の6つのサブユニットも含まれていたため,構造の位相をはじめから決定する必要があった.分解能の改善をはかるため,結晶を段階的に高分子ポリマー溶液に浸し結晶の水分含有量を制御するデハイドレーション処理を試みたところ結晶のパッキングが強固になり分解能は4Åまで向上した.くわえて,シンクロトロンの高輝度ビームを多段屈折レンズにより集光させることにより,X線による損傷を最小限にして並進性の高いビームを発生させ,分解能はさらに0.6Åほど改善した.高分解能のデータセットが得られたのちにも,すべてのサブユニットを帰属させるためには結晶への重原子マーカーの導入,ネイティブな結晶に含まれるMetやCysの硫黄の位置情報を異常散乱により測定するS-SAD法のデータが必要であり,構造のモデリングの完了には1年以上かかった.
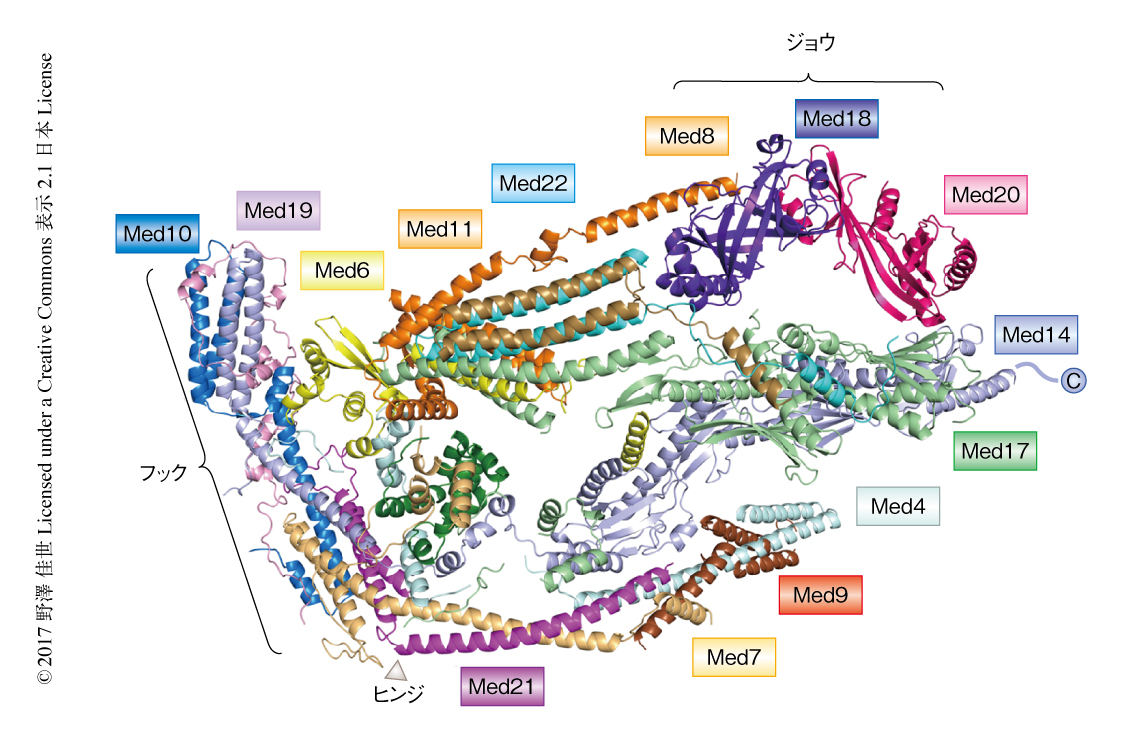
構造解析の結果,Med1は結晶のパッキングの際に欠損してしまったが,最終的には,分裂酵母の生存に必要不可欠な11のサブユニットすべてを含む分子量約400,000のコアMediatorの結晶構造を3.4Åの分解能で決定した(PDB ID:5N9J,図1).
結晶構造から分裂酵母のコアMediatorは13のサブモジュールから構成されることが可視化され,ヘッドモジュールとミドルモジュールの相互作用は,保存された4つの結合面と2つのタンパク質連結領域からなることが明らかにされた.なかでも,Med14はユビキチン結合酵素を構成するUBCフォールドが2つ連なったようなユニークなコンフォメーションにより,ヘッドモジュールのMed17に対し結合面を提示していた.また,近年の低分解能のクライオ電子顕微鏡解析により5),Med19が欠損するとヘッドモジュールとミドルモジュールとのあいだの相互作用が消失することが知られていたが,これを裏づけるように,結晶構造においてMed19はなわのような構造でMed14のN末端側とMed10のへリックスをぐるりと一周してフックサブモジュール全体を安定化していた.
これまでの出芽酵母における遺伝学的な研究結果を説明するため,出芽酵母,分裂酵母,それらに近縁の5つの生物種についてMediatorのアミノ酸配列のアライメントを比較し,2次構造予測のデータと分裂酵母における結晶構造とを照らし合わせることにより出芽酵母のコアMediatorのホモロジーモデルを作製した.得られたモデルは,これまで報告された架橋-質量分析実験にもとづく相互作用マップとよく一致する信頼性の高いもので,先行研究において出芽酵母に生育異常をもたらした10個の変異はヘッドモジュールとミドルモジュールとの結合面に配置されることが明らかにされた.また,ミドルモジュールの構造のみを破壊する変異を導入した3種類の出芽酵母株を作製し細胞におけるmRNAの合成量を測定したところ,変異株においては有意に減少していた.このことから,ヘッドモジュールとミドルモジュールとがそろってはじめてコアMediatorとして機能し転写開始前複合体の形成を促進することが裏づけられた.
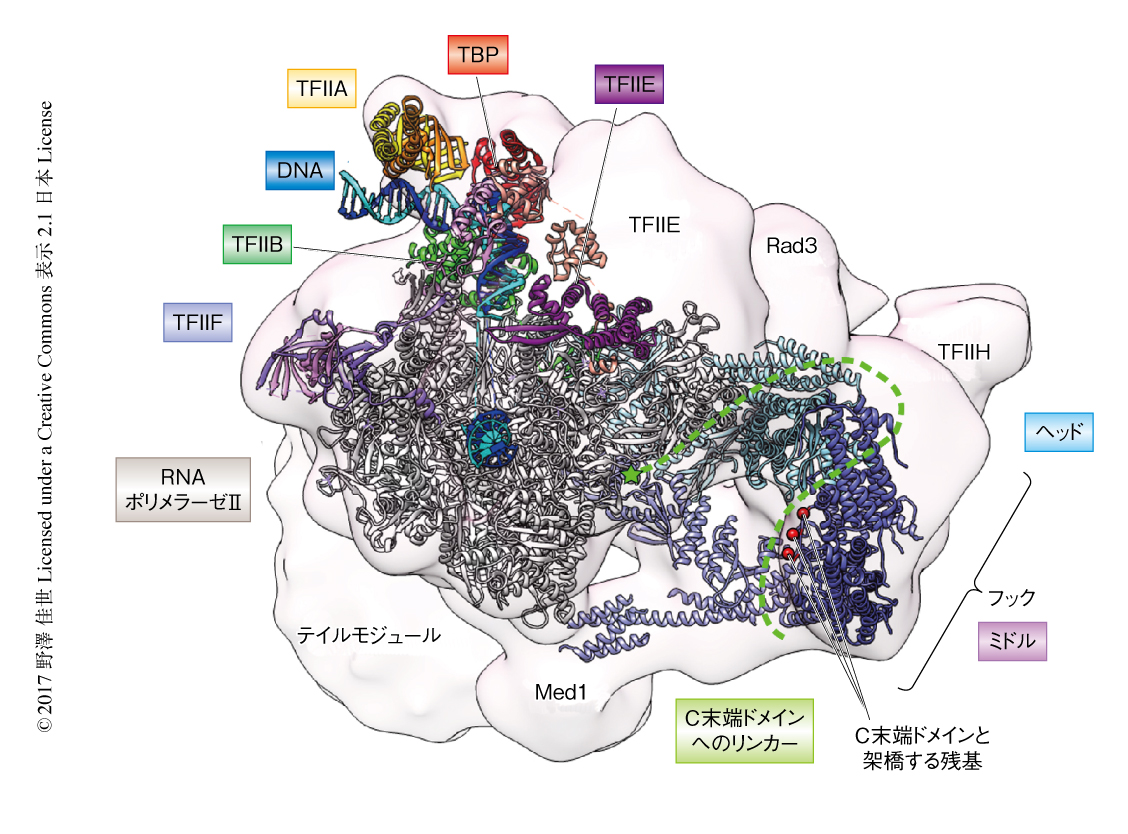
出芽酵母のコアMediatorのホモロジーモデルを転写開始前複合体の部分構造6,7) およびクライオ電子顕微鏡像8) にあてはめることにより,35のポリペプチドからなるRNAポリメラーゼII,TBP,TFIIA,TFIIB,TFIIE,TFIIF,コアMediatorの詳細な相互作用を可視化した.また,構造の決定されていないTFIIHおよびテイルモジュールについてはおおまかな位置関係が明らかにされた(図2).このフィッティングモデルにより,結晶化に用いなかったMed14のC末端側はテイルモジュールにむかって伸びており,Med14が3つのモジュールを橋渡ししていることがわかった.また,Med14のユビキチン結合酵素様のフォールドが可動性のジョウサブモジュールを安定化し,ヘッドモジュールとRNAポリメラーゼIIおよびTFIIBとの結合を可能にすることが示唆された.さらに,このモデルから,プランクサブモジュールのMed4-Med9二量体とRNAポリメラーゼIIとのあいだに新たな結合面がみつかり,コアMediatorのフックサブモジュールがTFIIHのキナーゼモジュールおよびヘリカーゼ活性をもつRad3と近接することがはじめて明らかにされた.
今回の結晶構造と比べると,クライオ電子顕微鏡像においてフックサブモジュールはTFIIHと接するため30度ほど傾いた構造をとっており8),その構造変化の起点となるヒンジは,欠損するとMediatorとRNAポリメラーゼIIとの相互作用がさまたげられることが示されている9).また,フックサブモジュールにはRNAポリメラーゼIIのC末端ドメインと架橋する残基が存在することから,RNAポリメラーゼIIはC末端ドメインを介してフックサブモジュールと相互作用する可能性が示唆された.くわえて,ヘッドモジュールとRNAポリメラーゼIIのC末端ドメインのペプチドとの複合体の結晶構造10) を今回のコアMediatorの結晶構造と重ね合わせると,C末端ドメインがミドルモジュールのMed4とぶつかることがわかった.このことから,MediatorはRNAポリメラーゼIIと結合する際には構造変化を起こし,フックサブモジュールを介してTFIIHにC末端ドメインを提示することによりそのリン酸化を促進するという機構が推測された.
近年,RNAポリメラーゼIIによるRNA合成反応が原子分解能のレベルで明らかにされつつある一方で,Mediatorに依存的な転写制御機構の多くはよくわかっていない.1990年代にMediatorの概念が提唱されて以来,Mediatorのテイルモジュールがエンハンサーをひき寄せるDNAループ機構は注目の的であるが,現在では,Mediatorの全体が開始,伸長,終結からなる転写サイクルのすべてにおいてより積極的な役割をはたすことがわかっている.最近では,Mediatorとヒトの疾患との関係性についても報告され,疾患マーカーや薬剤の分子標的としての利用にも期待が高まっている.Mediatorはじつに10種類以上の異なる組織のがんに関係しており,なかでも,多数のMediatorがクラスターを形成して異常ながん原遺伝子の活性化を誘発するスーパーエンハンサー構造が注目されている11).Mediatorの構成タンパク質のインタラクトーム解析からも,ヒストンタンパク質,ヒストンシャペロン,クロマチンリモデリングタンパク質との相互作用が明らかにされ,Mediatorのかかわるエピジェネティックな遺伝子制御は複雑さをきわめている.転写反応と数々の疾患との関連性を解明するにはまだまだ地道な基礎研究が必要であるが,この研究がその基盤を構築する助けになれば幸いである.
略歴:2012年 東京大学大学院理学系研究科博士課程 修了,同年 ドイツMunich大学 博士研究員を経て,2014年よりドイツMax Planck Institute for Biophysical Chemistry博士研究員.
研究テーマ:真核生物における転写制御機構の構造生物学.
関心事:X線結晶構造解析とクライオ電子顕微鏡解析とを用いたハイブリットなアプローチ.ドイツの化石.
© 2017 野澤 佳世 Licensed under CC 表示 2.1 日本
(ドイツMax Planck Institute for Biophysical Chemistry,Department of Molecular Biology)
email:野澤佳世
DOI: 10.7875/first.author.2017.046
Core Mediator structure at 3.4Å extends model of transcription initiation complex.
Kayo Nozawa, Thomas R. Schneider, Patrick Cramer
Nature, 545, 248-251 (2017)
要 約
Mediatorは転写開始領域から遠く離れたアクチベーターと転写マシーナリーとを直接につなぐことによりほとんどすべての遺伝子プロモーターに活性化シグナルを伝達する.この研究においては,X線結晶構造解析法により,生存に必要不可欠なサブユニットすべてを含む分裂酵母のコアMediatorの立体構造を原子分解能レベルで解明した.コアMediatorは,13のサブモジュールから構成され,なかでも,Med14はユビキチン結合酵素に似たユニークなコンフォメーションをとっていた.分裂酵母のコアMediatorの結晶構造を出芽酵母の転写開始前複合体のクライオ電子顕微鏡像にあてはめることにより,MediatorとRNAポリメラーゼIIおよび基本転写因子TFIIHとのあいだに新たな結合面が可視化され,転写制御機構の一端が明らかにされた.
はじめに
生物の部品であるタンパク質の情報はDNAに書き込まれた遺伝子に保存され,RNAポリメラーゼIIのはたらきによりその設計図となるmRNAへと書き出される.mRNAや遺伝子発現のオンオフを切り替える小分子RNAの合成は,RNAポリメラーゼIIが基本転写因子であるTFIIA,TFIIB,TFIID,TFIIE,TFIIF,TFIIHのセットと協同しながら,開始,伸長,終結の転写サイクルを制御することにより可能となる.しかし,RNAポリメラーゼIIや基本転写因子だけでは細胞の分化や分裂,生育状態に応じた転写制御はできない.Mediatorは転写開始領域から数kbpも離れたアクチベーター(高等生物におけるエンハンサー)と転写マシーナリーとを直接につなぐことにより,真核生物のほとんどすべての遺伝子プロモーターに対し活性化シグナルを伝達する1).出芽酵母において,Mediatorは分子量が約1,400,000,25のサブユニットから構成される超分子複合体であり,ヘッドモジュール,ミドルモジュール,テイルモジュール,キナーゼモジュールからなるその構造のほとんどは,菌類,植物,動物において高度に保存されている.テイルモジュールおよびキナーゼモジュールはアクチベーターとの相互作用や転写の抑制と関係して高度な発現制御を担うにもかかわらず,その構成サブユニットは出芽酵母の生存には必須でない.一方,ヘッドモジュールあるいはミドルモジュールの変異はmRNAの合成不全をひき起こすことから,ヘッドモジュールおよびミドルモジュールのセットはコアMediatorとよばれている2).
近年の生化学的および構造生物学的なアプローチにより,コアMediatorがRNAポリメラーゼIIおよび基本転写因子と直接に作用し,転写開始前複合体(pre-initiation complex)を遺伝子プロモーターへとリクルートする機構が明らかにされてきた3).さらに,転写開始前複合体のアセンブリーが完了すると,コアMediatorはTFIIHによるRNAポリメラーゼIIのC末端ドメインのリン酸化を促進し,RNAポリメラーゼIIの遺伝子プロモーターからの遊離および伸長状態への変換をもたらす4).RNAポリメラーゼIIのC末端ドメインはTrp-Ser-Pro-Thr-Ser-Pro-Serというアミノ酸残基のくり返し構造(出芽酵母では26回,ヒトでは52回のくり返し)から構成され,リン酸化されたC末端ドメインは転写サイクルのあいだ種々の転写因子の足場として機能する.
これまで,多くの研究室がMediatorの構造解析および機能解析に取り組んできたが,その巨大さ複雑さゆえに研究は難航しており,Mediatorがどのようにして綿密に転写を制御するかは長いあいだ不明であった.
1.コアMediatorの発現系の構築および結晶構造の決定
転写開始前複合体の解析が困難だった背景には試料の調製法があった.ほとんどの場合は,ゲノムに存在する目的とするタンパク質をコードする遺伝子に直接にタグを導入し数百リットルというスケールで培養した出芽酵母から内因性のタンパク質を精製していたため,試料に多量のコンタミネーションやプロテアーゼが含まれてしまうのであった.この問題を克服するため,Mediatorを構成するサブユニットそれぞれの長さが出芽酵母より短い分裂酵母に由来するMediatorを選択し,大腸菌においてコアMediatorの組換え発現系を確立した.具体的には,ヘッドモジュールおよびミドルモジュールを構成する14のサブユニット,Med19,Med14を3つの大腸菌ベクターにポリシストロニックに組み込んだ共発現系を構築し,150種類以上のコンストラクトを作製した.条件を検討した結果,Med1の存在下において複合体の収量が増大することを見い出し,4リットルの培養液からmg単位の高純度のタンパク質の獲得に成功した.この発現トライアルをつうじ,単体では非常に不安定なMed14は,N末端側でミドルモジュール,C末端側でヘッドモジュールと結合してコアMediatorの全体をつなぎとめる,要のような役割をはたすことが明らかにされた.
しかし,精製タンパク質のスクリーニングにより得られた結晶は初期の段階では8Åまでの分解能の回折像しか得られず,構造がまったく未知の6つのサブユニットも含まれていたため,構造の位相をはじめから決定する必要があった.分解能の改善をはかるため,結晶を段階的に高分子ポリマー溶液に浸し結晶の水分含有量を制御するデハイドレーション処理を試みたところ結晶のパッキングが強固になり分解能は4Åまで向上した.くわえて,シンクロトロンの高輝度ビームを多段屈折レンズにより集光させることにより,X線による損傷を最小限にして並進性の高いビームを発生させ,分解能はさらに0.6Åほど改善した.高分解能のデータセットが得られたのちにも,すべてのサブユニットを帰属させるためには結晶への重原子マーカーの導入,ネイティブな結晶に含まれるMetやCysの硫黄の位置情報を異常散乱により測定するS-SAD法のデータが必要であり,構造のモデリングの完了には1年以上かかった.
構造解析の結果,Med1は結晶のパッキングの際に欠損してしまったが,最終的には,分裂酵母の生存に必要不可欠な11のサブユニットすべてを含む分子量約400,000のコアMediatorの結晶構造を3.4Åの分解能で決定した(PDB ID:5N9J,図1).
2.コアMediatorの全体構造およびヘッドモジュールとミドルモジュールの結合様式
結晶構造から分裂酵母のコアMediatorは13のサブモジュールから構成されることが可視化され,ヘッドモジュールとミドルモジュールの相互作用は,保存された4つの結合面と2つのタンパク質連結領域からなることが明らかにされた.なかでも,Med14はユビキチン結合酵素を構成するUBCフォールドが2つ連なったようなユニークなコンフォメーションにより,ヘッドモジュールのMed17に対し結合面を提示していた.また,近年の低分解能のクライオ電子顕微鏡解析により5),Med19が欠損するとヘッドモジュールとミドルモジュールとのあいだの相互作用が消失することが知られていたが,これを裏づけるように,結晶構造においてMed19はなわのような構造でMed14のN末端側とMed10のへリックスをぐるりと一周してフックサブモジュール全体を安定化していた.
3.出芽酵母のコアMediatorのホモロジーモデルの作製および出芽酵母における変異体の解析
これまでの出芽酵母における遺伝学的な研究結果を説明するため,出芽酵母,分裂酵母,それらに近縁の5つの生物種についてMediatorのアミノ酸配列のアライメントを比較し,2次構造予測のデータと分裂酵母における結晶構造とを照らし合わせることにより出芽酵母のコアMediatorのホモロジーモデルを作製した.得られたモデルは,これまで報告された架橋-質量分析実験にもとづく相互作用マップとよく一致する信頼性の高いもので,先行研究において出芽酵母に生育異常をもたらした10個の変異はヘッドモジュールとミドルモジュールとの結合面に配置されることが明らかにされた.また,ミドルモジュールの構造のみを破壊する変異を導入した3種類の出芽酵母株を作製し細胞におけるmRNAの合成量を測定したところ,変異株においては有意に減少していた.このことから,ヘッドモジュールとミドルモジュールとがそろってはじめてコアMediatorとして機能し転写開始前複合体の形成を促進することが裏づけられた.
4.転写開始前複合体の全体像
出芽酵母のコアMediatorのホモロジーモデルを転写開始前複合体の部分構造6,7) およびクライオ電子顕微鏡像8) にあてはめることにより,35のポリペプチドからなるRNAポリメラーゼII,TBP,TFIIA,TFIIB,TFIIE,TFIIF,コアMediatorの詳細な相互作用を可視化した.また,構造の決定されていないTFIIHおよびテイルモジュールについてはおおまかな位置関係が明らかにされた(図2).このフィッティングモデルにより,結晶化に用いなかったMed14のC末端側はテイルモジュールにむかって伸びており,Med14が3つのモジュールを橋渡ししていることがわかった.また,Med14のユビキチン結合酵素様のフォールドが可動性のジョウサブモジュールを安定化し,ヘッドモジュールとRNAポリメラーゼIIおよびTFIIBとの結合を可能にすることが示唆された.さらに,このモデルから,プランクサブモジュールのMed4-Med9二量体とRNAポリメラーゼIIとのあいだに新たな結合面がみつかり,コアMediatorのフックサブモジュールがTFIIHのキナーゼモジュールおよびヘリカーゼ活性をもつRad3と近接することがはじめて明らかにされた.
今回の結晶構造と比べると,クライオ電子顕微鏡像においてフックサブモジュールはTFIIHと接するため30度ほど傾いた構造をとっており8),その構造変化の起点となるヒンジは,欠損するとMediatorとRNAポリメラーゼIIとの相互作用がさまたげられることが示されている9).また,フックサブモジュールにはRNAポリメラーゼIIのC末端ドメインと架橋する残基が存在することから,RNAポリメラーゼIIはC末端ドメインを介してフックサブモジュールと相互作用する可能性が示唆された.くわえて,ヘッドモジュールとRNAポリメラーゼIIのC末端ドメインのペプチドとの複合体の結晶構造10) を今回のコアMediatorの結晶構造と重ね合わせると,C末端ドメインがミドルモジュールのMed4とぶつかることがわかった.このことから,MediatorはRNAポリメラーゼIIと結合する際には構造変化を起こし,フックサブモジュールを介してTFIIHにC末端ドメインを提示することによりそのリン酸化を促進するという機構が推測された.
おわりに
近年,RNAポリメラーゼIIによるRNA合成反応が原子分解能のレベルで明らかにされつつある一方で,Mediatorに依存的な転写制御機構の多くはよくわかっていない.1990年代にMediatorの概念が提唱されて以来,Mediatorのテイルモジュールがエンハンサーをひき寄せるDNAループ機構は注目の的であるが,現在では,Mediatorの全体が開始,伸長,終結からなる転写サイクルのすべてにおいてより積極的な役割をはたすことがわかっている.最近では,Mediatorとヒトの疾患との関係性についても報告され,疾患マーカーや薬剤の分子標的としての利用にも期待が高まっている.Mediatorはじつに10種類以上の異なる組織のがんに関係しており,なかでも,多数のMediatorがクラスターを形成して異常ながん原遺伝子の活性化を誘発するスーパーエンハンサー構造が注目されている11).Mediatorの構成タンパク質のインタラクトーム解析からも,ヒストンタンパク質,ヒストンシャペロン,クロマチンリモデリングタンパク質との相互作用が明らかにされ,Mediatorのかかわるエピジェネティックな遺伝子制御は複雑さをきわめている.転写反応と数々の疾患との関連性を解明するにはまだまだ地道な基礎研究が必要であるが,この研究がその基盤を構築する助けになれば幸いである.
文 献
- Asturias, F. J., Jiang, Y. W., Myers, L. C. et al.: Conserved structures of mediator and RNA polymerase II holoenzyme. Science, 283, 985-987 (1999)[PubMed]
- Liu, Y., Ranish, J. A., Aebersold, R. et al.: Yeast nuclear extract contains two major forms of RNA polymerase II mediator complexes. J. Biol. Chem., 276, 7169-7175 (2001)[PubMed]
- Imasaki, T., Calero, G., Cai, G. et al.: Architecture of the Mediator head module. Nature, 475, 240-243 (2011)[PubMed] [新着論文レビュー]
- Wong, K. H., Jin, Y. & Struhl, K.: TFIIH phosphorylation of the Pol II CTD stimulates mediator dissociation from the preinitiation complex and promoter escape. Mol. Cell, 54, 601-612 (2014)[PubMed]
- Tsai, K. L., Tomomori-Sato, C., Sato, S. et al.: Subunit architecture and functional modular rearrangements of the transcriptional mediator complex. Cell, 157, 1430-1444 (2014)[PubMed]
- Plaschka, C., Lariviere, L., Wenzeck, L. et al.: Architecture of the RNA polymerase II-Mediator core initiation complex. Nature, 518, 376-380 (2015)[PubMed]
- Plaschka, C., Hantsche, M., Dienemann, C. et al.: Transcription initiation complex structures elucidate DNA opening. Nature, 533, 353-358 (2016)[PubMed]
- Robinson, P. J., Trnka, M. J., Bushnell, D. A. et al.: Structure of a complete Mediator-RNA polymerase II pre-initiation complex. Cell, 166, 1411-1422 (2016)[PubMed]
- Sato, S., Tomomori-Sato, C., Tsai, K. L. et al.: Role for the MED21-MED7 hinge in assembly of the Mediator-RNA polymerase II holoenzyme. J. Biol. Chem., 291, 26886-26898 (2016)[PubMed]
- Robinson, P. J., Bushnell, D. A., Trnka, M. J. et al.: Structure of the Mediator head module bound to the carboxy-terminal domain of RNA polymerase II. Proc. Natl. Acad. Sci. USA, 109, 17931-17935 (2012)[PubMed]
- Schiano, C., Casamassimi, A., Rienzo, M. et al.: Involvement of Mediator complex in malignancy. Biochim. Biophys. Acta, 1845, 66-83 (2014)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2012年 東京大学大学院理学系研究科博士課程 修了,同年 ドイツMunich大学 博士研究員を経て,2014年よりドイツMax Planck Institute for Biophysical Chemistry博士研究員.
研究テーマ:真核生物における転写制御機構の構造生物学.
関心事:X線結晶構造解析とクライオ電子顕微鏡解析とを用いたハイブリットなアプローチ.ドイツの化石.
© 2017 野澤 佳世 Licensed under CC 表示 2.1 日本