ヒトにおけるmRNA前駆体とlincRNAの異なる運命
野島 孝之
(英国Oxford大学Sir William Dunn School of Pathology)
email:野島孝之
DOI: 10.7875/first.author.2017.004
Distinctive patterns of transcription and RNA processing for human lincRNAs.
Margarita Schlackow, Takayuki Nojima, Tomas Gomes, Ashish Dhir, Maria Carmo-Fonseca, Nick J. Proudfoot
Molecular Cell, 65, 25-38 (2017)
ヒトのゲノムからはRNAポリメラーゼIIにより多くのlncRNAが転写される.いくつかのlncRNAは生理的な機能をもつと報告されているが,その発現の機構および安定性についてはよくわかっていない.今回,筆者らは,ヒトのHeLa細胞に発現するlincRNA遺伝子を体系的に解析し,タンパク質をコードするmRNA前駆体遺伝子との発現様式の違いについて調べた.哺乳類NET-seq法を中心とした種々の大規模なRNA-seq法による解析により,lincRNA遺伝子は効率的なプロセンシングをうけないことが示され,転写反応においてRNAの切断にともなう転写終結が生じることが示唆された.核質に放出されたlincRNAはRNAエクソソームにおいてすみやかに分解されるが,この分解は適切なプロセンシングにより回避されるというモデルが示された.
ヒトのゲノムには約2万ものタンパク質をコードする遺伝子が存在し,RNAポリメラーゼIIにより転写される.その転写産物はmRNA前駆体とよばれ,プロセシングにより安定なmRNAへと成熟し,細胞質まで輸送されてタンパク質に翻訳される.ヒトのRNAポリメラーゼIIの最大サブユニットのC末端ドメインは特徴的な7アミノ酸残基が52回くり返した配列をもち,この配列は転写においてダイナミックにリン酸化され,そのリン酸化の変化がプロセシングの精密な制御を実現すると考えられている.
ヒトのゲノムにおいてタンパク質をコードする領域は非常にかぎられる一方,潜在的にタンパク質をコードしない領域は広大である1).その領域からの転写産物のうち200塩基以上かつ潜在的なORFが100アミノ酸残基以下のRNAはlncRNA(long non-coding RNA,長鎖非コードRNA)とよばれ,そこには,遺伝子間領域に独立した転写ユニットをもつlincRNA(long intergenic/intervening non-coding RNA,長鎖遺伝子間非コードRNA)も含まれる.現在,いくつかのlincRNAは生理学的な機能をもつことが知られているが,多くのlincRNAについてはその生理学的な機能は不明であり,RNAポリメラーゼIIによる転写にともなうノイズとも考えられている.
これまで,多くの研究グループにより多くのlincRNAが同定されたが,そのほとんどはスプライシングをうけポリA鎖の付加された安定なmRNA様のlincRNAであり2,3),プロセシングをうけていないlincRNAはみのがされている可能性がある.また,lincRNAの5’末端の同定にはCAGE法やCap-seq法が利用されているが,不安定なRNAをみのがしている可能性があり,分解されたlincRNA産物の5’末端を検出している可能性も考えられる.3’末端の同定についても問題があり,mRNA前駆体遺伝子の下流の転写終結領域に存在する,いわゆる読み過ごし産物をlincRNAとして同定している場合もある4).3’末端の同定は3P-seq法などオリゴdTを用いたポリA鎖の検出によるが5),マイクロRNAの前駆体遺伝子の多くはDROSHAによるRNAの切断を転写終結の引き金としておりポリA鎖の切断に依存しないため,その3’末端は検出されない6).さらには,細胞種により発現するlincRNAやそのアイソフォームは異なることから,lincRNAの発現制御の包括的な理解は進んでいない.
この研究においては,種々の大規模なRNA-seq法による解析により,ヒトのHeLa細胞に発現するlincRNAを再精査し,mRNA前駆体遺伝子とlincRNA遺伝子との発現様式の違いに注目した.とくに,RNAポリメラーゼIIによる転写活性,および,転写と共役したプロセシングを正確に解析するため,哺乳類NET-seq法を用いた.さらに,主成分分析法によりlincRNAの発現について体系的に解析した.
RNAポリメラーゼIIの最大サブユニットのC末端ドメインのリン酸化はダイナミックかつ正確に制御され,転写開始点,遺伝子コード領域,転写終結点において重要な役割をはたすと考えられている7).しかしながら,これまでの知見は抗RNAポリメラーゼII抗体を用いたクロマチン免疫沈降法をもとに得られており,低解像度であったり転写の方向に非特異的であったりといった問題をかかえている.さらに,免疫沈降法はRNAポリメラーゼII転写複合体が新生RNAを保持するかどうかを判別できない.そこで,筆者らは,RNAポリメラーゼIIの最大サブユニットのC末端ドメインのリン酸化に特異的な転写活性およびRNAポリメラーゼII転写複合体と相互作用する新生RNAを1塩基の解像度かつ転写の方向に特異的に解析することが可能な哺乳類NET-seq法を用いて,lincRNA遺伝子の転写について詳細に解析した8,9)(文献9) は新着論文レビュー でも掲載).以前の報告においては,RNAポリメラーゼIIの最大サブユニットのC末端ドメインに対し,その全体,脱リン酸化したもの,Ser2がリン酸化したもの,Ser5がリン酸化したものを特異的に認識する抗体を哺乳類NET-seq法に用いてmRNA前駆体遺伝子から転写されたRNAを解析したが9),この研究においてはさらに,Tyr1がリン酸化したもの,Thr4がリン酸化したもの,Ser7がリン酸化したものを特異的に認識する抗体を用いて,lincRNA遺伝子についても同様に解析した.
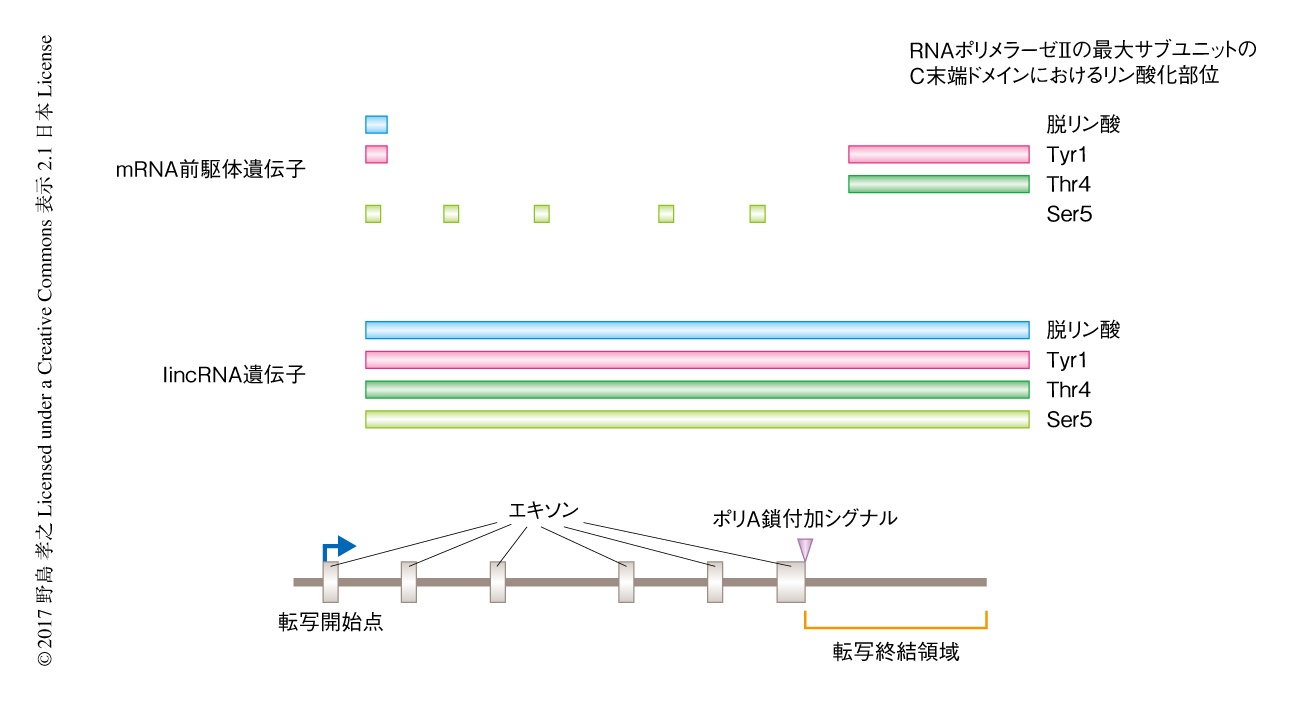
mRNA前駆体遺伝子においては転写開始点にて脱リン酸化およびリン酸化Tyr1に対するシグナルの蓄積が有意に検出されたが,lincRNA遺伝子においてその蓄積はみられなかった(図1).このことから,遺伝子発現の重要なチェックポイントであるプロモーターの近傍におけるRNAポリメラーゼIIの一時停止はlincRNA遺伝子の転写においては起こらないことが示唆された.リン酸化Thr4のシグナルはmRNA前駆体遺伝子の転写終結点において特異的であったことから,このリン酸化が転写終結の目印として重要であると考えられた.一方,lincRNA遺伝子においてこのシグナルは転写終結点に特異的ではなく,遺伝子コード領域においても有意に検出された(図1).これらの結果から,lincRNA遺伝子においてはRNAポリメラーゼIIの未成熟な転写終結が頻繁に起こることが示唆された.
哺乳類NET-seq法は細胞においてスプライシング中間体を検出できる唯一の方法であり,RNAポリメラーゼIIとスプライシング中間体との相互作用はリン酸化Ser5のシグナルに特異的である9).lincRNAのスプライシング中間体のシグナルはmRNA前駆体に比べ有意に低かったことから,lincRNAの転写と共役したスプライシングは効率的ではないことが示された.転写後産物である核質RNAのスプライシングの効率についても同様であり,lincRNAの多くはイントロンを保持していた.
RNAポリメラーゼIIはmRNA前駆体遺伝子の転写終結部位においてポリA鎖複合体に依存的に一時停止し,この一時停止は転写終結において重要である9).lincRNA遺伝子の転写終結について調べたところ,転写終結部位におけるRNAポリメラーゼIIの一時停止は,ポリA鎖複合体に依存的ではなかった.さらに,ポリA鎖をもつ転写産物を統計学的に調べた結果,lincRNAにポリA鎖の付加される効率はmRNA前駆体に比べ有意に低いことが示された.
ここまでの解析により,HeLa細胞に発現するlincRNAは適切なプロセシングをうけないことが示された.プロセシングはRNAを安定化することが知られているため,多くのlincRNAは核において分解される可能性が考えられた.そこで,クロマチンと核質を生化学的に単離し,そこに含まれる転写産物をRNA-seq法により統計学的に調べた.クロマチン画分におけるRNAのレベルはlincRNAとmRNA前駆体とで同様であったが,核質の画分においてlincRNAのレベルはmRNA前駆体に比べ有意に低かった.つまり,lincRNAはmRNA前駆体と同等のレベルで転写されるが,転写ののち核質において分解されると考えられた.そこで,核におけるRNA分解複合体であるRNAエクソソームの主要な構成タンパク質であるEXOSC3をノックダウンすることにより,核における転写産物の安定性について調べた.クロマチン画分におけるlincRNAのレベルはEXOSC3のノックダウンにより影響されなかったが,核質の画分においてはlincRNAのレベルが大幅に上昇した.また,ほかのlncRNAも同様の挙動を示したが,mRNA前駆体は影響がなかった.これらの結果から,lincRNAを含むlncRNAは転写ののち核において分解されることが示された.
RNAエクソソームがRNAを効率よく分解するためには,3’末端がポリA鎖などによる保護をうけていない必要がある.lincRNA遺伝子においては未成熟な転写終結が頻繁に起こることから,RNAポリメラーゼIIによる転写においてlincRNAが切断されており,その切断が未成熟な転写終結をひき起こす可能性を考えた.哺乳類NET-seq法においてリン酸化Ser5のシグナルによりスプライシング中間体の3’末端が検出されることから9),RNAポリメラーゼIIの転写活性中心だけでなくRNAポリメラーゼII転写複合体と相互作用する切断されたRNAも同時に検出されると考えられた.RNAポリメラーゼIIとの相互作用によるシグナルを同定するため,強力な界面活性剤であるEmpigenによりRNAポリメラーゼIIと相互作用するRNAを分離した.その結果,スプライシング複合体のシグナルや転写において切断されたRNAのシグナルは特異的に失われ,さらに,lincRNA遺伝子のシグナルも同様に失われた.それらのシグナルはスプライシング阻害剤の影響をうけず,リン酸化Ser5のシグナルだけでなくリン酸化Ser2のシグナルにより検出されたことから,Empigenの処理により影響をうけたlincRNAのシグナルはスプライシング以外のRNAの切断により生じたことが強く示唆された.
マイクロプロセッサー複合体がこのRNAの切断にかかわると考え,その構成タンパク質であるDGCR8をノックダウンしたところ,Empigenの処理により影響をうけたlincRNAのシグナルは減少せずに増加した.マイクロRNAの生合成にかかわるDICER1のノックダウンでは変化はなかったことから,DGCR8のノックダウンの影響はマイクロRNAを介していないと考えられた.最近,DGCR8はDROSHAとの複合体だけではなく,RNAエクソソームとも複合体を形成し核小体低分子RNAの分解にかかわることが明らかにされた10).この知見から,lincRNAの分解においてもDGCR8は転写反応においてRNA切断部位によび込まれ,核質においてRNAエクソソームに対するアダプターとして機能する可能性が考えられた.現在のところ,lincRNAに対するRNA切断タンパク質は不明である.
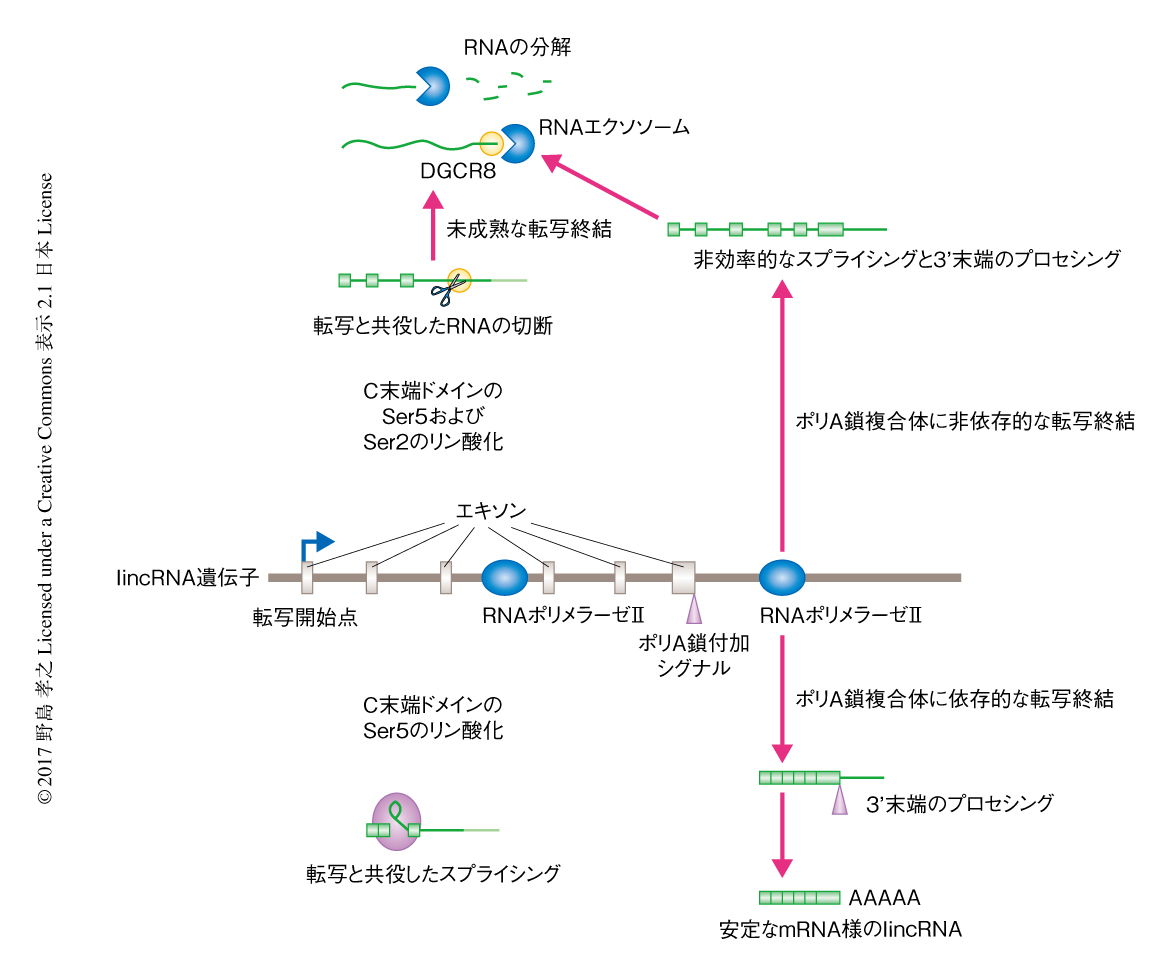
この研究においては,lincRNA遺伝子の発現制御はmRNA前駆体遺伝子とどのように異なるのか注目した.HeLa細胞に発現する遺伝子を主成分分析法により体系的に解析することにより,lincRNAはmRNA前駆体とは大きく異なる特徴をもつことが示された.とくに,lincRNAはプロセシングの効率および核質における安定性が顕著に低かった.機能の報告されているいくつかのlincRNAはmRNA前駆体と似た特徴を示し,mRNA様のlincRNAとして分類され,核質において安定であった(図2).神経や筋肉細胞などにおいて特定のlincRNAの機能が報告されていることから,細胞に特異的な転写制御タンパク質やプロセシングタンパク質がlincRNAの運命を決定することが示唆される.とくに,スプライシングはlincRNAの切断を回避する重要な反応であると考えられる.今後,異なる細胞における同様の解析により,lincRNA遺伝子の発現制御の理解がさらに深まると思われる.
略歴:2006年 東京医科歯科大学大学院生命情報教育部博士課程 修了,同年 同 特任助教,2010年 英国Oxford大学 博士研究員を経て,2014年より同 シニアリサーチフェロー.
研究テーマ:哺乳類細胞におけるRNAポリメラーゼIIによる転写およびプロセシング.
抱負:生化学な手法,システムバイオロジー,構造生物学的な手法を駆使し,哺乳類におけるRNAポリメラーゼIIによる転写の制御機構を多面的に明らかにしたい.
© 2017 野島 孝之 Licensed under CC 表示 2.1 日本
(英国Oxford大学Sir William Dunn School of Pathology)
email:野島孝之
DOI: 10.7875/first.author.2017.004
Distinctive patterns of transcription and RNA processing for human lincRNAs.
Margarita Schlackow, Takayuki Nojima, Tomas Gomes, Ashish Dhir, Maria Carmo-Fonseca, Nick J. Proudfoot
Molecular Cell, 65, 25-38 (2017)
この論文に出現する遺伝子・タンパク質のUniprot ID
RNAポリメラーゼII, RNAエクソソーム, DROSHA(Q9NRR4), クロマチン, EXOSC3(Q9NQT5), マイクロプロセッサー複合体, DGCR8(Q8WYQ5), DICER1(Q9UPY3)
要 約
ヒトのゲノムからはRNAポリメラーゼIIにより多くのlncRNAが転写される.いくつかのlncRNAは生理的な機能をもつと報告されているが,その発現の機構および安定性についてはよくわかっていない.今回,筆者らは,ヒトのHeLa細胞に発現するlincRNA遺伝子を体系的に解析し,タンパク質をコードするmRNA前駆体遺伝子との発現様式の違いについて調べた.哺乳類NET-seq法を中心とした種々の大規模なRNA-seq法による解析により,lincRNA遺伝子は効率的なプロセンシングをうけないことが示され,転写反応においてRNAの切断にともなう転写終結が生じることが示唆された.核質に放出されたlincRNAはRNAエクソソームにおいてすみやかに分解されるが,この分解は適切なプロセンシングにより回避されるというモデルが示された.
はじめに
ヒトのゲノムには約2万ものタンパク質をコードする遺伝子が存在し,RNAポリメラーゼIIにより転写される.その転写産物はmRNA前駆体とよばれ,プロセシングにより安定なmRNAへと成熟し,細胞質まで輸送されてタンパク質に翻訳される.ヒトのRNAポリメラーゼIIの最大サブユニットのC末端ドメインは特徴的な7アミノ酸残基が52回くり返した配列をもち,この配列は転写においてダイナミックにリン酸化され,そのリン酸化の変化がプロセシングの精密な制御を実現すると考えられている.
ヒトのゲノムにおいてタンパク質をコードする領域は非常にかぎられる一方,潜在的にタンパク質をコードしない領域は広大である1).その領域からの転写産物のうち200塩基以上かつ潜在的なORFが100アミノ酸残基以下のRNAはlncRNA(long non-coding RNA,長鎖非コードRNA)とよばれ,そこには,遺伝子間領域に独立した転写ユニットをもつlincRNA(long intergenic/intervening non-coding RNA,長鎖遺伝子間非コードRNA)も含まれる.現在,いくつかのlincRNAは生理学的な機能をもつことが知られているが,多くのlincRNAについてはその生理学的な機能は不明であり,RNAポリメラーゼIIによる転写にともなうノイズとも考えられている.
これまで,多くの研究グループにより多くのlincRNAが同定されたが,そのほとんどはスプライシングをうけポリA鎖の付加された安定なmRNA様のlincRNAであり2,3),プロセシングをうけていないlincRNAはみのがされている可能性がある.また,lincRNAの5’末端の同定にはCAGE法やCap-seq法が利用されているが,不安定なRNAをみのがしている可能性があり,分解されたlincRNA産物の5’末端を検出している可能性も考えられる.3’末端の同定についても問題があり,mRNA前駆体遺伝子の下流の転写終結領域に存在する,いわゆる読み過ごし産物をlincRNAとして同定している場合もある4).3’末端の同定は3P-seq法などオリゴdTを用いたポリA鎖の検出によるが5),マイクロRNAの前駆体遺伝子の多くはDROSHAによるRNAの切断を転写終結の引き金としておりポリA鎖の切断に依存しないため,その3’末端は検出されない6).さらには,細胞種により発現するlincRNAやそのアイソフォームは異なることから,lincRNAの発現制御の包括的な理解は進んでいない.
この研究においては,種々の大規模なRNA-seq法による解析により,ヒトのHeLa細胞に発現するlincRNAを再精査し,mRNA前駆体遺伝子とlincRNA遺伝子との発現様式の違いに注目した.とくに,RNAポリメラーゼIIによる転写活性,および,転写と共役したプロセシングを正確に解析するため,哺乳類NET-seq法を用いた.さらに,主成分分析法によりlincRNAの発現について体系的に解析した.
1.mRNA前駆体遺伝子とlincRNA遺伝子とで異なるRNAポリメラーゼIIの最大サブユニットのC末端ドメインのリン酸化
RNAポリメラーゼIIの最大サブユニットのC末端ドメインのリン酸化はダイナミックかつ正確に制御され,転写開始点,遺伝子コード領域,転写終結点において重要な役割をはたすと考えられている7).しかしながら,これまでの知見は抗RNAポリメラーゼII抗体を用いたクロマチン免疫沈降法をもとに得られており,低解像度であったり転写の方向に非特異的であったりといった問題をかかえている.さらに,免疫沈降法はRNAポリメラーゼII転写複合体が新生RNAを保持するかどうかを判別できない.そこで,筆者らは,RNAポリメラーゼIIの最大サブユニットのC末端ドメインのリン酸化に特異的な転写活性およびRNAポリメラーゼII転写複合体と相互作用する新生RNAを1塩基の解像度かつ転写の方向に特異的に解析することが可能な哺乳類NET-seq法を用いて,lincRNA遺伝子の転写について詳細に解析した8,9)(文献9) は新着論文レビュー でも掲載).以前の報告においては,RNAポリメラーゼIIの最大サブユニットのC末端ドメインに対し,その全体,脱リン酸化したもの,Ser2がリン酸化したもの,Ser5がリン酸化したものを特異的に認識する抗体を哺乳類NET-seq法に用いてmRNA前駆体遺伝子から転写されたRNAを解析したが9),この研究においてはさらに,Tyr1がリン酸化したもの,Thr4がリン酸化したもの,Ser7がリン酸化したものを特異的に認識する抗体を用いて,lincRNA遺伝子についても同様に解析した.
mRNA前駆体遺伝子においては転写開始点にて脱リン酸化およびリン酸化Tyr1に対するシグナルの蓄積が有意に検出されたが,lincRNA遺伝子においてその蓄積はみられなかった(図1).このことから,遺伝子発現の重要なチェックポイントであるプロモーターの近傍におけるRNAポリメラーゼIIの一時停止はlincRNA遺伝子の転写においては起こらないことが示唆された.リン酸化Thr4のシグナルはmRNA前駆体遺伝子の転写終結点において特異的であったことから,このリン酸化が転写終結の目印として重要であると考えられた.一方,lincRNA遺伝子においてこのシグナルは転写終結点に特異的ではなく,遺伝子コード領域においても有意に検出された(図1).これらの結果から,lincRNA遺伝子においてはRNAポリメラーゼIIの未成熟な転写終結が頻繁に起こることが示唆された.
2.lincRNAにおけるスプライシングの効率は低い
哺乳類NET-seq法は細胞においてスプライシング中間体を検出できる唯一の方法であり,RNAポリメラーゼIIとスプライシング中間体との相互作用はリン酸化Ser5のシグナルに特異的である9).lincRNAのスプライシング中間体のシグナルはmRNA前駆体に比べ有意に低かったことから,lincRNAの転写と共役したスプライシングは効率的ではないことが示された.転写後産物である核質RNAのスプライシングの効率についても同様であり,lincRNAの多くはイントロンを保持していた.
3.ポリA鎖複合体に非依存的なlincRNA遺伝子における転写終結
RNAポリメラーゼIIはmRNA前駆体遺伝子の転写終結部位においてポリA鎖複合体に依存的に一時停止し,この一時停止は転写終結において重要である9).lincRNA遺伝子の転写終結について調べたところ,転写終結部位におけるRNAポリメラーゼIIの一時停止は,ポリA鎖複合体に依存的ではなかった.さらに,ポリA鎖をもつ転写産物を統計学的に調べた結果,lincRNAにポリA鎖の付加される効率はmRNA前駆体に比べ有意に低いことが示された.
4.核におけるlincRNAの安定性
ここまでの解析により,HeLa細胞に発現するlincRNAは適切なプロセシングをうけないことが示された.プロセシングはRNAを安定化することが知られているため,多くのlincRNAは核において分解される可能性が考えられた.そこで,クロマチンと核質を生化学的に単離し,そこに含まれる転写産物をRNA-seq法により統計学的に調べた.クロマチン画分におけるRNAのレベルはlincRNAとmRNA前駆体とで同様であったが,核質の画分においてlincRNAのレベルはmRNA前駆体に比べ有意に低かった.つまり,lincRNAはmRNA前駆体と同等のレベルで転写されるが,転写ののち核質において分解されると考えられた.そこで,核におけるRNA分解複合体であるRNAエクソソームの主要な構成タンパク質であるEXOSC3をノックダウンすることにより,核における転写産物の安定性について調べた.クロマチン画分におけるlincRNAのレベルはEXOSC3のノックダウンにより影響されなかったが,核質の画分においてはlincRNAのレベルが大幅に上昇した.また,ほかのlncRNAも同様の挙動を示したが,mRNA前駆体は影響がなかった.これらの結果から,lincRNAを含むlncRNAは転写ののち核において分解されることが示された.
5.RNAポリメラーゼIIによる転写におけるlincRNAの切断
RNAエクソソームがRNAを効率よく分解するためには,3’末端がポリA鎖などによる保護をうけていない必要がある.lincRNA遺伝子においては未成熟な転写終結が頻繁に起こることから,RNAポリメラーゼIIによる転写においてlincRNAが切断されており,その切断が未成熟な転写終結をひき起こす可能性を考えた.哺乳類NET-seq法においてリン酸化Ser5のシグナルによりスプライシング中間体の3’末端が検出されることから9),RNAポリメラーゼIIの転写活性中心だけでなくRNAポリメラーゼII転写複合体と相互作用する切断されたRNAも同時に検出されると考えられた.RNAポリメラーゼIIとの相互作用によるシグナルを同定するため,強力な界面活性剤であるEmpigenによりRNAポリメラーゼIIと相互作用するRNAを分離した.その結果,スプライシング複合体のシグナルや転写において切断されたRNAのシグナルは特異的に失われ,さらに,lincRNA遺伝子のシグナルも同様に失われた.それらのシグナルはスプライシング阻害剤の影響をうけず,リン酸化Ser5のシグナルだけでなくリン酸化Ser2のシグナルにより検出されたことから,Empigenの処理により影響をうけたlincRNAのシグナルはスプライシング以外のRNAの切断により生じたことが強く示唆された.
マイクロプロセッサー複合体がこのRNAの切断にかかわると考え,その構成タンパク質であるDGCR8をノックダウンしたところ,Empigenの処理により影響をうけたlincRNAのシグナルは減少せずに増加した.マイクロRNAの生合成にかかわるDICER1のノックダウンでは変化はなかったことから,DGCR8のノックダウンの影響はマイクロRNAを介していないと考えられた.最近,DGCR8はDROSHAとの複合体だけではなく,RNAエクソソームとも複合体を形成し核小体低分子RNAの分解にかかわることが明らかにされた10).この知見から,lincRNAの分解においてもDGCR8は転写反応においてRNA切断部位によび込まれ,核質においてRNAエクソソームに対するアダプターとして機能する可能性が考えられた.現在のところ,lincRNAに対するRNA切断タンパク質は不明である.
おわりに
この研究においては,lincRNA遺伝子の発現制御はmRNA前駆体遺伝子とどのように異なるのか注目した.HeLa細胞に発現する遺伝子を主成分分析法により体系的に解析することにより,lincRNAはmRNA前駆体とは大きく異なる特徴をもつことが示された.とくに,lincRNAはプロセシングの効率および核質における安定性が顕著に低かった.機能の報告されているいくつかのlincRNAはmRNA前駆体と似た特徴を示し,mRNA様のlincRNAとして分類され,核質において安定であった(図2).神経や筋肉細胞などにおいて特定のlincRNAの機能が報告されていることから,細胞に特異的な転写制御タンパク質やプロセシングタンパク質がlincRNAの運命を決定することが示唆される.とくに,スプライシングはlincRNAの切断を回避する重要な反応であると考えられる.今後,異なる細胞における同様の解析により,lincRNA遺伝子の発現制御の理解がさらに深まると思われる.
文 献
- St Laurent, G., Wahlestedt, C. & Kapranov, P.: The landscape of long noncoding RNA classification. Trends Genet., 31, 239-251 (2015)[PubMed]
- Cabili, M. N., Trapnell, C., Goff, L. et al.: Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev., 25, 1915-1927 (2011)[PubMed]
- Grabherr, M. G., Haas, B. J., Yassour, M. et al.: Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nat. Biotechnol., 29, 644-652 (2011)[PubMed]
- Werner, M. S. & Ruthenburg, A. J.: Nuclear fractionation reveals thousands of chromatin-tethered noncoding RNAs adjacent to active genes. Cell Rep., 12, 1089-1098 (2015)[PubMed]
- Ulitsky, I. & Bartel, D. P.: lincRNAs: genomics, evolution, and mechanisms. Cell, 154, 26-46 (2013)[PubMed]
- Dhir, A., Dhir, S., Proudfoot, N. J. et al.: Microprocessor mediates transcriptional termination of long noncoding RNA transcripts hosting microRNAs. Nat.Struct. Mol. Biol., 22, 319-327 (2015)[PubMed]
- Heidemann, M., Hintermair, C., Voss, K. et al.: Dynamic phosphorylation patterns of RNA polymerase II CTD during transcription. Biochim. Biophys. Acta, 1829, 55-62 (2013)[PubMed]
- Nojima, T., Gomes, T., Carmo-Fonseca, M. et al.: Mammalian NET-seq analysis defines nascent RNA profiles and associated RNA processing genome-wide. Nat. Protoc., 11, 413-428 (2016)[PubMed]
- Nojima, T., Gomes, T., Grosso, A. R. et al.: Mammalian NET-Seq reveals genome-wide nascent transcription coupled to RNA processing. Cell, 161, 526-540 (2015)[PubMed] [新着論文レビュー]
- Macias, S., Cordiner, R. A., Gautier, P. et al.: DGCR8 acts as an adaptor for the exosome complex to degrade double-stranded structured RNAs. Mol. Cell, 60, 873-885 (2015)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2006年 東京医科歯科大学大学院生命情報教育部博士課程 修了,同年 同 特任助教,2010年 英国Oxford大学 博士研究員を経て,2014年より同 シニアリサーチフェロー.
研究テーマ:哺乳類細胞におけるRNAポリメラーゼIIによる転写およびプロセシング.
抱負:生化学な手法,システムバイオロジー,構造生物学的な手法を駆使し,哺乳類におけるRNAポリメラーゼIIによる転写の制御機構を多面的に明らかにしたい.
© 2017 野島 孝之 Licensed under CC 表示 2.1 日本