遺伝子発現解析の基準となるデータを快適に検索できるウェブツールRefEx
小野浩雅・坊農秀雅
(ライフサイエンス統合データベースセンター)
email:小野浩雅
DOI: 10.7875/first.author.2017.093
Hiromasa Ono, Osamu Ogasawara, Kosaku Okubo, Hidemasa Bono
Scientific Data, 4, 170105 (2017)
目 次
要 約
RefEx(URL:http://refex.dbcls.jp)は,遺伝子発現解析の基準となるおのおのの遺伝子の発現量を簡単に検索し閲覧できるウェブツールである.複数の計測手法により得られた,哺乳類の正常な組織や細胞などにおける遺伝子発現データを収集し並列に表現することにより,おのおのの組織における遺伝子の発現量を,計測手法のあいだの差異とともに直感的に比較することができる.とくに,遺伝子発現解析の過程においてしばしば比較の対象とされる,組織に特異的に発現する遺伝子を瞬時に検索できるほか,リスト機能を用いて遺伝子の発現量を比較しつつ機能に関する注釈情報を見比べることが容易であり,遺伝子発現解析により見い出された遺伝子のあいだの関係性を知るためのツールとして有用である.RefExの提供するすべてのデータは生命科学データの共有および再利用の活用例のひとつであり,データ駆動型研究のためのツールとして,だれでも無償で自由に使うことができる.
はじめに
DNAマイクロアレイの開発によりゲノム規模での遺伝子の発現量の測定が可能となって以来,遺伝子発現データはさまざまな研究グループにより異なる測定手法を用いて産生され,公共データベースに蓄積しつづけている.これらのデータは,仮説の構築,研究計画の立案,実験データの解釈など,さまざまな状況において幅広い分野の研究者に利用される汎用的なデータであるが,その膨大さや多様さのため,それらを自らの研究に利用することは困難な状況にある.そこで,筆者らは,大量の遺伝子発現データのなかから,まずどれをみればよいのか指針になりうる代表的な遺伝子発現データセットを選び出して整理し,それらをならべて閲覧できるウェブツールを構築したいと考え,RefEx(URL:http://refex.dbcls.jp)を開発した.
1.RefExウェブサイトの概要
RefExは,トップページ(検索フォーム),検索結果一覧ページ,遺伝子発現の詳細情報ページ,の3つを柱として構成されている.
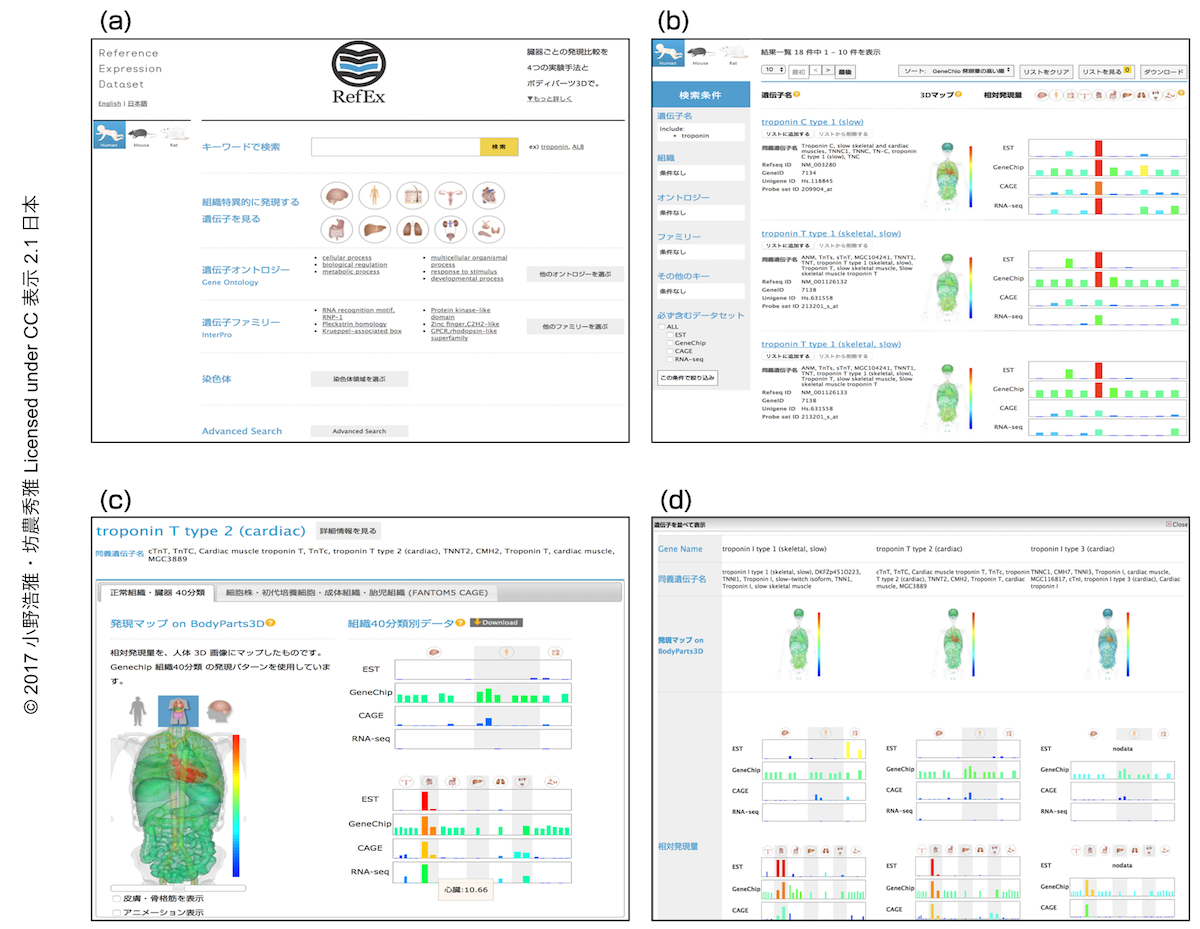
トップページ(図1a)には,左上部にRefExの対応する3つの生物種,ヒト,マウス,ラットのアイコンがあり,これをクリックすることにより切り替える.もっとも基本的な“キーワードで検索”においては,検索窓に文字を入力するたびに検索語の候補が提示されるので,それらから選択することより容易にキーワードを入力できる.また,“遺伝子オントロジー”,“遺伝子ファミリー”,“染色体”のそれぞれから検索できるよう整理されているので,たとえば,それぞれ,“転写因子”,“Gタンパク質共役受容体”,“2番染色体”などのように,ある分類に属する遺伝子についてまとめて検索し比較できる.さらに,遺伝子発現解析における比較対照としてしばしば用いられる組織に特異的な遺伝子を測定データから独自に算出しており,これらは“組織特異的に発現する遺伝子を見る”の組織および臓器のアイコンをクリックするだけで簡単に一覧することができる.さらに,“Advanced Search”では複雑な検索条件をいちどに指定することが可能であり,あらかじめID情報などが手元にある場合には目的とするデータに簡単にいきつくことができる.
検索結果一覧ページ(図1b)においては,項目別のソートおよび絞り込み検索が可能で,検索条件を柔軟に入れ替えながら検索結果を閲覧し比較することができる.検索結果一覧ページ,および,遺伝子発現の詳細情報ページ(図1c)では,組織あるいは臓器のあいだの比較と,EST 1,2),GeneChip 3),CAGE 4),RNA-seq 5) など測定手法のあいだの比較を両立させた相対発現量が棒グラフで示されるとともに,人体の3次元モデルであるBodyParts3D/Anatomography 6)(URL:http://lifesciencedb.jp/bp3d/)に発現量を反映させたヒートマップが表示される.詳細情報ページに記載された種々のIDにはそれぞれ,RefExの内部リンクやオリジナルのデータベースのサイトへの外部リンクが貼られており,同じ分類に属する遺伝子を再検索したり,RefEx自体を遺伝子検索の起点としたりすることもできる.また,リスト機能(図1d)を使うと,検索結果として表示された個々の遺伝子について一時的に保存しておくことができる.リストに追加した遺伝子は,最大で3つについて,組織あるいは臓器における発現量を比較しながら,Gene Ontology 7) などにより遺伝子に付与された機能に関する注釈情報を見比べることができる.
最近,理化学研究所のFANTOMプロジェクト5(FANTOM5)により大量の遺伝子発現データが公開されたが8,9),これらもRefExに収載されている.FANTOM5のデータは,ゲノムにコードされている遺伝子プロモーターおよび転写因子の制御ネットワークを明らかにすることを目的として得られたもので,それらを閲覧できるウェブサイトも公開されてはいるが,生命科学の多くの研究者にとって,その規模の大きさと複雑さから再利用のむずかしいものであった.RefExをつうじ,FANTOM5のヒトで556種にもおよぶ広範囲な組織や臓器における高精度な遺伝子発現データについても,可視化および比較が簡単になった.
なお,RefExの使い方を解説した動画をライフサイエンス統合データベースセンターが提供する統合TV 10) から公開している(URL:http://togotv.dbcls.jp/ja/20140222.html)ので,そちらも参考にされたい.
2.RefExの活用事例
RefExを利用することにより,研究の対象とする遺伝子が平常時にどの組織あるいは細胞にどのくらい発現しているのか,自ら実験することなく確認できる.また,なじみのない遺伝子に遭遇したときには,ふつうは個別の研究論文における実験データや記述などからそれらの生物学的な特徴を類推するが,RefExによれば実験のデザインに左右されない大規模かつ網羅的な測定データから自身の目でそれらを簡単に確認することができる.さらに,用意した複数の遺伝子IDについて一括で検索できる機能を備えているほか,リスト機能を用いて遺伝子の詳細データを並列に比較することができるため,遺伝子発現解析などにより見い出された遺伝子のあいだの関係性を知るためのツールとしても有用である.このような活用により,RefExは遺伝子発現解析のための強力なウェブツールとして,生命科学および医学の研究に幅広く貢献することが期待される.
一般の社会においても,新聞の見出しなどに“やせる遺伝子,発見”のような表現がみかけられるようになり,ひとつひとつの遺伝子がどのようなはたらきをもつのか,科学の研究に裏打ちされた正確な情報がもとめられている.将来的には,研究者だけでなく,一般の人も遺伝子について検索することが日常的になり,RefExがその第1の選択肢として使われることをめざしている.
3.データの出力機能およびデータレポジトリ
RefExの提供するすべてのデータは,クリエイティブ・コモンズライセンスのもとで,オープンデータとして自由にダウンロードおよび再利用することができる.検索結果一覧ページや詳細情報ページのデータはいずれもダウンロードが可能で,自身のデータと参照することも,それらを使った再解析も自由にできる.
また,外部の研究データレポジトリ“figshare”にも,すべてのデータがDOIつきで公開されている(URL:https://doi.org/10.6084/m9.figshare.c.3812815).さらに,ソフトウェア開発プロジェクトのための共有ウェブサービス“GitHub”にも,公開データの再解析に用いたプログラムやドキュメントを整理しており(URL:https://github.com/dbcls/RefEx),RefExの提供する再解析データについては,ある一定の評価品質および再現性を担保している.RefExは生命科学におけるデータの共有および再利用の活用例のひとつであり,データ駆動型研究のためのデータセットあるいはウェブツールとしてだれでも自由に使うことができる.
おわりに
RefExは,ライフサイエンス統合データベースセンターのミッションのひとつである“公共データベースの再利用の促進”の実例のひとつである.各種の遺伝子発現データを並列に整理しそれらを簡単に検索できる現在のインターフェースは,2011年にRefExとして公開をはじめたときから変わらない.RefExという名称はreference expression datasetの略であるが,米国NCBIの提供する高品質な遺伝子配列データベースRefSeq(NCBI reference sequence database)のように,誰もが参照し使われるような遺伝子発現データベースを構築したいという開発当初の目標に由来する.
RefExは公開ののちすぐには論文にせず,サービスとしての使いやすさを向上させることを優先して開発を進めた.統合データベース講習会AJACS(URL:http://togotv.dbcls.jp/ajacs_text.html)などにおいて積極的に紹介するうちしだいに利用されるようになり,さまざまな論文においてURLを引用される機会も増えてきた.このように,遺伝子発現解析に資するサービスとしてある一定の完成度に達したことから論文としてまとめることにし,投稿先を探した.ちょうど,Scientific Data誌においてFANTOM5 collection(URL:http://www.nature.com/collections/fantom5)という特集が組まれることになり,FANTOM5のデータがRefExに収載されたことから,RefExの論文もこの特集に組み込むことになった.当初は,1次データセットについて測定の対象,方法,品質を“Data Descriptor”という形式で出版し,ウェブツールとしての部分はとりあげない方針だったが,Technical Validationを記述することや,再解析したデータのすべてを公共アーカイブサイトに公開することなどにより,新設された“Article”という形式で出版された.
今後は,世界各地で進められているFANTOMやGTEx 11) などの遺伝子発現に関する大規模研究プロジェクトを中心として,高精度かつ広範囲な遺伝子発現データを収集し統合することにより,より有用性の高い参照データを作成する予定である.また,それらの参照データを簡単に検索したり,発現データどうしを詳細に比較したりすることを可能にする,直感的なウェブインターフェースの開発を進めていきたいと考えている.
文 献
- Okubo, K., Hori, N., Matoba, R. et al.: Large scale cDNA sequencing for analysis of quantitative and qualitative aspects of gene expression. Nat. Genet., 2, 173-179 (1992)[PubMed]
- Ogasawara, O., Otsuji, M., Watanabe, K. et al.: BodyMap-Xs: anatomical breakdown of 17 million animal ESTs for cross-species comparison of gene expression. Nucleic Acids Res., 34, D628-D631 (2006)[PubMed]
- Wu, C., Jin, X., Tsueng, G. et al.: BioGPS: building your own mash-up of gene annotations and expression profiles. Nucleic Acids Res., 44, D313-D316 (2016)[PubMed]
- Shiraki, T., Kondo, S., Katayama, S. et al.: Cap analysis gene expression for high-throughput analysis of transcriptional starting point and identification of promoter usage. Proc. Natl. Acad. Sci, USA, 100, 15776-15781 (2003)[PubMed]
- Sudmant, P. H., Alexis, M. S. & Burge, C. B.: Meta-analysis of RNA-seq expression data across species, tissues and studies. Genome Biol., 16, 287 (2015)[PubMed]
- Mitsuhashi, N., Fujieda, K., Tamura, T. et al.: BodyParts3D: 3D structure database for anatomical concepts. Nucleic Acids Res., 37, D782-D785 (2009)[PubMed]
- Ashburner, M., Ball, C. A., Blake, J. A. et al.: Gene ontology: tool for the unification of biology. Nat. Genet., 25, 25-29 (2000)[PubMed]
- The FANTOM Consortium and the RIKEN PMI and CLST (DGT): A promoter-level mammalian expression atlas. Nature, 507, 462-470 (2014)[PubMed]
- Lizio, M., Harshbarger, J., Shimoji, H. et al.: Gateways to the FANTOM5 promoter level mammalian expression atlas. Genome Biol., 16, 22 (2015)[PubMed]
- Kawano, S., Ono, H., Takagi, T. et al.: Tutorial videos of bioinformatics resources: online distribution trial in Japan named TogoTV. Brief. Bioinform., 13, 258-268 (2012)[PubMed]
- GTEx Consortium: The Genotype-Tissue Expression (GTEx) pilot analysis: multitissue gene regulation in humans. Science, 348, 648-660 (2015)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
生命科学の教科書における関連するセクションへのリンク
東京大学 大学院総合文化研究科・教養学部附属教養教育高度化機構自然科学教育高度化部門から公開されている生命科学の教科書 “A Comprehensive Approach To LIFE SCIENCE”(羊土社『理系総合のための生命科学 第2版』の英語版)における関連するセクションへのリンクです.
- 24.1 What is the Genome?
- 12.4 Protein Transport in Prokaryotic Cells
- 11.2 Characteristic Structures in Prokaryotes
- 1.7 Major Classification of Organisms
著者プロフィール
略歴:2010年 ライフサイエンス統合データベースセンター 特任技術専門員を経て,2012年より同 特任助教.
坊農 秀雅(Hidemasa Bono)
ライフサイエンス統合データベースセンター 特任准教授.
© 2017 小野浩雅・坊農秀雅 Licensed under CC 表示 2.1 日本