植物におけるマイクロRNAを介した翻訳抑制機構
岩川弘宙・泊 幸秀
(東京大学分子細胞生物学研究所 RNA機能研究分野)
email:岩川弘宙,泊 幸秀
DOI: 10.7875/first.author.2013.153
Molecular insights into microRNA-mediated translational repression in plants.
Hiro-oki Iwakawa, Yukihide Tomari
Molecular Cell, 52, 591-601 (2013)
小分子の非コードRNAであるマイクロRNAは,ArgonauteとともにRISCとよばれるRNA-タンパク質複合体を形成し,相補的な配列をもつmRNAの発現を抑制する.植物のマイクロRNAは標的mRNAを切断するとともに翻訳を抑制することが知られているが,どのようにして標的mRNAの翻訳を抑制するのか詳細な分子機構はこれまで明らかにされていなかった.この研究では,植物培養細胞に由来するin vitro系を用いることにより,シロイヌナズナのRISCのもつ遺伝子発現の抑制機能を生化学的に切り分けることに成功した.その結果,シロイヌナズナのRISCは動物のRISCとは異なり,標的mRNAのポリA鎖を分解することなく翻訳開始の段階を抑制した.興味深いことに,シロイヌナズナのRISCがタンパク質をコードする領域に結合した場合,翻訳開始を抑制するだけではなく,翻訳伸長,つまり,リボソームの進行を物理的に阻害することが明らかになった.また,シロイヌナズナのRISCが翻訳を抑制するには,動物のRISCと比べマイクロRNAと標的mRNAとのあいだに高い相補性が必要であった.これらの結果は,これまでほとんど理解の進んでいなかった植物のマイクロRNAに依存した翻訳抑制機構を解明する手がかりになると考えられる.
ここ十数年で,タンパク質の情報が書き込まれていない非コードRNAが動物および植物の遺伝子のはたらきを広く制御していることが明らかになってきた.約20塩基の長さをもつマイクロRNA(miRNA)は,もっとも重要な非コードRNAのひとつである.たとえば,ヒトのゲノムには2000種以上のmiRNAが存在し,ヒトのすべての遺伝子の半分程度を制御している可能性があると示唆されている.一方,植物研究のモデルとしてよく使われているシロイヌナズナのゲノムには,現在までに約300種のmiRNAがデータベースに登録されており,それらは発生,分化,ストレス応答など,さまざまな生命現象に深くかかわっている.
約20塩基の長さからなるmiRNAは,ヘアピン構造をもつ長い前駆体RNAが,RNaseIII型酵素であるDicerによりプロセシングされることにより,二本鎖RNAのかたちで生み出される.二本鎖miRNAはArgonaute(AGO)に取り込まれ,一方のRNA鎖だけがAGOに残り,RISC(RNA-induced silencing complex)とよばれるRNA-タンパク質複合体を形成する.AGOはRNaseH様の活性部位をもち,miRNAと相補的な標的mRNAを切断することができる.一方,miRNAと標的mRNAとが形成する二本鎖RNAの中央部分にミスマッチの存在する場合には,標的mRNAを切断することはできない1).
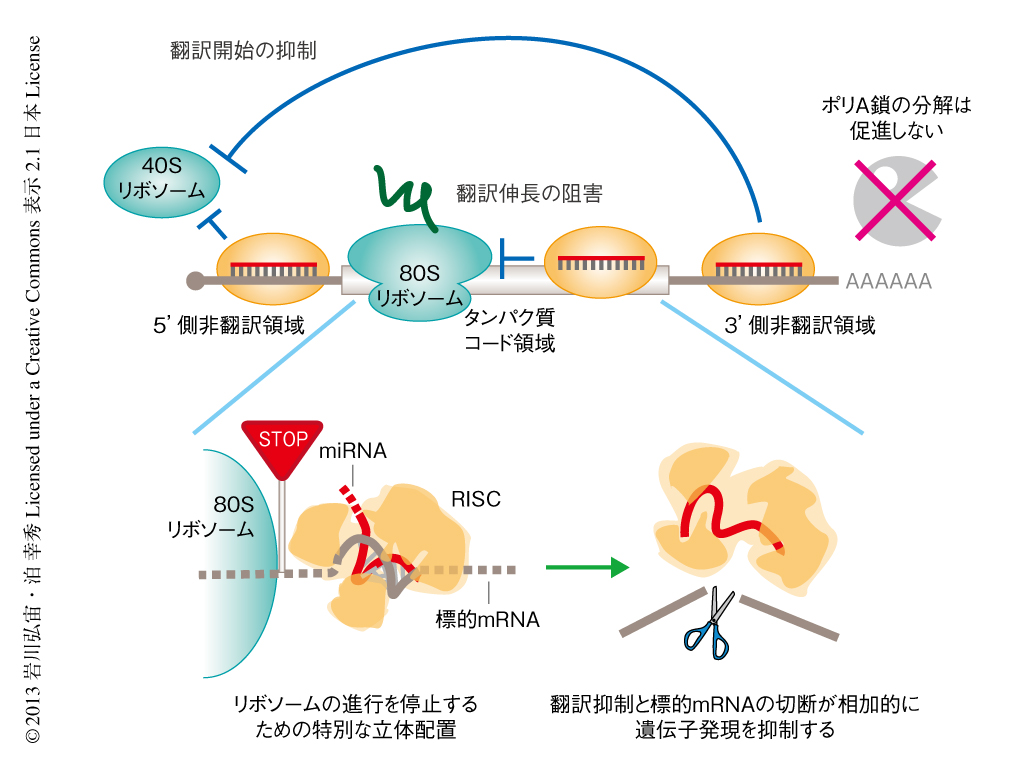
動物の標的mRNAはmiRNAと広範囲に相互作用する配列をもつことはまれであり,多くの場合,miRNAの5’側から数えて2~8番目までの配列(シード配列)に対し相補的な配列をもつ.標的配列の多くはmRNAの3’側非翻訳領域に集中しており,複数の標的配列が存在することも多い2).多くの場合,動物のRISCはmiRNAのシード領域においてのみ標的mRNAと結合し,中央部分にはミスマッチを含むため標的mRNAを切断することはない.その代わり,GW182というタンパク質をリクルートすることにより標的mRNAの翻訳を抑制し,mRNAの3’末端に存在し翻訳の促進およびmRNAの安定化にかかわるポリA鎖の分解を促進する3)(図1).
植物にはGW182のホモログは存在しない.また,これまで明らかにされている標的mRNAは,miRNAとほぼ完全に相補的な配列を,3’側非翻訳領域にかぎらず5’側非翻訳領域およびタンパク質をコードする領域にもつ4).これらの理由から,植物のRISCは標的mRNAを切断することにより遺伝子発現を抑制していると考えられてきたが,近年の遺伝学的な解析より,植物のRISCも翻訳を抑制していることが明らかにされた5).一方で,GW182の存在しない植物において,どのようにして標的mRNAの翻訳が抑制されるかという分子機構に関する報告は現在まで皆無であった(図1).
なお,RISCによる標的RNAの認識と切断の機構については,佐々木 浩・泊 幸秀, 領域融合レビュー, 1, e001, 2012 も参照されたい.
植物のRISCは標的mRNAの切断と翻訳抑制の2つの遺伝子発現の抑制経路をもつため,翻訳抑制活性のみを評価することが困難である.さらに,10種類以上も存在するAGOホモログの存在が解析をよりむずかしくしている.これらの問題を解決するには,1種類の切断活性欠損変異AGOのみに任意のmiRNAを結合させて機能を解析しなければならないが,植物体や培養細胞を用いた実験では現実性に乏しい.そこで,生体における翻訳の機構を忠実に再現でき,かつ,単一のRISCと結合することのできるタバコの培養細胞であるBY-2細胞に由来するin vitro系6,7),および,モデル植物であるシロイヌナズナの培養細胞であるMM2d細胞に由来するin vitro系8) を応用して,植物のRISCによる翻訳抑制を生化学的に解析する基盤を構築した.具体的には,in vitroにおいて発現させた活性欠損変異をもつシロイヌナズナAGO1に,化学合成したmiRNAを結合させ,切断活性を欠損したRISCを形成させたのち,標的配列をもつレポーターmRNAをくわえることにより翻訳抑制活性を評価した.この系では合成miRNAは内在性のAGOには取り込まれないため,in vitroにおいて発現させたRISCの機能のみを解析することができる.
動物における典型的な標的mRNAと同じ,3’側非翻訳領域に複数の標的配列をもつレポーターmRNAがシロイヌナズナAGO1を含むRISCにより翻訳を抑制されるかどうか調べるため,ルシフェラーゼアッセイを行った.その結果,標的mRNAの翻訳は標的配列の個数に依存して40%から60%ほど抑制された.以上より,シロイヌナズナAGO1を含むRISCは,動物のRISCと同様に,3’側非翻訳領域から標的mRNAの翻訳を抑制する能力のあることが明らかになった.一方で,シロイヌナズナAGO1を含むRISCは,動物にみられるような標的mRNAのもつポリA鎖の分解を促進しなかった.この動物と植物のあいだのRISCの機能の違いは,ポリA鎖分解酵素をAGOにリクルートするGW182のホモログが植物には存在しないことに起因すると考えられた.
植物の標的配列は3’側非翻訳領域だけでなくmRNAのさまざまな位置に存在する.そこで,5’側非翻訳領域およびタンパク質コード領域にそれぞれ標的配列をもつレポーターmRNAを作製しルシフェラーゼアッセイを行った.その結果,標的配列に完全に相補的なmiRNAを結合させた場合は5’側非翻訳領域およびタンパク質コード領域から強い翻訳抑制が認められた.一方で,シード領域でのみ標的配列との相補性をもつmiRNAはレポーターmRNAのどの領域に結合してもほとんど翻訳を抑制できなかった.標的mRNAと免疫沈降したシロイヌナズナAGO1を含むRISCとの結合親和性をフィルター結合アッセイにより調べたところ,標的配列と完全に相補的なmiRNAを結合させたRISCはpMオーダーという高い親和性を示したのに対し,シード領域でのみ標的配列と相補性をもつmiRNAを結合させたRISCは標的mRNAとほとんど結合しなかった.以上より,植物のmiRNAが標的mRNAを認識し翻訳を抑制するには,動物より高い相補性が必要であることが明らかになった.興味深いことに,標的mRNAの中央部分とのあいだに2塩基のミスマッチをもつmiRNAは,5’側非翻訳領域あるいは3’側非翻訳領域に結合した場合は効率よく標的mRNAの翻訳を抑制したものの,タンパク質コード領域に結合した場合にはほとんど翻訳を抑制できなかった.この結果は,RISCが結合する位置により標的mRNAの翻訳抑制に必要な相補性は異なることを示しており,RISCがタンパク質コード領域と相互作用した場合は,非翻訳領域と相互作用した場合とは異なる分子機構により翻訳は抑制されている可能性が示唆された.
シロイヌナズナAGO1を含むRISCは翻訳のどの段階を阻害しているか解析した.翻訳の過程は大きく分けて,開始,伸長,終結,の3つの段階からなる.翻訳開始は翻訳の律速段階であり,これまで知られているさまざまな翻訳制御機構は翻訳開始を抑制(または,促進)する場合が大半である.また最近の研究から,動物のRISCも翻訳開始を阻害することが知られている.シロイヌナズナAGO1を含むRISCが標的mRNAに結合した場合に翻訳開始を阻害するかどうか調べるため,スクロース密度勾配遠心法を用いたリボソーム-標的mRNA複合体の解析を行った.この方法は,放射線同位体により標識した標的mRNAを,翻訳伸長の阻害剤をくわえた植物の抽出液にRISCの存在下または非存在下で恒温にて静置し,スクロース密度勾配遠心法によりリボソームと結合した標的mRNAの量を解析するものである.RISCにより翻訳開始が阻害された場合,リボソームと結合した標的mRNAの量は減少し,リボソームと結合していない標的mRNAの量は増加する.実験には,5’側非翻訳領域,タンパク質コード領域,3’側非翻訳領域に標的配列をもつmRNAを用いた.これらどの標的mRNAを用いた場合においても,シロイヌナズナAGO1を含むRISCの存在下ではリボソーム-mRNA複合体の形成率は低下した.これより,シロイヌナズナAGO1を含むRISCは翻訳開始を阻害することが明らかになった.シロイヌナズナAGO1を含むRISCが5’側非翻訳領域または3’側非翻訳領域に結合した場合,さきのルシフェラーゼアッセイにおける翻訳抑制の度合いと,リボソーム-標的mRNA複合体の解析における複合体の減少の度合いがおおよそ合致したため,翻訳開始の阻害によりRISCによる翻訳抑制を説明することができる.一方,タンパク質コード領域に結合した植物のRISCがひき起こした95%にも及ぶ強い翻訳抑制は,40%程度の翻訳抑制を示した翻訳開始の阻害だけでは説明がつかなかった.この結果は,シロイヌナズナAGO1を含むRISCがタンパク質コード領域に結合した場合,翻訳開始よりあとの段階も阻害することを示唆した.
シロイヌナズナAGO1を含むRISCが5’側非翻訳領域に結合した場合,3’側非翻訳領域に結合した場合よりもはるかに強く翻訳開始を阻害した.この結果は,RISCが結合する位置により異なる機構で翻訳開始を抑制することを示唆した.この仮説を検証するため,真核生物のmRNAの翻訳において必須な5’末端のキャップ構造がなくてもリボソームをmRNAにリクルートすることのできるウイルスの特殊なRNA構造であるIRES(internal ribosome entry site)を用いた.5’側非翻訳領域にIRESをもち,3’側非翻訳領域に1つあるいは複数の標的配列をもつようなmRNAは,RISCによりまったく翻訳が抑制されなかった.つまり,IRESをもつmRNAはキャップ構造をもつ真核生物のmRNAとは異なり,3’側非翻訳領域に結合したRISCに対し抵抗性をもつことがわかった.一方で,5’側非翻訳領域にIRESとその下流に標的配列をもつmRNAは,真核生物のmRNAと同様に効率よく翻訳が抑制された.これらの結果より,5’側非翻訳領域に存在する標的配列と3’側非翻訳領域に存在する標的配列による翻訳抑制機構は異なることが示された.動物のRISCもIRESをもつmRNAの翻訳は抑制できないことから,3’側非翻訳領域に結合したシロイヌナズナAGO1を含むRISCは,動物のRISCと同じような機構により翻訳を抑制しているのかもしれない.一方,5’側非翻訳領域にRISCが結合した場合,リボソームのリクルートあるいは進行を物理的に阻害している可能性が高いと考えられた.
RISCがタンパク質コード領域に結合した場合に翻訳伸長を阻害しているかどうか調べるため,N末端にFLAGタグをもちタンパク質コード領域の異なる位置に標的配列をもつmRNAを設計した.もし,RISCがタンパク質コード領域においてリボソームの進行を停止するのであれば,FLAGタグに対するウェスタンブロット解析により,N末端から標的配列までのアミノ酸残基からなる不完全な長さをもつペプチドの蓄積を検出できるはずである.この仮説を支持するように,標的配列に完全に相補的なmiRNAを結合させたRISCをくわえた場合,完全長のタンパク質の蓄積は減少した一方,おおよそ標的配列までの不完全な長さのペプチドの蓄積が認められた.反対に,中央部分に2塩基のミスマッチをもつようなmiRNAを結合させたRISCをくわえても,完全長のタンパク質の減少および不完全長のペプチドの蓄積は観察されなかった.この結果は,中央部分に2塩基のミスマッチをもつmiRNAはタンパク質コード領域に結合しても翻訳を抑制しなかったという,さきのルシフェラーゼアッセイの結果と一致した.以上より,RISCが完全に相補的なmiRNAを介してタンパク質コード領域に結合した場合,翻訳伸長,つまり,リボソームの進行を阻害している可能性が強く示唆された.
なぜmiRNAと標的配列の中央部分とのあいだに2塩基のミスマッチが存在するとシロイヌナズナAGO1を含むRISCはタンパク質コード領域からの翻訳を抑制できないのだろうか.動物における研究により,中央部分の2塩基は標的mRNAとの親和性に影響しないと報告されている9).実際に,免疫沈降したシロイヌナズナAGO1を含むRISCとの親和性は,完全に相補的な標的mRNAと中央部分にミスマッチをもつ標的mRNAとのあいだでまったく差はなかった.解離速度定数を調べた結果,miRNAに完全に相補的な標的mRNAは,中央部分に2塩基のミスマッチをもつ標的mRNAよりも遅く結合し,遅く解離することが明らかになった.これより,完全に相補的な標的mRNAと中央部分に2塩基のミスマッチをもつ標的mRNAとでは,RISCとの結合様式が異なることが示された.実際に,高度好熱菌Thermus thermophilusのAGO,小分子DNA(T. thermophilusのAGOにはRNAではなくDNAが取り込まれる),標的mRNAとの三者複合体の結晶構造から,中央部分に2塩基のミスマッチが存在する場合にはRISCの立体配置が変化することがわかっている10).これらのことを考えあわせると,シロイヌナズナAGO1を含むRISCがタンパク質コード領域においてリボソームの進行をさまたげるには,mRNAとの親和性が高いだけでは不十分で,ある特別な立体配置をもつことが必要だと考えられた(図2).
これまでは,切断活性欠損変異をもつシロイヌナズナAGO1,合成miRNA,レポーターmRNAを用いることにより,miRNAによる翻訳抑制機構を厳密に切り分けてきた.最後に,植物のmiRNA,そして,実際の標的mRNAを用いて,生体に近い条件において実験を行った.標的mRNAとして,タンパク質コード領域にmiR156と相補的な配列をもつSPL13 mRNAを用いた.SPL13 mRNAを野生型のRISCとin vitroにおいて恒温にて静置した場合,完全長のタンパク質は蓄積しなかったのに対し,不完全な長さのペプチドは蓄積した.このペプチドがRISCにより切断された短いmRNAからの翻訳産物なのか,それとも,RISCがSPL mRNAのタンパク質コード領域に結合することにより翻訳伸長が停止したことによる短い翻訳産物なのかを明らかにするため,RISCによる予想される切断部位までの長さをもつ短いSPL13 mRNAをin vitroにおいて恒温にて静置し,ペプチドが蓄積するかどうかを調べた.その結果,この短いSPL13 mRNAはほとんど翻訳されないことが明らかになった.これらの結果より,in vitroにおいて蓄積したペプチドは,RISCがSPL13 mRNAを切断するまえに,タンパク質コード領域においてリボソームの進行を阻害することにより蓄積したものであると考えられた.この結果を支持するように,SPL13 mRNAと野生型のRISCをin vitroにおいて恒温にて静置し経時変化をおったところ,きわめて早い時間からペプチドの蓄積が観察された.これらの結果より,野生型シロイヌナズナAGO1を含むRISCは標的mRNAの切断と翻訳抑制の2つの経路を使うことにより,迅速かつ強力に遺伝子発現を抑制していると考えられた.
この研究では,植物培養細胞の抽出液から構築したin vitro系において“mRNAの切断”と“翻訳抑制”という植物のRISCがもつ2つの機能を切り分けることにより,翻訳抑制のみを抽出しその分子機構を解析した.その結果,1)翻訳抑制にはポリA鎖の分解はともなわないこと,2)翻訳抑制にはmiRNAの高い相補性を必要とすること,3)標的配列の位置により異なる翻訳抑制機構がはたらくこと,4)タンパク質コード領域においてはリボソームの進行の停止が起こること,5)標的mRNAの切断と翻訳抑制とが相加的に遺伝子発現を抑制すること,が明らかになった(図2).どれも動物のRISCはもたない性質であり,植物のRISCに特有の翻訳抑制機構であるといえる.
今後,植物体においてmRNAの切断と翻訳抑制とがどのように遺伝子発現の抑制に貢献しているかを明らかとするとともに,生体においてRISCによりリボソームの進行が停止したあとリボソームはmRNAから外れるのか,また,生合成されたペプチドや標的mRNAは分解されるのか,という翻訳抑制ののちの運命を調べることが課題となるだろう.
略歴:2010年 京都大学大学院農学研究科博士課程 修了,同年 東京大学分子細胞生物学研究所 研究員を経て,2013年より同 助教.
研究テーマ:小さなRNAの機能を分子レベルで理解する.
関心事:麺類,核酸,万歩計.
泊 幸秀(Yukihide Tomari)
東京大学分子細胞生物学研究所 教授.
研究室URL:http://www.iam.u-tokyo.ac.jp/tomari/
© 2013 岩川弘宙・泊 幸秀 Licensed under CC 表示 2.1 日本
(東京大学分子細胞生物学研究所 RNA機能研究分野)
email:岩川弘宙,泊 幸秀
DOI: 10.7875/first.author.2013.153
Molecular insights into microRNA-mediated translational repression in plants.
Hiro-oki Iwakawa, Yukihide Tomari
Molecular Cell, 52, 591-601 (2013)
要 約
小分子の非コードRNAであるマイクロRNAは,ArgonauteとともにRISCとよばれるRNA-タンパク質複合体を形成し,相補的な配列をもつmRNAの発現を抑制する.植物のマイクロRNAは標的mRNAを切断するとともに翻訳を抑制することが知られているが,どのようにして標的mRNAの翻訳を抑制するのか詳細な分子機構はこれまで明らかにされていなかった.この研究では,植物培養細胞に由来するin vitro系を用いることにより,シロイヌナズナのRISCのもつ遺伝子発現の抑制機能を生化学的に切り分けることに成功した.その結果,シロイヌナズナのRISCは動物のRISCとは異なり,標的mRNAのポリA鎖を分解することなく翻訳開始の段階を抑制した.興味深いことに,シロイヌナズナのRISCがタンパク質をコードする領域に結合した場合,翻訳開始を抑制するだけではなく,翻訳伸長,つまり,リボソームの進行を物理的に阻害することが明らかになった.また,シロイヌナズナのRISCが翻訳を抑制するには,動物のRISCと比べマイクロRNAと標的mRNAとのあいだに高い相補性が必要であった.これらの結果は,これまでほとんど理解の進んでいなかった植物のマイクロRNAに依存した翻訳抑制機構を解明する手がかりになると考えられる.
はじめに
ここ十数年で,タンパク質の情報が書き込まれていない非コードRNAが動物および植物の遺伝子のはたらきを広く制御していることが明らかになってきた.約20塩基の長さをもつマイクロRNA(miRNA)は,もっとも重要な非コードRNAのひとつである.たとえば,ヒトのゲノムには2000種以上のmiRNAが存在し,ヒトのすべての遺伝子の半分程度を制御している可能性があると示唆されている.一方,植物研究のモデルとしてよく使われているシロイヌナズナのゲノムには,現在までに約300種のmiRNAがデータベースに登録されており,それらは発生,分化,ストレス応答など,さまざまな生命現象に深くかかわっている.
約20塩基の長さからなるmiRNAは,ヘアピン構造をもつ長い前駆体RNAが,RNaseIII型酵素であるDicerによりプロセシングされることにより,二本鎖RNAのかたちで生み出される.二本鎖miRNAはArgonaute(AGO)に取り込まれ,一方のRNA鎖だけがAGOに残り,RISC(RNA-induced silencing complex)とよばれるRNA-タンパク質複合体を形成する.AGOはRNaseH様の活性部位をもち,miRNAと相補的な標的mRNAを切断することができる.一方,miRNAと標的mRNAとが形成する二本鎖RNAの中央部分にミスマッチの存在する場合には,標的mRNAを切断することはできない1).
動物の標的mRNAはmiRNAと広範囲に相互作用する配列をもつことはまれであり,多くの場合,miRNAの5’側から数えて2~8番目までの配列(シード配列)に対し相補的な配列をもつ.標的配列の多くはmRNAの3’側非翻訳領域に集中しており,複数の標的配列が存在することも多い2).多くの場合,動物のRISCはmiRNAのシード領域においてのみ標的mRNAと結合し,中央部分にはミスマッチを含むため標的mRNAを切断することはない.その代わり,GW182というタンパク質をリクルートすることにより標的mRNAの翻訳を抑制し,mRNAの3’末端に存在し翻訳の促進およびmRNAの安定化にかかわるポリA鎖の分解を促進する3)(図1).
植物にはGW182のホモログは存在しない.また,これまで明らかにされている標的mRNAは,miRNAとほぼ完全に相補的な配列を,3’側非翻訳領域にかぎらず5’側非翻訳領域およびタンパク質をコードする領域にもつ4).これらの理由から,植物のRISCは標的mRNAを切断することにより遺伝子発現を抑制していると考えられてきたが,近年の遺伝学的な解析より,植物のRISCも翻訳を抑制していることが明らかにされた5).一方で,GW182の存在しない植物において,どのようにして標的mRNAの翻訳が抑制されるかという分子機構に関する報告は現在まで皆無であった(図1).
なお,RISCによる標的RNAの認識と切断の機構については,佐々木 浩・泊 幸秀, 領域融合レビュー, 1, e001, 2012 も参照されたい.
1.植物のRISCによる翻訳抑制を再現することのできるin vitro系の構築
植物のRISCは標的mRNAの切断と翻訳抑制の2つの遺伝子発現の抑制経路をもつため,翻訳抑制活性のみを評価することが困難である.さらに,10種類以上も存在するAGOホモログの存在が解析をよりむずかしくしている.これらの問題を解決するには,1種類の切断活性欠損変異AGOのみに任意のmiRNAを結合させて機能を解析しなければならないが,植物体や培養細胞を用いた実験では現実性に乏しい.そこで,生体における翻訳の機構を忠実に再現でき,かつ,単一のRISCと結合することのできるタバコの培養細胞であるBY-2細胞に由来するin vitro系6,7),および,モデル植物であるシロイヌナズナの培養細胞であるMM2d細胞に由来するin vitro系8) を応用して,植物のRISCによる翻訳抑制を生化学的に解析する基盤を構築した.具体的には,in vitroにおいて発現させた活性欠損変異をもつシロイヌナズナAGO1に,化学合成したmiRNAを結合させ,切断活性を欠損したRISCを形成させたのち,標的配列をもつレポーターmRNAをくわえることにより翻訳抑制活性を評価した.この系では合成miRNAは内在性のAGOには取り込まれないため,in vitroにおいて発現させたRISCの機能のみを解析することができる.
2.シロイヌナズナAGO1を含むRISCはポリA鎖を分解することなく翻訳を抑制する
動物における典型的な標的mRNAと同じ,3’側非翻訳領域に複数の標的配列をもつレポーターmRNAがシロイヌナズナAGO1を含むRISCにより翻訳を抑制されるかどうか調べるため,ルシフェラーゼアッセイを行った.その結果,標的mRNAの翻訳は標的配列の個数に依存して40%から60%ほど抑制された.以上より,シロイヌナズナAGO1を含むRISCは,動物のRISCと同様に,3’側非翻訳領域から標的mRNAの翻訳を抑制する能力のあることが明らかになった.一方で,シロイヌナズナAGO1を含むRISCは,動物にみられるような標的mRNAのもつポリA鎖の分解を促進しなかった.この動物と植物のあいだのRISCの機能の違いは,ポリA鎖分解酵素をAGOにリクルートするGW182のホモログが植物には存在しないことに起因すると考えられた.
3.シロイヌナズナAGO1を含むRISCによる翻訳抑制にはmiRNAと標的mRNAとの高い相補性が必要である
植物の標的配列は3’側非翻訳領域だけでなくmRNAのさまざまな位置に存在する.そこで,5’側非翻訳領域およびタンパク質コード領域にそれぞれ標的配列をもつレポーターmRNAを作製しルシフェラーゼアッセイを行った.その結果,標的配列に完全に相補的なmiRNAを結合させた場合は5’側非翻訳領域およびタンパク質コード領域から強い翻訳抑制が認められた.一方で,シード領域でのみ標的配列との相補性をもつmiRNAはレポーターmRNAのどの領域に結合してもほとんど翻訳を抑制できなかった.標的mRNAと免疫沈降したシロイヌナズナAGO1を含むRISCとの結合親和性をフィルター結合アッセイにより調べたところ,標的配列と完全に相補的なmiRNAを結合させたRISCはpMオーダーという高い親和性を示したのに対し,シード領域でのみ標的配列と相補性をもつmiRNAを結合させたRISCは標的mRNAとほとんど結合しなかった.以上より,植物のmiRNAが標的mRNAを認識し翻訳を抑制するには,動物より高い相補性が必要であることが明らかになった.興味深いことに,標的mRNAの中央部分とのあいだに2塩基のミスマッチをもつmiRNAは,5’側非翻訳領域あるいは3’側非翻訳領域に結合した場合は効率よく標的mRNAの翻訳を抑制したものの,タンパク質コード領域に結合した場合にはほとんど翻訳を抑制できなかった.この結果は,RISCが結合する位置により標的mRNAの翻訳抑制に必要な相補性は異なることを示しており,RISCがタンパク質コード領域と相互作用した場合は,非翻訳領域と相互作用した場合とは異なる分子機構により翻訳は抑制されている可能性が示唆された.
4.シロイヌナズナAGO1を含むRISCは翻訳開始を阻害する
シロイヌナズナAGO1を含むRISCは翻訳のどの段階を阻害しているか解析した.翻訳の過程は大きく分けて,開始,伸長,終結,の3つの段階からなる.翻訳開始は翻訳の律速段階であり,これまで知られているさまざまな翻訳制御機構は翻訳開始を抑制(または,促進)する場合が大半である.また最近の研究から,動物のRISCも翻訳開始を阻害することが知られている.シロイヌナズナAGO1を含むRISCが標的mRNAに結合した場合に翻訳開始を阻害するかどうか調べるため,スクロース密度勾配遠心法を用いたリボソーム-標的mRNA複合体の解析を行った.この方法は,放射線同位体により標識した標的mRNAを,翻訳伸長の阻害剤をくわえた植物の抽出液にRISCの存在下または非存在下で恒温にて静置し,スクロース密度勾配遠心法によりリボソームと結合した標的mRNAの量を解析するものである.RISCにより翻訳開始が阻害された場合,リボソームと結合した標的mRNAの量は減少し,リボソームと結合していない標的mRNAの量は増加する.実験には,5’側非翻訳領域,タンパク質コード領域,3’側非翻訳領域に標的配列をもつmRNAを用いた.これらどの標的mRNAを用いた場合においても,シロイヌナズナAGO1を含むRISCの存在下ではリボソーム-mRNA複合体の形成率は低下した.これより,シロイヌナズナAGO1を含むRISCは翻訳開始を阻害することが明らかになった.シロイヌナズナAGO1を含むRISCが5’側非翻訳領域または3’側非翻訳領域に結合した場合,さきのルシフェラーゼアッセイにおける翻訳抑制の度合いと,リボソーム-標的mRNA複合体の解析における複合体の減少の度合いがおおよそ合致したため,翻訳開始の阻害によりRISCによる翻訳抑制を説明することができる.一方,タンパク質コード領域に結合した植物のRISCがひき起こした95%にも及ぶ強い翻訳抑制は,40%程度の翻訳抑制を示した翻訳開始の阻害だけでは説明がつかなかった.この結果は,シロイヌナズナAGO1を含むRISCがタンパク質コード領域に結合した場合,翻訳開始よりあとの段階も阻害することを示唆した.
5.異なる機構による翻訳開始の阻害
シロイヌナズナAGO1を含むRISCが5’側非翻訳領域に結合した場合,3’側非翻訳領域に結合した場合よりもはるかに強く翻訳開始を阻害した.この結果は,RISCが結合する位置により異なる機構で翻訳開始を抑制することを示唆した.この仮説を検証するため,真核生物のmRNAの翻訳において必須な5’末端のキャップ構造がなくてもリボソームをmRNAにリクルートすることのできるウイルスの特殊なRNA構造であるIRES(internal ribosome entry site)を用いた.5’側非翻訳領域にIRESをもち,3’側非翻訳領域に1つあるいは複数の標的配列をもつようなmRNAは,RISCによりまったく翻訳が抑制されなかった.つまり,IRESをもつmRNAはキャップ構造をもつ真核生物のmRNAとは異なり,3’側非翻訳領域に結合したRISCに対し抵抗性をもつことがわかった.一方で,5’側非翻訳領域にIRESとその下流に標的配列をもつmRNAは,真核生物のmRNAと同様に効率よく翻訳が抑制された.これらの結果より,5’側非翻訳領域に存在する標的配列と3’側非翻訳領域に存在する標的配列による翻訳抑制機構は異なることが示された.動物のRISCもIRESをもつmRNAの翻訳は抑制できないことから,3’側非翻訳領域に結合したシロイヌナズナAGO1を含むRISCは,動物のRISCと同じような機構により翻訳を抑制しているのかもしれない.一方,5’側非翻訳領域にRISCが結合した場合,リボソームのリクルートあるいは進行を物理的に阻害している可能性が高いと考えられた.
6.シロイヌナズナAGO1を含むRISCによる翻訳伸長の阻害
RISCがタンパク質コード領域に結合した場合に翻訳伸長を阻害しているかどうか調べるため,N末端にFLAGタグをもちタンパク質コード領域の異なる位置に標的配列をもつmRNAを設計した.もし,RISCがタンパク質コード領域においてリボソームの進行を停止するのであれば,FLAGタグに対するウェスタンブロット解析により,N末端から標的配列までのアミノ酸残基からなる不完全な長さをもつペプチドの蓄積を検出できるはずである.この仮説を支持するように,標的配列に完全に相補的なmiRNAを結合させたRISCをくわえた場合,完全長のタンパク質の蓄積は減少した一方,おおよそ標的配列までの不完全な長さのペプチドの蓄積が認められた.反対に,中央部分に2塩基のミスマッチをもつようなmiRNAを結合させたRISCをくわえても,完全長のタンパク質の減少および不完全長のペプチドの蓄積は観察されなかった.この結果は,中央部分に2塩基のミスマッチをもつmiRNAはタンパク質コード領域に結合しても翻訳を抑制しなかったという,さきのルシフェラーゼアッセイの結果と一致した.以上より,RISCが完全に相補的なmiRNAを介してタンパク質コード領域に結合した場合,翻訳伸長,つまり,リボソームの進行を阻害している可能性が強く示唆された.
7.シロイヌナズナAGO1を含むRISCは完全に相補的な標的配列には遅く結合し遅く解離する
なぜmiRNAと標的配列の中央部分とのあいだに2塩基のミスマッチが存在するとシロイヌナズナAGO1を含むRISCはタンパク質コード領域からの翻訳を抑制できないのだろうか.動物における研究により,中央部分の2塩基は標的mRNAとの親和性に影響しないと報告されている9).実際に,免疫沈降したシロイヌナズナAGO1を含むRISCとの親和性は,完全に相補的な標的mRNAと中央部分にミスマッチをもつ標的mRNAとのあいだでまったく差はなかった.解離速度定数を調べた結果,miRNAに完全に相補的な標的mRNAは,中央部分に2塩基のミスマッチをもつ標的mRNAよりも遅く結合し,遅く解離することが明らかになった.これより,完全に相補的な標的mRNAと中央部分に2塩基のミスマッチをもつ標的mRNAとでは,RISCとの結合様式が異なることが示された.実際に,高度好熱菌Thermus thermophilusのAGO,小分子DNA(T. thermophilusのAGOにはRNAではなくDNAが取り込まれる),標的mRNAとの三者複合体の結晶構造から,中央部分に2塩基のミスマッチが存在する場合にはRISCの立体配置が変化することがわかっている10).これらのことを考えあわせると,シロイヌナズナAGO1を含むRISCがタンパク質コード領域においてリボソームの進行をさまたげるには,mRNAとの親和性が高いだけでは不十分で,ある特別な立体配置をもつことが必要だと考えられた(図2).
8.天然の標的mRNAに対する翻訳抑制
これまでは,切断活性欠損変異をもつシロイヌナズナAGO1,合成miRNA,レポーターmRNAを用いることにより,miRNAによる翻訳抑制機構を厳密に切り分けてきた.最後に,植物のmiRNA,そして,実際の標的mRNAを用いて,生体に近い条件において実験を行った.標的mRNAとして,タンパク質コード領域にmiR156と相補的な配列をもつSPL13 mRNAを用いた.SPL13 mRNAを野生型のRISCとin vitroにおいて恒温にて静置した場合,完全長のタンパク質は蓄積しなかったのに対し,不完全な長さのペプチドは蓄積した.このペプチドがRISCにより切断された短いmRNAからの翻訳産物なのか,それとも,RISCがSPL mRNAのタンパク質コード領域に結合することにより翻訳伸長が停止したことによる短い翻訳産物なのかを明らかにするため,RISCによる予想される切断部位までの長さをもつ短いSPL13 mRNAをin vitroにおいて恒温にて静置し,ペプチドが蓄積するかどうかを調べた.その結果,この短いSPL13 mRNAはほとんど翻訳されないことが明らかになった.これらの結果より,in vitroにおいて蓄積したペプチドは,RISCがSPL13 mRNAを切断するまえに,タンパク質コード領域においてリボソームの進行を阻害することにより蓄積したものであると考えられた.この結果を支持するように,SPL13 mRNAと野生型のRISCをin vitroにおいて恒温にて静置し経時変化をおったところ,きわめて早い時間からペプチドの蓄積が観察された.これらの結果より,野生型シロイヌナズナAGO1を含むRISCは標的mRNAの切断と翻訳抑制の2つの経路を使うことにより,迅速かつ強力に遺伝子発現を抑制していると考えられた.
おわりに
この研究では,植物培養細胞の抽出液から構築したin vitro系において“mRNAの切断”と“翻訳抑制”という植物のRISCがもつ2つの機能を切り分けることにより,翻訳抑制のみを抽出しその分子機構を解析した.その結果,1)翻訳抑制にはポリA鎖の分解はともなわないこと,2)翻訳抑制にはmiRNAの高い相補性を必要とすること,3)標的配列の位置により異なる翻訳抑制機構がはたらくこと,4)タンパク質コード領域においてはリボソームの進行の停止が起こること,5)標的mRNAの切断と翻訳抑制とが相加的に遺伝子発現を抑制すること,が明らかになった(図2).どれも動物のRISCはもたない性質であり,植物のRISCに特有の翻訳抑制機構であるといえる.
今後,植物体においてmRNAの切断と翻訳抑制とがどのように遺伝子発現の抑制に貢献しているかを明らかとするとともに,生体においてRISCによりリボソームの進行が停止したあとリボソームはmRNAから外れるのか,また,生合成されたペプチドや標的mRNAは分解されるのか,という翻訳抑制ののちの運命を調べることが課題となるだろう.
文 献
- Carthew, R. W. & Sontheimer, E. J.: Origins and mechanisms of miRNAs and siRNAs. Cell, 136, 642-655 (2009)[PubMed]
- Bartel, D. P.: MicroRNAs: target recognition and regulatory functions. Cell, 136, 215-233 (2009)[PubMed]
- Huntzinger, E. & Izaurralde, E.: Gene silencing by microRNAs: contributions of translational repression and mRNA decay. Nat. Rev. Genet., 12, 99-110 (2011)[PubMed]
- Axtell, M. J., Westholm, J. O. & Lai, E. C.: Vive la difference: biogenesis and evolution of microRNAs in plants and animals. Genome Biol., 12, 221 (2011)[PubMed]
- Brodersen, P., Sakvarelidze-Achard, L., Bruun-Rasmussen, M. et al.: Widespread translational inhibition by plant miRNAs and siRNAs. Science, 320, 1185-1190 (2008)[PubMed]
- Iki, T., Yoshikawa, M., Nishikiori, M. et al.: In vitro assembly of plant RNA-induced silencing complexes facilitated by molecular chaperone HSP90. Mol Cell, 39, 282-291 (2010)[PubMed] [新着論文レビュー]
- Komoda, K., Naito, S., Ishikawa, M.: Replication of plant RNA virus genomes in a cell-free extract of evacuolated plant protoplasts. Proc. Natl. Acad. Sci. USA, 101, 1863-1867 (2004)[PubMed]
- Iwakawa, H. O, Tajima, Y., Taniguchi, T. et al.: Poly(A)-binding protein facilitates translation of an uncapped/nonpolyadenylated viral RNA by binding to the 3’ untranslated region. J. Virol., 86, 7836-7849 (2012)[PubMed]
- Wee, L. M., Flores-Jasso, C. F, Salomon, W. E. et al.: Argonaute divides its RNA guide into domains with distinct functions and RNA-binding properties. Cell, 151, 1055-1067 (2012)[PubMed]
- Wang, Y., Juranek, S., Li, H. et al.: Nucleation, propagation and cleavage of target RNAs in Ago silencing complexes. Nature, 461, 754-761 (2009)[PubMed]
著者プロフィール
略歴:2010年 京都大学大学院農学研究科博士課程 修了,同年 東京大学分子細胞生物学研究所 研究員を経て,2013年より同 助教.
研究テーマ:小さなRNAの機能を分子レベルで理解する.
関心事:麺類,核酸,万歩計.
泊 幸秀(Yukihide Tomari)
東京大学分子細胞生物学研究所 教授.
研究室URL:http://www.iam.u-tokyo.ac.jp/tomari/
© 2013 岩川弘宙・泊 幸秀 Licensed under CC 表示 2.1 日本