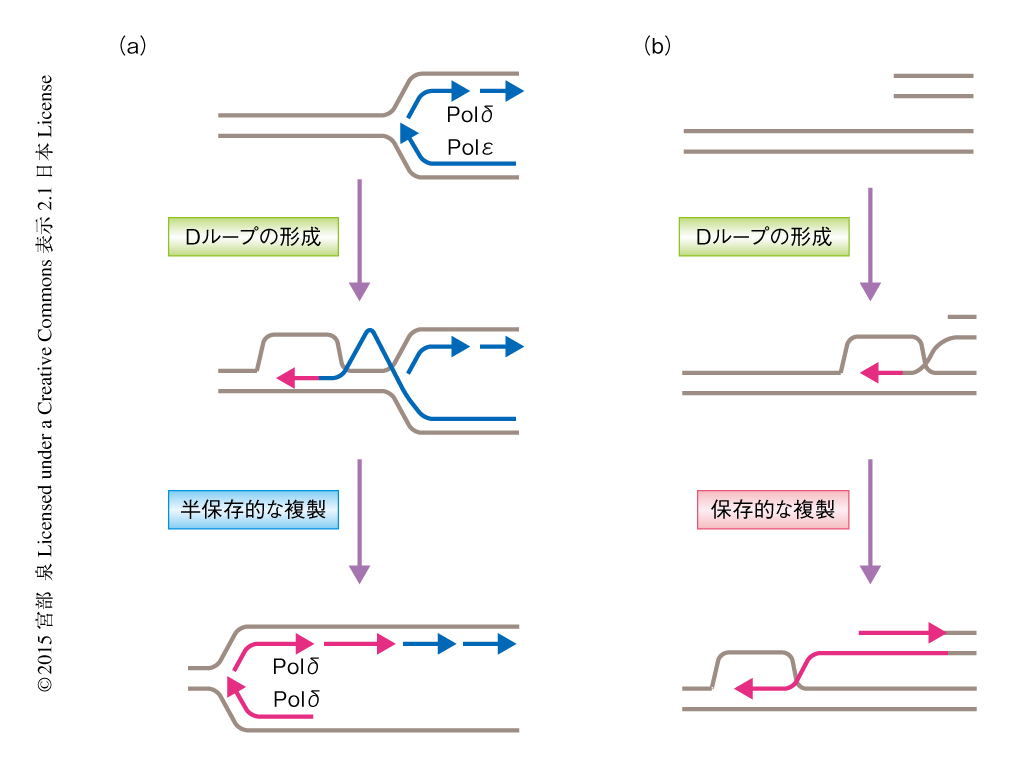
相同組換えにより再開された複製においてはDNAポリメラーゼδがリーディング鎖およびラギング鎖を合成する
宮部 泉
(英国Sussex大学Genome Damage and Stability Centre)
email:宮部 泉
DOI: 10.7875/first.author.2015.120
Polymeraseδreplicates both strands after homologous recombination-dependent fork restart.
Izumi Miyabe, Ken'Ichi Mizuno, Andrea Keszthelyi, Yasukazu Daigaku, Meliti Skouteri, Saed Mohebi, Thomas A. Kunkel, Johanne M. Murray, Antony M. Carr
Nature Structural & Molecular Biology, 22, 932-938 (2015)
ゲノムを安定に維持するため,複製は細胞周期においていちどだけ正確に起こらなければならない.しかし,複製フォークはさまざまな要因により障害され不活性化される.複製フォークの不活性化は高い頻度で起こるため,隣接した複製フォークからの複製だけではこれを補うことはできない.そのため,真核生物は不活性状態にある複製フォークを相同組換えに依存して再開する機構を進化させてきた.再開された複製は細胞の生存率を上げる一方,その複製は不正確で変異を起こしやすい.このことから,再開された複製は通常の複製とは異なる機構によりDNAを合成していることが示唆される.筆者らは,分裂酵母において相同組換えに依存的に再開された複製は通常の複製と同様に半保存的であるが,リーディング鎖およびラギング鎖ともDNAポリメラーゼδにより合成されていることを証明した.
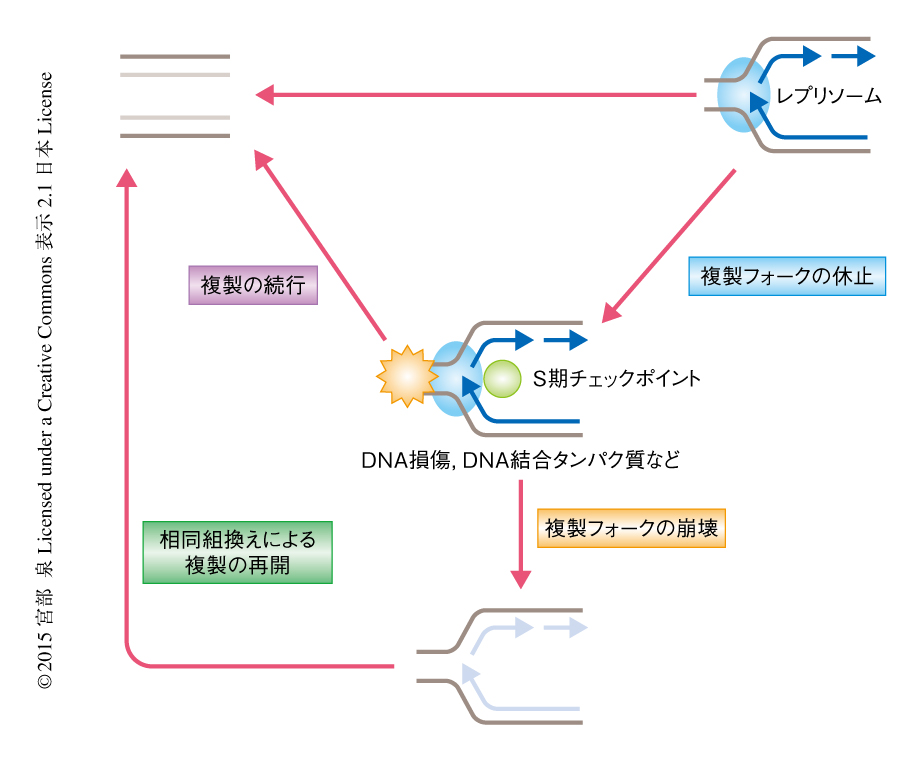
複製が正確に起こることはゲノムの維持において必要不可欠である.複製への障害は,がん細胞やある種の遺伝病の細胞においてみられる転座やコピー数多型など,種々の遺伝的な変化の原因になる.複製は細胞において,DNAの損傷,非ヒストンタンパク質とDNAとの結合,DNAの2次構造,転写装置との衝突などによりつねに障害されている.複製フォークがそれらの障害物に衝突すると複製は一時的に停止し,S期チェックポイントにより保護される.休止した複製フォークから障害が除去されると複製はそのまま続行される.しかし,複製フォークの一部は障害物の性質やS期チェックポイントによる保護の不首尾などにより複製を続行できなくなる.真核生物においては,崩壊した複製フォークから複製を再開するため,相同組換え機構が用いられている(図1).
相同組換えによる複製の開始の機構を解明するため,いくつかの部位特異的な実験系が構築されてきた.出芽酵母においては複製を開始させるのに2本鎖DNA切断が用いられており,これはDNA鎖切断により誘発される複製とよばれている.実験的には,DNA鎖切断により誘発される複製はS期以外で起こり,複製の開始には数時間がかかる.一方,分裂酵母においてはRTS1とよばれる複製停止配列が用いられている.RTS1による停止から再開された複製はS期に起こり,2本鎖DNA切断をともなわないことから,DNA鎖切断により誘発される複製とは区別される.
相同組換えによる複製の開始は複製の困難な状況においても複製を完了させることを可能にするが,そこにはリスクも存在する.1つ目のリスクは,相同組換えが異所的に起こり複製が本来とは異なる遺伝子座において開始されてしまう可能性のあることである.最近になり,2つ目のリスクとして,再開された複製はエラーを起こしやすいことが明らかにされた1,2).この複製のエラーの増加から,相同組換えにより開始される複製は通常の複製とは異なる機構によることが示唆された.
複製停止配列RTS1は約850塩基からなり,Rtf1の結合部位をいくつか含んでいる.Rtf1は複製の停止に必須であり欠失するとRTS1は通常どおりに複製される.つまり,RTS1の塩基配列それ自体に複製を停止させる効果はなく,複製の停止はRtf1に完全に依存する3).複製起点の近傍にRTS1を配置すると,複製起点からRTS1までの領域はRtf1のあるなしにかかわらず通常どおり複製される.RTS1の下流の領域は複製の阻害のないときには通常どおり複製されるが,複製の阻害があると相同組換えにより再開される4).
これまでに筆者らは,リボヌクレオチドを高頻度で取り込む変異型のDNAポリメラーゼを用いて,通常の複製においてはDNAポリメラーゼεがリーディング鎖を,DNAポリメラーゼδがラギング鎖を合成することを証明した5,6).RNaseH2の活性を欠失した細胞において,それら変異型のDNAポリメラーゼが合成したDNA鎖は過剰なリボヌクレオチドを含みアルカリ処理に対し高感受性になる.つまり,変異型のDNAポリメラーゼにより合成されたDNA鎖はアルカリ処理により特異的に分解される.通常の複製においては,変異型DNAポリメラーゼεをもつ細胞においてはリーディング鎖のみが,変異型DNAポリメラーゼδをもつ細胞においてはラギング鎖のみが,アルカリ処理により分解される.
これらの実験系を用いて,RTS1による停止から再開された複製に用いられるDNAポリメラーゼの同定を試みた.複製起点からRTS1までの領域をみると,複製の阻害の有無にかかわらず通常の複製と同じパターンを示した.この結果は,Rtf1は複製に用いられるDNAポリメラーゼに影響しないことを示した.そして,RTS1の下流においては,複製の阻害がないときは通常の複製と同じパターンを示したが,複製の阻害のあるときには変異型DNAポリメラーゼεをもつ細胞においてリーディング鎖およびラギング鎖ともアルカリ処理に耐性になり,変異型DNAポリメラーゼδをもつ細胞においてはリーディング鎖およびラギング鎖ともアルカリ処理に感受性になった.以上のことから,相同組換えにより再開された複製においては,リーディング鎖およびラギング鎖ともDNAポリメラーゼδにより合成され,DNAポリメラーゼεは複製にほとんど寄与しないことが示された.また,これまでの研究により,再開された複製は最初の約1 kbが非常に不安定であり,その下流の約1.5 kbにおいてある程度まで安定化されることが明らかにされている.そこで,RTS1のすぐ下流の約1.1 kb,および,さらにその下流の約1.8 kbにおけるアルカリ処理に対する感受性を変異型DNAポリメラーゼδをもつ細胞において比較したところ,差はみられなかった.この結果から,相同組換えによる複製の再開ののち少なくとも約3 kbはリーディング鎖およびラギング鎖ともDNAポリメラーゼδにより安定に複製されていることが示された.
DNAポリメラーゼαは複製の開始に必要とされ,岡崎フラグメントの合成はDNAポリメラーゼαにより開始される.つまり,DNAポリメラーゼαはおもにラギング鎖の合成に寄与している.RTS1による停止から再開された複製におけるDNAポリメラーゼαの役割を調べるため,高い頻度で変異を起こす変異型のDNAポリメラーゼαを用いて解析した.野生株では複製の阻害があるとRTS1の下流における変異の頻度は5倍ほど高くなるが,変異型DNAポリメラーゼαをもつ細胞では変異の頻度は逆に約1/5にまで下がった.このことから,相同組換えにより再開された複製においてDNAポリメラーゼαの使用頻度は通常の複製よりも顕著に低くなっていることが示唆された.
DNA鎖切断により誘発される複製においては,1本鎖DNAが相同な2本鎖DNAに入り込んだ構造であるDループが移動することにより複製が進行することが知られている7,8).RTS1による停止から再開された複製は2本鎖DNA切断をともなわないが,DNA鎖切断により誘発される複製と同様の機構により複製が進行している可能性は十分に考えられた.Dループの移動により複製が進行する場合,新生したリーディング鎖がラギング鎖の合成の鋳型になるため複製は保存的になる.この可能性をメセルソン-スタールの実験により検証した結果,相同組換えにより停止から再開された複製は半保存的であり,DNA鎖切断により誘発される複製とは異なる機構によることが示された.
停止ののち再開された複製は不安定であり,マイクロホモロジーの欠失や逆向きくり返し配列において染色体の再編成が高い頻度で誘発される.そこで,DNAポリメラーゼδによるリーディング鎖およびラギング鎖の複製がこの直接の原因なのかどうか検証した.DNAポリメラーゼεのDNA合成活性は細胞の生存に必須ではないことが知られており,DNAポリメラーゼεの活性部位を欠失した細胞においてバルクのDNA合成はDNAポリメラーゼδのみによると考えられている9).そこで,DNAポリメラーゼεの活性部位を欠失した細胞において実際にDNAポリメラーゼδがリーディング鎖およびラギング鎖を合成しているかどうか確かめるため,変異型DNAポリメラーゼδをもつ細胞においてアルカリ感受性試験を行った.その結果,リーディング鎖およびラギング鎖ともアルカリ処理による分解が観察され,DNAポリメラーゼεのDNA合成活性が欠失したときにはDNAポリメラーゼδにより代替されることが確認された.
マイクロホモロジーの欠失を測定するため,20 bpの重複配列をもつura4遺伝子を用いた.重複した20 bpが欠失するとウラシル原栄養能が回復する.RTS1の下流にこのura4遺伝子を配置して複製の阻害の有無により欠失の頻度を測定したところ,これまでの報告のとおり10),DNAポリメラーゼεをもつ細胞では複製を阻害すると欠失の頻度は約10倍に上昇した.DNAポリメラーゼεの活性部位を欠失した細胞においては,複製の阻害がない条件における欠失の頻度がDNAポリメラーゼεをもつ細胞と比べ約5倍に上昇したが,複製の阻害があるときには欠失の頻度はさらに上昇し,DNAポリメラーゼεをもつ細胞において複製の阻害があるときと同じくらいになった.したがって,DNAポリメラーゼδによるリーディング鎖およびラギング鎖の複製はマイクロホモロジーの欠失においてエラーをひき起こしやすいが,相同組換えにより再開された複製のエラーの頻度はそれだけでは説明できなかった.
また,RTS1の下流に逆向きくり返し配列を配置すると,複製の阻害に依存して染色体の再編成が増加する1).DNAポリメラーゼεの活性部位を欠失した細胞においてサザンブロット法により染色体の再編成を検出したところ,複製の阻害の有無にかかわらず,野生型の細胞とのあいだに大きな差はみられなかった.DNAポリメラーゼδによるリーディング鎖およびラギング鎖の複製は,このタイプの染色体の再編成にはほとんど寄与していないと考えられた.
停止ののち再開された複製は染色体異常をひき起こしやすいため,ゲノム不安定性の一因になりうる.もし,DNAポリメラーゼδによるリーディング鎖およびラギング鎖の複製が停止から再開された複製における共通の特徴であるなら,ゲノムにおいて染色体異常をひき起こしやすい領域においてはDNAポリメラーゼδの使用頻度が高くなっている可能性がある.つまり,DNAポリメラーゼδの使用頻度の高さが内在性の脆弱部位を反映すると推測された.これまでに筆者らは,多くの領域において,DNAポリメラーゼδの使用頻度がゲノム全体の平均よりも高くなっていることを報告している5).そのほとんどは複製起点と重なることから,複製の開始の際にリーディング鎖がDNAポリメラーゼδにより合成されることがあり,その場合は,複製の進行とともにDNAポリメラーゼεと入れ替わるとの仮説をたてた.
複製起点にくわえ,セントロメアや接合型変換のような特殊な領域を除き,DNAポリメラーゼδの使用頻度が高い領域がいくつかみつかった.そのうち4つの領域にそれぞれ逆向きくり返し配列を配置し,サザンブロット法によりその安定性を検証したところ,2つの領域においてゲノム再編成の有意な上昇がみられた.3つ目の領域は統計的に有意ではないものの同様の傾向がみられたが,残りの1つでは差はみられなかった.
筆者らは,分裂酵母の複製阻害配列RTS1を用いて,相同組換えにより再開された複製においては,DNAポリメラーゼδがリーディング鎖およびラギング鎖を合成すること,DNAポリメラーゼαの使用頻度が低くなること,複製は半保存的であること,を証明した(図2).しかしながら,DNAポリメラーゼδによるリーディング鎖およびラギング鎖の合成は,停止ののち再開された複製の不安定性の主要な原因ではないことがわかった.このことから,通常の複製と再開された複製とのあいだにそれ以外の様式の違いが存在し,それが不安定性の原因となることが示唆された.その様式の違いについてはさらなる解析が必要である.
出芽酵母におけるDNA鎖切断により誘発される複製の系では,2本鎖DNA切断から複製が開始される.分裂酵母におけるRTS1による停止から再開された複製の系とは異なり,複製はS期以外で起こりその開始には数時間もかかることが知られている.また,この場合,Dループが移動することにより複製が進行するため複製は保存的になる.これらの2つの系の違いが生物種の違いに由来するのか,あるいは,細胞周期やDNAの構造の違いによるのかは不明である.
がん化の初期の過程においては,がん遺伝子の活性化により複製ストレスが生じており,それに対する細胞の応答が破綻するとがんは悪性化すると考えられている11-13).また,がん細胞においてみられる染色体の再編成は,しばしば脆弱部位とよばれる特定の領域において起こることが知られている.そのような領域では複製フォークの崩壊が高い頻度で起こっており,相同組換えによる複製の再開の必要性が高くなっていると推測される.筆者らは,DNAポリメラーゼδの使用頻度がゲノムに内在する脆弱部位の指標になる可能性を示唆した.さらなる検証が必要であるが,がん細胞においてみられるある種のゲノム不安定性は,相同組換えによる複製の再開によりひき起こされているのかもしれない.
略歴:2001年 京都大学大学院理学研究科 修了,同年 大阪大学微生物病研究所 研究員を経て,2005年より英国Sussex大学 リサーチフェロー.
研究テーマ:複製の阻害に対する応答機構.
© 2015 宮部 泉 Licensed under CC 表示 2.1 日本
(英国Sussex大学Genome Damage and Stability Centre)
email:宮部 泉
DOI: 10.7875/first.author.2015.120
Polymeraseδreplicates both strands after homologous recombination-dependent fork restart.
Izumi Miyabe, Ken'Ichi Mizuno, Andrea Keszthelyi, Yasukazu Daigaku, Meliti Skouteri, Saed Mohebi, Thomas A. Kunkel, Johanne M. Murray, Antony M. Carr
Nature Structural & Molecular Biology, 22, 932-938 (2015)
要 約
ゲノムを安定に維持するため,複製は細胞周期においていちどだけ正確に起こらなければならない.しかし,複製フォークはさまざまな要因により障害され不活性化される.複製フォークの不活性化は高い頻度で起こるため,隣接した複製フォークからの複製だけではこれを補うことはできない.そのため,真核生物は不活性状態にある複製フォークを相同組換えに依存して再開する機構を進化させてきた.再開された複製は細胞の生存率を上げる一方,その複製は不正確で変異を起こしやすい.このことから,再開された複製は通常の複製とは異なる機構によりDNAを合成していることが示唆される.筆者らは,分裂酵母において相同組換えに依存的に再開された複製は通常の複製と同様に半保存的であるが,リーディング鎖およびラギング鎖ともDNAポリメラーゼδにより合成されていることを証明した.
はじめに
複製が正確に起こることはゲノムの維持において必要不可欠である.複製への障害は,がん細胞やある種の遺伝病の細胞においてみられる転座やコピー数多型など,種々の遺伝的な変化の原因になる.複製は細胞において,DNAの損傷,非ヒストンタンパク質とDNAとの結合,DNAの2次構造,転写装置との衝突などによりつねに障害されている.複製フォークがそれらの障害物に衝突すると複製は一時的に停止し,S期チェックポイントにより保護される.休止した複製フォークから障害が除去されると複製はそのまま続行される.しかし,複製フォークの一部は障害物の性質やS期チェックポイントによる保護の不首尾などにより複製を続行できなくなる.真核生物においては,崩壊した複製フォークから複製を再開するため,相同組換え機構が用いられている(図1).
相同組換えによる複製の開始の機構を解明するため,いくつかの部位特異的な実験系が構築されてきた.出芽酵母においては複製を開始させるのに2本鎖DNA切断が用いられており,これはDNA鎖切断により誘発される複製とよばれている.実験的には,DNA鎖切断により誘発される複製はS期以外で起こり,複製の開始には数時間がかかる.一方,分裂酵母においてはRTS1とよばれる複製停止配列が用いられている.RTS1による停止から再開された複製はS期に起こり,2本鎖DNA切断をともなわないことから,DNA鎖切断により誘発される複製とは区別される.
相同組換えによる複製の開始は複製の困難な状況においても複製を完了させることを可能にするが,そこにはリスクも存在する.1つ目のリスクは,相同組換えが異所的に起こり複製が本来とは異なる遺伝子座において開始されてしまう可能性のあることである.最近になり,2つ目のリスクとして,再開された複製はエラーを起こしやすいことが明らかにされた1,2).この複製のエラーの増加から,相同組換えにより開始される複製は通常の複製とは異なる機構によることが示唆された.
1.相同組換えにより再開された複製に用いられるDNAポリメラーゼ
複製停止配列RTS1は約850塩基からなり,Rtf1の結合部位をいくつか含んでいる.Rtf1は複製の停止に必須であり欠失するとRTS1は通常どおりに複製される.つまり,RTS1の塩基配列それ自体に複製を停止させる効果はなく,複製の停止はRtf1に完全に依存する3).複製起点の近傍にRTS1を配置すると,複製起点からRTS1までの領域はRtf1のあるなしにかかわらず通常どおり複製される.RTS1の下流の領域は複製の阻害のないときには通常どおり複製されるが,複製の阻害があると相同組換えにより再開される4).
これまでに筆者らは,リボヌクレオチドを高頻度で取り込む変異型のDNAポリメラーゼを用いて,通常の複製においてはDNAポリメラーゼεがリーディング鎖を,DNAポリメラーゼδがラギング鎖を合成することを証明した5,6).RNaseH2の活性を欠失した細胞において,それら変異型のDNAポリメラーゼが合成したDNA鎖は過剰なリボヌクレオチドを含みアルカリ処理に対し高感受性になる.つまり,変異型のDNAポリメラーゼにより合成されたDNA鎖はアルカリ処理により特異的に分解される.通常の複製においては,変異型DNAポリメラーゼεをもつ細胞においてはリーディング鎖のみが,変異型DNAポリメラーゼδをもつ細胞においてはラギング鎖のみが,アルカリ処理により分解される.
これらの実験系を用いて,RTS1による停止から再開された複製に用いられるDNAポリメラーゼの同定を試みた.複製起点からRTS1までの領域をみると,複製の阻害の有無にかかわらず通常の複製と同じパターンを示した.この結果は,Rtf1は複製に用いられるDNAポリメラーゼに影響しないことを示した.そして,RTS1の下流においては,複製の阻害がないときは通常の複製と同じパターンを示したが,複製の阻害のあるときには変異型DNAポリメラーゼεをもつ細胞においてリーディング鎖およびラギング鎖ともアルカリ処理に耐性になり,変異型DNAポリメラーゼδをもつ細胞においてはリーディング鎖およびラギング鎖ともアルカリ処理に感受性になった.以上のことから,相同組換えにより再開された複製においては,リーディング鎖およびラギング鎖ともDNAポリメラーゼδにより合成され,DNAポリメラーゼεは複製にほとんど寄与しないことが示された.また,これまでの研究により,再開された複製は最初の約1 kbが非常に不安定であり,その下流の約1.5 kbにおいてある程度まで安定化されることが明らかにされている.そこで,RTS1のすぐ下流の約1.1 kb,および,さらにその下流の約1.8 kbにおけるアルカリ処理に対する感受性を変異型DNAポリメラーゼδをもつ細胞において比較したところ,差はみられなかった.この結果から,相同組換えによる複製の再開ののち少なくとも約3 kbはリーディング鎖およびラギング鎖ともDNAポリメラーゼδにより安定に複製されていることが示された.
DNAポリメラーゼαは複製の開始に必要とされ,岡崎フラグメントの合成はDNAポリメラーゼαにより開始される.つまり,DNAポリメラーゼαはおもにラギング鎖の合成に寄与している.RTS1による停止から再開された複製におけるDNAポリメラーゼαの役割を調べるため,高い頻度で変異を起こす変異型のDNAポリメラーゼαを用いて解析した.野生株では複製の阻害があるとRTS1の下流における変異の頻度は5倍ほど高くなるが,変異型DNAポリメラーゼαをもつ細胞では変異の頻度は逆に約1/5にまで下がった.このことから,相同組換えにより再開された複製においてDNAポリメラーゼαの使用頻度は通常の複製よりも顕著に低くなっていることが示唆された.
2.相同組換えにより再開された複製は半保存的である
DNA鎖切断により誘発される複製においては,1本鎖DNAが相同な2本鎖DNAに入り込んだ構造であるDループが移動することにより複製が進行することが知られている7,8).RTS1による停止から再開された複製は2本鎖DNA切断をともなわないが,DNA鎖切断により誘発される複製と同様の機構により複製が進行している可能性は十分に考えられた.Dループの移動により複製が進行する場合,新生したリーディング鎖がラギング鎖の合成の鋳型になるため複製は保存的になる.この可能性をメセルソン-スタールの実験により検証した結果,相同組換えにより停止から再開された複製は半保存的であり,DNA鎖切断により誘発される複製とは異なる機構によることが示された.
3.相同組換えにより再開された複製の不安定性はDNAポリメラーゼδによる複製だけでは説明できない
停止ののち再開された複製は不安定であり,マイクロホモロジーの欠失や逆向きくり返し配列において染色体の再編成が高い頻度で誘発される.そこで,DNAポリメラーゼδによるリーディング鎖およびラギング鎖の複製がこの直接の原因なのかどうか検証した.DNAポリメラーゼεのDNA合成活性は細胞の生存に必須ではないことが知られており,DNAポリメラーゼεの活性部位を欠失した細胞においてバルクのDNA合成はDNAポリメラーゼδのみによると考えられている9).そこで,DNAポリメラーゼεの活性部位を欠失した細胞において実際にDNAポリメラーゼδがリーディング鎖およびラギング鎖を合成しているかどうか確かめるため,変異型DNAポリメラーゼδをもつ細胞においてアルカリ感受性試験を行った.その結果,リーディング鎖およびラギング鎖ともアルカリ処理による分解が観察され,DNAポリメラーゼεのDNA合成活性が欠失したときにはDNAポリメラーゼδにより代替されることが確認された.
マイクロホモロジーの欠失を測定するため,20 bpの重複配列をもつura4遺伝子を用いた.重複した20 bpが欠失するとウラシル原栄養能が回復する.RTS1の下流にこのura4遺伝子を配置して複製の阻害の有無により欠失の頻度を測定したところ,これまでの報告のとおり10),DNAポリメラーゼεをもつ細胞では複製を阻害すると欠失の頻度は約10倍に上昇した.DNAポリメラーゼεの活性部位を欠失した細胞においては,複製の阻害がない条件における欠失の頻度がDNAポリメラーゼεをもつ細胞と比べ約5倍に上昇したが,複製の阻害があるときには欠失の頻度はさらに上昇し,DNAポリメラーゼεをもつ細胞において複製の阻害があるときと同じくらいになった.したがって,DNAポリメラーゼδによるリーディング鎖およびラギング鎖の複製はマイクロホモロジーの欠失においてエラーをひき起こしやすいが,相同組換えにより再開された複製のエラーの頻度はそれだけでは説明できなかった.
また,RTS1の下流に逆向きくり返し配列を配置すると,複製の阻害に依存して染色体の再編成が増加する1).DNAポリメラーゼεの活性部位を欠失した細胞においてサザンブロット法により染色体の再編成を検出したところ,複製の阻害の有無にかかわらず,野生型の細胞とのあいだに大きな差はみられなかった.DNAポリメラーゼδによるリーディング鎖およびラギング鎖の複製は,このタイプの染色体の再編成にはほとんど寄与していないと考えられた.
4.DNAポリメラーゼδの使用頻度が染色体の不安定性の指標となる可能性
停止ののち再開された複製は染色体異常をひき起こしやすいため,ゲノム不安定性の一因になりうる.もし,DNAポリメラーゼδによるリーディング鎖およびラギング鎖の複製が停止から再開された複製における共通の特徴であるなら,ゲノムにおいて染色体異常をひき起こしやすい領域においてはDNAポリメラーゼδの使用頻度が高くなっている可能性がある.つまり,DNAポリメラーゼδの使用頻度の高さが内在性の脆弱部位を反映すると推測された.これまでに筆者らは,多くの領域において,DNAポリメラーゼδの使用頻度がゲノム全体の平均よりも高くなっていることを報告している5).そのほとんどは複製起点と重なることから,複製の開始の際にリーディング鎖がDNAポリメラーゼδにより合成されることがあり,その場合は,複製の進行とともにDNAポリメラーゼεと入れ替わるとの仮説をたてた.
複製起点にくわえ,セントロメアや接合型変換のような特殊な領域を除き,DNAポリメラーゼδの使用頻度が高い領域がいくつかみつかった.そのうち4つの領域にそれぞれ逆向きくり返し配列を配置し,サザンブロット法によりその安定性を検証したところ,2つの領域においてゲノム再編成の有意な上昇がみられた.3つ目の領域は統計的に有意ではないものの同様の傾向がみられたが,残りの1つでは差はみられなかった.
おわりに
筆者らは,分裂酵母の複製阻害配列RTS1を用いて,相同組換えにより再開された複製においては,DNAポリメラーゼδがリーディング鎖およびラギング鎖を合成すること,DNAポリメラーゼαの使用頻度が低くなること,複製は半保存的であること,を証明した(図2).しかしながら,DNAポリメラーゼδによるリーディング鎖およびラギング鎖の合成は,停止ののち再開された複製の不安定性の主要な原因ではないことがわかった.このことから,通常の複製と再開された複製とのあいだにそれ以外の様式の違いが存在し,それが不安定性の原因となることが示唆された.その様式の違いについてはさらなる解析が必要である.
出芽酵母におけるDNA鎖切断により誘発される複製の系では,2本鎖DNA切断から複製が開始される.分裂酵母におけるRTS1による停止から再開された複製の系とは異なり,複製はS期以外で起こりその開始には数時間もかかることが知られている.また,この場合,Dループが移動することにより複製が進行するため複製は保存的になる.これらの2つの系の違いが生物種の違いに由来するのか,あるいは,細胞周期やDNAの構造の違いによるのかは不明である.
がん化の初期の過程においては,がん遺伝子の活性化により複製ストレスが生じており,それに対する細胞の応答が破綻するとがんは悪性化すると考えられている11-13).また,がん細胞においてみられる染色体の再編成は,しばしば脆弱部位とよばれる特定の領域において起こることが知られている.そのような領域では複製フォークの崩壊が高い頻度で起こっており,相同組換えによる複製の再開の必要性が高くなっていると推測される.筆者らは,DNAポリメラーゼδの使用頻度がゲノムに内在する脆弱部位の指標になる可能性を示唆した.さらなる検証が必要であるが,がん細胞においてみられるある種のゲノム不安定性は,相同組換えによる複製の再開によりひき起こされているのかもしれない.
文 献
- Mizuno, K., Miyabe, I., Schalbetter, S. A. et al.: Recombination-restarted replication makes inverted chromosome fusions at inverted repeats. Nature, 493, 246-249 (2013)[PubMed] [新着論文レビュー]
- Smith, C. E., Llorente, B. & Symington, L. S.: Template switching during break-induced replication. Nature, 447, 102-105 (2007)[PubMed]
- Eydmann, T., Sommariva, E., Inagawa, T. et al.: Rtf1-mediated eukaryotic site-specific replication termination. Genetics, 180, 27-39 (2008)[PubMed]
- Lambert, S., Watson, A., Sheedy, D. M. et al.: Gross chromosomal rearrangements and elevated recombination at an inducible site-specific replication fork barrier. Cell, 121, 689-702 (2005)[PubMed]
- Daigaku, Y., Keszthelyi, A., Muller, C. A. et al.: A global profile of replicative polymerase usage. Nat. Struct. Mol. Biol., 22, 192-198 (2015)[PubMed]
- Miyabe, I., Kunkel, T. A. & Carr, A. M.: The major roles of DNA polymerases epsilon and delta at the eukaryotic replication fork are evolutionarily conserved. PLoS Genet., 7, e1002407 (2011)[PubMed]
- Saini, N., Ramakrishnan, S., Elango, R. et al.: Migrating bubble during break-induced replication drives conservative DNA synthesis. Nature, 502, 389-392 (2013)[PubMed]
- Wilson, M. A., Kwon, Y., Xu, Y. et al.: Pif1 helicase and Polδ promote recombination-coupled DNA synthesis via bubble migration. Nature, 502, 393-396 (2013)[PubMed]
- Kesti, T., Flick, K., Keranen, S. et al.: DNA polymerase ε catalytic domains are dispensable for DNA replication, DNA repair, and cell viability. Mol. Cell, 3, 679-685 (1999)[PubMed]
- Iraqui, I., Chekkal, Y., Jmari, N. et al.: Recovery of arrested replication forks by homologous recombination is error-prone. PLoS Genet., 8, e1002976 (2012)[PubMed]
- Bartkova, J., Horejsi, Z., Koed, K. et al.: DNA damage response as a candidate anti-cancer barrier in early human tumorigenesis. Nature, 434, 864-870 (2005)[PubMed]
- Bartkova, J., Rezaei, N., Liontos, M. et al.: Oncogene-induced senescence is part of the tumorigenesis barrier imposed by DNA damage checkpoints. Nature, 444, 633-637 (2006)[PubMed]
- Gorgoulis, V. G., Vassiliou, L. V., Karakaidos, P. et al.: Activation of the DNA damage checkpoint and genomic instability in human precancerous lesions. Nature, 434, 907-913 (2005)[PubMed]
著者プロフィール
略歴:2001年 京都大学大学院理学研究科 修了,同年 大阪大学微生物病研究所 研究員を経て,2005年より英国Sussex大学 リサーチフェロー.
研究テーマ:複製の阻害に対する応答機構.
© 2015 宮部 泉 Licensed under CC 表示 2.1 日本