組換えにより再開したDNAの複製は誤りがちで逆位反復配列において染色体の再編成をひき起こす
水野 健一
(英国Sussex大学Genome Damage and Stability Centre)
email:水野健一
DOI: 10.7875/first.author.2013.001
Recombination-restarted replication makes inverted chromosome fusions at inverted repeats.
Ken’Ichi Mizuno, Izumi Miyabe, Stephanie A. Schalbetter, Antony M. Carr, Johanne M. Murray
Nature, 493, 246-249 (2013)
DNAの複製は増殖している細胞にとり必須であり,通常の状態において複製装置は非常に精度の高いコピーマシンである.複製の効率あるいは精度の低下はゲノムの安定性に影響を及ぼす.そして,ゲノムの恒常性の破綻はがん化やヒトゲノムの構造変化に起因するゲノム障害につながる.しかし,複製の停止によるゲノム恒常性の破綻の分子機構については明らかでない部分が多い.複製の停止からの複製の再開には相同組換えの必要とされる場合がある.筆者らは,再開した複製の性質を分子レベルにおいて解析するため,分裂酵母を用い部位特異的に複製を停止したのち相同組換えにより複製を再開する系を開発した.そして,この再生複製は,通常の複製とは異なり誤りがちであることを明らかにした.再生複製は逆位反復配列において高い頻度で染色体の再編成をひき起こした.この再生複製の誤りがちな性質は遺伝子の増幅や欠失の起因となり,がん化やゲノム障害の原因になると考えられた.
DNAの複製は増殖している細胞にとり欠かせない細胞周期の一過程であり,複製の適切な制御はゲノムの安定性にとり必須である.通常は,複製フォークが他方からくる別の複製フォークと融合することで複製は完了する.がん化の初期段階では,原がん遺伝子の活性化またはがん抑制遺伝子の不活性化により,細胞周期の制御が撹乱される.それがDNA合成の効率を低下させ,しばしば複製フォークを停止させる.このような複製ストレスのもとでは休眠している複製開始点の活性化が複製完了の手段であるが,複製開始点はゲノムにおいて均一には分布していない.複製開始点のほとんど存在しない染色体の脆弱部位では1,2),停止した複製フォークの再始動が複製完了の手段となる.染色体の脆弱部位をはじめとするさまざまな部位は,複製ストレスのもとで染色体の再編成,遺伝子増幅,遺伝子欠失をひき起こす1,3).
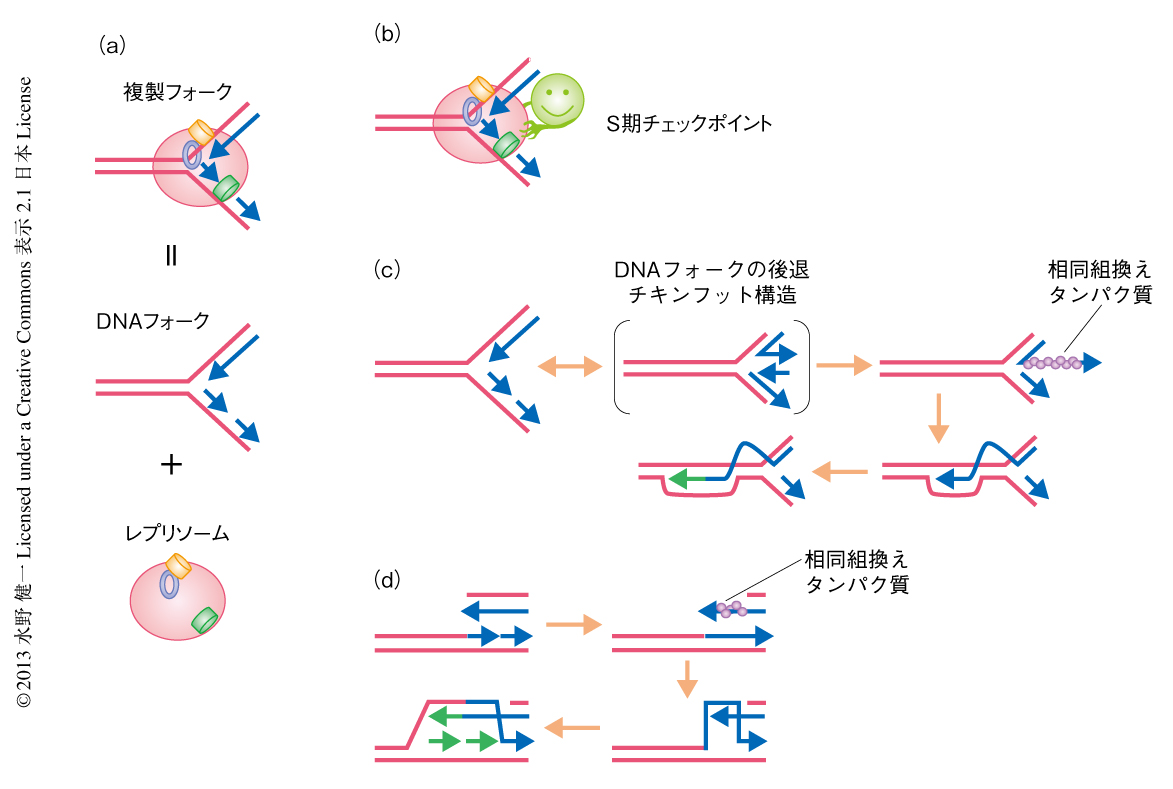
複製フォークはいろいろな要因により停止する.転写装置あるいはDNAに強く結合したタンパク質との衝突,基質であるdNTPの枯渇,DNA損傷による進行の阻害などである.停止した複製フォークは大きく3つに分類できる4)(図1).休止複製フォーク(paused fork),剥離複製フォーク(collapsed fork),切断複製フォーク(broken fork)である.休止複製フォークとは,複製フォークの進行は停止しているがDNAフォークにレプリソームが滞在したままの状態をいう(図1b).複製フォークの進行を停止させた障害が取り除かれるまで,S期チェックポイントによりレプリソームはDNAフォークにおいて保護される.障害が取り除かれたのち,レプリソームはDNAフォークに会合しているので,すぐにDNA合成を再開できる.休止複製フォークにおける複製の再開に相同組換えは必要ない.一方,剥離複製フォークと切断複製フォークは複製の再開に相同組換えを必要とする.剥離複製フォークとは,レプリソームがDNAフォークから解離した状態をいう4)(図1c).DNAフォークがあらわになるため,動的な構造変化が頻繁に起こりやすくなる.DNAフォークの後退が一時的にチキンフット状のDNA構造を形成する.その構造において露出した新生一本鎖DNAに相同組換えタンパク質が結合する.切断複製フォークとは,DNA鎖が切断され複製フォーク自体が壊れた状態をいう(図1d).複製フォークがニック(切れ目)のある二本鎖DNAに直面したときに起こる.このとき,レプリソームはDNAフォークから解離する.DNA切断末端が加工されることによりあらわになった一本鎖DNAに相同組換えタンパク質が結合する.一本鎖DNAと相同組換えタンパク質との複合体が二本鎖の鋳型DNAに侵入し相補DNA鎖と会合する.その会合の末端から新生DNA鎖が伸長することによりDNA合成は再開する.以下,相同組換えにより複製フォークが再始動した複製を再生複製とよぶ.
分裂酵母の接合型遺伝子座の近傍には複製フォークの停止配列であるRTS1配列が存在する.RTS1配列は方向性をもって複製フォークを停止させる.DNA結合タンパク質Rtf1はRTS1配列の複製フォーク停止活性に必要である.RTS1配列において停止した複製フォークは剥離複製フォークとなり複製の再開には相同組換えが必要となる5,6).複製フォークの停止と再始動の分子機構と,その再始動した複製フォークの性質を解明するため,RTS1配列を用いた部位特異的な複製フォークの停止系を開発した5-7).操作が安易な分裂酵母を使ったこの系の利点は,rtf1+遺伝子の転写発現の制御により,RTS1配列の複製フォーク停止活性を自由自在に制御できることである.これが,複製過程の分子レベルでの解析を可能にした.
1つのRTS1配列を用いてRTS1配列から再始動した複製フォークが逆位反復配列を複製するような系を構築した.用いた逆位反復配列の中央に14塩基対の介入配列を挿入した.この逆位反復配列はRTS1配列とはまったく異なる配列とした.RTS1配列のセントロメア側にある強力な複製開始点からの複製フォークは,RTS1配列において停止する.相同組換えを介し元来の鋳型DNA(相補DNA鎖)を用いて,停止した複製フォークは再始動する.
RTS1配列の下流に逆位反復配列が存在する場合,再生複製は染色体の再編成をひき起こした.複製フォークがRTS1配列において停止しない場合,この染色体の再編成は起こらなかった.この染色体の再編成は再生複製に依存した.また,この染色体の再編成は反復配列の長さに依存した.しかし,RTS1配列の下流に反復配列が存在しない場合は,再生複製はサザンブロッティング法により検出の可能なレベルの染色体の再編成を起こさなかった.この再編成した染色体の構造は,再生複製が逆位反復配列の中央において複製の方向を転換(Uターン)して逆位融合した構造に相当した(図2a).この逆位融合した染色体は余分なセントロメアをもつ染色体(dicentric chromosome,二連セントロメア染色体)であった(図2b).
逆位反復配列における介入配列の長さを変えて,染色体の再編成に対するその効果を観察した.介入配列が短いと逆位反復配列はDNA二次構造を形成しやすくなる.逆に,介入配列が長いとDNA二次構造を形成しにくい.予想されたとおり,介入配列が短いと染色体の逆位融合が頻発した.一方,介入配列が長いと染色体の逆位融合の頻度は低下した.しかしながら,それでもサザンブロッティング法により検出の可能な頻度であった.したがって,再生複製による染色体の再編成は,DNA二次構造を形成しやすい配列においてより頻繁に起こること,しかし,反復配列の相同性も染色体の再編成に寄与していることが明らかになった.
RTS1配列と逆位反復配列とのあいだに無関係なDNA配列を挿入し,染色体の再編成に対するRTS1配列から逆位反復配列までの距離の効果を観察した.逆位反復配列がRTS1配列に隣接しているとき,染色体の逆位融合の頻度は最高値を示した.距離を長くすると,その頻度は徐々に低下した.しかし,その距離が2400塩基対長であってもサザンブロッティング法により検出の可能な頻度であった.
2つのRTS1配列が近傍に存在すると,その相同な配列を鋳型として相同組換えの起こることがある.この誤った鋳型の選択により非対立遺伝子座の相同組換えが生じ染色体の再編成を起こす5).2つのRTS1配列が近傍で逆位にある場合は,誤った鋳型での再生複製は逆進し,姉妹染色体はRTS1配列において逆位融合する.生じた逆位融合した染色体は,セントロメアをもたない染色体(acentric chromosome,無セントロメア染色体)と二連セントロメア染色体となる(図2b).セントロメアは正確な染色体の分配に必須であるため,これら構造的に異常な染色体は染色体の分配に支障をきたし,一倍体である分裂酵母は生存に必須な遺伝子を失い死にいたる5).筆者らは,すでに,この相同組換えによる誤った鋳型の選択の機構について報告している5,6).
2つのRTS1配列が近傍において逆位にあり,なおかつ,そのあいだに逆位反復配列のある場合,染色体の逆位融合には2つの機構が考えられる.1つ目は,複製の再始動のときの相同組換えによる誤った鋳型の選択,2つ目は,再生複製のUターンである.そのどちらがおもな染色体の逆位融合の機構であるかを区別するため,サザンブロッティング法において反復配列でない2つのRTS1配列のあいだの配列をプローブとして解析した.その結果,再生複製のUターンが染色体の逆位融合のおもな機構であることが判明した(図2).
筆者らの研究は,元来の正しい鋳型DNAからの複製フォークの再始動は染色体の再編成をひき起こすことを明らかにした.再生複製は反復配列のあいだで鋳型DNAを変えながらDNA合成を進めていく.おそらく,新生DNA鎖は一時的な相補DNA鎖からの剥離と相補DNA鎖への再会合をくり返しているものと考えられる.染色体の逆位融合のモデルは以下のとおりである(図3).逆位反復配列における再生複製の誤った鋳型の選択が,反復配列の中央において“閉じられたY構造”を形成する.これに端を発して2つの逆位融合した染色体が生じる.その詳細な機構は今後の課題である.事実,二連セントロメア染色体の形成と同時に,無セントロメア染色体も形成された.近年,再生複製は短い相同配列のあいだでの重複や欠失も高頻度で起こすことが報告された8,9).ヒトの染色体の脆弱部位は複製開始点が非常に疎である1,2).複製フォークが停止するような環境では,複製開始点の活性化だけでは複製の完了はむずかしく,停止した複製フォークの再始動が必要である.その結果,再生複製の誤りがちな性質が染色体の再編成を起こし,遺伝子増幅と遺伝子欠失を促すと考えられる.
逆位融合した染色体は不安定である4,5).哺乳類のがん細胞やヒトのゲノム障害細胞では,逆位融合の痕跡が染色体の逆位重複末端欠失として観察される3,10).逆位重複した二連セントロメア染色体は染色体の分配のとき染色体の架橋という問題をかかえるが,染色体の分断は異常な組換えと複製をひき起こし二連セントロメア染色体を再形成する4,5)(これをbreakage-fusion-bridgeサイクルとよぶ).がん細胞ではこのサイクルを介する染色体の分断と重複が遺伝子増幅を促す4).染色体の分断ののち,セントロメアの1つの喪失と切断末端におけるテロメアの付加がその染色体を安定化する3,4,10).これが逆位重複末端の欠失である3,10).多くの逆位重複は複製フォークが前方または後方にスキップした痕跡をともなうため,逆位反復配列における複製の鋳型の切換えが原因と考えられている3,10).筆者らの発見は,ヒトのゲノム障害やがんで観察される染色体の逆位重複末端の欠失の発生機構をうまく説明するものと考えられる.
今後は,複製がどのように相同組換えを介し再生するのか? 再生複製はどのようにしてDNAを複製するのか? 再生複製はなぜ誤りがちなのか? 再生複製にどのヘリカーゼ,どのDNAポリメラーゼが用いられているのか? を明らかにしていきたい.
略歴:1995年 総合研究大学院大学大学院生命科学研究科 修了,1996年 理化学研究所遺伝生化学研究室 ポスドクを経て,2004年より英国Sussex大学 ポスドク.
研究テーマ:誤りがちな複製によるゲノム恒常性の破綻の分子機構.
抱負:誤りがちな複製によるゲノム恒常性の破綻の分子機構を解明することからの創薬.
© 2013 水野 健一 Licensed under CC 表示 2.1 日本
(英国Sussex大学Genome Damage and Stability Centre)
email:水野健一
DOI: 10.7875/first.author.2013.001
Recombination-restarted replication makes inverted chromosome fusions at inverted repeats.
Ken’Ichi Mizuno, Izumi Miyabe, Stephanie A. Schalbetter, Antony M. Carr, Johanne M. Murray
Nature, 493, 246-249 (2013)
要 約
DNAの複製は増殖している細胞にとり必須であり,通常の状態において複製装置は非常に精度の高いコピーマシンである.複製の効率あるいは精度の低下はゲノムの安定性に影響を及ぼす.そして,ゲノムの恒常性の破綻はがん化やヒトゲノムの構造変化に起因するゲノム障害につながる.しかし,複製の停止によるゲノム恒常性の破綻の分子機構については明らかでない部分が多い.複製の停止からの複製の再開には相同組換えの必要とされる場合がある.筆者らは,再開した複製の性質を分子レベルにおいて解析するため,分裂酵母を用い部位特異的に複製を停止したのち相同組換えにより複製を再開する系を開発した.そして,この再生複製は,通常の複製とは異なり誤りがちであることを明らかにした.再生複製は逆位反復配列において高い頻度で染色体の再編成をひき起こした.この再生複製の誤りがちな性質は遺伝子の増幅や欠失の起因となり,がん化やゲノム障害の原因になると考えられた.
はじめに
DNAの複製は増殖している細胞にとり欠かせない細胞周期の一過程であり,複製の適切な制御はゲノムの安定性にとり必須である.通常は,複製フォークが他方からくる別の複製フォークと融合することで複製は完了する.がん化の初期段階では,原がん遺伝子の活性化またはがん抑制遺伝子の不活性化により,細胞周期の制御が撹乱される.それがDNA合成の効率を低下させ,しばしば複製フォークを停止させる.このような複製ストレスのもとでは休眠している複製開始点の活性化が複製完了の手段であるが,複製開始点はゲノムにおいて均一には分布していない.複製開始点のほとんど存在しない染色体の脆弱部位では1,2),停止した複製フォークの再始動が複製完了の手段となる.染色体の脆弱部位をはじめとするさまざまな部位は,複製ストレスのもとで染色体の再編成,遺伝子増幅,遺伝子欠失をひき起こす1,3).
複製フォークはいろいろな要因により停止する.転写装置あるいはDNAに強く結合したタンパク質との衝突,基質であるdNTPの枯渇,DNA損傷による進行の阻害などである.停止した複製フォークは大きく3つに分類できる4)(図1).休止複製フォーク(paused fork),剥離複製フォーク(collapsed fork),切断複製フォーク(broken fork)である.休止複製フォークとは,複製フォークの進行は停止しているがDNAフォークにレプリソームが滞在したままの状態をいう(図1b).複製フォークの進行を停止させた障害が取り除かれるまで,S期チェックポイントによりレプリソームはDNAフォークにおいて保護される.障害が取り除かれたのち,レプリソームはDNAフォークに会合しているので,すぐにDNA合成を再開できる.休止複製フォークにおける複製の再開に相同組換えは必要ない.一方,剥離複製フォークと切断複製フォークは複製の再開に相同組換えを必要とする.剥離複製フォークとは,レプリソームがDNAフォークから解離した状態をいう4)(図1c).DNAフォークがあらわになるため,動的な構造変化が頻繁に起こりやすくなる.DNAフォークの後退が一時的にチキンフット状のDNA構造を形成する.その構造において露出した新生一本鎖DNAに相同組換えタンパク質が結合する.切断複製フォークとは,DNA鎖が切断され複製フォーク自体が壊れた状態をいう(図1d).複製フォークがニック(切れ目)のある二本鎖DNAに直面したときに起こる.このとき,レプリソームはDNAフォークから解離する.DNA切断末端が加工されることによりあらわになった一本鎖DNAに相同組換えタンパク質が結合する.一本鎖DNAと相同組換えタンパク質との複合体が二本鎖の鋳型DNAに侵入し相補DNA鎖と会合する.その会合の末端から新生DNA鎖が伸長することによりDNA合成は再開する.以下,相同組換えにより複製フォークが再始動した複製を再生複製とよぶ.
分裂酵母の接合型遺伝子座の近傍には複製フォークの停止配列であるRTS1配列が存在する.RTS1配列は方向性をもって複製フォークを停止させる.DNA結合タンパク質Rtf1はRTS1配列の複製フォーク停止活性に必要である.RTS1配列において停止した複製フォークは剥離複製フォークとなり複製の再開には相同組換えが必要となる5,6).複製フォークの停止と再始動の分子機構と,その再始動した複製フォークの性質を解明するため,RTS1配列を用いた部位特異的な複製フォークの停止系を開発した5-7).操作が安易な分裂酵母を使ったこの系の利点は,rtf1+遺伝子の転写発現の制御により,RTS1配列の複製フォーク停止活性を自由自在に制御できることである.これが,複製過程の分子レベルでの解析を可能にした.
1.再生複製は誤りがちで逆位反復配列において姉妹染色体を逆位融合する
1つのRTS1配列を用いてRTS1配列から再始動した複製フォークが逆位反復配列を複製するような系を構築した.用いた逆位反復配列の中央に14塩基対の介入配列を挿入した.この逆位反復配列はRTS1配列とはまったく異なる配列とした.RTS1配列のセントロメア側にある強力な複製開始点からの複製フォークは,RTS1配列において停止する.相同組換えを介し元来の鋳型DNA(相補DNA鎖)を用いて,停止した複製フォークは再始動する.
RTS1配列の下流に逆位反復配列が存在する場合,再生複製は染色体の再編成をひき起こした.複製フォークがRTS1配列において停止しない場合,この染色体の再編成は起こらなかった.この染色体の再編成は再生複製に依存した.また,この染色体の再編成は反復配列の長さに依存した.しかし,RTS1配列の下流に反復配列が存在しない場合は,再生複製はサザンブロッティング法により検出の可能なレベルの染色体の再編成を起こさなかった.この再編成した染色体の構造は,再生複製が逆位反復配列の中央において複製の方向を転換(Uターン)して逆位融合した構造に相当した(図2a).この逆位融合した染色体は余分なセントロメアをもつ染色体(dicentric chromosome,二連セントロメア染色体)であった(図2b).
2.逆位融合した染色体の形成はDNA二次構造の形成能に依存する
逆位反復配列における介入配列の長さを変えて,染色体の再編成に対するその効果を観察した.介入配列が短いと逆位反復配列はDNA二次構造を形成しやすくなる.逆に,介入配列が長いとDNA二次構造を形成しにくい.予想されたとおり,介入配列が短いと染色体の逆位融合が頻発した.一方,介入配列が長いと染色体の逆位融合の頻度は低下した.しかしながら,それでもサザンブロッティング法により検出の可能な頻度であった.したがって,再生複製による染色体の再編成は,DNA二次構造を形成しやすい配列においてより頻繁に起こること,しかし,反復配列の相同性も染色体の再編成に寄与していることが明らかになった.
3.複製フォークの進行距離にしたがい再生複製の精度は改善する
RTS1配列と逆位反復配列とのあいだに無関係なDNA配列を挿入し,染色体の再編成に対するRTS1配列から逆位反復配列までの距離の効果を観察した.逆位反復配列がRTS1配列に隣接しているとき,染色体の逆位融合の頻度は最高値を示した.距離を長くすると,その頻度は徐々に低下した.しかし,その距離が2400塩基対長であってもサザンブロッティング法により検出の可能な頻度であった.
4.再生複製のUターンが染色体の逆位融合のおもな機構である
2つのRTS1配列が近傍に存在すると,その相同な配列を鋳型として相同組換えの起こることがある.この誤った鋳型の選択により非対立遺伝子座の相同組換えが生じ染色体の再編成を起こす5).2つのRTS1配列が近傍で逆位にある場合は,誤った鋳型での再生複製は逆進し,姉妹染色体はRTS1配列において逆位融合する.生じた逆位融合した染色体は,セントロメアをもたない染色体(acentric chromosome,無セントロメア染色体)と二連セントロメア染色体となる(図2b).セントロメアは正確な染色体の分配に必須であるため,これら構造的に異常な染色体は染色体の分配に支障をきたし,一倍体である分裂酵母は生存に必須な遺伝子を失い死にいたる5).筆者らは,すでに,この相同組換えによる誤った鋳型の選択の機構について報告している5,6).
2つのRTS1配列が近傍において逆位にあり,なおかつ,そのあいだに逆位反復配列のある場合,染色体の逆位融合には2つの機構が考えられる.1つ目は,複製の再始動のときの相同組換えによる誤った鋳型の選択,2つ目は,再生複製のUターンである.そのどちらがおもな染色体の逆位融合の機構であるかを区別するため,サザンブロッティング法において反復配列でない2つのRTS1配列のあいだの配列をプローブとして解析した.その結果,再生複製のUターンが染色体の逆位融合のおもな機構であることが判明した(図2).
おわりに
筆者らの研究は,元来の正しい鋳型DNAからの複製フォークの再始動は染色体の再編成をひき起こすことを明らかにした.再生複製は反復配列のあいだで鋳型DNAを変えながらDNA合成を進めていく.おそらく,新生DNA鎖は一時的な相補DNA鎖からの剥離と相補DNA鎖への再会合をくり返しているものと考えられる.染色体の逆位融合のモデルは以下のとおりである(図3).逆位反復配列における再生複製の誤った鋳型の選択が,反復配列の中央において“閉じられたY構造”を形成する.これに端を発して2つの逆位融合した染色体が生じる.その詳細な機構は今後の課題である.事実,二連セントロメア染色体の形成と同時に,無セントロメア染色体も形成された.近年,再生複製は短い相同配列のあいだでの重複や欠失も高頻度で起こすことが報告された8,9).ヒトの染色体の脆弱部位は複製開始点が非常に疎である1,2).複製フォークが停止するような環境では,複製開始点の活性化だけでは複製の完了はむずかしく,停止した複製フォークの再始動が必要である.その結果,再生複製の誤りがちな性質が染色体の再編成を起こし,遺伝子増幅と遺伝子欠失を促すと考えられる.
逆位融合した染色体は不安定である4,5).哺乳類のがん細胞やヒトのゲノム障害細胞では,逆位融合の痕跡が染色体の逆位重複末端欠失として観察される3,10).逆位重複した二連セントロメア染色体は染色体の分配のとき染色体の架橋という問題をかかえるが,染色体の分断は異常な組換えと複製をひき起こし二連セントロメア染色体を再形成する4,5)(これをbreakage-fusion-bridgeサイクルとよぶ).がん細胞ではこのサイクルを介する染色体の分断と重複が遺伝子増幅を促す4).染色体の分断ののち,セントロメアの1つの喪失と切断末端におけるテロメアの付加がその染色体を安定化する3,4,10).これが逆位重複末端の欠失である3,10).多くの逆位重複は複製フォークが前方または後方にスキップした痕跡をともなうため,逆位反復配列における複製の鋳型の切換えが原因と考えられている3,10).筆者らの発見は,ヒトのゲノム障害やがんで観察される染色体の逆位重複末端の欠失の発生機構をうまく説明するものと考えられる.
今後は,複製がどのように相同組換えを介し再生するのか? 再生複製はどのようにしてDNAを複製するのか? 再生複製はなぜ誤りがちなのか? 再生複製にどのヘリカーゼ,どのDNAポリメラーゼが用いられているのか? を明らかにしていきたい.
文 献
- Arlt, M. F., Wilson, T. E. & Glover, T. W.: Replication stress and mechanisms of CNV formation. Curr. Opin. Genet. Dev., 22, 204-210 (2012)[PubMed]
- Letessier, A., Millot, G. A., Koundrioukoff, S. et al.: Cell-type-specific replication initiation programs set fragility of the FRA3B fragile site. Nature, 470, 120-123 (2011)[PubMed]
- Liu, P., Carvalho, C. M., Hastings, P. J. et al.: Mechanisms for recurrent and complex human genomic rearrangements. Curr. Opin. Genet. Dev., 22, 211-220 (2012)[PubMed]
- Carr, A. M., Paek, A. L. & Weinert, T.: DNA replication: failures and inverted fusions. Semin. Cell Dev. Biol., 22, 866-874 (2011)[PubMed]
- Mizuno, K., Lambert, S., Baldacci, G. et al.: Nearby inverted repeats fuse to generate acentric and dicentric palindromic chromosomes by a replication template exchange mechanism. Genes Dev., 23, 2876-2886 (2009)[PubMed]
- Lambert, S., Mizuno, K., Blaisonneau, J. et al.: Homologous recombination restarts blocked replication forks at the expense of genome rearrangements by template exchange. Mol. Cell, 39, 346-359 (2010)[PubMed]
- Lambert, S., Watson, A., Sheedy, D. M. et al.: Gross chromosomal rearrangements and elevated recombination at an inducible site-specific replication fork barrier. Cell, 121, 689-702 (2005)[PubMed]
- Iraqui, I., Chekkal, Y., Jmari, N. et al.: Recovery of arrested replication forks by homologous recombination is error-prone. PLoS Genet., 8, e1002976 (2012)[PubMed]
- Malkova, A. & Haber J. E.: Mutations arising during repair of chromosome breaks. Annu. Rev. Genet., 46, 455-73 (2012)[PubMed]
- Carvalho, C. M., Ramocki, M. B., Pehlivan, D. et al.: Inverted genomic segments and complex triplication rearrangements are mediated by inverted repeats in the human genome. Nat. Genet., 43, 1074-1081 (2011)[PubMed]
著者プロフィール
略歴:1995年 総合研究大学院大学大学院生命科学研究科 修了,1996年 理化学研究所遺伝生化学研究室 ポスドクを経て,2004年より英国Sussex大学 ポスドク.
研究テーマ:誤りがちな複製によるゲノム恒常性の破綻の分子機構.
抱負:誤りがちな複製によるゲノム恒常性の破綻の分子機構を解明することからの創薬.
© 2013 水野 健一 Licensed under CC 表示 2.1 日本