標的アナログと結合したCRISPR-Cas系のCmr複合体の結晶構造
沼田倫征・大澤拓生
(産業技術総合研究所バイオメディカル研究部門 RNAプロセシング研究特別チーム)
email:沼田倫征
DOI: 10.7875/first.author.2015.061
Crystal structure of the CRISPR-Cas RNA silencing Cmr complex bound to a target analog.
Takuo Osawa, Hideko Inanaga, Chikara Sato, Tomoyuki Numata
Molecular Cell, 58, 418-430 (2015)
原核生物のもつ獲得免疫機構CRISPR-Cas系では,エフェクター複合体がウイルスのもつ核酸を分解しその増殖を抑制する.エフェクター複合体のひとつであるCmr複合体はCmr1~Cmr6とcrRNAからなりウイルスのもつRNAを切断するが,そのしくみは不明であった.今回,筆者らは,標的アナログと結合したCmr複合体の結晶構造を分解能2.1Åで解析した.Cmr複合体は,Cmr4を3分子,Cmr5を2分子,それ以外のサブユニットを1分子ずつ含んでいた.crRNAのタグ配列はCmr3により認識されていた.また,標的アナログはcrRNAのガイド配列と2本鎖を形成していたが,Cmr4が6塩基周期で2本鎖のあいだに入り込んでいた.このため,Cmr複合体はcrRNAの5’側から一定の距離にある塩基対合の形成を阻害し,標的RNAのかたちを3カ所で不安定化させそのリン酸結合を切断することが判明した.Cmr複合体が標的となる核酸を捕捉するしくみはDNAを標的とするCascadeと類似しており,両者が共通の祖先から進化したと考えられた.
現在,CRISPR-Cas系はさまざまな生物種においてゲノム編集ツールとして利用されており注目をあびている.自然界ではCRISPR-Cas系は原核生物において獲得免疫系として機能している.原核生物はウイルスにはじめて感染すると,その核酸の一部をゲノムにあるCRISPR領域に組み込み,ウイルスの感染履歴をゲノムに記録する.同じウイルスに再感染するとCRISPR領域からcrRNAという小分子RNAが合成され,Casタンパク質と会合しエフェクター複合体を形成する.crRNAはウイルスの核酸と相補的な配列をもつので,エフェクター複合体はこれを認識して除去できる.エフェクター複合体は関与するCasタンパク質の種類により3つのタイプに分類される.タイプIおよびタイプIIのエフェクター複合体はウイルスのもつDNAを標的にするのに対し,タイプIIIのエフェクター複合体はウイルスのもつRNAを切断する.これまで,タイプIおよびタイプIIのエフェクター複合体の結晶構造が決定され,これらのエフェクター複合体がDNAを特異的に分解するしくみが解明されつつある1,2).一方,タイプIIIのエフェクター複合体についてはクライオ電子顕微鏡により構造が決定されているが3-5),その結晶構造は解析されていなかった.
タイプIIIエフェクター複合体はさらにタイプIII-AのCsm複合体とタイプIII-BのCmr複合体とに分かれる.アーキアPyrococcus furiosusに由来するCmr複合体は6種類のCasタンパク質Cmr1~Cmr6とcrRNAから構成される6).crRNAは5’側に8塩基のタグ配列,つづいて,31塩基あるいは37塩基のウイルスの核酸に由来するガイド配列をもち,Cmr複合体はガイド配列と相補的なRNAを捕捉し切断する6).質量分析法およびクライオ電子顕微鏡による解析から,Cmr複合体は複数個のCmr4およびCmr5を含み,一方,ほかのサブユニットは1分子ずつ含むことが示唆された3,4).また,生化学的な解析から,Cmr複合体は標的となるRNAを複数の部位で切断することが明らかにされ,Cmr4はRNAの切断にかかわる触媒サブユニットである可能性が指摘された3,7,8).しかしながら,Cmr複合体の結晶構造は決定されておらず,標的となるRNAを切断するしくみはほとんど理解されていなかった.
Cmr複合体の作動原理を解明するため,その結晶構造の決定をめざした.まず,個々のCmrサブユニットを調製してCmr複合体の再構成を試みたが,発現が困難なサブユニットもあり結晶化に適したCmr複合体の再構成にはいたらなかった.そこで,Cmrタンパク質をコードする遺伝子はオペロンを形成していることに注目し,発現ベクターにオペロン遺伝子をクローニングし相互作用しあうサブユニットどうしの大腸菌における共発現について検討した.その結果,Cmr2-Cmr3およびCmr4-Cmr5-Cmr6をそれぞれサブ複合体として(Cmr1は単独で)調製した.得られたP. furiosusに由来するCmrタンパク質を用いて複合体を再構成し標的RNAの切断活性を確認したが,Cmr複合体の結晶を得ることはできなかった.アーキアArchaeoglobus fulgidusに由来するCmr複合体についても検討したが,やはりCmr複合体の結晶化にはいたらなかった.
P. furiosusに由来するCmrタンパク質とA. fulgidusに由来するCmrタンパク質とのあいだにはアミノ酸配列の相同性(27.1~42.2%)がある.そこで,Cmr複合体の異なる種のあいだでの再構成について検討した.その結果,P. furiosusに由来するCmr2-Cmr3サブ複合体とA. fulgidusに由来するCmr4-Cmr5-Cmr6サブ複合体は相互作用することが判明した.さらに,この複合体はP. furiosusに由来する39塩基のcrRNAと会合し,Cmr1がなくてもガイド配列と相補的な標的RNAを切断することが明らかにされた.このキメラCmr複合体の結晶化条件をスクリーニングし,標的アナログとして標的となるRNAと同じ配列をもつ31塩基のDNAと結合したキメラCmr複合体の結晶を得た.X線回折データを測定し,分子置換法と単波長異常分散法の組合せにより結晶の位相を決定して,分解能2.1Åで結晶構造を決定した(PDB ID:3X1L).
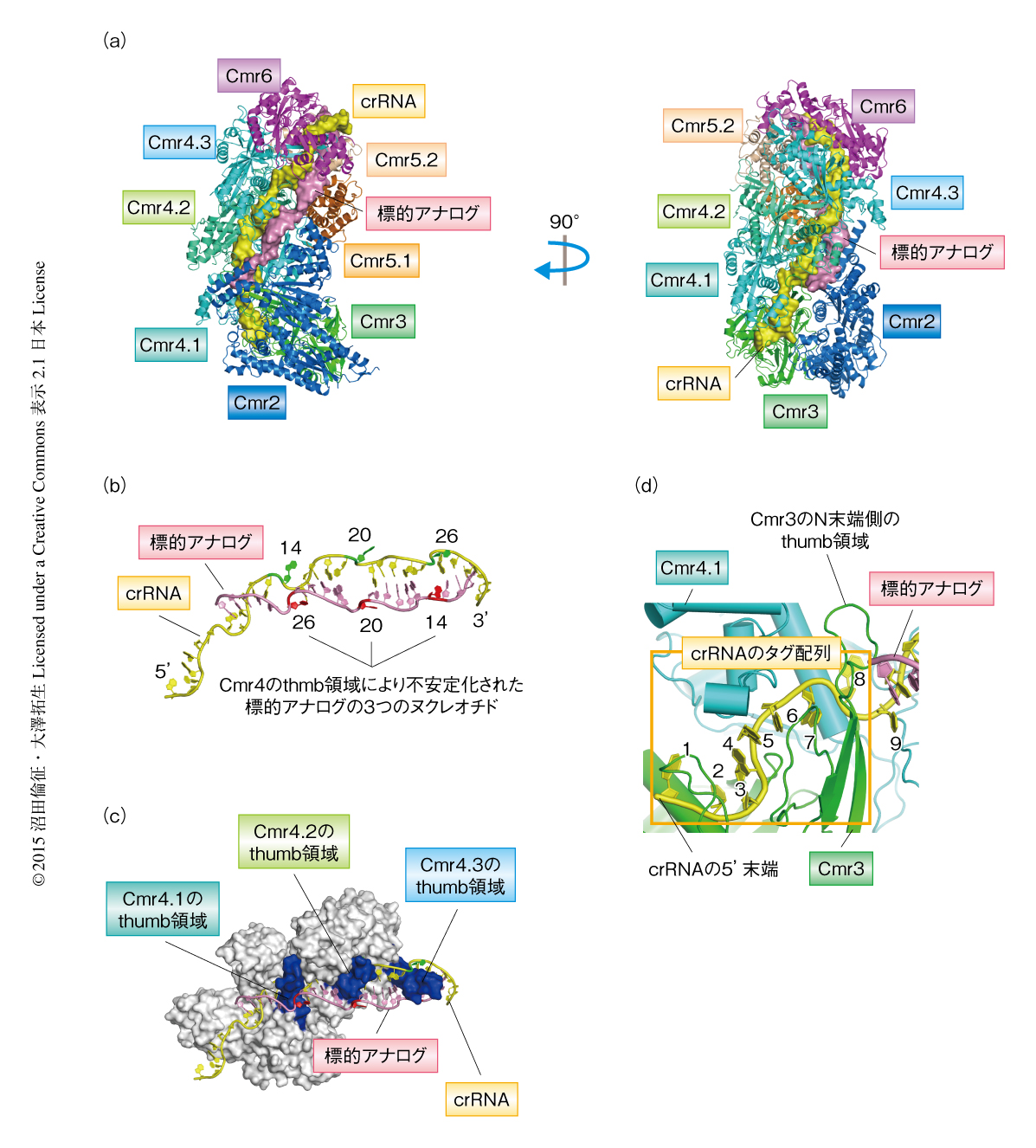
キメラCmr複合体は,Cmr4を3分子(Cmr4.1~Cmr4.3),Cmr5を2分子(Cmr5.1およびCmr5.2),それ以外のサブユニットについては1分子ずつ含むことが判明した(図1a).Cmr2-Cmr3サブ複合体はキメラCmr複合体の基底部をなし,crRNAの5’側に存在するタグ配列を特異的に認識していた.一方,Cmr4-Cmr5-Cmr6サブ複合体はキメラCmr複合体の上層部を形成しており,そこでは3分子のCmr4が規則的に積み重なって2分子のCmr5とともにらせん状の溝を形成していた.crRNAのガイド配列はこの溝にそって配置しており,標的アナログを配列に相補的に捕捉していた.
crRNAのガイド配列はおもに3分子のCmr4と相互作用していた.RRMフォールドをもつCmr4の構造は“右手”のかたちに類似しており,palm領域,finger領域,thumb領域に分割することができる.crRNAのガイド配列と標的アナログは2本鎖を形成していたが,一般的な核酸の二重らせん構造とは異なり,ほどけたリボン状のかたちをしていた(図1b).これは,Cmr4のthumb領域が6塩基対ごとに2本鎖のあいだに入り込み,塩基対の形成を周期的に阻害しているためであった(図1c).キメラCmr複合体は3分子のCmr4を含むため,crRNAのガイド配列と標的アナログの2本鎖は3カ所で塩基対の形成が阻害されていた.その結果,crRNAの5’側から数えて14番目,20番目,26番目(標的アナログの26番目,20番目,14番目)のヌクレオチドの構造が大きくひずんでいた.
これまでの実験から,Cmr複合体は標的となるRNAを複数の部位で切断することが指摘されていた3,7,8).そこで,Cmr複合体はヌクレオチドのかたちが不安定化していた標的RNAの14番目,20番目,26番目の3’側のリン酸結合を切断するのではないかと仮説をたてた.標的RNAのこれら3カ所のリボースを2’-デオキシ化した誘導体を作製しその分解のパターンを解析した結果,推定されたように,Cmr複合体は6塩基周期でこれら3カ所を切断することがわかった.加水分解されるこれら3カ所のリン酸結合の近くにはCmr4において高度に保存されているAsp31が配置されており,変異体の解析の結果,Asp31が標的RNAの切断にかかわることが判明した.
2’-デオキシリボースを含むRNA誘導体を用いた解析から,Cmr複合体によるRNAの切断にはリボースの2’-OH基が不可欠であることが判明した.したがって,標的RNAはRNase Aなどに代表される酸塩基触媒機構により切断されることが示唆された.これは,Cmr複合体による分解産物は3’リン酸末端(あるいは,2’,3’環状リン酸末端)および5’-OH末端をもつというこれまでの結果と一致した6).Cmr複合体の構造解析から,Cmr4のAsp31は一般酸触媒として機能し,分解産物の5’末端にプロトンを供与することが示唆された.一方,加水分解されるリン酸結合の周辺にはAsp31以外に保存されたアミノ酸残基は存在せず,リボースの2’-OH基の脱プロトン化を触媒する一般塩基触媒については明らかにされなかった.標的となるRNAの詳細な加水分解機構の解明は今後の課題である.
crRNAの5’側に存在するタグ配列は水素結合を介してCmr3およびCmr3に隣接したCmr4(Cmr4.1)と相互作用していた(図1d).タグ配列の8つのヌクレオチドのうち,2番目のウリジンはCmr3により塩基に特異的に認識されていた.このウリジンをアデノシンと置換したcrRNAを含むCmr複合体はRNA切断活性を消失したことから,この相互作用が重要であることが確認された.また,タグ配列の5’末端のOH基はCmr3のポケットに収容され,その主鎖と水素結合していた.これまでの研究から,crRNAの5’末端にリン酸基を付加するとCmr複合体のRNA切断活性は消失することが知られていた8).これは,crRNAの5’末端へのリン酸基の付加によりCmr3とのあいだに立体障害が起こり,crRNAがCmr複合体に取り込まれなくなった結果と考えられた.以上の結果より,crRNAのタグ配列は塩基に特異的に認識され,Cmr複合体において固定されていることが明らかにされた.
Cmr3はN末端側およびC末端側にそれぞれRRMフォールド様のドメインをもち,いずれもCmr4の構造とよく類似していたが,finger領域は欠落しておりpalm領域およびthumb領域から構成されていた.Cmr3の2つのthumb領域はcrRNAのタグ配列と強固に相互作用していた.興味深いことに,Cmr3のN末端側のthumb領域とCmr4のthumb領域は機能的によく類似しており,Cmr3のN末端側のthumb領域との相互作用によりcrRNAのタグ配列の8番目のヌクレオチドの構造は大きくひずみCmr4.1のポケットに配置されていた(図1d).その結果,crRNAの9番目のヌクレオチド(ガイド配列の最初のヌクレオチド)が標的RNAと塩基対を形成する環境が整い,crRNAのガイド配列と標的RNAの形成する2本鎖の開始位置が厳密に規定されていた.この構造的な特徴およびCmr複合体におけるcrRNAのタグ配列の固定により,のちに述べるように,Cmr複合体はcrRNAの5’側から一定の距離にある標的RNAのリン酸結合を長さに特異的に切断することが明らかにされた.
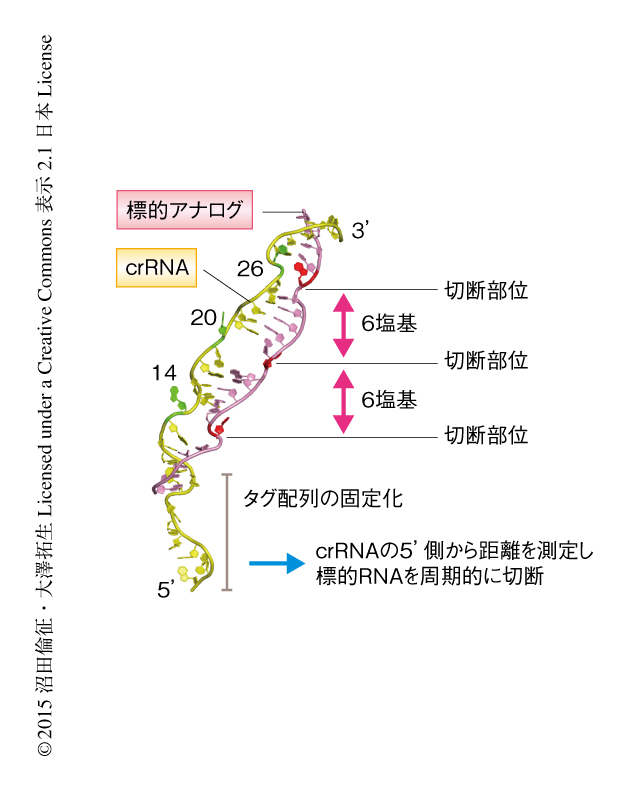
今回の解析から,crRNAのタグ配列がCmr複合体において固定されていることが判明した.このため,Cmr複合体はcrRNAの5’側から数えて一定の距離にある14番目,20番目,26番目の塩基対の形成を阻害する.その結果,これら3カ所において標的RNAの構造が大きくひずんでCmr複合体の活性部位に配置され,6塩基周期で切断されるしくみが明らかにされた(図2).
キメラCmr複合体には3分子のCmr4が含まれており,触媒残基であるAsp31は約24Åの間隔で配置されていた.これは6塩基の核酸に相当する長さであった.Cmr複合体から単離されたcrRNAには39塩基あるいは45塩基の長さの異なる2種類が存在する6).したがって,39塩基のcrRNAを含むCmr複合体は3分子のCmr4を,一方,45塩基のcrRNAを含むCmr複合体は4分子のCmr4をもち,それぞれ,標的RNAを3カ所または4カ所で切断すると考えられた.実際,45塩基のcrRNAを含むCmr複合体は標的となるRNAを4カ所で切断する7,8).
タイプIII-Aエフェクター複合体のCsm複合体は5種類のCasタンパク質Csm1~Csm5とcrRNAから構成される.Csm3の構造はCmr4と類似しており9),thumb領域に相当する領域(結晶構造では構造をとっていなかった)をもつことが示唆されるとともに,触媒反応にかかわるCmr4のAspはCsm3においても保存されていた10).また,Csm複合体のクライオ電子顕微鏡による解析から,複数のCsm3が規則的に積み重なってらせん状の溝を形成していることが明らかにされていた5).さらに,Csm4の構造はCmr3に類似していたことから9),Csm4がcrRNAのタグ配列の認識にかかわることが示唆された.したがって,Csm複合体も,タイプIII-Bエフェクター複合体であるCmr複合体とまったく同じしくみにより標的となるRNAの切断部位を選びだすと考えられ,標的となるRNAを分解するしくみはタイプIIIエフェクター複合体の種類にかかわらず共通することが示唆された.
2014年に,1本鎖DNAと結合したCascadeの結晶構造が報告されている1).タイプIエフェクター複合体であるCascadeは,1:2:1:1:6のストイキオメトリーで会合した5種類のCasタンパク質Cse1,Cse2,Cas5,Cas6,Cas7とcrRNAから構成される.立体構造の比較から,Cmr4とCas7の構造,また,Cmr3とCas5の構造がそれぞれ類似していることがわかった.また,Cmr複合体の場合と同様に,CascadeではCas5がcrRNAのタグ配列を認識し,6分子のCas7がcrRNAのガイド配列との相互作用に関与している1).このため,CascadeとCmr複合体においてこれらサブユニットの空間的な配置は類似しており,その結果,タイプIエフェクター複合体とタイプIIIエフェクター複合体はよく似た構造をとることがわかった.さらに興味深いことに,Cas7にもthumb領域が存在し,Cascadeもガイド配列と標的DNAとの塩基対の形成を6塩基周期で阻害する1).その結果,CascadeおよびCmr複合体において,ガイド配列と標的となる核酸の2本鎖はほどけたリボン状の構造をとる.リボン状の構造の形成はこれらエフェクター複合体が標的となる核酸と効率よく結合するうえで非常に重要であると考えられる.CascadeにDNA切断活性はなく,標的となるDNAを捕捉したのち,ヌクレアーゼ/ヘリカーゼであるCas3がリクルートされ標的となるDNAを分解する.
このように,CascadeとCmr複合体では標的となる核酸と結合するしくみはよく類似しているが,それを切断する機構はまったく異なっていた.したがって,タイプIエフェクター複合体とタイプIIIエフェクター複合体は共通の祖先から派生したと考えられた.今後,タイプIエフェクター複合体およびタイプIIIエフェクター複合体のさらなる解析により,それぞれの複合体が標的となる核酸を分解するしくみの詳細が明らかにされるとともに,生物がDNAとRNAの違いを区別してそれぞれに対応するエフェクター複合体をつくりだした進化戦略に対し,科学的な考察が可能になると期待される.
略歴:2003年 九州大学大学院生物資源環境科学府博士課程 修了,同年 同 博士研究員,2004年 東京工業大学大学院生命理工学研究科 博士研究員,2006年 科学技術振興機構 さきがけ研究者,2007年 産業技術総合研究所生物機能工学研究部門 研究員を経て,2012年より産業技術総合研究所バイオメディカル研究部門 主任研究員.
研究テーマ:RNAのかかわる生体高分子複合体の機能構造解析.
抱負:RNAやRNAとタンパク質からなる分子装置の作動原理を,原子分解能のレベルで明らかにしていきたい.
大澤 拓生(Takuo Osawa)
微生物化学研究所 博士研究員.
© 2015 沼田倫征・大澤拓生 Licensed under CC 表示 2.1 日本
(産業技術総合研究所バイオメディカル研究部門 RNAプロセシング研究特別チーム)
email:沼田倫征
DOI: 10.7875/first.author.2015.061
Crystal structure of the CRISPR-Cas RNA silencing Cmr complex bound to a target analog.
Takuo Osawa, Hideko Inanaga, Chikara Sato, Tomoyuki Numata
Molecular Cell, 58, 418-430 (2015)
要 約
原核生物のもつ獲得免疫機構CRISPR-Cas系では,エフェクター複合体がウイルスのもつ核酸を分解しその増殖を抑制する.エフェクター複合体のひとつであるCmr複合体はCmr1~Cmr6とcrRNAからなりウイルスのもつRNAを切断するが,そのしくみは不明であった.今回,筆者らは,標的アナログと結合したCmr複合体の結晶構造を分解能2.1Åで解析した.Cmr複合体は,Cmr4を3分子,Cmr5を2分子,それ以外のサブユニットを1分子ずつ含んでいた.crRNAのタグ配列はCmr3により認識されていた.また,標的アナログはcrRNAのガイド配列と2本鎖を形成していたが,Cmr4が6塩基周期で2本鎖のあいだに入り込んでいた.このため,Cmr複合体はcrRNAの5’側から一定の距離にある塩基対合の形成を阻害し,標的RNAのかたちを3カ所で不安定化させそのリン酸結合を切断することが判明した.Cmr複合体が標的となる核酸を捕捉するしくみはDNAを標的とするCascadeと類似しており,両者が共通の祖先から進化したと考えられた.
はじめに
現在,CRISPR-Cas系はさまざまな生物種においてゲノム編集ツールとして利用されており注目をあびている.自然界ではCRISPR-Cas系は原核生物において獲得免疫系として機能している.原核生物はウイルスにはじめて感染すると,その核酸の一部をゲノムにあるCRISPR領域に組み込み,ウイルスの感染履歴をゲノムに記録する.同じウイルスに再感染するとCRISPR領域からcrRNAという小分子RNAが合成され,Casタンパク質と会合しエフェクター複合体を形成する.crRNAはウイルスの核酸と相補的な配列をもつので,エフェクター複合体はこれを認識して除去できる.エフェクター複合体は関与するCasタンパク質の種類により3つのタイプに分類される.タイプIおよびタイプIIのエフェクター複合体はウイルスのもつDNAを標的にするのに対し,タイプIIIのエフェクター複合体はウイルスのもつRNAを切断する.これまで,タイプIおよびタイプIIのエフェクター複合体の結晶構造が決定され,これらのエフェクター複合体がDNAを特異的に分解するしくみが解明されつつある1,2).一方,タイプIIIのエフェクター複合体についてはクライオ電子顕微鏡により構造が決定されているが3-5),その結晶構造は解析されていなかった.
タイプIIIエフェクター複合体はさらにタイプIII-AのCsm複合体とタイプIII-BのCmr複合体とに分かれる.アーキアPyrococcus furiosusに由来するCmr複合体は6種類のCasタンパク質Cmr1~Cmr6とcrRNAから構成される6).crRNAは5’側に8塩基のタグ配列,つづいて,31塩基あるいは37塩基のウイルスの核酸に由来するガイド配列をもち,Cmr複合体はガイド配列と相補的なRNAを捕捉し切断する6).質量分析法およびクライオ電子顕微鏡による解析から,Cmr複合体は複数個のCmr4およびCmr5を含み,一方,ほかのサブユニットは1分子ずつ含むことが示唆された3,4).また,生化学的な解析から,Cmr複合体は標的となるRNAを複数の部位で切断することが明らかにされ,Cmr4はRNAの切断にかかわる触媒サブユニットである可能性が指摘された3,7,8).しかしながら,Cmr複合体の結晶構造は決定されておらず,標的となるRNAを切断するしくみはほとんど理解されていなかった.
1.Cmr複合体の再構成および結晶構造の決定
Cmr複合体の作動原理を解明するため,その結晶構造の決定をめざした.まず,個々のCmrサブユニットを調製してCmr複合体の再構成を試みたが,発現が困難なサブユニットもあり結晶化に適したCmr複合体の再構成にはいたらなかった.そこで,Cmrタンパク質をコードする遺伝子はオペロンを形成していることに注目し,発現ベクターにオペロン遺伝子をクローニングし相互作用しあうサブユニットどうしの大腸菌における共発現について検討した.その結果,Cmr2-Cmr3およびCmr4-Cmr5-Cmr6をそれぞれサブ複合体として(Cmr1は単独で)調製した.得られたP. furiosusに由来するCmrタンパク質を用いて複合体を再構成し標的RNAの切断活性を確認したが,Cmr複合体の結晶を得ることはできなかった.アーキアArchaeoglobus fulgidusに由来するCmr複合体についても検討したが,やはりCmr複合体の結晶化にはいたらなかった.
P. furiosusに由来するCmrタンパク質とA. fulgidusに由来するCmrタンパク質とのあいだにはアミノ酸配列の相同性(27.1~42.2%)がある.そこで,Cmr複合体の異なる種のあいだでの再構成について検討した.その結果,P. furiosusに由来するCmr2-Cmr3サブ複合体とA. fulgidusに由来するCmr4-Cmr5-Cmr6サブ複合体は相互作用することが判明した.さらに,この複合体はP. furiosusに由来する39塩基のcrRNAと会合し,Cmr1がなくてもガイド配列と相補的な標的RNAを切断することが明らかにされた.このキメラCmr複合体の結晶化条件をスクリーニングし,標的アナログとして標的となるRNAと同じ配列をもつ31塩基のDNAと結合したキメラCmr複合体の結晶を得た.X線回折データを測定し,分子置換法と単波長異常分散法の組合せにより結晶の位相を決定して,分解能2.1Åで結晶構造を決定した(PDB ID:3X1L).
キメラCmr複合体は,Cmr4を3分子(Cmr4.1~Cmr4.3),Cmr5を2分子(Cmr5.1およびCmr5.2),それ以外のサブユニットについては1分子ずつ含むことが判明した(図1a).Cmr2-Cmr3サブ複合体はキメラCmr複合体の基底部をなし,crRNAの5’側に存在するタグ配列を特異的に認識していた.一方,Cmr4-Cmr5-Cmr6サブ複合体はキメラCmr複合体の上層部を形成しており,そこでは3分子のCmr4が規則的に積み重なって2分子のCmr5とともにらせん状の溝を形成していた.crRNAのガイド配列はこの溝にそって配置しており,標的アナログを配列に相補的に捕捉していた.
2.crRNAのガイド配列と標的アナログの構造
crRNAのガイド配列はおもに3分子のCmr4と相互作用していた.RRMフォールドをもつCmr4の構造は“右手”のかたちに類似しており,palm領域,finger領域,thumb領域に分割することができる.crRNAのガイド配列と標的アナログは2本鎖を形成していたが,一般的な核酸の二重らせん構造とは異なり,ほどけたリボン状のかたちをしていた(図1b).これは,Cmr4のthumb領域が6塩基対ごとに2本鎖のあいだに入り込み,塩基対の形成を周期的に阻害しているためであった(図1c).キメラCmr複合体は3分子のCmr4を含むため,crRNAのガイド配列と標的アナログの2本鎖は3カ所で塩基対の形成が阻害されていた.その結果,crRNAの5’側から数えて14番目,20番目,26番目(標的アナログの26番目,20番目,14番目)のヌクレオチドの構造が大きくひずんでいた.
3.Cmr複合体は6塩基周期で標的RNAを切断する
これまでの実験から,Cmr複合体は標的となるRNAを複数の部位で切断することが指摘されていた3,7,8).そこで,Cmr複合体はヌクレオチドのかたちが不安定化していた標的RNAの14番目,20番目,26番目の3’側のリン酸結合を切断するのではないかと仮説をたてた.標的RNAのこれら3カ所のリボースを2’-デオキシ化した誘導体を作製しその分解のパターンを解析した結果,推定されたように,Cmr複合体は6塩基周期でこれら3カ所を切断することがわかった.加水分解されるこれら3カ所のリン酸結合の近くにはCmr4において高度に保存されているAsp31が配置されており,変異体の解析の結果,Asp31が標的RNAの切断にかかわることが判明した.
2’-デオキシリボースを含むRNA誘導体を用いた解析から,Cmr複合体によるRNAの切断にはリボースの2’-OH基が不可欠であることが判明した.したがって,標的RNAはRNase Aなどに代表される酸塩基触媒機構により切断されることが示唆された.これは,Cmr複合体による分解産物は3’リン酸末端(あるいは,2’,3’環状リン酸末端)および5’-OH末端をもつというこれまでの結果と一致した6).Cmr複合体の構造解析から,Cmr4のAsp31は一般酸触媒として機能し,分解産物の5’末端にプロトンを供与することが示唆された.一方,加水分解されるリン酸結合の周辺にはAsp31以外に保存されたアミノ酸残基は存在せず,リボースの2’-OH基の脱プロトン化を触媒する一般塩基触媒については明らかにされなかった.標的となるRNAの詳細な加水分解機構の解明は今後の課題である.
4.crRNAのタグ配列の認識
crRNAの5’側に存在するタグ配列は水素結合を介してCmr3およびCmr3に隣接したCmr4(Cmr4.1)と相互作用していた(図1d).タグ配列の8つのヌクレオチドのうち,2番目のウリジンはCmr3により塩基に特異的に認識されていた.このウリジンをアデノシンと置換したcrRNAを含むCmr複合体はRNA切断活性を消失したことから,この相互作用が重要であることが確認された.また,タグ配列の5’末端のOH基はCmr3のポケットに収容され,その主鎖と水素結合していた.これまでの研究から,crRNAの5’末端にリン酸基を付加するとCmr複合体のRNA切断活性は消失することが知られていた8).これは,crRNAの5’末端へのリン酸基の付加によりCmr3とのあいだに立体障害が起こり,crRNAがCmr複合体に取り込まれなくなった結果と考えられた.以上の結果より,crRNAのタグ配列は塩基に特異的に認識され,Cmr複合体において固定されていることが明らかにされた.
Cmr3はN末端側およびC末端側にそれぞれRRMフォールド様のドメインをもち,いずれもCmr4の構造とよく類似していたが,finger領域は欠落しておりpalm領域およびthumb領域から構成されていた.Cmr3の2つのthumb領域はcrRNAのタグ配列と強固に相互作用していた.興味深いことに,Cmr3のN末端側のthumb領域とCmr4のthumb領域は機能的によく類似しており,Cmr3のN末端側のthumb領域との相互作用によりcrRNAのタグ配列の8番目のヌクレオチドの構造は大きくひずみCmr4.1のポケットに配置されていた(図1d).その結果,crRNAの9番目のヌクレオチド(ガイド配列の最初のヌクレオチド)が標的RNAと塩基対を形成する環境が整い,crRNAのガイド配列と標的RNAの形成する2本鎖の開始位置が厳密に規定されていた.この構造的な特徴およびCmr複合体におけるcrRNAのタグ配列の固定により,のちに述べるように,Cmr複合体はcrRNAの5’側から一定の距離にある標的RNAのリン酸結合を長さに特異的に切断することが明らかにされた.
5.タイプIIIのエフェクター複合体が標的RNAを切断するしくみ
今回の解析から,crRNAのタグ配列がCmr複合体において固定されていることが判明した.このため,Cmr複合体はcrRNAの5’側から数えて一定の距離にある14番目,20番目,26番目の塩基対の形成を阻害する.その結果,これら3カ所において標的RNAの構造が大きくひずんでCmr複合体の活性部位に配置され,6塩基周期で切断されるしくみが明らかにされた(図2).
キメラCmr複合体には3分子のCmr4が含まれており,触媒残基であるAsp31は約24Åの間隔で配置されていた.これは6塩基の核酸に相当する長さであった.Cmr複合体から単離されたcrRNAには39塩基あるいは45塩基の長さの異なる2種類が存在する6).したがって,39塩基のcrRNAを含むCmr複合体は3分子のCmr4を,一方,45塩基のcrRNAを含むCmr複合体は4分子のCmr4をもち,それぞれ,標的RNAを3カ所または4カ所で切断すると考えられた.実際,45塩基のcrRNAを含むCmr複合体は標的となるRNAを4カ所で切断する7,8).
タイプIII-Aエフェクター複合体のCsm複合体は5種類のCasタンパク質Csm1~Csm5とcrRNAから構成される.Csm3の構造はCmr4と類似しており9),thumb領域に相当する領域(結晶構造では構造をとっていなかった)をもつことが示唆されるとともに,触媒反応にかかわるCmr4のAspはCsm3においても保存されていた10).また,Csm複合体のクライオ電子顕微鏡による解析から,複数のCsm3が規則的に積み重なってらせん状の溝を形成していることが明らかにされていた5).さらに,Csm4の構造はCmr3に類似していたことから9),Csm4がcrRNAのタグ配列の認識にかかわることが示唆された.したがって,Csm複合体も,タイプIII-Bエフェクター複合体であるCmr複合体とまったく同じしくみにより標的となるRNAの切断部位を選びだすと考えられ,標的となるRNAを分解するしくみはタイプIIIエフェクター複合体の種類にかかわらず共通することが示唆された.
おわりに
2014年に,1本鎖DNAと結合したCascadeの結晶構造が報告されている1).タイプIエフェクター複合体であるCascadeは,1:2:1:1:6のストイキオメトリーで会合した5種類のCasタンパク質Cse1,Cse2,Cas5,Cas6,Cas7とcrRNAから構成される.立体構造の比較から,Cmr4とCas7の構造,また,Cmr3とCas5の構造がそれぞれ類似していることがわかった.また,Cmr複合体の場合と同様に,CascadeではCas5がcrRNAのタグ配列を認識し,6分子のCas7がcrRNAのガイド配列との相互作用に関与している1).このため,CascadeとCmr複合体においてこれらサブユニットの空間的な配置は類似しており,その結果,タイプIエフェクター複合体とタイプIIIエフェクター複合体はよく似た構造をとることがわかった.さらに興味深いことに,Cas7にもthumb領域が存在し,Cascadeもガイド配列と標的DNAとの塩基対の形成を6塩基周期で阻害する1).その結果,CascadeおよびCmr複合体において,ガイド配列と標的となる核酸の2本鎖はほどけたリボン状の構造をとる.リボン状の構造の形成はこれらエフェクター複合体が標的となる核酸と効率よく結合するうえで非常に重要であると考えられる.CascadeにDNA切断活性はなく,標的となるDNAを捕捉したのち,ヌクレアーゼ/ヘリカーゼであるCas3がリクルートされ標的となるDNAを分解する.
このように,CascadeとCmr複合体では標的となる核酸と結合するしくみはよく類似しているが,それを切断する機構はまったく異なっていた.したがって,タイプIエフェクター複合体とタイプIIIエフェクター複合体は共通の祖先から派生したと考えられた.今後,タイプIエフェクター複合体およびタイプIIIエフェクター複合体のさらなる解析により,それぞれの複合体が標的となる核酸を分解するしくみの詳細が明らかにされるとともに,生物がDNAとRNAの違いを区別してそれぞれに対応するエフェクター複合体をつくりだした進化戦略に対し,科学的な考察が可能になると期待される.
文 献
- Mulepati, S., Heroux, A. & Bailey, S.: Crystal structure of a CRISPR RNA-guided surveillance complex bound to a ssDNA target. Science, 345, 1479-1484 (2014)[PubMed]
- Nishimasu, H., Ran, F. A., Hsu, P. D. et al.: Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell, 156, 935-949 (2014)[PubMed] [新着論文レビュー]
- Staals, R. H., Agari, Y., Maki-Yonekura, S. et al.: Structure and activity of the RNA-targeting Type III-B CRISPR-Cas complex of Thermus thermophilus. Mol. Cell, 52, 135-145 (2013)[PubMed] [新着論文レビュー]
- Spilman, M., Cocozaki, A., Hale, C. et al.: Structure of an RNA silencing complex of the CRISPR-Cas immune system. Mol. Cell, 52, 146-152 (2013)[PubMed]
- Staals, R. H., Zhu, Y., Taylor, D. W. et al.: RNA targeting by the type III-A CRISPR-Cas Csm complex of Thermus thermophilus. Mol. Cell, 56, 518-530 (2014)[PubMed]
- Hale, C. R., Zhao, P., Olson, S. et al.: RNA-guided RNA cleavage by a CRISPR RNA-Cas protein complex. Cell, 139, 945-956 (2009)[PubMed]
- Benda, C., Ebert, J., Scheltema, R. A. et al.: Structural model of a CRISPR RNA-silencing complex reveals the RNA-target cleavage activity in Cmr4. Mol. Cell, 56, 43-54 (2014)[PubMed]
- Hale, C. R., Cocozaki, A., Li, H. et al.: Target RNA capture and cleavage by the Cmr type III-B CRISPR-Cas effector complex. Genes Dev., 28, 2432-2443 (2014)[PubMed]
- Numata, T., Inanaga, H., Sato, C. et al.: Crystal structure of the Csm3-Csm4 subcomplex in the type III-A CRISPR-Cas interference complex. J. Mol. Biol., 427, 259-273 (2015)[PubMed]
- Tamulaitis, G., Kazlauskiene, M., Manakova, E. et al.: Programmable RNA shredding by the type III-A CRISPR-Cas system of Streptococcus thermophilus. Mol. Cell, 56, 506-517 (2014)[PubMed]
著者プロフィール
略歴:2003年 九州大学大学院生物資源環境科学府博士課程 修了,同年 同 博士研究員,2004年 東京工業大学大学院生命理工学研究科 博士研究員,2006年 科学技術振興機構 さきがけ研究者,2007年 産業技術総合研究所生物機能工学研究部門 研究員を経て,2012年より産業技術総合研究所バイオメディカル研究部門 主任研究員.
研究テーマ:RNAのかかわる生体高分子複合体の機能構造解析.
抱負:RNAやRNAとタンパク質からなる分子装置の作動原理を,原子分解能のレベルで明らかにしていきたい.
大澤 拓生(Takuo Osawa)
微生物化学研究所 博士研究員.
© 2015 沼田倫征・大澤拓生 Licensed under CC 表示 2.1 日本