RNAポリメラーゼのもつ複数の機能はラチェット型およびラチェット型に移行しやすいコンホメーションによりささえられている
関根俊一・村山祐子・横山茂之
(理化学研究所ライフサイエンス技術基盤研究センター 超分子構造解析研究チーム)
email:関根俊一,横山茂之
DOI: 10.7875/first.author.2015.022
The ratcheted and ratchetable structural states of RNA polymerase underlie multiple transcriptional functions.
Shun-ichi Sekine, Yuko Murayama, Vladimir Svetlov, Evgeny Nudler, Shigeyuki Yokoyama
Molecular Cell, 57, 408-421 (2015)
RNAポリメラーゼはDNAのうえを移動しながらその塩基配列をRNAに転写する役割を担う.転写は必ずしもスムーズな過程ではなく,RNAポリメラーゼはDNAのうえで,転写を一時休止したり,後退したり,転写のエラーを校正したりなど,さまざまな過程をへながら転写を遂行する.そのときどきに応じて必要な機能を発揮するため,RNAポリメラーゼはその立体構造を変化させることにより機能あるいは活性を切り替えると考えられているが,その構造の変化あるいは制御の具体的な分子機構は不明であった.RNAポリメラーゼはタイト型とラチェット型の2つのコンホメーションをとりうることが知られている.タイト型は従来から知られていたコンホメーションでRNA伸長反応に必須である.一方,ラチェット型は,最近,筆者らが発見したコンホメーションであるが,その機能や転写における役割はよくわかっていなかった.この研究においては,ラチェット型のコンホメーションの機能を明らかにし2つのコンホメーションが存在する意義を解明するためにCysペア架橋法を開発し,さまざまな転写複合体におけるRNAポリメラーゼのコンホメーションを調べるとともに,後退状態および転写因子Greと結合した状態のRNAポリメラーゼのX線結晶構造解析を行った.その結果,ラチェット型のコンホメーションは,RNAポリメラーゼの後退,Greに依存したRNAの切断,RNAのヘアピン構造に依存した転写の休止および終結をささえる重要な構造であることが明らかにされた.一方,タイト型のコンホメーションはRNA伸長反応だけでなく,ミスマッチ塩基の取り込みによる転写の休止,1塩基分の後退,転写因子に依存しないRNAの切断に関与していることがわかった.興味深いことに,休止状態あるいは後退状態にあるタイト型の転写複合体は,RNA伸長反応を行う転写複合体と比べラチェット型に移行しやすい性質をもつことが示され,タイト型には機能的に異なる2つの状態が含まれることが示唆された.新生RNA鎖の3’末端やRNAのヘアピン構造がRNAポリメラーゼのトリガーループの構造に影響を及ぼすことにより,この性質を左右していると考えられた.これらの結果は,RNAポリメラーゼが転写因子や新生RNAなどに応じてタイト型とラチェット型とのあいだでコンホメーションを切り替え,そのときどきに最適な機能を発揮できるしくみを備えていることを示した.
RNAポリメラーゼはDNAの塩基配列をRNAに転写する役割を担う.転写は複雑かつ高度に制御された多段階の過程であり,開始,伸長,終結の段階をへる.RNAポリメラーゼは転写のおのおのの段階に必要とされる複数の機能をはたすため,そのコンホメーションを動的に変化させつつ転写を遂行すると考えられている.また,さまざまな転写因子やDNA配列あるいはRNA配列などがときに応じてRNAポリメラーゼの構造および機能を制御していると考えられている.しかしながら,具体的にRNAポリメラーゼのどのようなコンホメーションが転写のおのおのの機能に対応しているのか,転写因子やDNAあるいはRNAはどのようにRNAポリメラーゼを制御しているのか,それらの分子機構は今日でもよくわかっていない.
RNAポリメラーゼは複数のサブユニットからなる巨大なタンパク質複合体である.いくつかのモジュールから構成されており,全体としてカニのはさみのような形状をしている1,2).RNAポリメラーゼの中心部はコアモジュールとシェルフモジュールからなり,それらのあいだに2つのチャネルが形成されている.大きい1次チャネルはDNAおよびRNAとの結合に,小さい2次チャネルは基質であるNTPの取り込みに用いられる.1次チャネルの中央部には触媒活性に必要なMg2+が結合し,トリガーループやブリッジヘリックスといった特徴的な構造とともに活性部位を形成している.
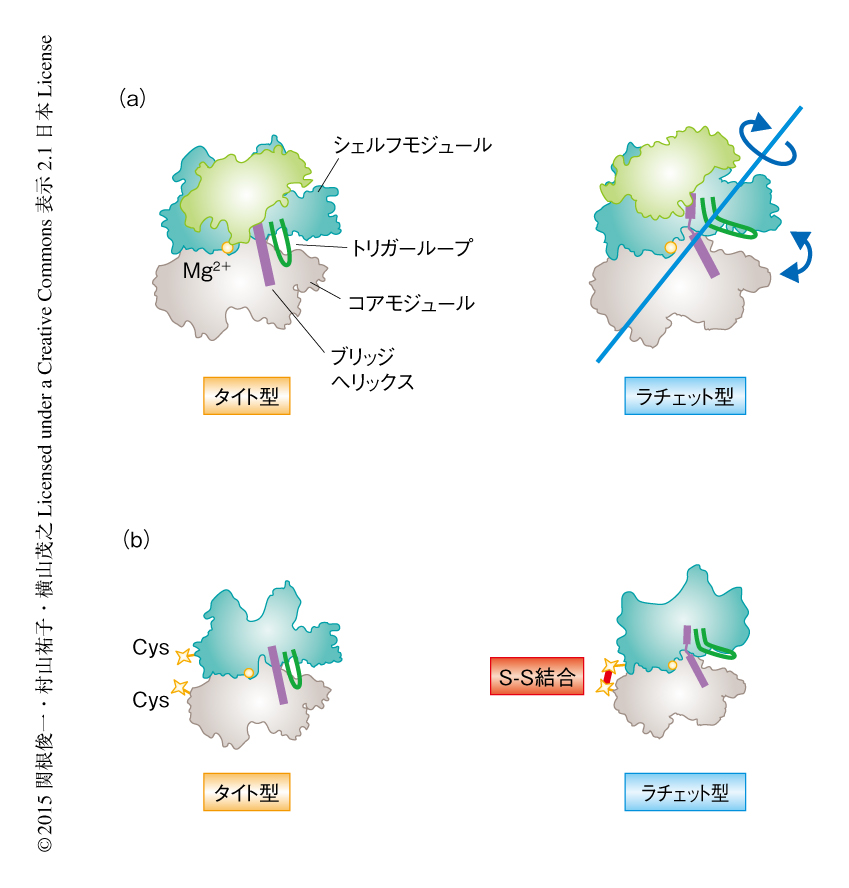
以前に,筆者らは,高度好熱菌のRNAポリメラーゼのはたらきを阻害するタンパク質Gfh1と高度好熱菌RNAポリメラーゼとの複合体の結晶構造解析に成功し,RNAポリメラーゼが従来から知られていたものとは異なる新規のコンホメーションをとることを見い出した3)(新着論文レビュー でも掲載).シェルフモジュールがコアモジュールに対し回転してずれた位置関係にあり,1次チャネルおよび2次チャネルの拡張やブリッジヘリックスの屈曲を含め,活性部位が再編成されていた.そこで,この新規のコンホメーションを既知のタイト型と区別してラチェット型と命名した(図1a).しかし,ラチェット型のコンホメーションの存在意義や転写における役割については不明であった.
RNAポリメラーゼは,NTPを基質として新生RNA鎖の3’末端に鋳型に依存的にヌクレオチドを1つずつ付加することによりRNAを合成する.転写は必ずしもスムーズに進行するわけではなく,RNAポリメラーゼはDNAのうえで頻繁に休止や後退をくり返しながら転写を遂行する.典型的な例として,転写のエラーにより新生RNA鎖の3’末端にミスマッチのヌクレオチドが取り込まれたとき,RNAポリメラーゼはRNAの3’末端が鋳型DNAからほつれた状態で転写を休止する.さらに,RNAポリメラーゼはほつれたRNAの3’末端を2次チャネルの内部にはみ出させながらDNAのうえを1塩基から数塩基ほど後退する4).後退状態のRNAポリメラーゼにはエンドヌクレアーゼ活性があり,RNA合成と同じ活性部位においてRNAのはみ出した部分を切断し転写を再開できる5).こうした転写の休止,後退,RNA切断は,転写のエラーが生じた際にかぎらず頻繁に起こっていると考えられるが,とくに,転写のエラーの場合におけるRNA切断ではエラーが修正されるため,校正が行われていることになる.さらに,RNAポリメラーゼのRNA切断活性は転写因子(細菌ではGreAあるいはGreB,真核生物ではTFIIS)により飛躍的に増強される6).一方,転写の休止は新生RNA鎖に形成されるヘアピン構造によってもひき起こされることが知られている7).転写終結シグナルにおいては新生RNA鎖のヘアピン構造がRNAポリメラーゼからのDNAおよびRNAの解離において重要な役割をはたしている8).
筆者らは,RNAポリメラーゼのラチェット型のコンホメーションが転写の休止,後退,RNA切断,終結などに深く関与しているのではないか,また,RNAポリメラーゼの機能の切り替えおよび制御はタイト型とラチェット型とのコンホメーションの切り替えにより説明できるのではないかと考えた.この仮説を検証するため,生化学的な手法およびX線結晶構造解析を用いてRNAポリメラーゼのコンホメーションと機能との相関を解析した.
転写のさまざまな状態においてRNAポリメラーゼがタイト型とラチェット型のどちらのコンホメーションをとっているかを調べるため,高度好熱菌のRNAポリメラーゼのCysペア架橋変異体を作製した(図1b).この変異体には,RNAポリメラーゼのαサブユニットとβ’サブユニットに1か所ずつCysを導入してある.タイト型のコンホメーションではこれらのCysは離れた位置関係にあるがラチェット型のコンホメーションでは互いに近づくため,酸化条件においてはRNAポリメラーゼがラチェット型をとっているときサブユニットのあいだにS-S結合がより高い効率で形成される.このCysペア架橋法を用いてさまざまな転写複合体におけるRNAポリメラーゼのコンホメーションについて調べた.
Cysペア架橋変異体を用いて再構成した転写伸長複合体にGfh1をくわえるとS-S結合の形成効率がいちじるしく上昇した.この結果は,Gfh1がRNAポリメラーゼをラチェット型のコンホメーションにシフトさせるタンパク質であることを示しており(図2),以前のRNAポリメラーゼとGfh1との複合体の結晶構造解析3) の結果とよく一致した.また,RNAポリメラーゼは核酸と結合していない遊離の状態ではラチェット型のコンホメーションをとっており,DNAおよびRNAとの結合によりタイト型の転写伸長複合体が安定化されることもわかった.
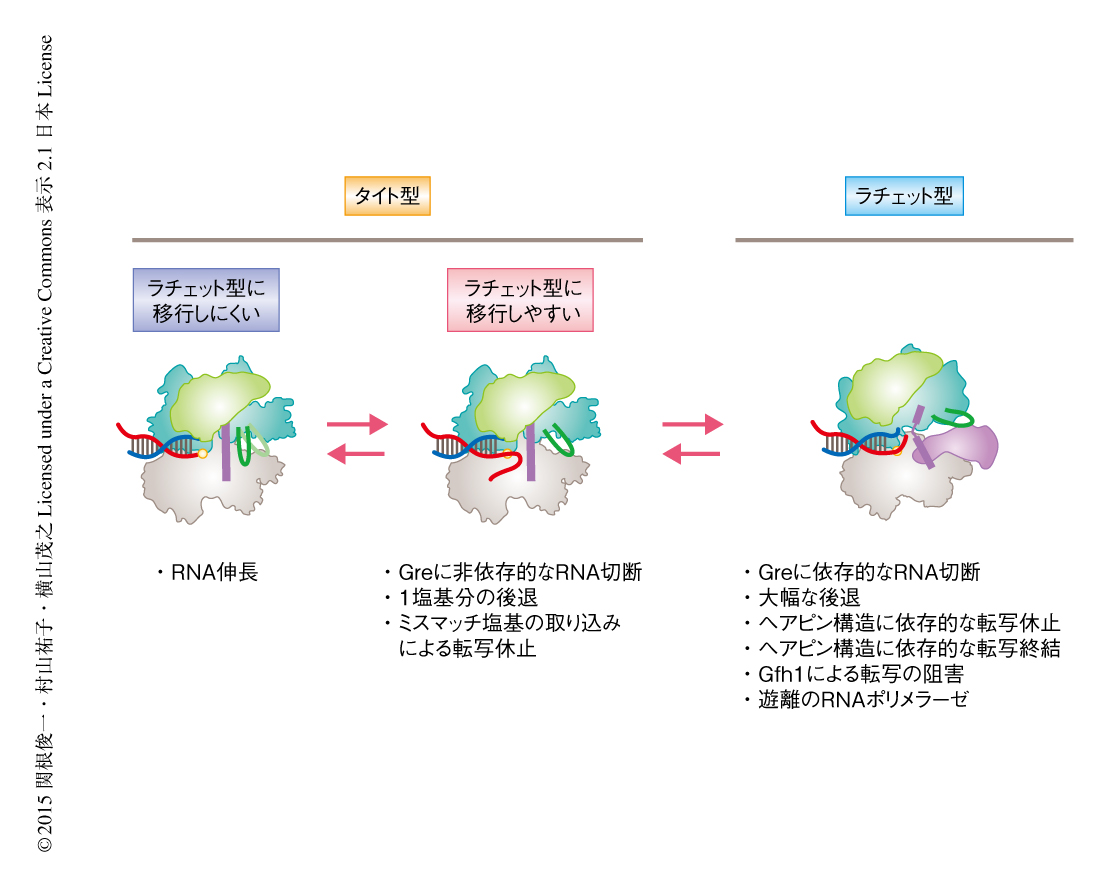
ミスマッチ塩基によりDNAのうえを後退した状態の転写複合体について調べたところ,RNAポリメラーゼは,DNAのうえを1塩基分だけ後退した状態ではタイト型のコンホメーションを,より大きく8塩基分だけ後退した状態ではラチェット型のコンホメーションをとることが示された(図2).さらに,1塩基分だけ後退した転写複合体に結合してRNAポリメラーゼによるRNA切断の活性を大幅に促進する転写因子GreAは,RNAポリメラーゼをラチェット型のコンホメーションにシフトさせることが明らかにされた.これらの結果から,GreAに依存しないRNA切断反応はタイト型のRNAポリメラーゼ,GreA依存的なRNA切断反応はラチェット型のRNAポリメラーゼにより行われることが示唆された.実際に,S-S結合によりコンホメーションをラチェット型にかたよらせたCysペア架橋変異体によるRNA切断の活性を測定することにより,2種類のRNA切断反応がそれぞれ異なるRNAポリメラーゼのコンホメーションに依拠していることが実証された.
興味深いことに,1塩基分だけ後退した状態の転写後退複合体はGreAによりラチェット型のコンホメーションにシフトするのに対し,転写伸長複合体は抵抗性を示した.これは,GreAは転写伸長複合体による転写のじゃまをせず,転写後退複合体にのみ選択的に作用して転写休止状態からの復旧や校正を手助けできることを示唆しており,効率的かつ正確な転写の観点から理にかなっていた.また,同様に,ミスマッチによりRNAの3’末端がほつれた状態の転写休止複合体もラチェット型のコンホメーションにシフトしやすい性質をもつことがわかった.以上のことは,タイト型のコンホメーションにはラチェット型にシフトしやすい状態としにくい状態の2つの状態があり,それぞれ,転写の機能と密接な関連のあることを示唆した(図2).
1塩基分だけ後退した状態のRNAポリメラーゼおよび転写因子Greと結合した状態のRNAポリメラーゼのX線結晶構造解析を行い,それらの構造を詳細に調べた.全体構造については,転写後退複合体はタイト型,Greと結合した転写複合体はラチェット型のコンホメーションをとっており,Cysペア架橋法による解析の結果とよく一致した.
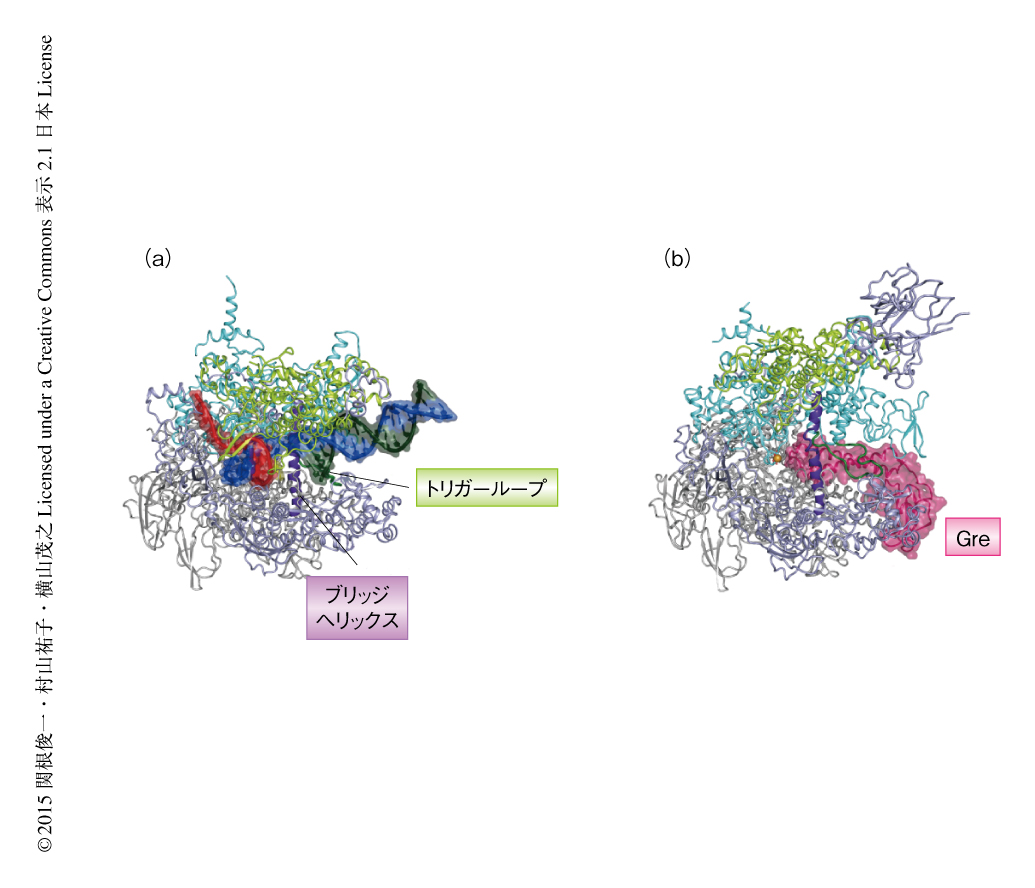
転写後退複合体では,RNAの3’末端はDNAから離れ,向きを変えて校正部位とよばれるくぼみに収まっていた.また,RNAポリメラーゼの活性部位を構成するトリガーループはその根元で折れ曲がった構造をとっていた(PDB ID:4WQS,図3a).トリガーループはRNA伸長反応の基質であるNTPの結合部位の一部を構成するループで,反応のときにはNTPと結合してまっすぐな構造をとる9,10).転写後退複合体ではRNAポリメラーゼの後退により突出したRNAの3’末端が校正部位にあるため,トリガーループはまっすぐな構造をとれず折れ曲がった状態に固定されていると考えられた.
Greと結合した転写複合体では,GreのN末端のコイルドコイルドメインがRNAポリメラーゼの2次チャネルと結合していた(PDB ID:4WQT,図3b).コンホメーションはラチェット型であったが,それはタイト型の狭い2次チャネルではかさ高いコイルドコイルドメインを受け入れられないため,ラチェット型に切り替えて2次チャネルを拡張した結果と考えられた.RNAポリメラーゼの構造の変化にともなう活性部位の再編成とGreの提供する活性に重要な残基により,校正部位とは別にRNAの3’末端との新たな結合部位が形成され,より高い効率のRNA切断を実現していると考えられた.
Cysペア架橋法による解析により,休止した状態あるいは1塩基分だけ後退した状態のRNAポリメラーゼは,転写伸長複合体と比較してより容易にラチェット型のコンホメーションへとシフトできることが明らかにされたが,休止あるいは後退した状態の転写複合体ではRNAの3’末端が校正部位にあるため,トリガーループが折れ曲がって2次チャネルが半分だけ開いた状態にあり,Greのアクセスが可能になりラチェット型へのコンホメーションの変化がひき起こされると考えられた.
RNAポリメラーゼによる転写の休止や後退は,転写のエラーが起こったときだけでなく,DNA配列などに依存して頻繁に起こる現象である.よく知られている転写休止の例では,新生RNA鎖がヘアピン構造をとる場合,それによりRNAポリメラーゼは安定な転写休止の状態に入る7).また,RNAのヘアピン構造に依存した転写終結の場合には,RNAポリメラーゼは転写を休止した状態からDNAおよびRNAを解離して転写を終結する8).RNAポリメラーゼのコンホメーションに対するRNAのヘアピン構造の影響をCysペア架橋法により調べたところ,ヘアピン構造にはコンホメーションをラチェット型にシフトさせる効果のあることが明らかにされた(図2).この結果は,ラチェット型のコンホメーションは,RNAポリメラーゼの後退やRNA切断だけでなく,RNAのヘアピン構造に依存した転写休止や終結においても重要な役割をはたしていることを示した.
以上の結果から,RNAポリメラーゼのとるラチェット型のコンホメーションは,RNAのヘアピン構造に依存的な転写休止あるいは転写終結,RNAポリメラーゼの後退および転写因子Greに依存的なRNA切断など,転写における多くの重要な機能をささえる必須の構造であることが明らかにされた.一方,タイト型のコンホメーションには機能的に異なる2つの状態があり,RNA伸長反応を行うRNAポリメラーゼと転写休止状態あるいは後退状態にあるRNAポリメラーゼでは,後者のほうがラチェット型にシフトしやすい.転写休止状態あるいは後退状態にあるRNAポリメラーゼは,ラチェット型と関連づけられた機能に切り替えるための前段階と考えることもできる.この結果は,RNA合成,転写休止,後退,転写終結,RNA切断など,おのおのの転写の機能がタイト型とラチェット型の2つのコンホメーションに割り当てられており,RNAポリメラーゼの機能がそれらの切り替えによりたくみに制御されていることを示唆した.さらに,RNAポリメラーゼにより生み出されつつある新生RNA鎖の3’末端やヘアピン構造がコンホメーションの切り替えにおいて重要な役割をはたしており,転写の機能の制御に深く関与していることが示唆された.
巨大なタンパク質複合体であるRNAポリメラーゼが転写の状況や種々のシグナルに応じて転写を休止あるいは終結したり,その活性あるいは機能を切り替えたりすることは知られていたが,その具体的な分子機構はよくわかっていなかった.この研究は,RNAポリメラーゼの2つのコンホメーションにそれぞれ異なる機能が割り当てられていることを明らかにし,転写の機能の制御が2つのコンホメーションのあいだの切り替えにより説明できることを提案した.転写および転写制御における基本原理の解明へとつながる重要な一歩と考えている.この発見をもとに,普遍的な転写機構のさらなる理解やそれにもとづいた応用へと研究が発展することを期待する.
略歴:1997年 東京大学大学院理学系研究科にて博士号取得,同年 理化学研究所 研究員,2004年 東京大学大学院理学系研究科 講師,特任准教授,2012年 理化学研究所 上級研究員を経て,2013年より理化学研究所ライフサイエンス技術基盤研究センター チームリーダー.
村山 祐子(Yuko Murayama)
略歴:2013年 東京大学大学院理学系研究科にて博士号取得,同年より理化学研究所ライフサイエンス技術基盤研究センター 特別研究員.
横山 茂之(Shigeyuki Yokoyama)
理化学研究所横山構造生物学研究室 上席研究員.
© 2015 関根俊一・村山祐子・横山茂之 Licensed under CC 表示 2.1 日本
(理化学研究所ライフサイエンス技術基盤研究センター 超分子構造解析研究チーム)
email:関根俊一,横山茂之
DOI: 10.7875/first.author.2015.022
The ratcheted and ratchetable structural states of RNA polymerase underlie multiple transcriptional functions.
Shun-ichi Sekine, Yuko Murayama, Vladimir Svetlov, Evgeny Nudler, Shigeyuki Yokoyama
Molecular Cell, 57, 408-421 (2015)
要 約
RNAポリメラーゼはDNAのうえを移動しながらその塩基配列をRNAに転写する役割を担う.転写は必ずしもスムーズな過程ではなく,RNAポリメラーゼはDNAのうえで,転写を一時休止したり,後退したり,転写のエラーを校正したりなど,さまざまな過程をへながら転写を遂行する.そのときどきに応じて必要な機能を発揮するため,RNAポリメラーゼはその立体構造を変化させることにより機能あるいは活性を切り替えると考えられているが,その構造の変化あるいは制御の具体的な分子機構は不明であった.RNAポリメラーゼはタイト型とラチェット型の2つのコンホメーションをとりうることが知られている.タイト型は従来から知られていたコンホメーションでRNA伸長反応に必須である.一方,ラチェット型は,最近,筆者らが発見したコンホメーションであるが,その機能や転写における役割はよくわかっていなかった.この研究においては,ラチェット型のコンホメーションの機能を明らかにし2つのコンホメーションが存在する意義を解明するためにCysペア架橋法を開発し,さまざまな転写複合体におけるRNAポリメラーゼのコンホメーションを調べるとともに,後退状態および転写因子Greと結合した状態のRNAポリメラーゼのX線結晶構造解析を行った.その結果,ラチェット型のコンホメーションは,RNAポリメラーゼの後退,Greに依存したRNAの切断,RNAのヘアピン構造に依存した転写の休止および終結をささえる重要な構造であることが明らかにされた.一方,タイト型のコンホメーションはRNA伸長反応だけでなく,ミスマッチ塩基の取り込みによる転写の休止,1塩基分の後退,転写因子に依存しないRNAの切断に関与していることがわかった.興味深いことに,休止状態あるいは後退状態にあるタイト型の転写複合体は,RNA伸長反応を行う転写複合体と比べラチェット型に移行しやすい性質をもつことが示され,タイト型には機能的に異なる2つの状態が含まれることが示唆された.新生RNA鎖の3’末端やRNAのヘアピン構造がRNAポリメラーゼのトリガーループの構造に影響を及ぼすことにより,この性質を左右していると考えられた.これらの結果は,RNAポリメラーゼが転写因子や新生RNAなどに応じてタイト型とラチェット型とのあいだでコンホメーションを切り替え,そのときどきに最適な機能を発揮できるしくみを備えていることを示した.
はじめに
RNAポリメラーゼはDNAの塩基配列をRNAに転写する役割を担う.転写は複雑かつ高度に制御された多段階の過程であり,開始,伸長,終結の段階をへる.RNAポリメラーゼは転写のおのおのの段階に必要とされる複数の機能をはたすため,そのコンホメーションを動的に変化させつつ転写を遂行すると考えられている.また,さまざまな転写因子やDNA配列あるいはRNA配列などがときに応じてRNAポリメラーゼの構造および機能を制御していると考えられている.しかしながら,具体的にRNAポリメラーゼのどのようなコンホメーションが転写のおのおのの機能に対応しているのか,転写因子やDNAあるいはRNAはどのようにRNAポリメラーゼを制御しているのか,それらの分子機構は今日でもよくわかっていない.
RNAポリメラーゼは複数のサブユニットからなる巨大なタンパク質複合体である.いくつかのモジュールから構成されており,全体としてカニのはさみのような形状をしている1,2).RNAポリメラーゼの中心部はコアモジュールとシェルフモジュールからなり,それらのあいだに2つのチャネルが形成されている.大きい1次チャネルはDNAおよびRNAとの結合に,小さい2次チャネルは基質であるNTPの取り込みに用いられる.1次チャネルの中央部には触媒活性に必要なMg2+が結合し,トリガーループやブリッジヘリックスといった特徴的な構造とともに活性部位を形成している.
以前に,筆者らは,高度好熱菌のRNAポリメラーゼのはたらきを阻害するタンパク質Gfh1と高度好熱菌RNAポリメラーゼとの複合体の結晶構造解析に成功し,RNAポリメラーゼが従来から知られていたものとは異なる新規のコンホメーションをとることを見い出した3)(新着論文レビュー でも掲載).シェルフモジュールがコアモジュールに対し回転してずれた位置関係にあり,1次チャネルおよび2次チャネルの拡張やブリッジヘリックスの屈曲を含め,活性部位が再編成されていた.そこで,この新規のコンホメーションを既知のタイト型と区別してラチェット型と命名した(図1a).しかし,ラチェット型のコンホメーションの存在意義や転写における役割については不明であった.
RNAポリメラーゼは,NTPを基質として新生RNA鎖の3’末端に鋳型に依存的にヌクレオチドを1つずつ付加することによりRNAを合成する.転写は必ずしもスムーズに進行するわけではなく,RNAポリメラーゼはDNAのうえで頻繁に休止や後退をくり返しながら転写を遂行する.典型的な例として,転写のエラーにより新生RNA鎖の3’末端にミスマッチのヌクレオチドが取り込まれたとき,RNAポリメラーゼはRNAの3’末端が鋳型DNAからほつれた状態で転写を休止する.さらに,RNAポリメラーゼはほつれたRNAの3’末端を2次チャネルの内部にはみ出させながらDNAのうえを1塩基から数塩基ほど後退する4).後退状態のRNAポリメラーゼにはエンドヌクレアーゼ活性があり,RNA合成と同じ活性部位においてRNAのはみ出した部分を切断し転写を再開できる5).こうした転写の休止,後退,RNA切断は,転写のエラーが生じた際にかぎらず頻繁に起こっていると考えられるが,とくに,転写のエラーの場合におけるRNA切断ではエラーが修正されるため,校正が行われていることになる.さらに,RNAポリメラーゼのRNA切断活性は転写因子(細菌ではGreAあるいはGreB,真核生物ではTFIIS)により飛躍的に増強される6).一方,転写の休止は新生RNA鎖に形成されるヘアピン構造によってもひき起こされることが知られている7).転写終結シグナルにおいては新生RNA鎖のヘアピン構造がRNAポリメラーゼからのDNAおよびRNAの解離において重要な役割をはたしている8).
筆者らは,RNAポリメラーゼのラチェット型のコンホメーションが転写の休止,後退,RNA切断,終結などに深く関与しているのではないか,また,RNAポリメラーゼの機能の切り替えおよび制御はタイト型とラチェット型とのコンホメーションの切り替えにより説明できるのではないかと考えた.この仮説を検証するため,生化学的な手法およびX線結晶構造解析を用いてRNAポリメラーゼのコンホメーションと機能との相関を解析した.
1.Cysペア架橋法によるRNAポリメラーゼのコンホメーションの解析
転写のさまざまな状態においてRNAポリメラーゼがタイト型とラチェット型のどちらのコンホメーションをとっているかを調べるため,高度好熱菌のRNAポリメラーゼのCysペア架橋変異体を作製した(図1b).この変異体には,RNAポリメラーゼのαサブユニットとβ’サブユニットに1か所ずつCysを導入してある.タイト型のコンホメーションではこれらのCysは離れた位置関係にあるがラチェット型のコンホメーションでは互いに近づくため,酸化条件においてはRNAポリメラーゼがラチェット型をとっているときサブユニットのあいだにS-S結合がより高い効率で形成される.このCysペア架橋法を用いてさまざまな転写複合体におけるRNAポリメラーゼのコンホメーションについて調べた.
2.転写後退複合体および転写休止複合体におけるRNAポリメラーゼのコンホメーションと転写因子の影響
Cysペア架橋変異体を用いて再構成した転写伸長複合体にGfh1をくわえるとS-S結合の形成効率がいちじるしく上昇した.この結果は,Gfh1がRNAポリメラーゼをラチェット型のコンホメーションにシフトさせるタンパク質であることを示しており(図2),以前のRNAポリメラーゼとGfh1との複合体の結晶構造解析3) の結果とよく一致した.また,RNAポリメラーゼは核酸と結合していない遊離の状態ではラチェット型のコンホメーションをとっており,DNAおよびRNAとの結合によりタイト型の転写伸長複合体が安定化されることもわかった.
ミスマッチ塩基によりDNAのうえを後退した状態の転写複合体について調べたところ,RNAポリメラーゼは,DNAのうえを1塩基分だけ後退した状態ではタイト型のコンホメーションを,より大きく8塩基分だけ後退した状態ではラチェット型のコンホメーションをとることが示された(図2).さらに,1塩基分だけ後退した転写複合体に結合してRNAポリメラーゼによるRNA切断の活性を大幅に促進する転写因子GreAは,RNAポリメラーゼをラチェット型のコンホメーションにシフトさせることが明らかにされた.これらの結果から,GreAに依存しないRNA切断反応はタイト型のRNAポリメラーゼ,GreA依存的なRNA切断反応はラチェット型のRNAポリメラーゼにより行われることが示唆された.実際に,S-S結合によりコンホメーションをラチェット型にかたよらせたCysペア架橋変異体によるRNA切断の活性を測定することにより,2種類のRNA切断反応がそれぞれ異なるRNAポリメラーゼのコンホメーションに依拠していることが実証された.
興味深いことに,1塩基分だけ後退した状態の転写後退複合体はGreAによりラチェット型のコンホメーションにシフトするのに対し,転写伸長複合体は抵抗性を示した.これは,GreAは転写伸長複合体による転写のじゃまをせず,転写後退複合体にのみ選択的に作用して転写休止状態からの復旧や校正を手助けできることを示唆しており,効率的かつ正確な転写の観点から理にかなっていた.また,同様に,ミスマッチによりRNAの3’末端がほつれた状態の転写休止複合体もラチェット型のコンホメーションにシフトしやすい性質をもつことがわかった.以上のことは,タイト型のコンホメーションにはラチェット型にシフトしやすい状態としにくい状態の2つの状態があり,それぞれ,転写の機能と密接な関連のあることを示唆した(図2).
3.転写後退複合体および転写因子Greと結合した転写複合体のX線結晶構造解析
1塩基分だけ後退した状態のRNAポリメラーゼおよび転写因子Greと結合した状態のRNAポリメラーゼのX線結晶構造解析を行い,それらの構造を詳細に調べた.全体構造については,転写後退複合体はタイト型,Greと結合した転写複合体はラチェット型のコンホメーションをとっており,Cysペア架橋法による解析の結果とよく一致した.
転写後退複合体では,RNAの3’末端はDNAから離れ,向きを変えて校正部位とよばれるくぼみに収まっていた.また,RNAポリメラーゼの活性部位を構成するトリガーループはその根元で折れ曲がった構造をとっていた(PDB ID:4WQS,図3a).トリガーループはRNA伸長反応の基質であるNTPの結合部位の一部を構成するループで,反応のときにはNTPと結合してまっすぐな構造をとる9,10).転写後退複合体ではRNAポリメラーゼの後退により突出したRNAの3’末端が校正部位にあるため,トリガーループはまっすぐな構造をとれず折れ曲がった状態に固定されていると考えられた.
Greと結合した転写複合体では,GreのN末端のコイルドコイルドメインがRNAポリメラーゼの2次チャネルと結合していた(PDB ID:4WQT,図3b).コンホメーションはラチェット型であったが,それはタイト型の狭い2次チャネルではかさ高いコイルドコイルドメインを受け入れられないため,ラチェット型に切り替えて2次チャネルを拡張した結果と考えられた.RNAポリメラーゼの構造の変化にともなう活性部位の再編成とGreの提供する活性に重要な残基により,校正部位とは別にRNAの3’末端との新たな結合部位が形成され,より高い効率のRNA切断を実現していると考えられた.
Cysペア架橋法による解析により,休止した状態あるいは1塩基分だけ後退した状態のRNAポリメラーゼは,転写伸長複合体と比較してより容易にラチェット型のコンホメーションへとシフトできることが明らかにされたが,休止あるいは後退した状態の転写複合体ではRNAの3’末端が校正部位にあるため,トリガーループが折れ曲がって2次チャネルが半分だけ開いた状態にあり,Greのアクセスが可能になりラチェット型へのコンホメーションの変化がひき起こされると考えられた.
4.RNAポリメラーゼのコンホメーションにおけるRNAのヘアピン構造の影響
RNAポリメラーゼによる転写の休止や後退は,転写のエラーが起こったときだけでなく,DNA配列などに依存して頻繁に起こる現象である.よく知られている転写休止の例では,新生RNA鎖がヘアピン構造をとる場合,それによりRNAポリメラーゼは安定な転写休止の状態に入る7).また,RNAのヘアピン構造に依存した転写終結の場合には,RNAポリメラーゼは転写を休止した状態からDNAおよびRNAを解離して転写を終結する8).RNAポリメラーゼのコンホメーションに対するRNAのヘアピン構造の影響をCysペア架橋法により調べたところ,ヘアピン構造にはコンホメーションをラチェット型にシフトさせる効果のあることが明らかにされた(図2).この結果は,ラチェット型のコンホメーションは,RNAポリメラーゼの後退やRNA切断だけでなく,RNAのヘアピン構造に依存した転写休止や終結においても重要な役割をはたしていることを示した.
おわりに
以上の結果から,RNAポリメラーゼのとるラチェット型のコンホメーションは,RNAのヘアピン構造に依存的な転写休止あるいは転写終結,RNAポリメラーゼの後退および転写因子Greに依存的なRNA切断など,転写における多くの重要な機能をささえる必須の構造であることが明らかにされた.一方,タイト型のコンホメーションには機能的に異なる2つの状態があり,RNA伸長反応を行うRNAポリメラーゼと転写休止状態あるいは後退状態にあるRNAポリメラーゼでは,後者のほうがラチェット型にシフトしやすい.転写休止状態あるいは後退状態にあるRNAポリメラーゼは,ラチェット型と関連づけられた機能に切り替えるための前段階と考えることもできる.この結果は,RNA合成,転写休止,後退,転写終結,RNA切断など,おのおのの転写の機能がタイト型とラチェット型の2つのコンホメーションに割り当てられており,RNAポリメラーゼの機能がそれらの切り替えによりたくみに制御されていることを示唆した.さらに,RNAポリメラーゼにより生み出されつつある新生RNA鎖の3’末端やヘアピン構造がコンホメーションの切り替えにおいて重要な役割をはたしており,転写の機能の制御に深く関与していることが示唆された.
巨大なタンパク質複合体であるRNAポリメラーゼが転写の状況や種々のシグナルに応じて転写を休止あるいは終結したり,その活性あるいは機能を切り替えたりすることは知られていたが,その具体的な分子機構はよくわかっていなかった.この研究は,RNAポリメラーゼの2つのコンホメーションにそれぞれ異なる機能が割り当てられていることを明らかにし,転写の機能の制御が2つのコンホメーションのあいだの切り替えにより説明できることを提案した.転写および転写制御における基本原理の解明へとつながる重要な一歩と考えている.この発見をもとに,普遍的な転写機構のさらなる理解やそれにもとづいた応用へと研究が発展することを期待する.
文 献
- Zhang, G., Campbell, E. A., Minakhin, L. et al.: Crystal structure of Thermus aquaticus core RNA polymerase at 3.3Å resolution. Cell, 98, 811-824 (1999)[PubMed]
- Cramer, P., Bushnell, D. A. & Kornberg, R. D.: Structural basis of transcription: RNA polymerase II at 2.8 angstrom resolution. Science, 292, 1863-1876 (2001)[PubMed]
- Tagami, S., Sekine, S., Kumarevel, T. et al.: Crystal structure of bacterial RNA polymerase bound with a transcription inhibitor protein. Nature, 468, 978-982 (2010)[PubMed] [新着論文レビュー]
- Komissarova, N. & Kashlev, M.: Transcriptional arrest: Escherichia coli RNA polymerase translocates backward, leaving the 3' end of the RNA intact and extruded. Proc. Natl. Acad. Sci. USA, 94, 1755-1760 (1997)[PubMed]
- Fish, R. N. & Kane, C. M.: Promoting elongation with transcript cleavage stimulatory factors. Biochim. Biophys. Acta, 1577, 287-307 (2002)[PubMed]
- Borukhov, S., Polyakov, A., Nikiforov, V. et al.: GreA protein: a transcription elongation factor from Escherichia coli. Proc. Natl. Acad. Sci. USA, 89, 8899-8902 (1992)[PubMed]
- Landick, R.: The regulatory roles and mechanism of transcriptional pausing. Biochem. Soc. Trans., 34, 1062-1066 (2006)[PubMed]
- Epshtein, V., Cardinale, C. J., Ruckenstein, A. E. et al.: An allosteric path to transcription termination. Mol. Cell, 28, 991-1001 (2007)[PubMed]
- Vassylyev, D. G., Vassylyeva, M. N., Zhang, J. et al.: Structural basis for substrate loading in bacterial RNA polymerase. Nature, 448, 163-168 (2007)[PubMed]
- Wang, D., Bushnell, D. A., Westover, K. D. et al.: Structural basis of transcription: role of the trigger loop in substrate specificity and catalysis. Cell, 127, 941-954 (2006)[PubMed]
著者プロフィール
略歴:1997年 東京大学大学院理学系研究科にて博士号取得,同年 理化学研究所 研究員,2004年 東京大学大学院理学系研究科 講師,特任准教授,2012年 理化学研究所 上級研究員を経て,2013年より理化学研究所ライフサイエンス技術基盤研究センター チームリーダー.
村山 祐子(Yuko Murayama)
略歴:2013年 東京大学大学院理学系研究科にて博士号取得,同年より理化学研究所ライフサイエンス技術基盤研究センター 特別研究員.
横山 茂之(Shigeyuki Yokoyama)
理化学研究所横山構造生物学研究室 上席研究員.
© 2015 関根俊一・村山祐子・横山茂之 Licensed under CC 表示 2.1 日本