ゲノムワイドな解析により解き明かされたDicerの新たな標的RNAと機能
村川 泰裕
(ドイツMax Delbrück Center for Molecular Medicine,Laboratory for RNA Biology and Posttranscriptional Regulation)
email:村川泰裕
DOI: 10.7875/first.author.2014.155
A variety of Dicer substrates in human and C. elegans.
Agnieszka Rybak-Wolf, Marvin Jens, Yasuhiro Murakawa, Margareta Herzog, Markus Landthaler, Nikolaus Rajewsky
Cell, 159, 1153-1167 (2014)
Dicerは複数のドメインからなるRNA切断酵素として知られ,miRNAやsiRNAなど小分子RNAの生成において中心的な役割をもつが,その標的となるRNAの全貌は不明であった.今回,筆者らは,次世代シークエンサーを活用した網羅的な手法を用い,Dicerと直接的に結合する標的RNAをヒトおよび線虫においてゲノムワイドに同定した.その結果,数千ものDicer結合部位が同定され,Dicerの基質である既知のmiRNA前駆体にくわえ,ヒトにおい数百もの新規のmiRNAが同定された.さらに,Dicerの標的RNAには,miRNAのみならずtRNAやミトコンドリアRNAなども含まれていた.ヒトにおいても線虫においても,大多数のDicer結合部位はmRNAに存在し,小分子RNAは生成していなかった.DicerはこれらのmRNAに直接的に結合することによりその安定性などを制御していることが明らかになり,Dicerの新たな機能が示された.
ゲノムDNAに存在する遺伝子はRNAポリメラーゼによりmRNAへと転写され,転写されたmRNAはさらにリボソームによりタンパク質へと翻訳される1).ヒトでは,約2万の遺伝子がこのおのおのの段階において精巧な発現制御をうけ,同一のゲノムから多様な細胞や機能が生み出される.より具体的には,DNAからmRNAへの転写のレベルはエピジェネティック制御タンパク質や転写因子などにより制御されているが2),転写されたmRNAも多くのRNA結合タンパク質により,スプライシング,局在,安定性の制御,翻訳の制御など,さまざまな転写後制御をうける.最近の研究により,mRNAに結合するRNA結合タンパク質は約1000種類も存在すると考えられており,転写後制御の重要性が示唆される3).
近年,タンパク質をコードしない非コードRNAが多く発見され,それらのなかでも,miRNA(microRNA)とよばれる約22塩基長の小分子RNAが転写後制御を担っていることが明らかにされた4).現在,ヒトでは2000種類近くのmiRNAがmiRBaseデータベースに登録されている.miRNAは長鎖非コードRNAやイントロンに存在するヘアピンループ構造をとるRNAから生成される.ヘアピンループ構造は核においてDGCR8とDROSHAからなるMicroprocessor複合体により切り出され,miRNA前駆体となったのち細胞質へと輸送され,RNA切断酵素であるDicerによりさらにループ構造が取り除かれる.この際,DicerのPAZドメインがmiRNA前駆体の5’末端のリン酸基および3’側の突出末端を認識し,RNA切断活性をもつRNase IIIドメインまでの物理的な距離を利用して約22塩基長に切断する5).このようにして成熟したmiRNAは,AGO(Argonaute)を中心とするRISC(RNA-induced silencing complex)にうけわたされ,RISCはmiRNAに相補的な配列をもつmRNAと結合し,mRNAの不安定化や翻訳の抑制により転写後のレベルにおいて遺伝子発現を抑制することが知られている4)(AGOによる遺伝子発現の抑制の構造基盤については,佐々木 浩・泊 幸秀, 領域融合レビュー, 1, e001, 2012 も参照されたい).
次世代シークエンサーの普及によりRNA結合タンパク質の標的となるRNAがゲノムワイドに解析されるにつれ,ひとつのRNA結合タンパク質がRNAに対し数百から数万もの結合部位をもち多彩な機能を発揮しうることが明らかになりつつある6,7).これまで,DicerはmiRNAやsiRNA(small interfering RNA)などの小分子RNAの生成において中心的なRNA切断酵素として知られてきたが,Dicerが核8),核膜孔9),さらに,生殖細胞においてRNA顆粒に存在するとの報告10) などから,Dicerのもつ未知の機能も示唆されており,Dicerの標的となるRNAの全貌は不明であった.
Dicerと結合するRNAをゲノムワイドに同定するため,PAR-CLIP(photoactivatable-ribonucleoside-enhanced crosslinking and immunoprecipitation)法6) とよばれる網羅的な手法を用いた.光励起性の人工のウリジン類似体である4-チオウリジンは培地に添加すると生合成されているRNAに取り込まれる.そののち,波長365 nmの紫外線を照射すると4-チオウリジンは励起し,RNA結合タンパク質とその標的となるRNAは共有結合により架橋される.FLAGタグを付加したDicerを発現したヒトHEK293細胞株に4-チオウリジンを添加し紫外線を照射し,その抽出液からDicerと標的RNAとの複合体を抗FLAG抗体を用いた免疫沈降法により濃縮し,RNAをヌクレアーゼにより約19~35塩基長に切断,さらに,SDS-ポリアクリルアミドゲル電気泳動により精製した.Dicerに直接的に結合しているRNAを回収したのち,cDNAライブラリーを作製し,次世代シークエンサーにより塩基配列を解読することによりDicerの標的となるRNAを同定した.PAR-CLIP法の利点として,cDNAライブラリーの作製の過程でタンパク質とRNAとの架橋の部位においてTからCへの塩基置換の起こることが知られており,配列データを解析することによりRNA結合タンパク質と標的RNAとの結合部位を1塩基のレベルで同定できる.同様のiPAR-CLIP(in vivo PAR-CLIP)法7) とよばれる手法を線虫の個体に対し用いることにより,線虫においてもDicerの標的となるRNAを同定し,Dicerの標的RNAのヒトと線虫とのあいだでの進化的な保存度を評価した.
その結果,これまで知られていたDicerの基質であるmiRNA前駆体はPAR-CLIP法により高感度に同定された.興味深いことに,miRNAはDicer結合部位の一部にすぎず,tRNA,ミトコンドリアRNA,プロモーターRNA,mRNAのコード領域,3’側非翻訳領域,イントロンにも多数のDicer結合部位が同定され,ヒトで約8500箇所,線虫で約2500箇所のDicer結合部位がゲノムワイドに同定された.
同定されたおのおののDicer結合部位においてDicerのRNA切断活性を評価する目的で,ヒトにおいてAGOに取り込まれた小分子RNAの塩基配列を次世代シークエンサーにより解読し,Dicer結合部位と照らし合わせた.その結果,小分子RNAを生成していたDicer結合部位は既知のmiRNAを含めわずか約24%であった.これら1978箇所のDicer結合部位はDicerに認識されAGOに取り込まれる小分子RNAを生成することを意味し,DicerのRNA切断活性が示唆されたことから活性結合部位と名づけた.しかし,生成された小分子RNAのディープシークエンシングデータを詳細に調べたところ,古典的なmiRNAの特徴は必ずしも認められず,Dicerは基質ごとにさまざまな忠実度によりRNAを切断し古典的なmiRNA以外の小分子RNAも生成している可能性が考えられた.この傾向は,tRNAやミトコンドリアRNAのDicer結合部位において多く認められ,Dicerがこれらの代謝にも関与している可能性が示唆された.
1978箇所の活性結合部位はmiRBaseデータベースに登録されていた300の既知のmiRNAを含んでいたが,in silicoにおいてmiRDeep2 11) とよばれるプログラムを利用することにより,残りの1678箇所の活性結合部位からも新規のmiRNAを探索した.すなわち,AGOに取り込まれた小分子RNAのディープシークエンシングデータからmiRNAの候補をリストアップし,Dicer結合部位と重ね合わせることにより,確信度の高い212の新規のmiRNAを同定した.また,PAR-CLIP法という生化学的な実験により得られたDicer結合部位はin vivoにおけるヘアピンステムループ構造を示唆していたため,in silicoにおける予測によるヘアピンステムループ構造のスコアを取り除き,さらに155の新規のmiRNAの同定にいたった.近年,小分子RNAの塩基配列のデータを膨大に蓄積し,その解析により新規のmiRNAを同定しようとする試みがなされているが,in silicoにおける予測では発見の困難なmiRNAがPAR-CLIP法により高感度に同定できることが示された.ほかの組織や細胞株における同様のアプローチにより,多くの新規のmiRNAが同定されると期待される.
今回,ヒトにおいて合計で367の確信度の高い新規のmiRNAを発見した.これらmiRNAのHEK293細胞における発現量は総じて低かったが,Dicerのノックダウンにより多くの発現量が減少したことから,これらmiRNAのDicerに対する依存性がさらに確認された.また,これらのmiRNAは,mRNAの5’非翻訳領域,コード領域,3’側非翻訳領域を含むゲノムのさまざまな領域から生成されていた.
ヒトにおいて7割以上のDicer結合部位からは,実質的に小分子RNAは生成されていないことが明らかになり,これを受動性結合部位と命名した.受動性結合部位の多くはmRNAのコード領域や3’非翻訳領域に存在した.同様に,線虫においても8割以上ものDicer結合部位は受動性結合部位であった.
生化学的な実験により,Dicerは受動性結合部位においてmRNAのヘアピンループ構造に直接的に結合していることが示された.さらに,受動性結合部位の2次構造を詳細に解析した.既知のmiRNAを生成する部位の構造は,成熟したmiRNAの22塩基長より11塩基ほど長いステム領域をもち,このステム領域はMicroprocessor複合体による認識およびmiRNA前駆体の切り出しに必要である12).それに対して,受動性結合部位では概してヘアピンループ構造を形成する傾向が認められたものの,ステム領域はより短いと予測された.また,受動性結合部位はMicroprocessor複合体により切り出されてはいないことが確認された.PAR-CLIP法にてタンパク質とRNAとの架橋部位において生じるTからCへの塩基置換の頻度の分布を解析することによりDicerと標的RNAとの結合様式を詳細に解析した.その結果,既知のmiRNAの生成部位に関しては前駆体の末端に対応する位置に数多くの塩基置換が認められ,これはDicerのPAZドメインによるmiRNA前駆体との結合を反映していると考えられた.それに対し,受動性結合部位ではヘアピンループ構造のループ領域に塩基置換が認められたのみで,おもにDicerのヘリカーゼドメインにより認識されていると推測された.これらの解析により,受動性結合部位においてはmRNAに存在する短いヘアピンループ構造が異なる様式でDicerにより直接的に認識されていることが示された.
受動性結合部位が生物学的な機能をもちうるかどうか検討した.ヒトの細胞において受動性結合部位を強制発現することによりmiRNAの発現量が減少したことから,受動性結合部位がmiRNAの生成を競合的に阻害する可能性が示唆された.また,ヒトにおいても線虫においても,受動性結合部位しかもたないmRNAにはRNA顆粒の構成タンパク質をコードするmRNAの濃縮が認められた.線虫の生殖系の細胞においてはDicerがRNA顆粒に存在しその形成にも必要であることから,RNA顆粒の構成タンパク質をコードするmRNAに対しDicerがなんらかの制御していることが示唆された.さらに,Dicerをノックダウンしたのちのトランスクリプトームを解析したところ,受動性結合部位しかもたないmRNAは全体としてわずかではあるが不安定化しており,この傾向は線虫とのあいだで進化的に保存されたmRNAにおいてより顕著であった.したがって,DicerがmRNAを直接的に認識しその安定性を制御するといった新しい機能も明らかにされた.
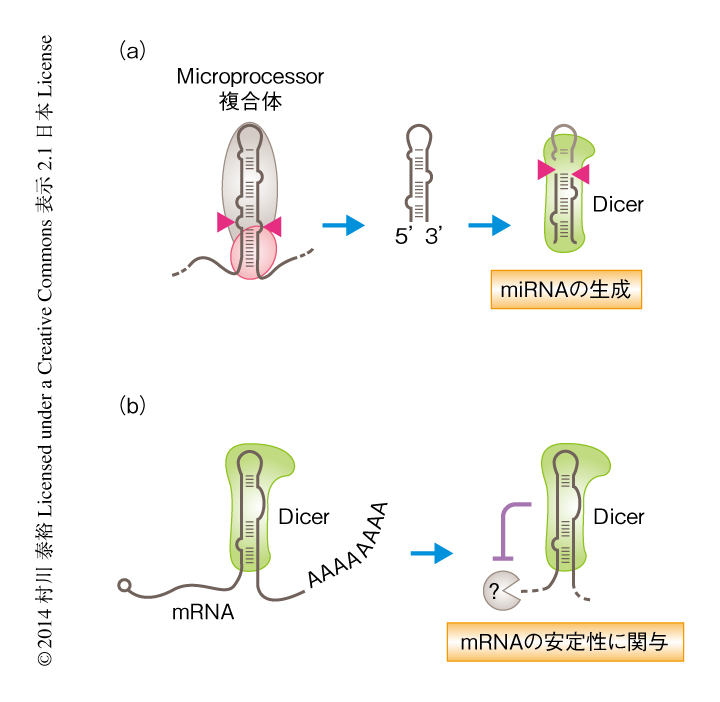
これまで,DicerはmiRNAやsiRNAなどの小分子RNAの生成において中心的な役割をもつRNA切断酵素として知られていたが(図1a),Dicerの結合部位をゲノムワイドに解析することにより,ヒトのひとつの細胞株において数百もの新規のmiRNAが同定されただけでなく,DicerがmRNAを直接的に認識しその安定性に関与するといった新しい機能も明らかになった(図1b).さらに,Dicerの標的となるRNAとしてtRNAやミトコンドリアRNAなども同定されており,Dicerのより多くの機能が示唆された.同様に,最近の研究により,Microprocessor複合体の構成タンパク質であるDGCR8においても網羅的な結合部位の同定によりmiRNAの生成以外の経路における新しい機能が示された13).網羅的な解析手法の進歩により,ひとつのRNA結合タンパク質が多くのRNAの構造を柔軟かつ精巧に認識することによりさまざまな機能を発揮しうることが示されつつあるが,これら転写後制御ネットワークの解明は細胞の多様性や高次機能を理解するうえで重要であると思われる.
略歴:2014年 ドイツFree University of Berlinにて博士号取得,ドイツMax Delbrück Center for Molecular Medicine研究員.
関心事:人はどのようにして生まれ年齢を重ねるのか? がんはなぜ生じるのか? そして,寿命はどのように規定されるのか? 生老病死の四天使を遺伝子発現制御という観点からどこまで理解そして制御できるのかに興味がある.
© 2014 村川 泰裕 Licensed under CC 表示 2.1 日本
(ドイツMax Delbrück Center for Molecular Medicine,Laboratory for RNA Biology and Posttranscriptional Regulation)
email:村川泰裕
DOI: 10.7875/first.author.2014.155
A variety of Dicer substrates in human and C. elegans.
Agnieszka Rybak-Wolf, Marvin Jens, Yasuhiro Murakawa, Margareta Herzog, Markus Landthaler, Nikolaus Rajewsky
Cell, 159, 1153-1167 (2014)
要 約
Dicerは複数のドメインからなるRNA切断酵素として知られ,miRNAやsiRNAなど小分子RNAの生成において中心的な役割をもつが,その標的となるRNAの全貌は不明であった.今回,筆者らは,次世代シークエンサーを活用した網羅的な手法を用い,Dicerと直接的に結合する標的RNAをヒトおよび線虫においてゲノムワイドに同定した.その結果,数千ものDicer結合部位が同定され,Dicerの基質である既知のmiRNA前駆体にくわえ,ヒトにおい数百もの新規のmiRNAが同定された.さらに,Dicerの標的RNAには,miRNAのみならずtRNAやミトコンドリアRNAなども含まれていた.ヒトにおいても線虫においても,大多数のDicer結合部位はmRNAに存在し,小分子RNAは生成していなかった.DicerはこれらのmRNAに直接的に結合することによりその安定性などを制御していることが明らかになり,Dicerの新たな機能が示された.
はじめに
ゲノムDNAに存在する遺伝子はRNAポリメラーゼによりmRNAへと転写され,転写されたmRNAはさらにリボソームによりタンパク質へと翻訳される1).ヒトでは,約2万の遺伝子がこのおのおのの段階において精巧な発現制御をうけ,同一のゲノムから多様な細胞や機能が生み出される.より具体的には,DNAからmRNAへの転写のレベルはエピジェネティック制御タンパク質や転写因子などにより制御されているが2),転写されたmRNAも多くのRNA結合タンパク質により,スプライシング,局在,安定性の制御,翻訳の制御など,さまざまな転写後制御をうける.最近の研究により,mRNAに結合するRNA結合タンパク質は約1000種類も存在すると考えられており,転写後制御の重要性が示唆される3).
近年,タンパク質をコードしない非コードRNAが多く発見され,それらのなかでも,miRNA(microRNA)とよばれる約22塩基長の小分子RNAが転写後制御を担っていることが明らかにされた4).現在,ヒトでは2000種類近くのmiRNAがmiRBaseデータベースに登録されている.miRNAは長鎖非コードRNAやイントロンに存在するヘアピンループ構造をとるRNAから生成される.ヘアピンループ構造は核においてDGCR8とDROSHAからなるMicroprocessor複合体により切り出され,miRNA前駆体となったのち細胞質へと輸送され,RNA切断酵素であるDicerによりさらにループ構造が取り除かれる.この際,DicerのPAZドメインがmiRNA前駆体の5’末端のリン酸基および3’側の突出末端を認識し,RNA切断活性をもつRNase IIIドメインまでの物理的な距離を利用して約22塩基長に切断する5).このようにして成熟したmiRNAは,AGO(Argonaute)を中心とするRISC(RNA-induced silencing complex)にうけわたされ,RISCはmiRNAに相補的な配列をもつmRNAと結合し,mRNAの不安定化や翻訳の抑制により転写後のレベルにおいて遺伝子発現を抑制することが知られている4)(AGOによる遺伝子発現の抑制の構造基盤については,佐々木 浩・泊 幸秀, 領域融合レビュー, 1, e001, 2012 も参照されたい).
次世代シークエンサーの普及によりRNA結合タンパク質の標的となるRNAがゲノムワイドに解析されるにつれ,ひとつのRNA結合タンパク質がRNAに対し数百から数万もの結合部位をもち多彩な機能を発揮しうることが明らかになりつつある6,7).これまで,DicerはmiRNAやsiRNA(small interfering RNA)などの小分子RNAの生成において中心的なRNA切断酵素として知られてきたが,Dicerが核8),核膜孔9),さらに,生殖細胞においてRNA顆粒に存在するとの報告10) などから,Dicerのもつ未知の機能も示唆されており,Dicerの標的となるRNAの全貌は不明であった.
1.Dicerの標的となるRNAのゲノムワイドな同定
Dicerと結合するRNAをゲノムワイドに同定するため,PAR-CLIP(photoactivatable-ribonucleoside-enhanced crosslinking and immunoprecipitation)法6) とよばれる網羅的な手法を用いた.光励起性の人工のウリジン類似体である4-チオウリジンは培地に添加すると生合成されているRNAに取り込まれる.そののち,波長365 nmの紫外線を照射すると4-チオウリジンは励起し,RNA結合タンパク質とその標的となるRNAは共有結合により架橋される.FLAGタグを付加したDicerを発現したヒトHEK293細胞株に4-チオウリジンを添加し紫外線を照射し,その抽出液からDicerと標的RNAとの複合体を抗FLAG抗体を用いた免疫沈降法により濃縮し,RNAをヌクレアーゼにより約19~35塩基長に切断,さらに,SDS-ポリアクリルアミドゲル電気泳動により精製した.Dicerに直接的に結合しているRNAを回収したのち,cDNAライブラリーを作製し,次世代シークエンサーにより塩基配列を解読することによりDicerの標的となるRNAを同定した.PAR-CLIP法の利点として,cDNAライブラリーの作製の過程でタンパク質とRNAとの架橋の部位においてTからCへの塩基置換の起こることが知られており,配列データを解析することによりRNA結合タンパク質と標的RNAとの結合部位を1塩基のレベルで同定できる.同様のiPAR-CLIP(in vivo PAR-CLIP)法7) とよばれる手法を線虫の個体に対し用いることにより,線虫においてもDicerの標的となるRNAを同定し,Dicerの標的RNAのヒトと線虫とのあいだでの進化的な保存度を評価した.
その結果,これまで知られていたDicerの基質であるmiRNA前駆体はPAR-CLIP法により高感度に同定された.興味深いことに,miRNAはDicer結合部位の一部にすぎず,tRNA,ミトコンドリアRNA,プロモーターRNA,mRNAのコード領域,3’側非翻訳領域,イントロンにも多数のDicer結合部位が同定され,ヒトで約8500箇所,線虫で約2500箇所のDicer結合部位がゲノムワイドに同定された.
2.ヒトにおける数百の新規のmiRNAの同定
同定されたおのおののDicer結合部位においてDicerのRNA切断活性を評価する目的で,ヒトにおいてAGOに取り込まれた小分子RNAの塩基配列を次世代シークエンサーにより解読し,Dicer結合部位と照らし合わせた.その結果,小分子RNAを生成していたDicer結合部位は既知のmiRNAを含めわずか約24%であった.これら1978箇所のDicer結合部位はDicerに認識されAGOに取り込まれる小分子RNAを生成することを意味し,DicerのRNA切断活性が示唆されたことから活性結合部位と名づけた.しかし,生成された小分子RNAのディープシークエンシングデータを詳細に調べたところ,古典的なmiRNAの特徴は必ずしも認められず,Dicerは基質ごとにさまざまな忠実度によりRNAを切断し古典的なmiRNA以外の小分子RNAも生成している可能性が考えられた.この傾向は,tRNAやミトコンドリアRNAのDicer結合部位において多く認められ,Dicerがこれらの代謝にも関与している可能性が示唆された.
1978箇所の活性結合部位はmiRBaseデータベースに登録されていた300の既知のmiRNAを含んでいたが,in silicoにおいてmiRDeep2 11) とよばれるプログラムを利用することにより,残りの1678箇所の活性結合部位からも新規のmiRNAを探索した.すなわち,AGOに取り込まれた小分子RNAのディープシークエンシングデータからmiRNAの候補をリストアップし,Dicer結合部位と重ね合わせることにより,確信度の高い212の新規のmiRNAを同定した.また,PAR-CLIP法という生化学的な実験により得られたDicer結合部位はin vivoにおけるヘアピンステムループ構造を示唆していたため,in silicoにおける予測によるヘアピンステムループ構造のスコアを取り除き,さらに155の新規のmiRNAの同定にいたった.近年,小分子RNAの塩基配列のデータを膨大に蓄積し,その解析により新規のmiRNAを同定しようとする試みがなされているが,in silicoにおける予測では発見の困難なmiRNAがPAR-CLIP法により高感度に同定できることが示された.ほかの組織や細胞株における同様のアプローチにより,多くの新規のmiRNAが同定されると期待される.
今回,ヒトにおいて合計で367の確信度の高い新規のmiRNAを発見した.これらmiRNAのHEK293細胞における発現量は総じて低かったが,Dicerのノックダウンにより多くの発現量が減少したことから,これらmiRNAのDicerに対する依存性がさらに確認された.また,これらのmiRNAは,mRNAの5’非翻訳領域,コード領域,3’側非翻訳領域を含むゲノムのさまざまな領域から生成されていた.
3.DicerはmRNAを直接的に認識しこれを制御しうる
ヒトにおいて7割以上のDicer結合部位からは,実質的に小分子RNAは生成されていないことが明らかになり,これを受動性結合部位と命名した.受動性結合部位の多くはmRNAのコード領域や3’非翻訳領域に存在した.同様に,線虫においても8割以上ものDicer結合部位は受動性結合部位であった.
生化学的な実験により,Dicerは受動性結合部位においてmRNAのヘアピンループ構造に直接的に結合していることが示された.さらに,受動性結合部位の2次構造を詳細に解析した.既知のmiRNAを生成する部位の構造は,成熟したmiRNAの22塩基長より11塩基ほど長いステム領域をもち,このステム領域はMicroprocessor複合体による認識およびmiRNA前駆体の切り出しに必要である12).それに対して,受動性結合部位では概してヘアピンループ構造を形成する傾向が認められたものの,ステム領域はより短いと予測された.また,受動性結合部位はMicroprocessor複合体により切り出されてはいないことが確認された.PAR-CLIP法にてタンパク質とRNAとの架橋部位において生じるTからCへの塩基置換の頻度の分布を解析することによりDicerと標的RNAとの結合様式を詳細に解析した.その結果,既知のmiRNAの生成部位に関しては前駆体の末端に対応する位置に数多くの塩基置換が認められ,これはDicerのPAZドメインによるmiRNA前駆体との結合を反映していると考えられた.それに対し,受動性結合部位ではヘアピンループ構造のループ領域に塩基置換が認められたのみで,おもにDicerのヘリカーゼドメインにより認識されていると推測された.これらの解析により,受動性結合部位においてはmRNAに存在する短いヘアピンループ構造が異なる様式でDicerにより直接的に認識されていることが示された.
受動性結合部位が生物学的な機能をもちうるかどうか検討した.ヒトの細胞において受動性結合部位を強制発現することによりmiRNAの発現量が減少したことから,受動性結合部位がmiRNAの生成を競合的に阻害する可能性が示唆された.また,ヒトにおいても線虫においても,受動性結合部位しかもたないmRNAにはRNA顆粒の構成タンパク質をコードするmRNAの濃縮が認められた.線虫の生殖系の細胞においてはDicerがRNA顆粒に存在しその形成にも必要であることから,RNA顆粒の構成タンパク質をコードするmRNAに対しDicerがなんらかの制御していることが示唆された.さらに,Dicerをノックダウンしたのちのトランスクリプトームを解析したところ,受動性結合部位しかもたないmRNAは全体としてわずかではあるが不安定化しており,この傾向は線虫とのあいだで進化的に保存されたmRNAにおいてより顕著であった.したがって,DicerがmRNAを直接的に認識しその安定性を制御するといった新しい機能も明らかにされた.
おわりに
これまで,DicerはmiRNAやsiRNAなどの小分子RNAの生成において中心的な役割をもつRNA切断酵素として知られていたが(図1a),Dicerの結合部位をゲノムワイドに解析することにより,ヒトのひとつの細胞株において数百もの新規のmiRNAが同定されただけでなく,DicerがmRNAを直接的に認識しその安定性に関与するといった新しい機能も明らかになった(図1b).さらに,Dicerの標的となるRNAとしてtRNAやミトコンドリアRNAなども同定されており,Dicerのより多くの機能が示唆された.同様に,最近の研究により,Microprocessor複合体の構成タンパク質であるDGCR8においても網羅的な結合部位の同定によりmiRNAの生成以外の経路における新しい機能が示された13).網羅的な解析手法の進歩により,ひとつのRNA結合タンパク質が多くのRNAの構造を柔軟かつ精巧に認識することによりさまざまな機能を発揮しうることが示されつつあるが,これら転写後制御ネットワークの解明は細胞の多様性や高次機能を理解するうえで重要であると思われる.
文 献
- Schwanhausser, B., Busse, D., Li, N. et al.: Global quantification of mammalian gene expression control. Nature, 473, 337-342 (2011)[PubMed]
- ENCODE Project Consortium: An integrated encyclopedia of DNA elements in the human genome. Nature, 489, 57-74 (2012)[PubMed]
- Baltz, A. G., Munschauer, M., Schwanhausser, B. et al.: The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts. Mol. Cell, 46, 674-690 (2012)[PubMed]
- Bartel, D. P.: MicroRNAs: target recognition and regulatory functions. Cell, 136, 215-233 (2009)[PubMed]
- Lau, P. W., Guiley, K. Z., De, N. et al.: The molecular architecture of human Dicer. Nat. Struct. Mol. Biol., 19, 436-440 (2012)[PubMed]
- Hafner, M., Landthaler, M., Burger, L. et al.: Transcriptome-wide identification of RNA-binding protein and microRNA target sites by PAR-CLIP. Cell, 141, 129-141 (2010)[PubMed]
- Jungkamp, A. C., Stoeckius, M., Mecenas, D. et al.: In vivo and transcriptome-wide identification of RNA binding protein target sites. Mol. Cell, 44, 828-840 (2011)[PubMed]
- Doyle, M., Badertscher, L., Jaskiewicz, L. et al.: The double-stranded RNA binding domain of human Dicer functions as a nuclear localization signal. RNA, 19, 1238-1252 (2013)[PubMed]
- Ando, Y., Tomaru, Y., Morinaga, A. et al.: Nuclear pore complex protein mediated nuclear localization of dicer protein in human cells. PLoS One, 6, e23385 (2011)[PubMed]
- Beshore, E. L., McEwen, T. J., Jud, M. C. et al.: C. elegans Dicer interacts with the P-granule component GLH-1 and both regulate germline RNPs. Dev. Biol., 350, 370-381 (2011)[PubMed]
- Friedlander, M. R., Mackowiak, S. D., Li, N. et al.: miRDeep2 accurately identifies known and hundreds of novel microRNA genes in seven animal clades. Nucleic Acids Res., 40, 37-52 (2012)[PubMed]
- Han, J., Lee, Y., Yeom, K. H. et al.: Molecular basis for the recognition of primary microRNAs by the Drosha-DGCR8 complex. Cell, 125, 887-901 (2006)[PubMed]
- Macias, S., Plass, M., Stajuda, A. et al.: DGCR8 HITS-CLIP reveals novel functions for the Microprocessor. Nat. Struct. Mol. Biol., 19, 760-766 (2012)[PubMed]
著者プロフィール
略歴:2014年 ドイツFree University of Berlinにて博士号取得,ドイツMax Delbrück Center for Molecular Medicine研究員.
関心事:人はどのようにして生まれ年齢を重ねるのか? がんはなぜ生じるのか? そして,寿命はどのように規定されるのか? 生老病死の四天使を遺伝子発現制御という観点からどこまで理解そして制御できるのかに興味がある.
© 2014 村川 泰裕 Licensed under CC 表示 2.1 日本