80Sリボソーム,リボソーム内部進入部位RNA,開始tRNA,翻訳開始因子eIF5Bからなる複合体のクライオ電子顕微鏡による構造解析
山本 紘・Anett Unbehaun・Christian M. T. Spahn
(ドイツCharité – Universitätsmedizin Berlin,Institute of Medical Physics and Biophysics)
email:山本 紘
DOI: 10.7875/first.author.2014.116
Structure of the mammalian 80S initiation complex with initiation factor 5B on HCV-IRES RNA.
Hiroshi Yamamoto, Anett Unbehaun, Justus Loerke, Elmar Behrmann, Marianne Collier, Jörg Bürger, Thorsten Mielke, Christian M. T. Spahn
Nature Structural & Molecular Biology, 21, 721-727 (2014)
真核生物の翻訳開始因子のひとつeIF5Bは,原核生物から真核生物まで高度に保存されたGTPase活性をもつタンパク質であり,翻訳開始反応に不可欠である.eIF5BはC型肝炎ウイルスに由来するリボソーム内部進入部位RNAを介した翻訳開始反応においてもはたらくことが知られている.eIF5Bは翻訳開始反応において,開始tRNAの結合した40Sサブユニットと60Sサブユニットとの会合を促進し80Sリボソームを形成する.しかしながら,eIF5BがどのようにGTPの加水分解を介しサブユニットの会合を促進するのかは解明されていない.今回,筆者らは,真核生物の80Sリボソーム,C型肝炎ウイルスに由来するリボソーム内部進入部位RNA,開始tRNA,翻訳開始因子eIF5Bからなる複合体について,GTPのアナログであるGMPPNPを用いることによりeIF5Bを固定し,クライオ電子顕微鏡によりその構造を解析した.得られた画像の解析から2つの構造が得られ,それらの構造は40Sサブユニットの回転運動,および,開始tRNAの位置の違いから区別することができた.この2つの構造を比較することにより,eIF5Bはリボソームにおける開始tRNAの位置を変えることによりサブユニットの会合を促進していることが予想され,eIF5BはtRNAの再配置にはたらくことが明らかにされた.
真核生物における翻訳開始反応は非常に厳密に制御されている.この反応は,40Sサブユニットと翻訳開始因子との複合体が60Sサブユニットと会合することにより80Sリボソームが形成されることで達成される.少なくとも12個の翻訳開始因子がこの反応に関与しており,開始tRNAおよび5’末端にキャップ構造をもつmRNAの40Sサブユニットへの結合,40SサブユニットがmRNAをスキャンする際の開始tRNAとの相互作用の制御,開始コドンの選択,40Sサブユニットと60Sサブユニットとの会合の促進,などの機能が知られている1).この反応の最後のステップにおいて,GTPase活性をもつ翻訳開始因子eIF5Bは40Sサブユニットと60Sサブユニットとの会合を促進する2).eIF5Bは原核生物の翻訳開始因子であるIF2のオーソログであり普遍的に保存されている.この遺伝子の欠損は出芽酵母においては生育の遅延をひき起こし,ショウジョウバエや哺乳類細胞では生育に必須である.また,eIF5Bはキャップ構造に依存しない,リボソーム内部進入部位(internal ribosomal entry site:IRES)を介した翻訳開始反応に必要なタンパク質のひとつでもある.リボソーム内部進入部位RNAは一部の翻訳開始因子のみを必要とする特殊な翻訳開始機構をもち,C型肝炎ウイルスやブタコレラウイルスに由来するものが知られている3).C型肝炎ウイルスに由来するリボソーム内部進入部位RNAは40Sサブユニットと高い親和性で会合し,GTPと結合したeIF2と開始tRNAとの複合体をリボソームのP部位に受け入れる.eIF5はeIF2のもつGTPase活性を促進することによりeIF2とGDPとの解離をひき起こし,eIF5Bを介したサブユニットの会合に進行させる4,5).生体におけるアミノ酸の不足による飢餓状態などにおいてはeIF2のαサブユニットはリン酸化されてその機能を失うが,C型肝炎ウイルスに由来するリボソーム内部進入部位RNAはeIF5Bが直接に作用する経路によりサブユニットの会合に進行することができる.リボソームと結合したeIF5Bについての構造的な知見は少なく,原核生物および出芽酵母における通常の翻訳開始反応を介したIF2あるいはeIF5Bとの複合体の構造は報告されているものの,eIF5BのもつGTPase活性を介したサブユニットの会合はどのように促進されるのか,また,開始tRNAのリボソームにおける適切な配置はどのようになされるのかなどについては,いまだ解明されていない6,7).そこで,筆者らは,80Sリボソーム,C型肝炎ウイルスに由来するリボソーム内部進入部位RNA,開始tRNA,翻訳開始因子eIF5Bからなる複合体をクライオ電子顕微鏡により構造解析することで,eIF5Bのリボソームにおいての役割について調べた.
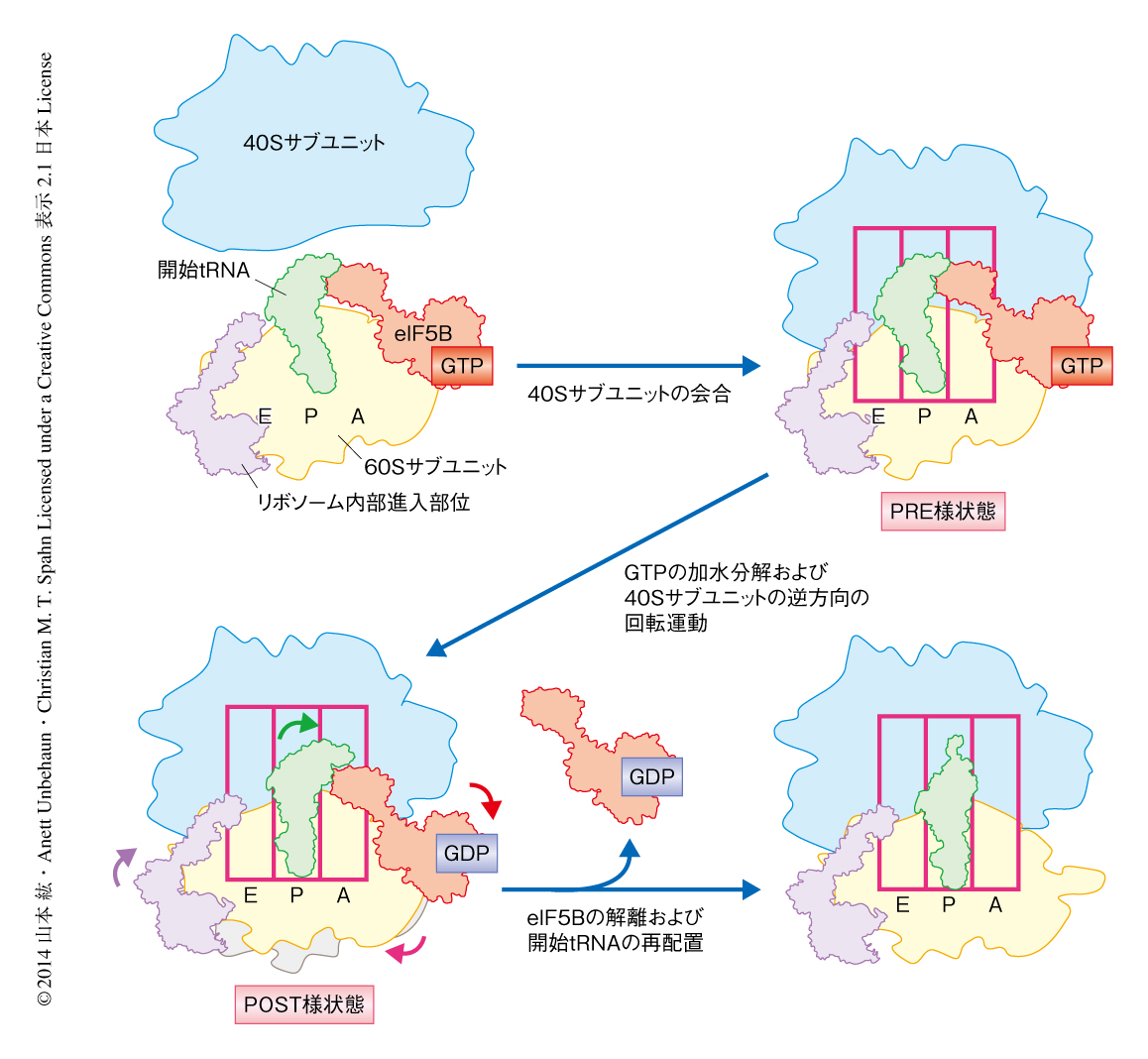
翻訳開始因子であるeIF2およびeIF5を介さない系においてリボソームを調製し,また,GTPの非加水分解アナログであるGMPPNPを用いることにより翻訳開始因子eIF5Bを固定した.電子顕微鏡により得られた画像をマルチパーティクルクライオ電子顕微鏡法により解析することにより8),2つの構造を分解能8.9Å(EMDB ID:2682,図1),および,分解能9.5Å(EMDB ID:2683)で得た.この2つの構造は,40Sサブユニットの回転運動の大きさ,開始tRNAの位置の違いにより区別された(図1b).興味深いことに,観察された40Sサブユニットの回転運動は,原核生物のリボソームにおいてよく知られているものとは異なり,最近,真核生物においてポリペプチド鎖の伸長段階のリボソームにて明らかにされたものと類似していた9).この回転運動は,40Sサブユニットに垂直な軸に対して直交した方向に回転するものであり約6度の回転が観察された.得られた2つの構造をポリペプチド鎖の伸長段階のリボソームの構造と比較した結果,伸長段階のPRE状態およびPOST状態と類似していた.このことから,この2つの構造をPRE様状態およびPOST様状態とよぶことにした.
PRE様状態とPOST様状態の構造を比較したところ,40Sサブユニットに結合しているeIF5B-開始tRNA複合体について,リボソームに対する構造変化はほとんどなく,逆方向の回転運動によりeIF5B-開始tRNA複合体の位置は40Sサブユニットといっしょに移動していることがわかった(図1b).観察されたPRE様状態における開始tRNAの位置は,原核生物においてよく知られている翻訳開始反応に特異的な位置をとっていたが,開始tRNAの“ひじ”およびアミノ酸受容末端の位置が少し異なっていた.それとは対照的に,POST様状態において開始tRNAは新規の位置をとっていた.最近の出芽酵母におけるeIF5Bのクライオ電子顕微鏡による解析により得られた構造は,PRE様状態に類似している.しかしながら,今回のPOST様状態は,出芽酵母7) あるいは原核生物6) において観察されておらず,まったく新規の状態であった.また,PRE様状態のeIF5BはGTPase活性に深く関与するリボソームRNAのサルシン-リシンループと結合していたのに対し,POST様状態ではeIF5Bはサルシン-リシンループから解離していた.このことから,PRE様状態はGTPの加水分解の起こるまえの状態であり,POST様状態はGTPの加水分解ののちの状態であることが推測された.
C型肝炎ウイルスに由来するリボソーム内部進入部位RNAのコアドメインは,ドメインII,ドメインIII,ドメインIV,シュードノットから構成されており3),翻訳開始因子に対し非依存的に調製した40Sサブユニットとリボソーム内部進入部位RNAとの複合体と比較すると,ドメインIIに大きな変化がみられた.80SリボソームではドメインIIbは40Sサブユニットから解離しており,40Sサブユニットにおいてみられた傾きは解消していることがわかった.また,解離したドメインIIbは開始tRNAの“ひじ”に結合していることが観察された.また,この結合はPOST様状態ではみられなかった.ドメインIIはリボソームとの結合能には関与しないが翻訳開始反応に不可欠であることが知られており4,5),今回の知見から,リボソーム内部進入部位RNAのドメインIIは開始tRNAの位置どりを助けていると推察された.
翻訳開始因子IF2およびeIF5Bはリボソームのサブユニットの会合を促進することが知られている1,2,6).ポリペプチド鎖の伸長段階のリボソームにおいてP部位にあるtRNAをみてみると,CCA末端はペプチジルトランスフェラーゼの活性中心のある溝に結合している.しかしながら,開始tRNAの直接的な結合は中央突起とヘリックス69により阻まれており,開始tRNAが通過するにはせますぎる.そのため,すでに40Sサブユニットと結合した開始tRNAは,60Sサブユニットと会合するためにこの構造的な立体障害をさけなければならない.実際に,eIF5Bの存在しない条件においては,eIF5に依存したeIF2によるGTPの加水分解ののち,40Sサブユニットは60Sサブユニットと会合できない4,5).PRE様状態の構造をみてみると,eIF5BのドメインIVは開始tRNAのCCA末端と結合し,開始tRNAの位置を変えることによりサブユニットの会合の際の立体障害をさけ,60Sサブユニットと結合しやすくしていることがわかった.この結果から,eIF5BをtRNA再配置因子と名づけた.
最近の蛍光共鳴エネルギー移動(fluorescence resonance energy transfer:FRET)解析から,原核生物におけるサブユニットの会合においては,翻訳開始因子IF2によるGTPの加水分解は30Sサブユニットが回転運動(50Sサブユニットに対し反時計回り)した状態で起こり,そののち回転していない状態に移行することがわかっている10).そのため,真核生物においては,回転運動の起こったPRE様状態においてサブユニットの会合が起こり,GTPの加水分解ののち逆方向の回転運動が起こり,POST様状態に移行することによりeIF5Bが解離し最初のアミノアシルtRNAを受け入れる状態になると考えられた(図2).また,出芽酵母におけるリボソームRNAへの変異の導入実験から,eIF5BとリボソームRNAのAループとの結合が翻訳開始反応の正確性において重要であることが報告されている.この結合は,POST様状態の構造において観察された.このため,今回の研究により得られたサブユニットの会合のモデルは,リボソーム内部進入部位RNAを介した翻訳開始反応だけでなく,キャップ構造に依存する翻訳開始反応にもあてはまると推測された.
今回の解析より,80Sリボソーム,リボソーム内部進入部位RNA,開始tRNA,翻訳開始因子eIF5Bからなる複合体には2つの状態があり,この2つの状態は真核生物において特異的な40Sサブユニットの回転運動により移行していることがわかった.また,この回転運動はGTPの加水分解により制御されていると考えられた.
解析技術の向上により,ひとつのリボソームの試料から複数の状態がみえるようになってきた.今後の技術のさらなる向上にともない,いままでみえなかった状態がみえるようになり,リボソームの機能をより詳細におえるようになるであろう.
略歴:2007年 新潟大学大学院自然科学研究科博士課程 修了,同年 新潟薬科大学 研究員,同年 ドイツMax Planck Institute for Molecular Genetics研究員を経て,2010年よりドイツCharité – Universitätsmedizin Berlin研究員.
研究テーマ:リボソーム結合RNAおよびタンパク質の生化学的な手法を用いた機能解析と,その複合体のクライオ電子顕微鏡による構造解析.
Anett Unbehaun
ドイツCharité – Universitätsmedizin Berlin研究員.
Christian M. T. Spahn
ドイツCharité – Universitätsmedizin Berlin教授.
© 2014 山本 紘・Anett Unbehaun・Christian M. T. Spahn Licensed under CC 表示 2.1 日本
(ドイツCharité – Universitätsmedizin Berlin,Institute of Medical Physics and Biophysics)
email:山本 紘
DOI: 10.7875/first.author.2014.116
Structure of the mammalian 80S initiation complex with initiation factor 5B on HCV-IRES RNA.
Hiroshi Yamamoto, Anett Unbehaun, Justus Loerke, Elmar Behrmann, Marianne Collier, Jörg Bürger, Thorsten Mielke, Christian M. T. Spahn
Nature Structural & Molecular Biology, 21, 721-727 (2014)
要 約
真核生物の翻訳開始因子のひとつeIF5Bは,原核生物から真核生物まで高度に保存されたGTPase活性をもつタンパク質であり,翻訳開始反応に不可欠である.eIF5BはC型肝炎ウイルスに由来するリボソーム内部進入部位RNAを介した翻訳開始反応においてもはたらくことが知られている.eIF5Bは翻訳開始反応において,開始tRNAの結合した40Sサブユニットと60Sサブユニットとの会合を促進し80Sリボソームを形成する.しかしながら,eIF5BがどのようにGTPの加水分解を介しサブユニットの会合を促進するのかは解明されていない.今回,筆者らは,真核生物の80Sリボソーム,C型肝炎ウイルスに由来するリボソーム内部進入部位RNA,開始tRNA,翻訳開始因子eIF5Bからなる複合体について,GTPのアナログであるGMPPNPを用いることによりeIF5Bを固定し,クライオ電子顕微鏡によりその構造を解析した.得られた画像の解析から2つの構造が得られ,それらの構造は40Sサブユニットの回転運動,および,開始tRNAの位置の違いから区別することができた.この2つの構造を比較することにより,eIF5Bはリボソームにおける開始tRNAの位置を変えることによりサブユニットの会合を促進していることが予想され,eIF5BはtRNAの再配置にはたらくことが明らかにされた.
はじめに
真核生物における翻訳開始反応は非常に厳密に制御されている.この反応は,40Sサブユニットと翻訳開始因子との複合体が60Sサブユニットと会合することにより80Sリボソームが形成されることで達成される.少なくとも12個の翻訳開始因子がこの反応に関与しており,開始tRNAおよび5’末端にキャップ構造をもつmRNAの40Sサブユニットへの結合,40SサブユニットがmRNAをスキャンする際の開始tRNAとの相互作用の制御,開始コドンの選択,40Sサブユニットと60Sサブユニットとの会合の促進,などの機能が知られている1).この反応の最後のステップにおいて,GTPase活性をもつ翻訳開始因子eIF5Bは40Sサブユニットと60Sサブユニットとの会合を促進する2).eIF5Bは原核生物の翻訳開始因子であるIF2のオーソログであり普遍的に保存されている.この遺伝子の欠損は出芽酵母においては生育の遅延をひき起こし,ショウジョウバエや哺乳類細胞では生育に必須である.また,eIF5Bはキャップ構造に依存しない,リボソーム内部進入部位(internal ribosomal entry site:IRES)を介した翻訳開始反応に必要なタンパク質のひとつでもある.リボソーム内部進入部位RNAは一部の翻訳開始因子のみを必要とする特殊な翻訳開始機構をもち,C型肝炎ウイルスやブタコレラウイルスに由来するものが知られている3).C型肝炎ウイルスに由来するリボソーム内部進入部位RNAは40Sサブユニットと高い親和性で会合し,GTPと結合したeIF2と開始tRNAとの複合体をリボソームのP部位に受け入れる.eIF5はeIF2のもつGTPase活性を促進することによりeIF2とGDPとの解離をひき起こし,eIF5Bを介したサブユニットの会合に進行させる4,5).生体におけるアミノ酸の不足による飢餓状態などにおいてはeIF2のαサブユニットはリン酸化されてその機能を失うが,C型肝炎ウイルスに由来するリボソーム内部進入部位RNAはeIF5Bが直接に作用する経路によりサブユニットの会合に進行することができる.リボソームと結合したeIF5Bについての構造的な知見は少なく,原核生物および出芽酵母における通常の翻訳開始反応を介したIF2あるいはeIF5Bとの複合体の構造は報告されているものの,eIF5BのもつGTPase活性を介したサブユニットの会合はどのように促進されるのか,また,開始tRNAのリボソームにおける適切な配置はどのようになされるのかなどについては,いまだ解明されていない6,7).そこで,筆者らは,80Sリボソーム,C型肝炎ウイルスに由来するリボソーム内部進入部位RNA,開始tRNA,翻訳開始因子eIF5Bからなる複合体をクライオ電子顕微鏡により構造解析することで,eIF5Bのリボソームにおいての役割について調べた.
1.80Sリボソーム,リボソーム内部進入部位RNA,開始tRNA,eIF5Bからなる複合体では40Sサブユニットに回転運動が起こる
翻訳開始因子であるeIF2およびeIF5を介さない系においてリボソームを調製し,また,GTPの非加水分解アナログであるGMPPNPを用いることにより翻訳開始因子eIF5Bを固定した.電子顕微鏡により得られた画像をマルチパーティクルクライオ電子顕微鏡法により解析することにより8),2つの構造を分解能8.9Å(EMDB ID:2682,図1),および,分解能9.5Å(EMDB ID:2683)で得た.この2つの構造は,40Sサブユニットの回転運動の大きさ,開始tRNAの位置の違いにより区別された(図1b).興味深いことに,観察された40Sサブユニットの回転運動は,原核生物のリボソームにおいてよく知られているものとは異なり,最近,真核生物においてポリペプチド鎖の伸長段階のリボソームにて明らかにされたものと類似していた9).この回転運動は,40Sサブユニットに垂直な軸に対して直交した方向に回転するものであり約6度の回転が観察された.得られた2つの構造をポリペプチド鎖の伸長段階のリボソームの構造と比較した結果,伸長段階のPRE状態およびPOST状態と類似していた.このことから,この2つの構造をPRE様状態およびPOST様状態とよぶことにした.
2.40Sサブユニットの逆方向の回転運動は開始tRNAおよびeIF5Bの位置を変える
PRE様状態とPOST様状態の構造を比較したところ,40Sサブユニットに結合しているeIF5B-開始tRNA複合体について,リボソームに対する構造変化はほとんどなく,逆方向の回転運動によりeIF5B-開始tRNA複合体の位置は40Sサブユニットといっしょに移動していることがわかった(図1b).観察されたPRE様状態における開始tRNAの位置は,原核生物においてよく知られている翻訳開始反応に特異的な位置をとっていたが,開始tRNAの“ひじ”およびアミノ酸受容末端の位置が少し異なっていた.それとは対照的に,POST様状態において開始tRNAは新規の位置をとっていた.最近の出芽酵母におけるeIF5Bのクライオ電子顕微鏡による解析により得られた構造は,PRE様状態に類似している.しかしながら,今回のPOST様状態は,出芽酵母7) あるいは原核生物6) において観察されておらず,まったく新規の状態であった.また,PRE様状態のeIF5BはGTPase活性に深く関与するリボソームRNAのサルシン-リシンループと結合していたのに対し,POST様状態ではeIF5Bはサルシン-リシンループから解離していた.このことから,PRE様状態はGTPの加水分解の起こるまえの状態であり,POST様状態はGTPの加水分解ののちの状態であることが推測された.
3.リボソーム内部進入部位RNAのドメインIIは翻訳開始反応において40Sサブユニットの傾きを解消し開始tRNAと結合する
C型肝炎ウイルスに由来するリボソーム内部進入部位RNAのコアドメインは,ドメインII,ドメインIII,ドメインIV,シュードノットから構成されており3),翻訳開始因子に対し非依存的に調製した40Sサブユニットとリボソーム内部進入部位RNAとの複合体と比較すると,ドメインIIに大きな変化がみられた.80SリボソームではドメインIIbは40Sサブユニットから解離しており,40Sサブユニットにおいてみられた傾きは解消していることがわかった.また,解離したドメインIIbは開始tRNAの“ひじ”に結合していることが観察された.また,この結合はPOST様状態ではみられなかった.ドメインIIはリボソームとの結合能には関与しないが翻訳開始反応に不可欠であることが知られており4,5),今回の知見から,リボソーム内部進入部位RNAのドメインIIは開始tRNAの位置どりを助けていると推察された.
4.eIF5Bは開始tRNAの再配置にはたらく
翻訳開始因子IF2およびeIF5Bはリボソームのサブユニットの会合を促進することが知られている1,2,6).ポリペプチド鎖の伸長段階のリボソームにおいてP部位にあるtRNAをみてみると,CCA末端はペプチジルトランスフェラーゼの活性中心のある溝に結合している.しかしながら,開始tRNAの直接的な結合は中央突起とヘリックス69により阻まれており,開始tRNAが通過するにはせますぎる.そのため,すでに40Sサブユニットと結合した開始tRNAは,60Sサブユニットと会合するためにこの構造的な立体障害をさけなければならない.実際に,eIF5Bの存在しない条件においては,eIF5に依存したeIF2によるGTPの加水分解ののち,40Sサブユニットは60Sサブユニットと会合できない4,5).PRE様状態の構造をみてみると,eIF5BのドメインIVは開始tRNAのCCA末端と結合し,開始tRNAの位置を変えることによりサブユニットの会合の際の立体障害をさけ,60Sサブユニットと結合しやすくしていることがわかった.この結果から,eIF5BをtRNA再配置因子と名づけた.
5.真核生物におけるリボソーム内部進入部位RNAを介したサブユニットの会合のモデル
最近の蛍光共鳴エネルギー移動(fluorescence resonance energy transfer:FRET)解析から,原核生物におけるサブユニットの会合においては,翻訳開始因子IF2によるGTPの加水分解は30Sサブユニットが回転運動(50Sサブユニットに対し反時計回り)した状態で起こり,そののち回転していない状態に移行することがわかっている10).そのため,真核生物においては,回転運動の起こったPRE様状態においてサブユニットの会合が起こり,GTPの加水分解ののち逆方向の回転運動が起こり,POST様状態に移行することによりeIF5Bが解離し最初のアミノアシルtRNAを受け入れる状態になると考えられた(図2).また,出芽酵母におけるリボソームRNAへの変異の導入実験から,eIF5BとリボソームRNAのAループとの結合が翻訳開始反応の正確性において重要であることが報告されている.この結合は,POST様状態の構造において観察された.このため,今回の研究により得られたサブユニットの会合のモデルは,リボソーム内部進入部位RNAを介した翻訳開始反応だけでなく,キャップ構造に依存する翻訳開始反応にもあてはまると推測された.
おわりに
今回の解析より,80Sリボソーム,リボソーム内部進入部位RNA,開始tRNA,翻訳開始因子eIF5Bからなる複合体には2つの状態があり,この2つの状態は真核生物において特異的な40Sサブユニットの回転運動により移行していることがわかった.また,この回転運動はGTPの加水分解により制御されていると考えられた.
解析技術の向上により,ひとつのリボソームの試料から複数の状態がみえるようになってきた.今後の技術のさらなる向上にともない,いままでみえなかった状態がみえるようになり,リボソームの機能をより詳細におえるようになるであろう.
文 献
- Aitken, C. E. & Lorsch, J. R.: A mechanistic overview of translation initiation in eukaryotes. Nat. Struct. Mol. Biol., 19, 568-576 (2012)[PubMed]
- Pestova, T. V., Lomakin, I. B., Lee, J. H. et al.: The joining of ribosomal subunits in eukaryotes requires eIF5B. Nature, 403, 332-335 (2000)[PubMed]
- Lukavsky, P. J.: Structure and function of HCV IRES domains. Virus Res., 139, 166-171 (2009)[PubMed]
- Pestova, T. V., de Breyne, S., Pisarev, A. V. et al.: eIF2-dependent and eIF2-independent modes of initiation on the CSFV IRES: a common role of domain II. EMBO J., 27, 1060-1072 (2008)[PubMed]
- Locker, N., Easton, L. E. & Lukavsky, P. J.: HCV and CSFV IRES domain II mediate eIF2 release during 80S ribosome assembly. EMBO J., 26, 795-805 (2007)[PubMed]
- Allen, G. S., Zavialov, A., Gursky, R. et al.: The cryo-EM structure of a translation initiation complex from Escherichia coli. Cell, 121, 703-712 (2005)[PubMed]
- Fernandez, I. S., Bai, X. C., Hussain, T. et al.: Molecular architecture of a eukaryotic translational initiation complex. Science, 342, 1240585 (2013)[PubMed]
- Loerke, J., Giesebrecht, J. & Spahn, C. M.: Multiparticle cryo-EM of ribosomes. Methods Enzymol., 483, 161-177 (2010)[PubMed]
- Budkevich, T. V., Giesebrecht, J., Behrmann, E. et al.: Regulation of the mammalian elongation cycle by subunit rolling: a eukaryotic-specific ribosome rearrangement. Cell, 158, 121-131 (2014)[PubMed]
- Marshall, R. A., Aitken, C. E. & Puglisi, J. D.: GTP hydrolysis by IF2 guides progression of the ribosome into elongation. Mol. Cell, 35, 37-47 (2009)[PubMed]
著者プロフィール
略歴:2007年 新潟大学大学院自然科学研究科博士課程 修了,同年 新潟薬科大学 研究員,同年 ドイツMax Planck Institute for Molecular Genetics研究員を経て,2010年よりドイツCharité – Universitätsmedizin Berlin研究員.
研究テーマ:リボソーム結合RNAおよびタンパク質の生化学的な手法を用いた機能解析と,その複合体のクライオ電子顕微鏡による構造解析.
Anett Unbehaun
ドイツCharité – Universitätsmedizin Berlin研究員.
Christian M. T. Spahn
ドイツCharité – Universitätsmedizin Berlin教授.
© 2014 山本 紘・Anett Unbehaun・Christian M. T. Spahn Licensed under CC 表示 2.1 日本