G・U塩基対に依存したtRNAの選択的なアミノアシル化の分子機構
永沼政広・横山茂之
(理化学研究所 横山構造生物学研究室)
email:永沼政広,横山茂之
DOI: 10.7875/first.author.2014.091
The selective tRNA aminoacylation mechanism based on a single G•U pair.
Masahiro Naganuma, Shun-ichi Sekine, Yeeting Esther Chong, Min Guo, Xiang-Lei Yang, Howard Gamper, Ya-Ming Hou, Paul Schimmel, Shigeyuki Yokoyama
Nature, 510, 507-511 (2014)
遺伝暗号のタンパク質への翻訳の過程において,アラニルtRNA合成酵素はアラニンおよびアラニン専用のtRNAであるtRNAAlaを認識してアラニルtRNAAlaを生成する.その際,アラニルtRNA合成酵素はtRNAAlaのアクセプターステムにあるG3・U70塩基対を目印とすることで正確にtRNAAlaを選択する.しかし,どのような分子機構により1つの塩基対に依存してtRNAAlaを選択的にアミノアシル化するのかはわからないままであった.この研究においては,アラニルtRNA合成酵素と野生型のtRNAAlaとの複合体,および,アラニルtRNA合成酵素とG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaとの複合体の結晶構造を解析し,tRNAAlaを選択する分子機構を原子レベルで明らかにした.G・U塩基対はワトソン-クリック型塩基対とは幾何学的に構造の異なる塩基対を形成するが,アラニルtRNA合成酵素は野生型のtRNAAlaのもつG3・U70塩基対の構造の違いをCCA末端の方向へ伝達し,アミノアシル化されるCCA末端を活性部位の方向に配置して,G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaのようにこの位置にワトソン-クリック型塩基対をもつtRNAのCCA末端をまったく異なる位置に配置することにより,選択的なtRNAアミノアシル化をなしとげていることが明らかになった.
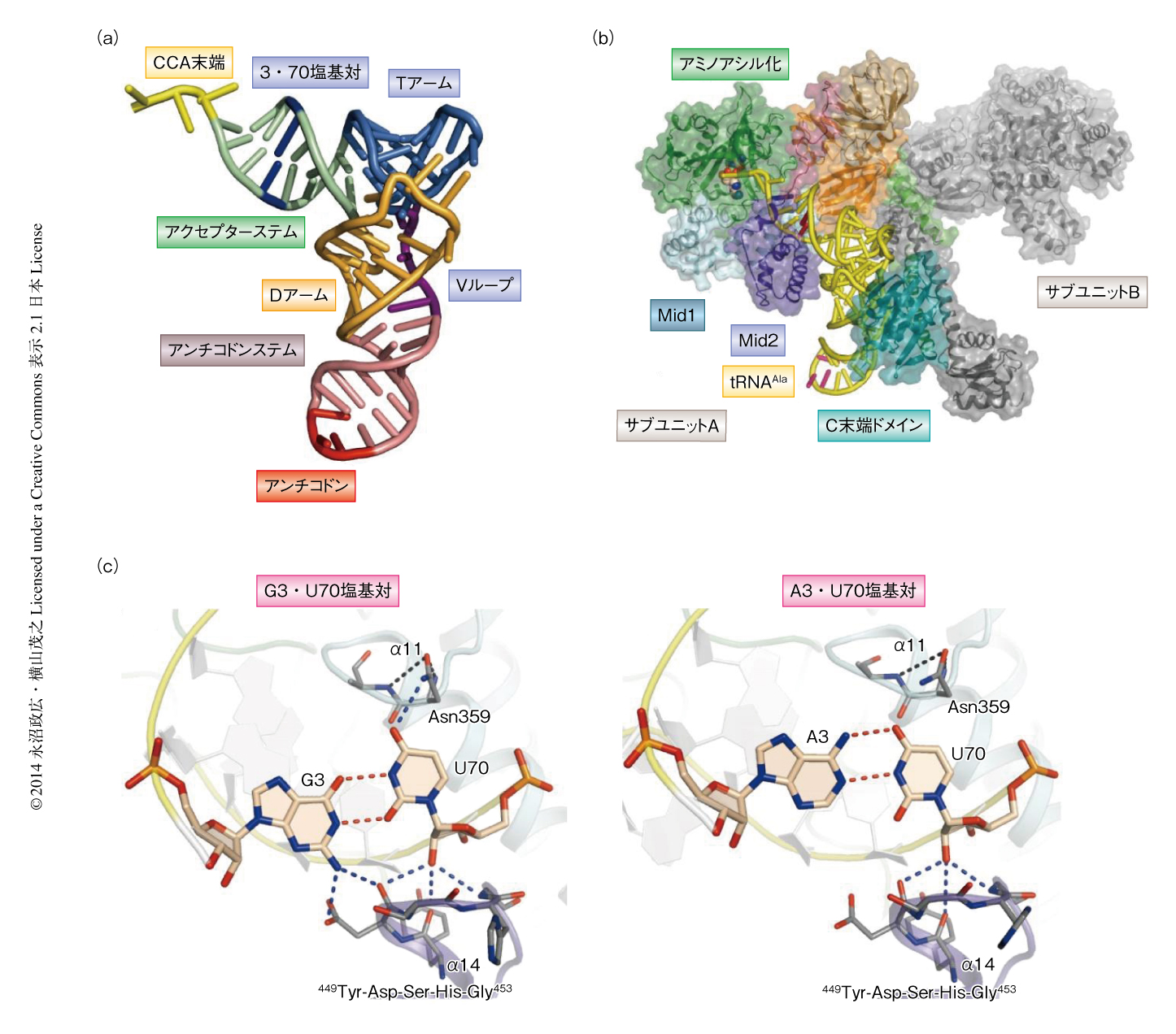
遺伝暗号においてコドンはそれぞれ20種類のアミノ酸のうち1種類に割り当てられている.このコドンとアミノ酸との対応づけは,20種類のアミノアシルtRNA合成酵素が専用のアミノ酸とそのコドンと相補的なアンチコドンをもつtRNAを選択し,アミノアシル化することにより達成されている.多くのアミノアシルtRNA合成酵素はそれぞれのtRNAに特徴的なアンチコドンを最大の目印として専用のtRNAを選択的にアミノアシル化する1).しかし,アラニンのアミノアシルtRNA合成酵素であるアラニルtRNA合成酵素は,アラニン専用のtRNAであるtRNAAlaのもつアンチコドンを目印にするのではなく,アクセプターステムにあるG3・U70塩基対を目印にしてtRNAAlaを選択的にアミノアシル化する1-3)(図1a).つまり,G3・U70塩基対をA3・U70塩基対やG3・C70塩基対などのワトソン-クリック型塩基対に置換したtRNAAlaはアラニル化されなくなり,反対に,tRNAAla以外のいくつかのtRNAでもG3・U70塩基対を導入するとアラニルtRNA合成酵素によりアラニル化されるようになる.しかし,アラニルtRNA合成酵素がどのような分子機構によりたったひとつの塩基対に強く依存してtRNAAlaを選択的にアミノアシル化しているのかはわからないままであった.
この研究では,アラニルtRNA合成酵素とG3・U70塩基対をもつ野生型tRNAAlaとの複合体の結晶構造を分解能3.3Å(PDB ID:3WQY),また,アラニルtRNA合成酵素とG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaとの複合体の結晶構造を分解能3.5Å(PDB ID:3WQZ)で解くことにより,アラニルtRNA合成酵素によるtRNAAlaの選択的なアミノアシル化の分子機構を解明した.
アラニルtRNA合成酵素と野生型のtRNAAlaとの複合体の結晶構造,および,アラニルtRNA合成酵素とG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaとの複合体の結晶構造において,結晶学的な非対称単位においてアラニルtRNA合成酵素は二量体を形成し片方のサブユニット(サブユニットA)にのみ1分子のtRNAAlaが結合していた(図1b).アミノアシル化ドメインはクラスIIアミノアシルtRNA合成酵素のあいだで共通のフォールドをとっていたが4),アミノアシル化ドメインに対するtRNAAlaの結合の位置はほかのクラスIIアミノアシルtRNA合成酵素のあいだで保存されている位置とは完全に異なっていた.tRNAAlaのアクセプターステムおよびTアームがtRNA認識ドメインおよびC末端ドメインによりとりかこまれるよう相互作用しており,一方,アンチコドンは相互作用していなかった(図1b).
G3・U70塩基対はワトソン-クリック型塩基対であるA3・U70塩基対と比較して,G3が副溝側,U70が主溝側へずれて塩基対を形成していた(図1c).野生型のtRNAAlaのアクセプターステムは副溝と主溝の両側からはさまれるようにtRNA認識ドメインと相互作用しており,Mid2のα14ヘリックスの先端の449Tyr-Asp-Ser-His-Gly453がアクセプターステムのG2・C71塩基対,G3・U70塩基対,C4・G69塩基対,U5・A68塩基対の副溝側と相互作用し,G3の2-アミノ基はAsp450の側鎖のカルボキシル基および主鎖のカルボニル基と水素結合できる距離にあり,U70の2'-ヒドロキシル基はAsp450,Tyr449,Gly453の主鎖のカルボニル基と水素結合できる距離にあった(図1c).一方,G1・C72塩基対,G2・C71塩基対,G3・U70塩基対の主溝側はMid1のα11ヘリックスのN末端側のヘリカルループと相互作用しており,U70の4-カルボニル基がAsn359の側鎖のアミノ基と水素結合できる距離にあった(図1c).G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaのアクセプターステムも,野生型のtRNAAlaと同様に副溝と主溝の両側からはさまれるように相互作用していた.しかし,A3はG3に比べ主溝側にずれてAsp450から離れていた(図1c).
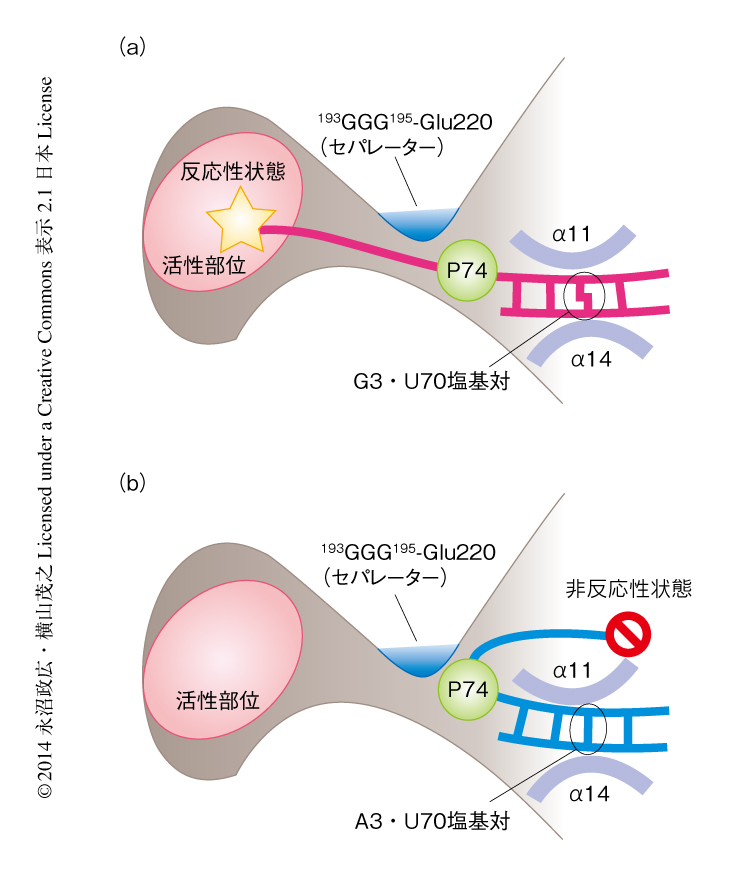
野生型のtRNAAlaにおいてはG3・U70塩基対のもつ幾何学的な構造の違いがCCA末端の方向へと伝達していた(図2a).G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaにおいてA3・U70塩基対はG3・U70塩基対に比べずれているため,G2・C71塩基対とG1・C72塩基対も連動してずれていた.これらのヌクレオチドの動きはターニングポイントになるC74の5'-リン酸基まで到達し,CCA末端の位置を大きく変化させていた(図2b).野生型のtRNAAlaのCCA末端の“反応性”の位置と,G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaのCCA末端の“非反応性”の位置は,193GGG195とGlu220の領域により分け隔てられていた.193GGG195とGlu220の領域がCCA末端の位置を分岐させる“セパレーター”としてはたらいていると考えられた.大腸菌のアラニルtRNA合成酵素において,193GGG195とGlu220の領域と空間的に近い位置にあるAsp174をGlyに置換したアラニルtRNA合成酵素変異体は,G3・U70塩基対をG3・C70塩基対に置換したtRNAをまちがってアミノアシル化してしまうという報告があり5),このアラニルtRNA合成酵素変異体はCCA末端を“非反応性”の位置に捕捉できないものと考えられる.このようなCCA末端の領域における構造変化の増幅は,Mid1とMid2がアクセプターステムを副溝と主溝の両側からしっかりはさみこみアクセプターステムの3番目の塩基対を正しく配置することにより可能になっていた(図1c).
クエンチフロー装置を用いて前定常状態の反応速度論の解析を行った.野生型のtRNAAlaとG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaは,約100倍のkcatの差をもってアラニルtRNA合成酵素により区別されていた.kcatは酵素1分子あたり1秒間に何個の基質を触媒するかというパラメーターである.シングルターンオーバー解析を行ったところ,G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaは野生型のtRNAAlaと比べ,“反応性”の状態でのAla-tRNAAlaの生成速度であるkchemが約2倍小さく解離定数Kdが約2倍大きかった.このように,G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaと野生型のtRNAAlaとのあいだのkchemとKdとの差は,kcatにおける差に比べ非常に小さかった.
これらの結果の解釈のため,非反応性の状態を考慮した酵素反応速度論にもとづいてシミュレーションを行ったところ,野生型のtRNAAlaおよびG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaの前定常状態の反応を再現することができた.シミュレーションにおいて,野生型のtRNAAlaではほとんどの複合体が“反応性”の状態になったが,G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaではほとんどが“非反応性”の状態に捕捉されており生成物の酵素からの解離が律速段階となっていた.この非反応性の状態はアラニルtRNA合成酵素とG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaとの複合体のCCA末端が捕捉された結晶構造を表わしていると考えられた.このように,アラニルtRNA合成酵素は反応性と非反応性の状態を用いる分子機構によりkcatに依存したtRNAの選択的なアミノアシル化をなしとげていた.
アラニルtRNA合成酵素と野生型のtRNAAlaとの複合体,および,アラニルtRNA合成酵素とG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaとの複合体の構造解析により,反応性と非反応性の状態を用いた分子活性に依存したtRNAの選択的なアミノアシル化の分子機構が明らかにされた.多くのアミノアシルtRNA合成酵素においては,G3・U70塩基対よりもさらに遠いアンチコドンの変異が,Kdを上げるのではなくkcatを劇的に低下させる1).しかし,そのようなアミノアシルtRNA合成酵素においても,今回,明らかにされたものと同様の反応機構が用いられているかどうかはいまだ謎である.今後,この反応性と非反応性の状態をとりいれた酵素の設計などにより人工塩基をもつtRNAの認識および選択が可能になれば,それらの人工コドンを人工アミノ酸に割り当てる遺伝暗号の拡張の技術の開発にもつながると予想され,有用な人工アミノ酸を活用するタンパク質工学を発展させることが期待される.
略歴:2012年 東京大学大学院理学系研究科博士課程 修了,同年より理化学研究所 特別研究員.
研究テーマ:アミノアシルtRNA合成酵素によるtRNAの認識の機構.
関心事:タンパク質によるRNAの認識.
横山 茂之(Shigeyuki Yokoyama)
理化学研究所 上席研究員.
研究室URL:http://sbl.riken.jp/
© 2014 永沼政広・横山茂之 Licensed under CC 表示 2.1 日本
(理化学研究所 横山構造生物学研究室)
email:永沼政広,横山茂之
DOI: 10.7875/first.author.2014.091
The selective tRNA aminoacylation mechanism based on a single G•U pair.
Masahiro Naganuma, Shun-ichi Sekine, Yeeting Esther Chong, Min Guo, Xiang-Lei Yang, Howard Gamper, Ya-Ming Hou, Paul Schimmel, Shigeyuki Yokoyama
Nature, 510, 507-511 (2014)
要 約
遺伝暗号のタンパク質への翻訳の過程において,アラニルtRNA合成酵素はアラニンおよびアラニン専用のtRNAであるtRNAAlaを認識してアラニルtRNAAlaを生成する.その際,アラニルtRNA合成酵素はtRNAAlaのアクセプターステムにあるG3・U70塩基対を目印とすることで正確にtRNAAlaを選択する.しかし,どのような分子機構により1つの塩基対に依存してtRNAAlaを選択的にアミノアシル化するのかはわからないままであった.この研究においては,アラニルtRNA合成酵素と野生型のtRNAAlaとの複合体,および,アラニルtRNA合成酵素とG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaとの複合体の結晶構造を解析し,tRNAAlaを選択する分子機構を原子レベルで明らかにした.G・U塩基対はワトソン-クリック型塩基対とは幾何学的に構造の異なる塩基対を形成するが,アラニルtRNA合成酵素は野生型のtRNAAlaのもつG3・U70塩基対の構造の違いをCCA末端の方向へ伝達し,アミノアシル化されるCCA末端を活性部位の方向に配置して,G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaのようにこの位置にワトソン-クリック型塩基対をもつtRNAのCCA末端をまったく異なる位置に配置することにより,選択的なtRNAアミノアシル化をなしとげていることが明らかになった.
はじめに
遺伝暗号においてコドンはそれぞれ20種類のアミノ酸のうち1種類に割り当てられている.このコドンとアミノ酸との対応づけは,20種類のアミノアシルtRNA合成酵素が専用のアミノ酸とそのコドンと相補的なアンチコドンをもつtRNAを選択し,アミノアシル化することにより達成されている.多くのアミノアシルtRNA合成酵素はそれぞれのtRNAに特徴的なアンチコドンを最大の目印として専用のtRNAを選択的にアミノアシル化する1).しかし,アラニンのアミノアシルtRNA合成酵素であるアラニルtRNA合成酵素は,アラニン専用のtRNAであるtRNAAlaのもつアンチコドンを目印にするのではなく,アクセプターステムにあるG3・U70塩基対を目印にしてtRNAAlaを選択的にアミノアシル化する1-3)(図1a).つまり,G3・U70塩基対をA3・U70塩基対やG3・C70塩基対などのワトソン-クリック型塩基対に置換したtRNAAlaはアラニル化されなくなり,反対に,tRNAAla以外のいくつかのtRNAでもG3・U70塩基対を導入するとアラニルtRNA合成酵素によりアラニル化されるようになる.しかし,アラニルtRNA合成酵素がどのような分子機構によりたったひとつの塩基対に強く依存してtRNAAlaを選択的にアミノアシル化しているのかはわからないままであった.
この研究では,アラニルtRNA合成酵素とG3・U70塩基対をもつ野生型tRNAAlaとの複合体の結晶構造を分解能3.3Å(PDB ID:3WQY),また,アラニルtRNA合成酵素とG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaとの複合体の結晶構造を分解能3.5Å(PDB ID:3WQZ)で解くことにより,アラニルtRNA合成酵素によるtRNAAlaの選択的なアミノアシル化の分子機構を解明した.
1.アラニルtRNA合成酵素とtRNAAlaとの複合体の結晶構造
アラニルtRNA合成酵素と野生型のtRNAAlaとの複合体の結晶構造,および,アラニルtRNA合成酵素とG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaとの複合体の結晶構造において,結晶学的な非対称単位においてアラニルtRNA合成酵素は二量体を形成し片方のサブユニット(サブユニットA)にのみ1分子のtRNAAlaが結合していた(図1b).アミノアシル化ドメインはクラスIIアミノアシルtRNA合成酵素のあいだで共通のフォールドをとっていたが4),アミノアシル化ドメインに対するtRNAAlaの結合の位置はほかのクラスIIアミノアシルtRNA合成酵素のあいだで保存されている位置とは完全に異なっていた.tRNAAlaのアクセプターステムおよびTアームがtRNA認識ドメインおよびC末端ドメインによりとりかこまれるよう相互作用しており,一方,アンチコドンは相互作用していなかった(図1b).
G3・U70塩基対はワトソン-クリック型塩基対であるA3・U70塩基対と比較して,G3が副溝側,U70が主溝側へずれて塩基対を形成していた(図1c).野生型のtRNAAlaのアクセプターステムは副溝と主溝の両側からはさまれるようにtRNA認識ドメインと相互作用しており,Mid2のα14ヘリックスの先端の449Tyr-Asp-Ser-His-Gly453がアクセプターステムのG2・C71塩基対,G3・U70塩基対,C4・G69塩基対,U5・A68塩基対の副溝側と相互作用し,G3の2-アミノ基はAsp450の側鎖のカルボキシル基および主鎖のカルボニル基と水素結合できる距離にあり,U70の2'-ヒドロキシル基はAsp450,Tyr449,Gly453の主鎖のカルボニル基と水素結合できる距離にあった(図1c).一方,G1・C72塩基対,G2・C71塩基対,G3・U70塩基対の主溝側はMid1のα11ヘリックスのN末端側のヘリカルループと相互作用しており,U70の4-カルボニル基がAsn359の側鎖のアミノ基と水素結合できる距離にあった(図1c).G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaのアクセプターステムも,野生型のtRNAAlaと同様に副溝と主溝の両側からはさまれるように相互作用していた.しかし,A3はG3に比べ主溝側にずれてAsp450から離れていた(図1c).
2.CCA末端の分岐
野生型のtRNAAlaにおいてはG3・U70塩基対のもつ幾何学的な構造の違いがCCA末端の方向へと伝達していた(図2a).G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaにおいてA3・U70塩基対はG3・U70塩基対に比べずれているため,G2・C71塩基対とG1・C72塩基対も連動してずれていた.これらのヌクレオチドの動きはターニングポイントになるC74の5'-リン酸基まで到達し,CCA末端の位置を大きく変化させていた(図2b).野生型のtRNAAlaのCCA末端の“反応性”の位置と,G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaのCCA末端の“非反応性”の位置は,193GGG195とGlu220の領域により分け隔てられていた.193GGG195とGlu220の領域がCCA末端の位置を分岐させる“セパレーター”としてはたらいていると考えられた.大腸菌のアラニルtRNA合成酵素において,193GGG195とGlu220の領域と空間的に近い位置にあるAsp174をGlyに置換したアラニルtRNA合成酵素変異体は,G3・U70塩基対をG3・C70塩基対に置換したtRNAをまちがってアミノアシル化してしまうという報告があり5),このアラニルtRNA合成酵素変異体はCCA末端を“非反応性”の位置に捕捉できないものと考えられる.このようなCCA末端の領域における構造変化の増幅は,Mid1とMid2がアクセプターステムを副溝と主溝の両側からしっかりはさみこみアクセプターステムの3番目の塩基対を正しく配置することにより可能になっていた(図1c).
3.分子活性によるtRNAAlaの選択の機構
クエンチフロー装置を用いて前定常状態の反応速度論の解析を行った.野生型のtRNAAlaとG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaは,約100倍のkcatの差をもってアラニルtRNA合成酵素により区別されていた.kcatは酵素1分子あたり1秒間に何個の基質を触媒するかというパラメーターである.シングルターンオーバー解析を行ったところ,G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaは野生型のtRNAAlaと比べ,“反応性”の状態でのAla-tRNAAlaの生成速度であるkchemが約2倍小さく解離定数Kdが約2倍大きかった.このように,G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaと野生型のtRNAAlaとのあいだのkchemとKdとの差は,kcatにおける差に比べ非常に小さかった.
これらの結果の解釈のため,非反応性の状態を考慮した酵素反応速度論にもとづいてシミュレーションを行ったところ,野生型のtRNAAlaおよびG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaの前定常状態の反応を再現することができた.シミュレーションにおいて,野生型のtRNAAlaではほとんどの複合体が“反応性”の状態になったが,G3・U70塩基対をA3・U70塩基対に置換したtRNAAlaではほとんどが“非反応性”の状態に捕捉されており生成物の酵素からの解離が律速段階となっていた.この非反応性の状態はアラニルtRNA合成酵素とG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaとの複合体のCCA末端が捕捉された結晶構造を表わしていると考えられた.このように,アラニルtRNA合成酵素は反応性と非反応性の状態を用いる分子機構によりkcatに依存したtRNAの選択的なアミノアシル化をなしとげていた.
おわりに
アラニルtRNA合成酵素と野生型のtRNAAlaとの複合体,および,アラニルtRNA合成酵素とG3・U70塩基対をA3・U70塩基対に置換したtRNAAlaとの複合体の構造解析により,反応性と非反応性の状態を用いた分子活性に依存したtRNAの選択的なアミノアシル化の分子機構が明らかにされた.多くのアミノアシルtRNA合成酵素においては,G3・U70塩基対よりもさらに遠いアンチコドンの変異が,Kdを上げるのではなくkcatを劇的に低下させる1).しかし,そのようなアミノアシルtRNA合成酵素においても,今回,明らかにされたものと同様の反応機構が用いられているかどうかはいまだ謎である.今後,この反応性と非反応性の状態をとりいれた酵素の設計などにより人工塩基をもつtRNAの認識および選択が可能になれば,それらの人工コドンを人工アミノ酸に割り当てる遺伝暗号の拡張の技術の開発にもつながると予想され,有用な人工アミノ酸を活用するタンパク質工学を発展させることが期待される.
文 献
- Giege, R., Sissler, M. & Florentz, C.: Universal rules and idiosyncratic features in tRNA identity. Nucleic Acids Res., 26, 5017-5035 (1998)[PubMed]
- Hou, Y. M. & Schimmel, P.: A simple structural feature is a major determinant of the identity of a transfer RNA. Nature, 333, 140-145 (1988)[PubMed]
- McClain, W. H. & Foss, K.: Changing the identity of a tRNA by introducing a G-U wobble pair near the 3' acceptor end. Science, 240, 793-796 (1988)[PubMed]
- Cavarelli, J. & Moras, D.: Recognition of tRNAs by aminoacyl-tRNA synthetases. FASEB J., 7, 79-86 (1993)[PubMed]
- Miller, W. T., Hou, Y. M. & Schimmel, P.: Mutant aminoacyl-tRNA synthetase that compensates for a mutation in the major identity determinant of its tRNA. Biochemistry, 30, 2635-2641 (1991)[PubMed]
著者プロフィール
略歴:2012年 東京大学大学院理学系研究科博士課程 修了,同年より理化学研究所 特別研究員.
研究テーマ:アミノアシルtRNA合成酵素によるtRNAの認識の機構.
関心事:タンパク質によるRNAの認識.
横山 茂之(Shigeyuki Yokoyama)
理化学研究所 上席研究員.
研究室URL:http://sbl.riken.jp/
© 2014 永沼政広・横山茂之 Licensed under CC 表示 2.1 日本