トリガー因子は変性状態のタンパク質とのダイナミックな相互作用により凝集を抑制する
斉尾智英・Charalampos G. Kalodimos
(米国Rutgers大学Center for Integrative Proteomics Research)
email:斉尾智英
DOI: 10.7875/first.author.2014.065
Structural basis for protein antiaggregation activity of the trigger factor chaperone.
Tomohide Saio, Xiao Guan, Paolo Rossi, Anastassios Economou, Charalampos G. Kalodimos
Science, 344, 1250494 (2014)
分子シャペロンはタンパク質の凝集やミスフォールディングを防ぐことにより,タンパク質の品質管理において主要な役割をはたすが,その分子機構はほとんど明らかにされていない.筆者らは,細菌における分子シャペロンのひとつであるトリガー因子とその基質であるPhoAに対し,NMR法を主体とした構造解析,相互作用解析,ダイナミクス解析を行った.その結果,トリガー因子は4箇所の基質結合部位により基質タンパク質の疎水性の領域と結合することにより,基質タンパク質を変性状態に保ち凝集を抑制していることが明らかになった.トリガー因子の基質結合部位は,結合する基質タンパク質により局所の構造を柔軟に変化させることによって基質タンパク質の幅広い認識を可能にしていた.
分子シャペロンは変性タンパク質の凝集を防ぐことにより,タンパク質のフォールディングや品質管理などにおいて重要な機能をはたす1).とくに,リボソームにより合成されたばかりの新生ポリペプチド鎖は立体構造をもたず,本来はタンパク質のコアを形成する疎水性の領域が露出しているため凝集の危険にさらされている.細菌における主要な分子シャペロンのひとつであるトリガー因子は,リボソームにおいて新生ポリペプチド鎖の出口となる領域に結合し,リボソームから出てきた変性状態のポリペプチド鎖に結合することによりその凝集を防ぐ2,3).新生ポリペプチド鎖はDnaK,DnaJ,GroEL-GroESなどといったほかの分子シャペロンにうけわたされ,分子シャペロンとの結合および解離をへて折りたたまれていく.また,トリガー因子は細胞質にも豊富に存在し,細胞質タンパク質の凝集の抑制による恒常性の維持,サブユニットタンパク質の集合体への組み込みの補助など,多様な機能をもつと考えられている2).これまでの研究により,トリガー因子単独3),トリガー因子-リボソーム複合体3),トリガー因子と折りたたまれた基質タンパク質との複合体4) の結晶構造が決定されているが,トリガー因子と変性状態の基質タンパク質との複合体の構造は決定されておらず,トリガー因子の主要な機能である変性状態のタンパク質との相互作用についての情報はごくかぎられていた.トリガー因子のみならず,ほかの分子シャペロンについても,変性状態の基質タンパク質の認識における分子機構はほとんど未解明であった.その最大の理由は,分子シャペロンと基質とのあいだのダイナミックかつ過渡的な相互作用,および,複合体の大きさにあった.分子を溶液状態で観測するNMR法は動的な系や弱い相互作用の解析に適しているが,従来のNMR法では分子量による制限が大きな壁になっていた.筆者らは,メチル基選択的安定同位体標識法5) やメチルTROSY法6) などを駆使した最新のNMR法により分子量の制約をのりこえ,変性状態の基質タンパク質と結合したトリガー因子の立体構造を決定した.
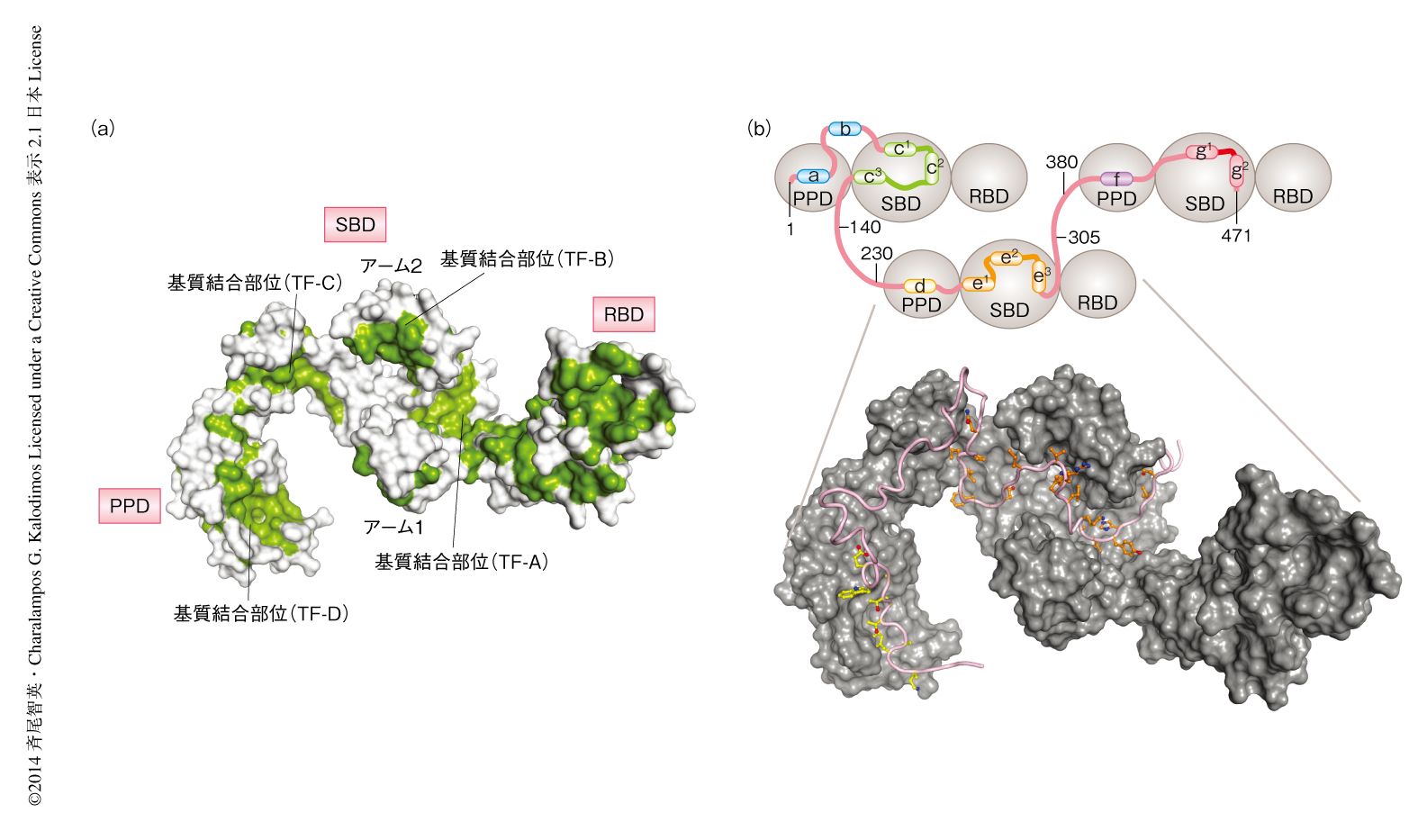
432アミノ酸残基からなる大腸菌のトリガー因子は,RBD,SBD,PPDの3つのドメインから構成され(図1a),その細長く特徴的な立体構造は,PPDを頭,SBDのアーム1およびアーム2を翼,RBDを尾にみたてて“dragon shape”とも形容される.トリガー因子は溶液においては100 kDaの二量体を形成し,NMRの対象としては高分子量であるが,高分子量タンパク質に特化した安定同位体標識法およびスペクトル測定法は,良好なNMRスペクトルを得ることを可能にした.なかでも,タンパク質に豊富に存在するメチル基を特異的に13C1H3により標識し,ほかを2Hにより標識するメチル基選択的安定同位体標識法5) は,重要な立体構造情報を失わずに横緩和を抑制することによりNMR信号を先鋭化かつ高感度化する強力な手法である.全長のトリガー因子のNMR信号の帰属のため,まず,おのおののドメインを分離したコンストラクトを作製し,それぞれに対し各種の3次元測定を行い主鎖アミドおよび側鎖のNMR信号を帰属した.ついで,NOESYスペクトル6) とおのおののドメインの帰属にもとづき,PPDとSBDを連結させたコンストラクト,RBDとSBDを連結させたコンストラクト,さらに,全長のトリガー因子の主鎖アミドおよび側鎖メチル基のNMR信号を帰属した.
分子シャペロンと基質タンパク質との複合体の構造生物学な研究におけるもうひとつの困難は,変性状態の基質タンパク質の扱いにくさである.通常,タンパク質を変性状態に保つには尿素やグアニジンなどの高濃度の変性剤が必要であるが,分子シャペロンとの複合体の形成のためには変性剤を取り除かなければならない.変性剤を取り除いたあとの基質タンパク質は凝集しやすく,構造解析に適した均一な試料を得ることは困難である.一方,天然変性タンパク質などは変性状態において可溶であるが,分子シャペロンとの相互作用に必要な疎水性クラスターをもたない.そこで,酸化還元状態によりそのフォールディングが制御され,さらに,細胞においてトリガー因子との相互作用が示されているPhoAを基質タンパク質として用いた7).PhoAは還元状態では変性状態となり,変性タンパク質に特徴的な分散の狭いNMRスペクトルを示した.変性状態のPhoAのシグナル強度は均一で,可溶性も高く,分子シャペロンとの複合体の解析に理想的な基質タンパク質であった.全長のPhoAを4つのフラグメントに分割し,それぞれに対して連鎖帰属を行い,ほぼすべての主鎖アミドのNMR信号の帰属を完了した.高次構造をもたない変性タンパク質としての特性から予想されたように,全長のPhoAとフラグメントのPhoAのNMR信号はほぼ完全に一致し,変性状態の全長PhoAはフラグメントPhoAの単純な足し合わせとして理解できることが示された.
トリガー因子とPhoAとの複合体の立体構造解析にさきだち,NMR滴定実験によりPhoAおよびトリガー因子における相互作用領域を決定した.安定同位体により標識された全長のPhoAおよびフラグメントのPhoAに対しトリガー因子を滴定し,PhoAにおける基質結合部位を決定した.さらに,安定同位体により標識されたトリガー因子に対し全長PhoAならびにフラグメントPhoAを滴定し,トリガー因子における基質結合部位および結合の特異性を決定した.その結果,トリガー因子において4箇所,PhoAにおいて7箇所の基質結合部位が存在し,3分子のトリガー因子が1分子のPhoAと結合することが明らかになった(図1b).短いPhoAフラグメントから長いPhoAフラグメントまで段階的に領域を変えて相互作用を解析した結果,PhoAにおけるいくつかの基質結合部位はトリガー因子における特定の基質結合部位と特異的に結合し,長いPhoAフラグメントはその特異性の組合せにより特定のコンフォメーションに落ち着くことが明らかになった(図1b).さらに,NMR緩和分散法によるダイナミクスの解析および等温滴定型熱量測定による解析の結果,PhoAフラグメントが長くなるにつれてトリガー因子との結合は強まり,結合-解離の交換も遅くなることが示された.しかし,長いPhoAフラグメントであってもトリガー因子とPhoAとの相互作用はダイナミックであり,比較的速い交換が起こっていることが明らかになった.このダイナミックな結合および解離が,タンパク質のフォールディングを補助するというトリガー因子の分子シャペロンとしてのもうひとつの機能を担っていると考えられた.
全長のPhoAに結合した3分子のトリガー因子はそれぞれ独立していたため,PhoAを3つのセグメントに分割してトリガー因子との複合体を決定した,メチル基選択的安定同位体標識法により標識された試料に対し各種のNOESYスペクトルを測定し束縛条件を取得した.構造決定の結果,トリガー因子に存在する4箇所の独立した基質結合部位がPhoAの疎水性の領域,とくに芳香族アミノ酸残基を含む領域を認識し,トリガー因子のアーム1およびアーム2により形成される空洞の内部にPhoAが格納されていることが明らかになった.また,複数の基質結合部位を介した結合によりPhoAは引き伸ばされた状態に保持されていた.このように,トリガー因子は基質タンパク質の疎水性の領域を互いに引き離した状態で保持することにより,その凝集およびミスフォールディングを防いでいることが明らかになった.3つのセグメント複合体の構造を比較したところ,トリガー因子の基質結合部位による基質タンパク質の認識はゆるく,認識するアミノ酸配列に傾向はあるものの,それほど厳密には制限されていなかった.トリガー因子の相互作用面の立体構造の比較から,相互作用面を形成する残基は基質タンパク質のアミノ酸配列の違いにより柔軟に変化し,それによって多様なアミノ酸配列を認識することが明らかになった.この柔軟性が,ほぼすべての新生ポリペプチド鎖と相互作用するトリガー因子の汎用性において重要であると考えられた.トリガー因子の相互作用面にある疎水性残基をAlaあるいは親水性残基に変異させると,変性状態のPhoAとの親和性は低下し,抗凝集活性も低下した.
トリガー因子がどのようにタンパク質の凝集を抑制するのかを調べるため,MBP変異体8) を用いたNMR実験を行った.このMBP変異体は野生型のMBPと同じ立体構造をとるものの,熱安定性が低く,高温(30℃以上)において多量の凝集体を形成する.温度を変化させながらトリガー因子とのNMR滴定実験を行った結果,22℃以下の低温ではMBP変異体とトリガー因子との相互作用は観測されなかったのに対し,30℃以上の高温ではMBP変異体とトリガー因子とは相互作用することが明らかになった.PhoAとトリガー因子との相互作用に用いられていたトリガー因子のもつ4箇所すべての基質結合部位が,MBP変異体との相互作用においても用いられていた.さらに,トリガー因子の存在のもとではMBP変異体は50℃まで立体構造を保持し可溶であることが明らかになった.以上より,MBP変異体は非変性と変性との平衡状態にあり,トリガー因子は変性状態のMBP変異体と相互作用して凝集を抑制しその熱安定性を高めていることが示された.一方で,トリガー因子とMBP変異体とを融合し単一のポリペプチド鎖としたコンストラクトを作製したところ,MBP変異体は変性状態で保持された.単一のポリペプチド鎖とすることは基質タンパク質とトリガー因子とが共局在した状態,つまり,リボソームにおいて伸長している新生ポリペプチド鎖とトリガー因子とが相互作用している状態を再現すると考えられた.この結果より,リボソームにおいてトリガー因子と基質タンパク質とが共局在した状態では,トリガー因子はそのアンフォールダーゼ活性により基質タンパク質のフォールディングをさまたげ,伸長している新生ポリペプチド鎖のミスフォールディングを防いでいることが示された.
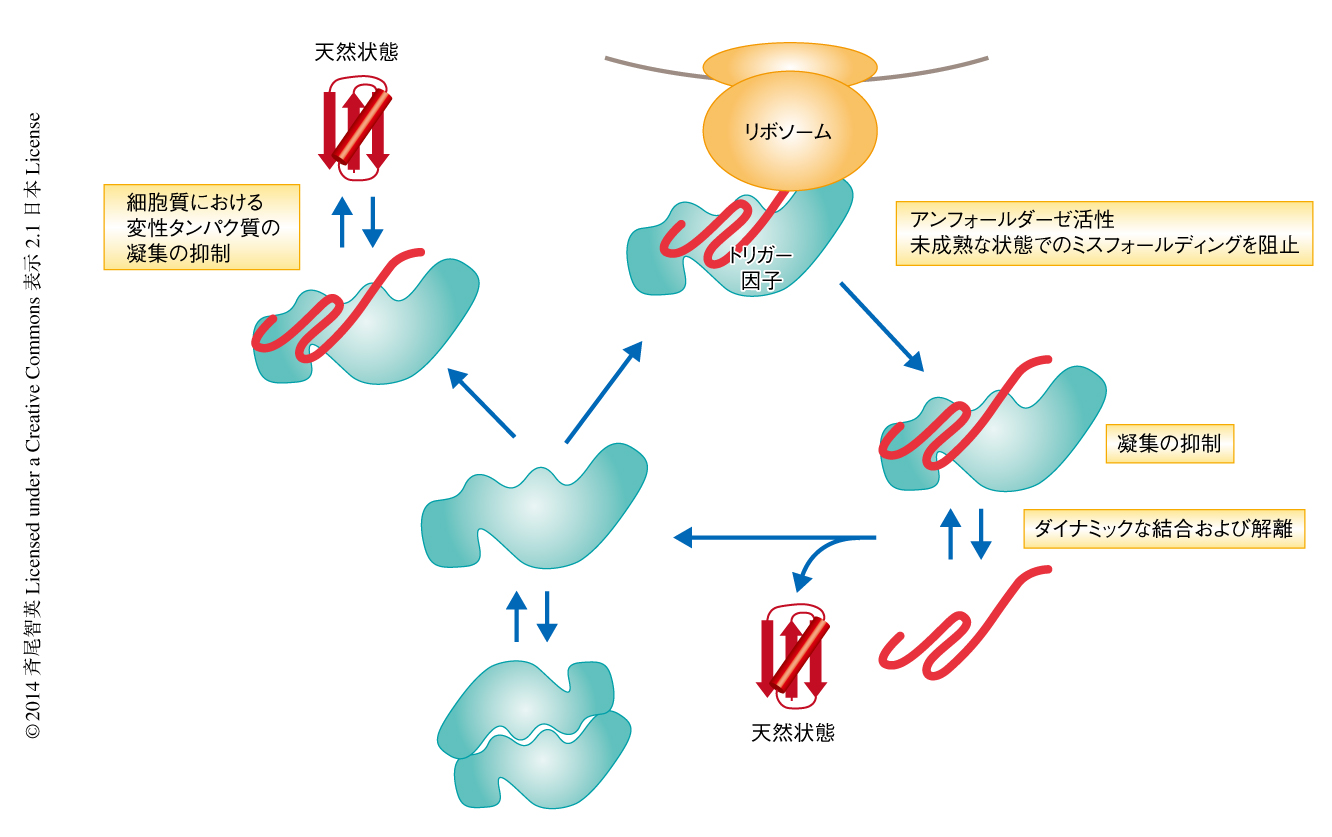
以上のように,トリガー因子はリボソームに結合した状態では基質タンパク質の尚早なフォールディングを抑制し,リボソームから解離したのちには基質タンパク質とのダイナミックな結合および解離によりその凝集を防ぎフォールディングを助ける(図2).細胞質においても,変性状態のタンパク質に一過的に結合することにより凝集を防ぎリフォールディングのチャンスをあたえる.トリガー因子はアンフォールダーゼ活性と抗凝集活性という異なる活性を使い分け,タンパク質の新生,フォールディングから細胞質におけるタンパク質の恒常性の維持まで多様な役割をはたしている.
30年ほどまえにトリガー因子の存在と変性タンパク質との相互作用が示されてから9),トリガー因子の機能や基質特異性がさまざまな手法により調べられ,細胞におけるタンパク質のフォールディング,Sec系と協調したタンパク質の輸送など,トリガー因子の多様な機能が明らかにされてきた.同時に,X線結晶解析を主体としたトリガー因子の立体構造解析も進められてきたが,トリガー因子の分子シャペロンとしての機能の解明においてもっとも重要な,変性状態の基質タンパク質との複合体の立体構造は長いあいだ明らかにされていなかった.変性状態のタンパク質の扱いにくさや運動性の高さ,弱くダイナミックな結合,高分子量などがさまたげとなり,X線結晶解析およびNMR解析を困難にしていた.これは,トリガー因子のみならず,すべての分子シャペロンと基質タンパク質との複合体の解析に共通した問題であった.筆者らは,高分子量タンパク質に特化した安定同位体標識法およびスペクトル測定法によりNMR法における分子量の制約をのりこえ,トリガー因子と変性状態の基質タンパク質との複合体の立体構造を決定した.もともと動的な分子や弱く過渡的な相互作用の解析にすぐれるNMR法が高分子量タンパク質に対しても応用できるようになったいま,NMR法は分子シャペロンの分子機構の解明においてもっとも強力なツールのひとつであるといえるだろう.今後,分子シャペロンの分子機構の解明,さらには,システム全体への理解へと研究を発展させるうえで,NMR法による研究は鍵をにぎると期待される.
略歴:2011年 北海道大学大学院生命科学院博士課程 修了,同年より米国Rutgers大学 博士研究員.
研究テーマ:NMR法による生体分子の作用機序の解明.
関心事:タンパク質のフォールディングおよび輸送における分子シャペロンの機能および作用の機序.
Charalampos G. Kalodimos
米国Rutgers大学 教授.
研究室URL:http://chem.rutgers.edu/~babis/
© 2014 斉尾智英・Charalampos G. Kalodimos Licensed under CC 表示 2.1 日本
(米国Rutgers大学Center for Integrative Proteomics Research)
email:斉尾智英
DOI: 10.7875/first.author.2014.065
Structural basis for protein antiaggregation activity of the trigger factor chaperone.
Tomohide Saio, Xiao Guan, Paolo Rossi, Anastassios Economou, Charalampos G. Kalodimos
Science, 344, 1250494 (2014)
要 約
分子シャペロンはタンパク質の凝集やミスフォールディングを防ぐことにより,タンパク質の品質管理において主要な役割をはたすが,その分子機構はほとんど明らかにされていない.筆者らは,細菌における分子シャペロンのひとつであるトリガー因子とその基質であるPhoAに対し,NMR法を主体とした構造解析,相互作用解析,ダイナミクス解析を行った.その結果,トリガー因子は4箇所の基質結合部位により基質タンパク質の疎水性の領域と結合することにより,基質タンパク質を変性状態に保ち凝集を抑制していることが明らかになった.トリガー因子の基質結合部位は,結合する基質タンパク質により局所の構造を柔軟に変化させることによって基質タンパク質の幅広い認識を可能にしていた.
はじめに
分子シャペロンは変性タンパク質の凝集を防ぐことにより,タンパク質のフォールディングや品質管理などにおいて重要な機能をはたす1).とくに,リボソームにより合成されたばかりの新生ポリペプチド鎖は立体構造をもたず,本来はタンパク質のコアを形成する疎水性の領域が露出しているため凝集の危険にさらされている.細菌における主要な分子シャペロンのひとつであるトリガー因子は,リボソームにおいて新生ポリペプチド鎖の出口となる領域に結合し,リボソームから出てきた変性状態のポリペプチド鎖に結合することによりその凝集を防ぐ2,3).新生ポリペプチド鎖はDnaK,DnaJ,GroEL-GroESなどといったほかの分子シャペロンにうけわたされ,分子シャペロンとの結合および解離をへて折りたたまれていく.また,トリガー因子は細胞質にも豊富に存在し,細胞質タンパク質の凝集の抑制による恒常性の維持,サブユニットタンパク質の集合体への組み込みの補助など,多様な機能をもつと考えられている2).これまでの研究により,トリガー因子単独3),トリガー因子-リボソーム複合体3),トリガー因子と折りたたまれた基質タンパク質との複合体4) の結晶構造が決定されているが,トリガー因子と変性状態の基質タンパク質との複合体の構造は決定されておらず,トリガー因子の主要な機能である変性状態のタンパク質との相互作用についての情報はごくかぎられていた.トリガー因子のみならず,ほかの分子シャペロンについても,変性状態の基質タンパク質の認識における分子機構はほとんど未解明であった.その最大の理由は,分子シャペロンと基質とのあいだのダイナミックかつ過渡的な相互作用,および,複合体の大きさにあった.分子を溶液状態で観測するNMR法は動的な系や弱い相互作用の解析に適しているが,従来のNMR法では分子量による制限が大きな壁になっていた.筆者らは,メチル基選択的安定同位体標識法5) やメチルTROSY法6) などを駆使した最新のNMR法により分子量の制約をのりこえ,変性状態の基質タンパク質と結合したトリガー因子の立体構造を決定した.
1.トリガー因子のNMR解析
432アミノ酸残基からなる大腸菌のトリガー因子は,RBD,SBD,PPDの3つのドメインから構成され(図1a),その細長く特徴的な立体構造は,PPDを頭,SBDのアーム1およびアーム2を翼,RBDを尾にみたてて“dragon shape”とも形容される.トリガー因子は溶液においては100 kDaの二量体を形成し,NMRの対象としては高分子量であるが,高分子量タンパク質に特化した安定同位体標識法およびスペクトル測定法は,良好なNMRスペクトルを得ることを可能にした.なかでも,タンパク質に豊富に存在するメチル基を特異的に13C1H3により標識し,ほかを2Hにより標識するメチル基選択的安定同位体標識法5) は,重要な立体構造情報を失わずに横緩和を抑制することによりNMR信号を先鋭化かつ高感度化する強力な手法である.全長のトリガー因子のNMR信号の帰属のため,まず,おのおののドメインを分離したコンストラクトを作製し,それぞれに対し各種の3次元測定を行い主鎖アミドおよび側鎖のNMR信号を帰属した.ついで,NOESYスペクトル6) とおのおののドメインの帰属にもとづき,PPDとSBDを連結させたコンストラクト,RBDとSBDを連結させたコンストラクト,さらに,全長のトリガー因子の主鎖アミドおよび側鎖メチル基のNMR信号を帰属した.
2.変性状態の基質タンパク質のNMR解析
分子シャペロンと基質タンパク質との複合体の構造生物学な研究におけるもうひとつの困難は,変性状態の基質タンパク質の扱いにくさである.通常,タンパク質を変性状態に保つには尿素やグアニジンなどの高濃度の変性剤が必要であるが,分子シャペロンとの複合体の形成のためには変性剤を取り除かなければならない.変性剤を取り除いたあとの基質タンパク質は凝集しやすく,構造解析に適した均一な試料を得ることは困難である.一方,天然変性タンパク質などは変性状態において可溶であるが,分子シャペロンとの相互作用に必要な疎水性クラスターをもたない.そこで,酸化還元状態によりそのフォールディングが制御され,さらに,細胞においてトリガー因子との相互作用が示されているPhoAを基質タンパク質として用いた7).PhoAは還元状態では変性状態となり,変性タンパク質に特徴的な分散の狭いNMRスペクトルを示した.変性状態のPhoAのシグナル強度は均一で,可溶性も高く,分子シャペロンとの複合体の解析に理想的な基質タンパク質であった.全長のPhoAを4つのフラグメントに分割し,それぞれに対して連鎖帰属を行い,ほぼすべての主鎖アミドのNMR信号の帰属を完了した.高次構造をもたない変性タンパク質としての特性から予想されたように,全長のPhoAとフラグメントのPhoAのNMR信号はほぼ完全に一致し,変性状態の全長PhoAはフラグメントPhoAの単純な足し合わせとして理解できることが示された.
3.PhoAはトリガー因子と特異的かつダイナミックに結合する
トリガー因子とPhoAとの複合体の立体構造解析にさきだち,NMR滴定実験によりPhoAおよびトリガー因子における相互作用領域を決定した.安定同位体により標識された全長のPhoAおよびフラグメントのPhoAに対しトリガー因子を滴定し,PhoAにおける基質結合部位を決定した.さらに,安定同位体により標識されたトリガー因子に対し全長PhoAならびにフラグメントPhoAを滴定し,トリガー因子における基質結合部位および結合の特異性を決定した.その結果,トリガー因子において4箇所,PhoAにおいて7箇所の基質結合部位が存在し,3分子のトリガー因子が1分子のPhoAと結合することが明らかになった(図1b).短いPhoAフラグメントから長いPhoAフラグメントまで段階的に領域を変えて相互作用を解析した結果,PhoAにおけるいくつかの基質結合部位はトリガー因子における特定の基質結合部位と特異的に結合し,長いPhoAフラグメントはその特異性の組合せにより特定のコンフォメーションに落ち着くことが明らかになった(図1b).さらに,NMR緩和分散法によるダイナミクスの解析および等温滴定型熱量測定による解析の結果,PhoAフラグメントが長くなるにつれてトリガー因子との結合は強まり,結合-解離の交換も遅くなることが示された.しかし,長いPhoAフラグメントであってもトリガー因子とPhoAとの相互作用はダイナミックであり,比較的速い交換が起こっていることが明らかになった.このダイナミックな結合および解離が,タンパク質のフォールディングを補助するというトリガー因子の分子シャペロンとしてのもうひとつの機能を担っていると考えられた.
4.トリガー因子は4箇所の基質結合部位により基質タンパク質を引き伸ばした状態で保持する
全長のPhoAに結合した3分子のトリガー因子はそれぞれ独立していたため,PhoAを3つのセグメントに分割してトリガー因子との複合体を決定した,メチル基選択的安定同位体標識法により標識された試料に対し各種のNOESYスペクトルを測定し束縛条件を取得した.構造決定の結果,トリガー因子に存在する4箇所の独立した基質結合部位がPhoAの疎水性の領域,とくに芳香族アミノ酸残基を含む領域を認識し,トリガー因子のアーム1およびアーム2により形成される空洞の内部にPhoAが格納されていることが明らかになった.また,複数の基質結合部位を介した結合によりPhoAは引き伸ばされた状態に保持されていた.このように,トリガー因子は基質タンパク質の疎水性の領域を互いに引き離した状態で保持することにより,その凝集およびミスフォールディングを防いでいることが明らかになった.3つのセグメント複合体の構造を比較したところ,トリガー因子の基質結合部位による基質タンパク質の認識はゆるく,認識するアミノ酸配列に傾向はあるものの,それほど厳密には制限されていなかった.トリガー因子の相互作用面の立体構造の比較から,相互作用面を形成する残基は基質タンパク質のアミノ酸配列の違いにより柔軟に変化し,それによって多様なアミノ酸配列を認識することが明らかになった.この柔軟性が,ほぼすべての新生ポリペプチド鎖と相互作用するトリガー因子の汎用性において重要であると考えられた.トリガー因子の相互作用面にある疎水性残基をAlaあるいは親水性残基に変異させると,変性状態のPhoAとの親和性は低下し,抗凝集活性も低下した.
5.トリガー因子は基質タンパク質と共局在したときにはこれをアンフォールディングする
トリガー因子がどのようにタンパク質の凝集を抑制するのかを調べるため,MBP変異体8) を用いたNMR実験を行った.このMBP変異体は野生型のMBPと同じ立体構造をとるものの,熱安定性が低く,高温(30℃以上)において多量の凝集体を形成する.温度を変化させながらトリガー因子とのNMR滴定実験を行った結果,22℃以下の低温ではMBP変異体とトリガー因子との相互作用は観測されなかったのに対し,30℃以上の高温ではMBP変異体とトリガー因子とは相互作用することが明らかになった.PhoAとトリガー因子との相互作用に用いられていたトリガー因子のもつ4箇所すべての基質結合部位が,MBP変異体との相互作用においても用いられていた.さらに,トリガー因子の存在のもとではMBP変異体は50℃まで立体構造を保持し可溶であることが明らかになった.以上より,MBP変異体は非変性と変性との平衡状態にあり,トリガー因子は変性状態のMBP変異体と相互作用して凝集を抑制しその熱安定性を高めていることが示された.一方で,トリガー因子とMBP変異体とを融合し単一のポリペプチド鎖としたコンストラクトを作製したところ,MBP変異体は変性状態で保持された.単一のポリペプチド鎖とすることは基質タンパク質とトリガー因子とが共局在した状態,つまり,リボソームにおいて伸長している新生ポリペプチド鎖とトリガー因子とが相互作用している状態を再現すると考えられた.この結果より,リボソームにおいてトリガー因子と基質タンパク質とが共局在した状態では,トリガー因子はそのアンフォールダーゼ活性により基質タンパク質のフォールディングをさまたげ,伸長している新生ポリペプチド鎖のミスフォールディングを防いでいることが示された.
以上のように,トリガー因子はリボソームに結合した状態では基質タンパク質の尚早なフォールディングを抑制し,リボソームから解離したのちには基質タンパク質とのダイナミックな結合および解離によりその凝集を防ぎフォールディングを助ける(図2).細胞質においても,変性状態のタンパク質に一過的に結合することにより凝集を防ぎリフォールディングのチャンスをあたえる.トリガー因子はアンフォールダーゼ活性と抗凝集活性という異なる活性を使い分け,タンパク質の新生,フォールディングから細胞質におけるタンパク質の恒常性の維持まで多様な役割をはたしている.
おわりに
30年ほどまえにトリガー因子の存在と変性タンパク質との相互作用が示されてから9),トリガー因子の機能や基質特異性がさまざまな手法により調べられ,細胞におけるタンパク質のフォールディング,Sec系と協調したタンパク質の輸送など,トリガー因子の多様な機能が明らかにされてきた.同時に,X線結晶解析を主体としたトリガー因子の立体構造解析も進められてきたが,トリガー因子の分子シャペロンとしての機能の解明においてもっとも重要な,変性状態の基質タンパク質との複合体の立体構造は長いあいだ明らかにされていなかった.変性状態のタンパク質の扱いにくさや運動性の高さ,弱くダイナミックな結合,高分子量などがさまたげとなり,X線結晶解析およびNMR解析を困難にしていた.これは,トリガー因子のみならず,すべての分子シャペロンと基質タンパク質との複合体の解析に共通した問題であった.筆者らは,高分子量タンパク質に特化した安定同位体標識法およびスペクトル測定法によりNMR法における分子量の制約をのりこえ,トリガー因子と変性状態の基質タンパク質との複合体の立体構造を決定した.もともと動的な分子や弱く過渡的な相互作用の解析にすぐれるNMR法が高分子量タンパク質に対しても応用できるようになったいま,NMR法は分子シャペロンの分子機構の解明においてもっとも強力なツールのひとつであるといえるだろう.今後,分子シャペロンの分子機構の解明,さらには,システム全体への理解へと研究を発展させるうえで,NMR法による研究は鍵をにぎると期待される.
文 献
- Hartl, F. U., Bracher, A. & Hayer-Hartl, M.: Molecular chaperones in protein folding and proteostasis. Nature, 475, 324-332 (2011)[PubMed]
- Hoffmann, A., Bukau, B. & Kramer, G.: Structure and function of the molecular chaperone Trigger Factor. Biochim. Biophys. Acta, 1803, 650-661 (2010)[PubMed]
- Ferbitz, L., Maier, T., Patzelt, H. et al.: Trigger factor in complex with the ribosome forms a molecular cradle for nascent proteins. Nature, 431, 590-596 (2004)[PubMed]
- Martinez-Hackert, E. & Hendrickson, W. A.: Promiscuous substrate recognition in folding and assembly activities of the trigger factor chaperone. Cell, 138, 923-934 (2009)[PubMed]
- Sprangers, R., Velyvis, A. & Kay, L. E.: Solution NMR of supramolecular complexes: providing new insights into function. Nat. Methods, 4, 697-703 (2007)[PubMed]
- Sprangers R. & Kay, L. E.: Quantitative dynamics and binding studies of the 20S proteasome by NMR. Nature, 445, 618-622 (2007)[PubMed]
- Gouridis, G., Karamanou, S., Gelis, I. et al.: Signal peptides are allosteric activators of the protein translocase. Nature, 462, 363-367 (2009)[PubMed]
- Betton, J. M. & Hofnung, M.: Folding of a mutant maltose-binding protein of Escherichia coli which forms inclusion bodies. J. Biol. Chem., 271, 8046-8052 (1996)[PubMed]
- Crooke, E. & Wickner, W.: Trigger factor: a soluble protein that folds pro-OmpA into a membrane-assembly-competent form. Proc. Natl. Acad. Sci. USA, 84, 5216-5220 (1987)[PubMed]
著者プロフィール
略歴:2011年 北海道大学大学院生命科学院博士課程 修了,同年より米国Rutgers大学 博士研究員.
研究テーマ:NMR法による生体分子の作用機序の解明.
関心事:タンパク質のフォールディングおよび輸送における分子シャペロンの機能および作用の機序.
Charalampos G. Kalodimos
米国Rutgers大学 教授.
研究室URL:http://chem.rutgers.edu/~babis/
© 2014 斉尾智英・Charalampos G. Kalodimos Licensed under CC 表示 2.1 日本