Cas9-ガイド鎖RNA-標的DNA三者複合体の結晶構造
西増弘志・濡木 理
(東京大学大学院理学系研究科 生物化学専攻)
email:西増弘志,濡木 理
DOI: 10.7875/first.author.2014.028
Crystal structure of Cas9 in complex with guide RNA and target DNA.
Hiroshi Nishimasu, F. Ann Ran, Patrick D. Hsu, Silvana Konermann, Soraya I. Shehata, Naoshi Dohmae, Ryuichiro Ishitani, Feng Zhang, Osamu Nureki
Cell, 156, 935-949 (2014)
原核生物のもつ獲得免疫機構CRISPR-Cas系にかかわるRNA依存性DNAヌクレアーゼCas9は,ガイド鎖RNAと協同し標的となる2本鎖DNAを切断する.Cas9を応用したゲノム編集技術が急速な進展をみせる一方で,Cas9の構造情報は不明であった.今回,筆者らは,Cas9-ガイド鎖RNA-標的DNA三者複合体の結晶構造を2.5Å分解能で解明した.結晶構造および変異体の解析から,1)Cas9は2つのローブから構成されること,2)ガイド鎖RNAと標的DNAからなるヘテロ2本鎖は2つのローブのあいだに結合すること,3)HNHドメインおよびRuvCドメインの2つのヌクレアーゼドメインはそれぞれ,標的2本鎖DNAの相補鎖および非相補鎖を切断するのに適した位置に存在すること,4)Cas9のC末端領域はDNA切断に必須の塩基配列であるPAMの認識にかかわること,が明らかになった.今回,明らかになったCas9-ガイド鎖RNA-標的DNA三者複合体の構造は,Cas9によるRNA依存性のDNA切断機構の構造基盤をあたえるのみならず,新たなゲノム編集ツールの合理的な設計の基盤ともなることが期待される.
CRISPR-Cas系(CRISPR:clustered regularly interspaced short palindromic repeat,Cas:CRISPR-associated protein)は原核生物のもつ獲得免疫機構であり,ファージやプラスミドなど外来DNAに対する防御を担う1).細胞に侵入した外来DNAはCasのはたらきによりゲノムに存在するCRISPR領域に取り込まれる.別のCasはCRISPR領域から転写されたガイド鎖RNAと複合体を形成し,ガイド鎖RNAと相補的な外来DNAを認識して切断する.CRISPR-Cas系には複数のCasが関与し,その違いなどにもとづきI型~III型に大別される.II型CRISPR-Cas系はさらにIIA型~IIC型に分けられる.
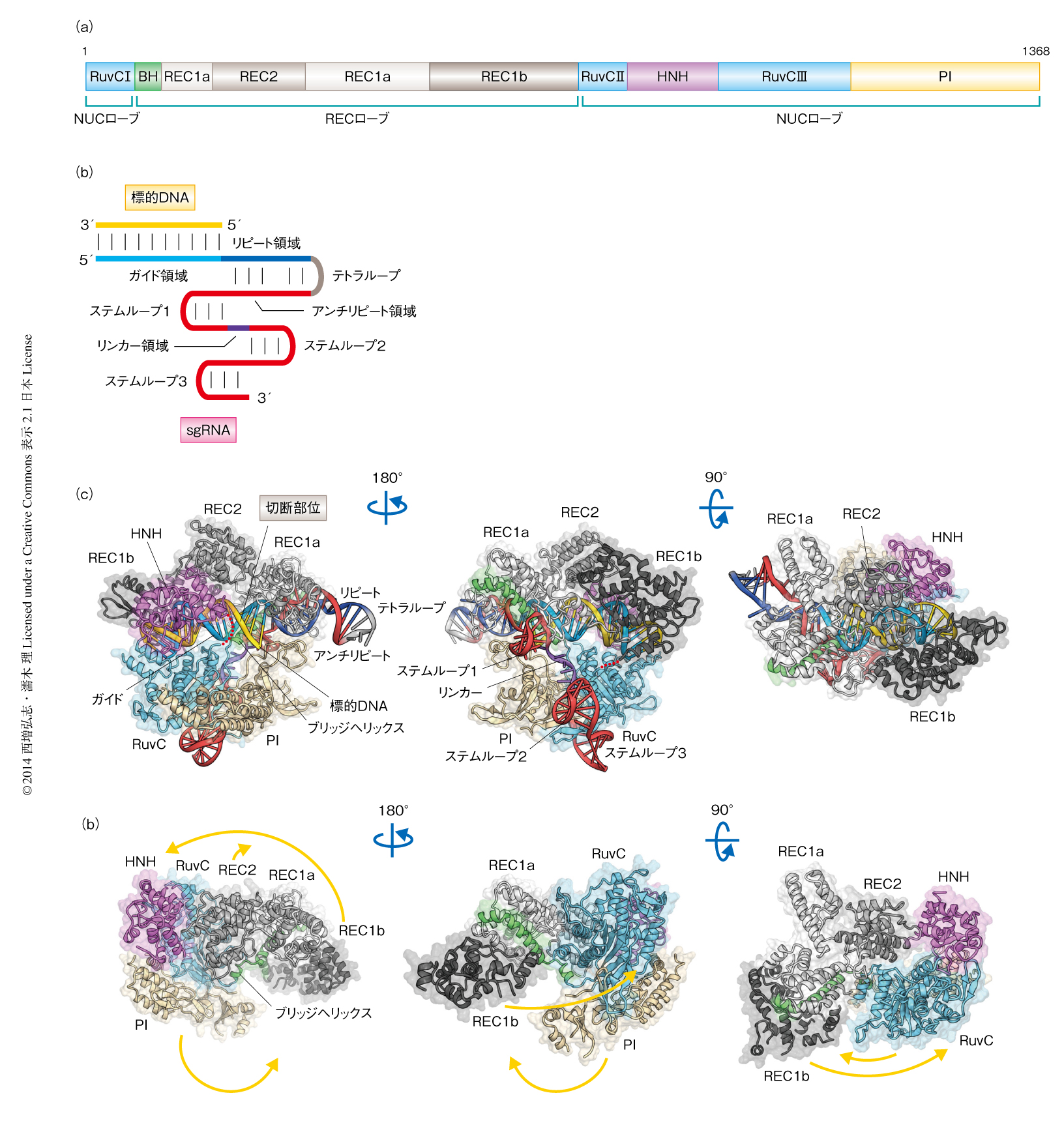
Cas9はII型CRISPR-Cas系にかかわる100~150 kDaのRNA依存性DNAエンドヌクレアーゼであり,RuvCドメインおよびHNHドメインの2つのヌクレアーゼドメインをもつ2,3)(図1a).それ以外の領域は既知のタンパク質との配列相同性をもたず,その機能はわかっていなかった.Cas9はcrRNA(CRISPR RNA)およびtracrRNA(trans-activating crRNA)とよばれる2種類のRNAと複合体を形成し,crRNAのもつ20塩基のガイド配列と相補的な標的となる2本鎖DNAを切断する.標的2本鎖DNAのうち,ガイド配列と相補的なDNA鎖はHNHドメインにより切断され,もう一方のDNA鎖はRuvCドメインにより切断される.標的2本鎖DNAの切断には,ガイド配列との相補性にくわえ,標的となる配列の近傍に存在するPAM(protospacer adjacent motif)とよばれる特定の塩基配列が必須である.crRNAとtracrRNAとを人工的なリンカーにより連結したsgRNA(single-guide RNA)も,ガイド鎖RNAとして機能する2)(図1b).
Cas9およびsgRNAを細胞に共発現させることによりゲノムDNAを配列特異的に切断できることから,Cas9はさまざまな生物種において利用可能なゲノム編集ツールとして注目されている4,5).Cas9を応用したゲノム編集技術により,これまで困難だった複数の遺伝子の同時ノックアウトや,霊長類におけるノックアウト動物の作製が迅速かつ簡便に可能であることが報告されている6,7).さらに,Cas9の応用はゲノム編集にとどまることなく,不活性型Cas9を用いた転写制御技術やライブイメージング技術など新規の技術が続々と報告されている.このように,Cas9を応用した新規の技術が生命科学に革命を起こしつつある一方,Cas9の作動する分子機構は不明であった.
Streptococcus pyogenesに由来するCas9(残基1~残基1368)とsgRNA(98塩基),および,標的DNA(23塩基)との三者複合体の結晶構造を2.5Å分解能で決定した(PDB ID:4OO8,図1c).Cas9の性状を改善するため,Cys80およびCys574の2つのシステイン残基をそれぞれロイシン残基およびグルタミン酸残基に置換した.DNA切断実験により,これらの変異は酵素活性に影響しないことが確認された.さらに,標的DNAの切断を防ぐため,RuvCドメインのAsp10およびHNHドメインのHis840の2つの活性残基をアラニン残基に置換した.
結晶構造から,Cas9はRECローブ(REC:recognition)およびNUCローブ(NUC:nuclease)と名づけた2つのローブからなることが明らかになった(図1 a, c).RECローブは,アルギニン残基に富むαヘリックス(ブリッジヘリックスと名づけた),REC1ドメイン,REC2ドメインから構成されていた(最近,報告されたCas9の単体の構造8) との比較から,REC1ドメインはREC1aおよびREC1bの2つのサブドメインに分けられることが明らかになった).NUCローブはRuvCドメイン,HNHドメイン,PIドメイン(PI:PAM-interacting)から構成されていた.1次構造において分散して存在する3つのRuvCモチーフ(RuvC I~RuvC III)は集合し,立体構造においてひとつのRuvCドメインを形成していた.RuvCドメインとPIドメインとは密に相互作用していた一方,HNHドメインはRuvC IIとRuvC IIIとのあいだに存在し,NUCローブとはほとんど相互作用していなかった.RECローブとNUCローブはブリッジヘリックス,および,REC1とRuvC IIとのあいだの天然変性リンカーにより連結していた.sgRNAと標的DNAはヘテロ2本鎖を形成し,2つのローブのあいだに収容されていた.
sgRNAはcrRNA配列とtracrRNA配列,および,それらを連結するテトラループから構成される(図1b).crRNA配列はガイド領域(20塩基)とリピート領域(12塩基)からなり,tracrRNA配列はアンチリピート領域(14塩基)と3'側テイル領域からなる.結晶構造から,sgRNAは標的DNAと結合し,ガイド領域:標的DNAヘテロ2本鎖,リピート領域:アンチリピート領域2本鎖,3つのステムループ(ステムループ1~ステムループ3),および,1本鎖のリンカー領域から構成されるT字型の構造をとることが明らかになった(図1c).
sgRNAのガイド領域と標的DNAは20対のWatson-Crick塩基対を介してガイド領域:標的DNAヘテロ2本鎖を形成し,sgRNAのリピート領域とアンチリピート領域は9対のWatson-Crick塩基対を介してリピート領域:アンチリピート領域2本鎖を形成していた(図1c).リピート領域:アンチリピート領域2本鎖にある6塩基,G27,A28,A41,A42,A43,U44は塩基対を形成せず,A28とU44はフリップアウトして特徴的な構造を形成していた.塩基配列から予想されたように,3'側テイル領域は2つのステムループ(ステムループ2およびステムループ3)を形成していた.さらに,3'側テイル領域は塩基配列からは予想されていなかったステムループ1を形成していることが明らかになった.
RECローブはおもにαヘリックスから構成されており,既知のタンパク質とは構造類似性をもたない新規な構造をとっていた(図1c).REC1ドメインはリピート領域:アンチリピート領域2本鎖を認識していた.変異体の解析の結果,REC1ドメインはCas9の機能に必須であることが示された.一方,REC2ドメインはsgRNAとは相互作用しておらず,変異体の解析の結果からもCas9の機能に必須ではないことが示された.この結果は,Cas9のオルソログの多くはREC2ドメインをもたないことと一致していた.REC2ドメインの役割は現時点では不明である.
RuvCドメインおよびHNHドメインの活性部位はそれぞれ,RNase Hフォールドおよびββα-メタルフォールドをとっていた(図1c).したがって,RuvCドメインおよびHNHドメインは既知のヌクレアーゼと同様の反応機構により,標的2本鎖DNAを形成する非相補鎖および相補鎖をそれぞれを切断することが示唆された.一方,RuvCドメインおよびHNHドメインには既知のヌクレアーゼとは異なる構造的な特徴も存在した.たとえば,RuvCヌクレアーゼは二量体を形成することによりHolliday構造を認識するのに対し,Cas9のもつRuvCドメインは二量体を形成せず,ほかのドメインとの相互作用にかかわる領域をもっていた.
RECローブにくわえ,PIドメインも新規な構造をとっていた(図1c).標的DNAおよびRuvCドメインの位置から,PIドメインはPAMの認識にかかわることが示唆された.S. pyogenesに由来するCas9とStreptococcus thermophilusに由来するCRISPR-3 Cas9は,59%の配列同一性をもちガイド鎖RNAの交換が可能である9).一方,2本鎖DNAの切断に必要なPAMの配列は異なっており,S. pyogenesのCas9は5'-NGG-3'という配列を必要とするのに対し,S. thermophilusのCas9は5'-NGGNG-3'という配列を必要とする.PIドメインがPAMの認識にかかわるのかどうかを調べるため,PIドメインを交換したキメラ変異体として,S. pyogenesのCas9のもつPIドメインをS. thermophilusのCas9のもつPIドメインに置換したCas9変異体,および,S. thermophilusのCas9のもつPIドメインをS. pyogenesのCas9のもつPIドメインに置換したCas9変異体を作製し,5'-NGG-3'あるいは5'-NGGNG-3'という配列のPAMをもつ標的2本鎖DNAに対する切断活性を測定した.その結果,S. thermophilusのCas9のもつPIドメインをS. pyogenesのCas9のものと置換したCas9変異体は,S. thermophilusのCas9とは異なり,5'-NGG-3'という配列のPAMをもつ標的2本鎖DNAを切断した.したがって,S. pyogenesのCas9のもつPIドメインは5'-NGG-3'という配列のPAMの認識に必要であり,S. thermophilusのもつCas9のPAMに対する特異性を変えるのに十分であることが示唆された.PIドメインを欠失したS. pyogenesのCas9はDNA切断活性を示さなかったことから,PIドメインはCas9の機能に必須であることが示された.これらの結果から,PIドメインはPAMの認識にかかわることが明らかになった.
HNHドメインは相補鎖DNAの3位と4位とのあいだのホスホジエステル結合を切断し,DNA切断活性にはガイド領域:標的DNAヘテロ2本鎖のうち“シード”領域とよばれるPAMの近傍の10~12塩基対が重要である2,10).結晶構造において,sgRNAのシード領域のリン酸骨格は,ブリッジヘリックスにある複数のアルギニン残基により認識されていた(図1c).変異体の解析の結果,これらのアルギニン残基はCas9の機能に重要であることが確認された.したがって,Cas9-sgRNA複合体において,sgRNAはシード領域の塩基を外側にむけてCas9と結合し,相補鎖DNAとWatson-Crick塩基対を形成することにより標的DNAを認識すると考えられた.ブリッジヘリックスのアルギニン残基は高度に保存されていることから,ブリッジヘリックスはCas9のオルソログに共通する構造的な特徴であることが示唆された.
ガイド領域:標的DNAヘテロ2本鎖はおもにRECローブにより配列非依存的に認識され,末端の塩基対はRuvCドメインにより認識されていた(図1c).これらの構造的な特徴は,Cas9はガイド配列の異なるsgRNAと結合すること2),20塩基以上のガイド配列は細胞内で分解され配列特異性の向上には貢献しないこと11),と一致していた.
ガイド領域:標的DNAヘテロ2本鎖とは対照的に,リピート領域:アンチリピート領域2本鎖はおもにRECローブにより配列依存的に認識されていた(図1c).これらの構造的な特徴は,IIA型~IIC型のCRISPR-Cas系において,リピート領域:アンチリピート領域2本鎖の塩基配列,および,RECローブのアミノ酸配列は多様であり,Cas9は近縁のsgRNAのみをガイド鎖RNAとして利用できるという報告9) と一致していた.
ステムループ1はRECローブとNUCローブの両方と相互作用していた一方,ステムループ2およびステムループ3はNUCローブのみと相互作用しCas9との結合を補強していた(図1c).48塩基のtracrRNA配列をもつsgRNAはin vitroにおけるDNA切断活性に必要な最小領域である一方2),67塩基あるいは85塩基の長いtracrRNA配列をもつsgRNAはin vivoにおけるDNA切断活性に重要であることが報告されている10).結晶構造から,48塩基,67塩基,85塩基のtracrRNA配列をもつsgRNAはそれぞれ,ステムループ1,ステムループ1およびステムループ2,ステムループ1~ステムループ3を含むことが明らかになった.これらの結果から,ステムループ1は機能的なCas9-sgRNA複合体の形成に必須である一方,ステムループ2およびステムループ3は複合体を安定化しin vivoにおけるDNA切断活性を上昇させると考えられた.
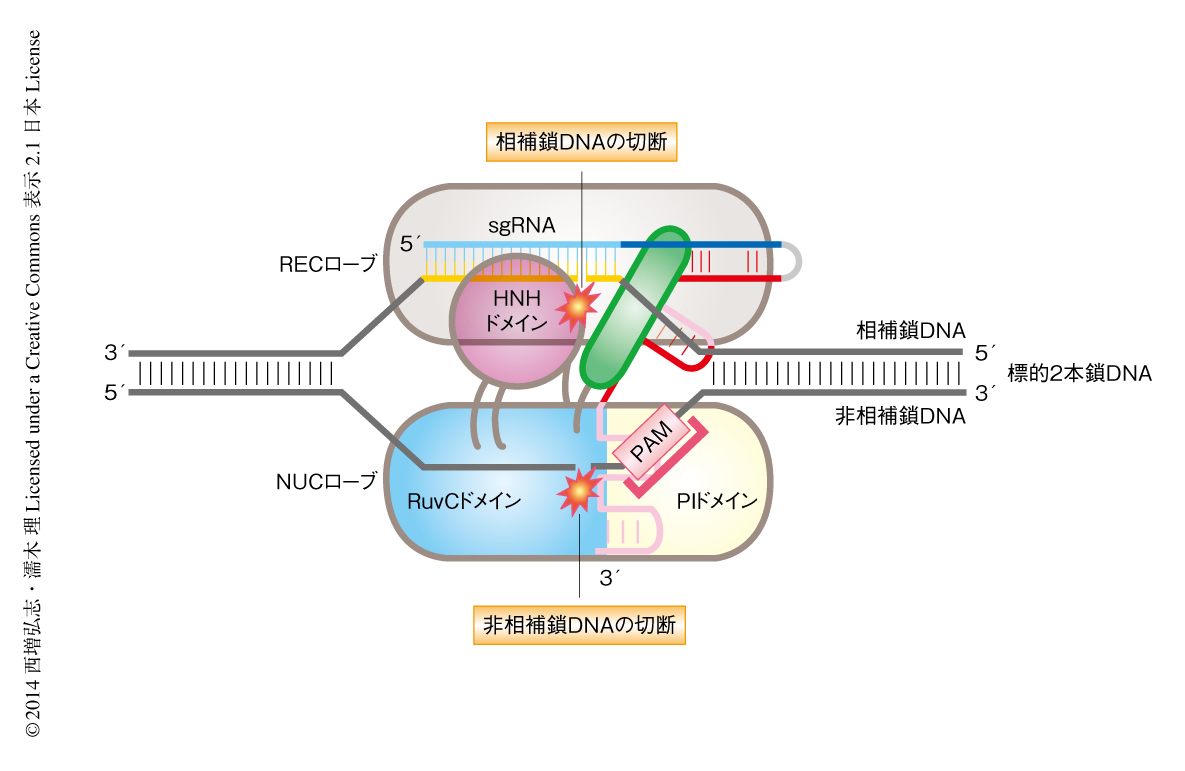
Cas9-sgRNA-標的DNA三者複合体の結晶構造においてHNHドメインは相補鎖DNAの切断部位と離れた位置に存在していた(図1c).結晶の単位格子にはコンフォメーションの異なる2つの三者複合体Mol AおよびMol Bが含まれており,それらの比較から,HNHドメインによる相補鎖DNAの切断機構を推測することができた.Mol AではHNHドメインはRuvC IIおよびRuvC IIIとそれぞれ天然変性リンカーおよびαヘリックスにより連結していた.一方,Mol BではHNHドメインの電子密度は観察されず,HNHドメインは特定の位置には固定されていないことが示唆された.さらに,Mol AにおいてHNHドメインとRuvC IIIとを連結しているαヘリックスは構造変化を起こし相補鎖DNAの方向をむいていた.これらの比較から,HNHドメインとRuvCドメインとを連結するαヘリックスの構造変化により,HNHドメインは相補鎖DNAに近づき標的DNAを切断することが示唆された(図2).最近の研究により,Cas9はPAMと結合すると,不活性型から活性型へと構造変化することが示唆された12).この結果は,今回,構造決定したCas9-sgRNA-標的DNA三者複合体はHNHドメインが切断部位と離れた不活性型の構造をとっていることと一致しており,PAMとの結合によりHNHドメインは切断部位に近づく可能性が考えられた.
Cas9がRNA依存性DNAエンドヌクレアーゼであるという2012年6月の報告2) を発端に,“CRISPR Craze”とも形容される激しい研究競争がはじまった.筆者らは,その半年のち,2013年1月に構造解析に着手した.この論文を発表した米国の研究グループは結晶構造解析のトップラボのひとつであり,すでに構造解析に着手しているのは明らかだった.しかし,筆者らにとり,競争のあることはCas9の構造解析に挑戦しない理由にはならなかった.Cas9があまりに魅力的なタンパク質だったからである.競争は激しいが勝算はゼロではないと思った.構造解析のターゲットはCas9の単体,Cas9-ガイド鎖RNA複合体,Cas9-ガイド鎖RNA-標的DNA三者複合体などいくつか存在し,さらに,核酸との複合体の結晶化には,用いる核酸の長さや塩基配列,複合体の調製法など,多くのパラメーターが影響すると予想された.したがって,ゴールはいくつもあり,さらに,それぞれのゴールまでの道筋もひとつではないため,競争は単純なスピード勝負ではないと考えた.
構造解析に着手してから1カ月,S. pyogenesに由来するCas9の単体の結晶を得ることに成功した.この結晶は構造決定には不十分だったが,半年あれば構造は決定できるだろうと感じた.しかしこの結果は,S. pyogenesに由来するCas9は結晶化に適していることにくわえ,競争相手も同様の結晶を得ているであろうことを示唆していた.そこで,Cas9単体の構造決定はあきらめ核酸との複合体に集中することにした.sgRNA:DNAヘテロ2本鎖の長さや塩基配列が核酸との複合体の結晶化に大きく影響したため,さまざまなヘテロ2本鎖とCas9との複合体を調製し,複合体ごとに結晶化スクリーニングを行った.さらに,巨大かつ新規なCas9-sgRNA-標的DNA三者複合体の構造決定は簡単ではなかった.
筆者らの論文がオンライン出版されるちょうど1週間前,競争相手と予想していた米国の研究グループがCas9の結晶構造を報告した8)(PDB ID:4CMP).予想どおり,彼らもS. pyogenesに由来するCas9を結晶化しており,結晶化の条件も筆者らが最初に見い出したものと類似していた.彼らは,Cas9の単体の構造とクライオ電子顕微鏡により決定した構造とを組み合わせることにより,Cas9単体は閉じた不活性型の構造をとること,および,Cas9はガイド鎖RNAとの結合により開いた活性型へと構造変化することを示した.このCas9単体の構造と筆者らの構造決定したCas9-sgRNA-標的DNA三者複合体の構造との比較から,この2つの構造は大きく異なることがわかった(図1 c, d).ガイド鎖RNAの結合による構造変化は想定していたが,これほど大きな構造変化は想定外だった.Cas9単体の構造ではブリッジヘリックスが折れ曲がることによりRECローブとNUCローブとが近接し密に相互作用していた(図1d).その結果,ガイド領域:標的DNAヘテロ2本鎖を収容する溝は存在していなかった.さらに,Cas9-sgRNA-標的DNA三者複合体の構造においてガイド領域:標的DNAヘテロ2本鎖を認識していたREC1bドメインは,Cas9単体の構造では大きく移動していた(図1d).Cas9単体の構造解析にさきをこされることは想定していたが,その時期は想定よりも遅かった.その理由として,Cas9単体は核酸との複合体よりもフレキシブルだったため,その構造決定には複合体とは別のむずかしさがあったこと,また,Cas9単体の構造は予想外にも閉じた構造をとっていたため,構造機能相関を説明するにはクライオ電子顕微鏡により決定した構造などの追加のデータが必要だったこと,などが考えられた.Cas9単体の構造解析をあきらめ,核酸との複合体に集中したのは正しい選択だった.もし,Cas9単体の構造解析を継続していたら完敗だった.
この研究は米国Massachusetts Institute of TechnologyのFeng Zhang博士のグループとの共同研究として行った.CRISPR研究の若きスーパースターFeng Zhangとともにしたこの1年間は,これまでの人生でもっともエキサイティングな1年だった.
略歴:2007年 東京大学大学院農学系研究科 修了,2013年より東京大学大学院理学系研究科 助教.2013年より科学技術振興機構 さきがけ研究者 兼任.
研究テーマ:RNAサイレンシング.
抱負:たった一度の人生,やりたいことを貫きたい.国際競争の激化のため研究にスピードが求められる今日だが,妥協せずにていねいな仕事をしていきたい.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究テーマ:遺伝暗号の翻訳,膜輸送,自然炎症.
抱負:細胞の生理機構を原子分解能で解明したい.
研究室URL:http://www.nurekilab.net/
© 2014 西増弘志・濡木 理 Licensed under CC 表示 2.1 日本
(東京大学大学院理学系研究科 生物化学専攻)
email:西増弘志,濡木 理
DOI: 10.7875/first.author.2014.028
Crystal structure of Cas9 in complex with guide RNA and target DNA.
Hiroshi Nishimasu, F. Ann Ran, Patrick D. Hsu, Silvana Konermann, Soraya I. Shehata, Naoshi Dohmae, Ryuichiro Ishitani, Feng Zhang, Osamu Nureki
Cell, 156, 935-949 (2014)
要 約
原核生物のもつ獲得免疫機構CRISPR-Cas系にかかわるRNA依存性DNAヌクレアーゼCas9は,ガイド鎖RNAと協同し標的となる2本鎖DNAを切断する.Cas9を応用したゲノム編集技術が急速な進展をみせる一方で,Cas9の構造情報は不明であった.今回,筆者らは,Cas9-ガイド鎖RNA-標的DNA三者複合体の結晶構造を2.5Å分解能で解明した.結晶構造および変異体の解析から,1)Cas9は2つのローブから構成されること,2)ガイド鎖RNAと標的DNAからなるヘテロ2本鎖は2つのローブのあいだに結合すること,3)HNHドメインおよびRuvCドメインの2つのヌクレアーゼドメインはそれぞれ,標的2本鎖DNAの相補鎖および非相補鎖を切断するのに適した位置に存在すること,4)Cas9のC末端領域はDNA切断に必須の塩基配列であるPAMの認識にかかわること,が明らかになった.今回,明らかになったCas9-ガイド鎖RNA-標的DNA三者複合体の構造は,Cas9によるRNA依存性のDNA切断機構の構造基盤をあたえるのみならず,新たなゲノム編集ツールの合理的な設計の基盤ともなることが期待される.
はじめに
CRISPR-Cas系(CRISPR:clustered regularly interspaced short palindromic repeat,Cas:CRISPR-associated protein)は原核生物のもつ獲得免疫機構であり,ファージやプラスミドなど外来DNAに対する防御を担う1).細胞に侵入した外来DNAはCasのはたらきによりゲノムに存在するCRISPR領域に取り込まれる.別のCasはCRISPR領域から転写されたガイド鎖RNAと複合体を形成し,ガイド鎖RNAと相補的な外来DNAを認識して切断する.CRISPR-Cas系には複数のCasが関与し,その違いなどにもとづきI型~III型に大別される.II型CRISPR-Cas系はさらにIIA型~IIC型に分けられる.
Cas9はII型CRISPR-Cas系にかかわる100~150 kDaのRNA依存性DNAエンドヌクレアーゼであり,RuvCドメインおよびHNHドメインの2つのヌクレアーゼドメインをもつ2,3)(図1a).それ以外の領域は既知のタンパク質との配列相同性をもたず,その機能はわかっていなかった.Cas9はcrRNA(CRISPR RNA)およびtracrRNA(trans-activating crRNA)とよばれる2種類のRNAと複合体を形成し,crRNAのもつ20塩基のガイド配列と相補的な標的となる2本鎖DNAを切断する.標的2本鎖DNAのうち,ガイド配列と相補的なDNA鎖はHNHドメインにより切断され,もう一方のDNA鎖はRuvCドメインにより切断される.標的2本鎖DNAの切断には,ガイド配列との相補性にくわえ,標的となる配列の近傍に存在するPAM(protospacer adjacent motif)とよばれる特定の塩基配列が必須である.crRNAとtracrRNAとを人工的なリンカーにより連結したsgRNA(single-guide RNA)も,ガイド鎖RNAとして機能する2)(図1b).
Cas9およびsgRNAを細胞に共発現させることによりゲノムDNAを配列特異的に切断できることから,Cas9はさまざまな生物種において利用可能なゲノム編集ツールとして注目されている4,5).Cas9を応用したゲノム編集技術により,これまで困難だった複数の遺伝子の同時ノックアウトや,霊長類におけるノックアウト動物の作製が迅速かつ簡便に可能であることが報告されている6,7).さらに,Cas9の応用はゲノム編集にとどまることなく,不活性型Cas9を用いた転写制御技術やライブイメージング技術など新規の技術が続々と報告されている.このように,Cas9を応用した新規の技術が生命科学に革命を起こしつつある一方,Cas9の作動する分子機構は不明であった.
1.Cas9-sgRNA-標的DNA三者複合体
Streptococcus pyogenesに由来するCas9(残基1~残基1368)とsgRNA(98塩基),および,標的DNA(23塩基)との三者複合体の結晶構造を2.5Å分解能で決定した(PDB ID:4OO8,図1c).Cas9の性状を改善するため,Cys80およびCys574の2つのシステイン残基をそれぞれロイシン残基およびグルタミン酸残基に置換した.DNA切断実験により,これらの変異は酵素活性に影響しないことが確認された.さらに,標的DNAの切断を防ぐため,RuvCドメインのAsp10およびHNHドメインのHis840の2つの活性残基をアラニン残基に置換した.
結晶構造から,Cas9はRECローブ(REC:recognition)およびNUCローブ(NUC:nuclease)と名づけた2つのローブからなることが明らかになった(図1 a, c).RECローブは,アルギニン残基に富むαヘリックス(ブリッジヘリックスと名づけた),REC1ドメイン,REC2ドメインから構成されていた(最近,報告されたCas9の単体の構造8) との比較から,REC1ドメインはREC1aおよびREC1bの2つのサブドメインに分けられることが明らかになった).NUCローブはRuvCドメイン,HNHドメイン,PIドメイン(PI:PAM-interacting)から構成されていた.1次構造において分散して存在する3つのRuvCモチーフ(RuvC I~RuvC III)は集合し,立体構造においてひとつのRuvCドメインを形成していた.RuvCドメインとPIドメインとは密に相互作用していた一方,HNHドメインはRuvC IIとRuvC IIIとのあいだに存在し,NUCローブとはほとんど相互作用していなかった.RECローブとNUCローブはブリッジヘリックス,および,REC1とRuvC IIとのあいだの天然変性リンカーにより連結していた.sgRNAと標的DNAはヘテロ2本鎖を形成し,2つのローブのあいだに収容されていた.
2.sgRNA:DNAヘテロ2本鎖の構造
sgRNAはcrRNA配列とtracrRNA配列,および,それらを連結するテトラループから構成される(図1b).crRNA配列はガイド領域(20塩基)とリピート領域(12塩基)からなり,tracrRNA配列はアンチリピート領域(14塩基)と3'側テイル領域からなる.結晶構造から,sgRNAは標的DNAと結合し,ガイド領域:標的DNAヘテロ2本鎖,リピート領域:アンチリピート領域2本鎖,3つのステムループ(ステムループ1~ステムループ3),および,1本鎖のリンカー領域から構成されるT字型の構造をとることが明らかになった(図1c).
sgRNAのガイド領域と標的DNAは20対のWatson-Crick塩基対を介してガイド領域:標的DNAヘテロ2本鎖を形成し,sgRNAのリピート領域とアンチリピート領域は9対のWatson-Crick塩基対を介してリピート領域:アンチリピート領域2本鎖を形成していた(図1c).リピート領域:アンチリピート領域2本鎖にある6塩基,G27,A28,A41,A42,A43,U44は塩基対を形成せず,A28とU44はフリップアウトして特徴的な構造を形成していた.塩基配列から予想されたように,3'側テイル領域は2つのステムループ(ステムループ2およびステムループ3)を形成していた.さらに,3'側テイル領域は塩基配列からは予想されていなかったステムループ1を形成していることが明らかになった.
3.RECローブの構造
RECローブはおもにαヘリックスから構成されており,既知のタンパク質とは構造類似性をもたない新規な構造をとっていた(図1c).REC1ドメインはリピート領域:アンチリピート領域2本鎖を認識していた.変異体の解析の結果,REC1ドメインはCas9の機能に必須であることが示された.一方,REC2ドメインはsgRNAとは相互作用しておらず,変異体の解析の結果からもCas9の機能に必須ではないことが示された.この結果は,Cas9のオルソログの多くはREC2ドメインをもたないことと一致していた.REC2ドメインの役割は現時点では不明である.
4.NUCローブの構造
RuvCドメインおよびHNHドメインの活性部位はそれぞれ,RNase Hフォールドおよびββα-メタルフォールドをとっていた(図1c).したがって,RuvCドメインおよびHNHドメインは既知のヌクレアーゼと同様の反応機構により,標的2本鎖DNAを形成する非相補鎖および相補鎖をそれぞれを切断することが示唆された.一方,RuvCドメインおよびHNHドメインには既知のヌクレアーゼとは異なる構造的な特徴も存在した.たとえば,RuvCヌクレアーゼは二量体を形成することによりHolliday構造を認識するのに対し,Cas9のもつRuvCドメインは二量体を形成せず,ほかのドメインとの相互作用にかかわる領域をもっていた.
RECローブにくわえ,PIドメインも新規な構造をとっていた(図1c).標的DNAおよびRuvCドメインの位置から,PIドメインはPAMの認識にかかわることが示唆された.S. pyogenesに由来するCas9とStreptococcus thermophilusに由来するCRISPR-3 Cas9は,59%の配列同一性をもちガイド鎖RNAの交換が可能である9).一方,2本鎖DNAの切断に必要なPAMの配列は異なっており,S. pyogenesのCas9は5'-NGG-3'という配列を必要とするのに対し,S. thermophilusのCas9は5'-NGGNG-3'という配列を必要とする.PIドメインがPAMの認識にかかわるのかどうかを調べるため,PIドメインを交換したキメラ変異体として,S. pyogenesのCas9のもつPIドメインをS. thermophilusのCas9のもつPIドメインに置換したCas9変異体,および,S. thermophilusのCas9のもつPIドメインをS. pyogenesのCas9のもつPIドメインに置換したCas9変異体を作製し,5'-NGG-3'あるいは5'-NGGNG-3'という配列のPAMをもつ標的2本鎖DNAに対する切断活性を測定した.その結果,S. thermophilusのCas9のもつPIドメインをS. pyogenesのCas9のものと置換したCas9変異体は,S. thermophilusのCas9とは異なり,5'-NGG-3'という配列のPAMをもつ標的2本鎖DNAを切断した.したがって,S. pyogenesのCas9のもつPIドメインは5'-NGG-3'という配列のPAMの認識に必要であり,S. thermophilusのもつCas9のPAMに対する特異性を変えるのに十分であることが示唆された.PIドメインを欠失したS. pyogenesのCas9はDNA切断活性を示さなかったことから,PIドメインはCas9の機能に必須であることが示された.これらの結果から,PIドメインはPAMの認識にかかわることが明らかになった.
5.sgRNA:DNAヘテロ2本鎖の認識
HNHドメインは相補鎖DNAの3位と4位とのあいだのホスホジエステル結合を切断し,DNA切断活性にはガイド領域:標的DNAヘテロ2本鎖のうち“シード”領域とよばれるPAMの近傍の10~12塩基対が重要である2,10).結晶構造において,sgRNAのシード領域のリン酸骨格は,ブリッジヘリックスにある複数のアルギニン残基により認識されていた(図1c).変異体の解析の結果,これらのアルギニン残基はCas9の機能に重要であることが確認された.したがって,Cas9-sgRNA複合体において,sgRNAはシード領域の塩基を外側にむけてCas9と結合し,相補鎖DNAとWatson-Crick塩基対を形成することにより標的DNAを認識すると考えられた.ブリッジヘリックスのアルギニン残基は高度に保存されていることから,ブリッジヘリックスはCas9のオルソログに共通する構造的な特徴であることが示唆された.
ガイド領域:標的DNAヘテロ2本鎖はおもにRECローブにより配列非依存的に認識され,末端の塩基対はRuvCドメインにより認識されていた(図1c).これらの構造的な特徴は,Cas9はガイド配列の異なるsgRNAと結合すること2),20塩基以上のガイド配列は細胞内で分解され配列特異性の向上には貢献しないこと11),と一致していた.
ガイド領域:標的DNAヘテロ2本鎖とは対照的に,リピート領域:アンチリピート領域2本鎖はおもにRECローブにより配列依存的に認識されていた(図1c).これらの構造的な特徴は,IIA型~IIC型のCRISPR-Cas系において,リピート領域:アンチリピート領域2本鎖の塩基配列,および,RECローブのアミノ酸配列は多様であり,Cas9は近縁のsgRNAのみをガイド鎖RNAとして利用できるという報告9) と一致していた.
ステムループ1はRECローブとNUCローブの両方と相互作用していた一方,ステムループ2およびステムループ3はNUCローブのみと相互作用しCas9との結合を補強していた(図1c).48塩基のtracrRNA配列をもつsgRNAはin vitroにおけるDNA切断活性に必要な最小領域である一方2),67塩基あるいは85塩基の長いtracrRNA配列をもつsgRNAはin vivoにおけるDNA切断活性に重要であることが報告されている10).結晶構造から,48塩基,67塩基,85塩基のtracrRNA配列をもつsgRNAはそれぞれ,ステムループ1,ステムループ1およびステムループ2,ステムループ1~ステムループ3を含むことが明らかになった.これらの結果から,ステムループ1は機能的なCas9-sgRNA複合体の形成に必須である一方,ステムループ2およびステムループ3は複合体を安定化しin vivoにおけるDNA切断活性を上昇させると考えられた.
6.Cas9のフレキシビリティ
Cas9-sgRNA-標的DNA三者複合体の結晶構造においてHNHドメインは相補鎖DNAの切断部位と離れた位置に存在していた(図1c).結晶の単位格子にはコンフォメーションの異なる2つの三者複合体Mol AおよびMol Bが含まれており,それらの比較から,HNHドメインによる相補鎖DNAの切断機構を推測することができた.Mol AではHNHドメインはRuvC IIおよびRuvC IIIとそれぞれ天然変性リンカーおよびαヘリックスにより連結していた.一方,Mol BではHNHドメインの電子密度は観察されず,HNHドメインは特定の位置には固定されていないことが示唆された.さらに,Mol AにおいてHNHドメインとRuvC IIIとを連結しているαヘリックスは構造変化を起こし相補鎖DNAの方向をむいていた.これらの比較から,HNHドメインとRuvCドメインとを連結するαヘリックスの構造変化により,HNHドメインは相補鎖DNAに近づき標的DNAを切断することが示唆された(図2).最近の研究により,Cas9はPAMと結合すると,不活性型から活性型へと構造変化することが示唆された12).この結果は,今回,構造決定したCas9-sgRNA-標的DNA三者複合体はHNHドメインが切断部位と離れた不活性型の構造をとっていることと一致しており,PAMとの結合によりHNHドメインは切断部位に近づく可能性が考えられた.
おわりに
Cas9がRNA依存性DNAエンドヌクレアーゼであるという2012年6月の報告2) を発端に,“CRISPR Craze”とも形容される激しい研究競争がはじまった.筆者らは,その半年のち,2013年1月に構造解析に着手した.この論文を発表した米国の研究グループは結晶構造解析のトップラボのひとつであり,すでに構造解析に着手しているのは明らかだった.しかし,筆者らにとり,競争のあることはCas9の構造解析に挑戦しない理由にはならなかった.Cas9があまりに魅力的なタンパク質だったからである.競争は激しいが勝算はゼロではないと思った.構造解析のターゲットはCas9の単体,Cas9-ガイド鎖RNA複合体,Cas9-ガイド鎖RNA-標的DNA三者複合体などいくつか存在し,さらに,核酸との複合体の結晶化には,用いる核酸の長さや塩基配列,複合体の調製法など,多くのパラメーターが影響すると予想された.したがって,ゴールはいくつもあり,さらに,それぞれのゴールまでの道筋もひとつではないため,競争は単純なスピード勝負ではないと考えた.
構造解析に着手してから1カ月,S. pyogenesに由来するCas9の単体の結晶を得ることに成功した.この結晶は構造決定には不十分だったが,半年あれば構造は決定できるだろうと感じた.しかしこの結果は,S. pyogenesに由来するCas9は結晶化に適していることにくわえ,競争相手も同様の結晶を得ているであろうことを示唆していた.そこで,Cas9単体の構造決定はあきらめ核酸との複合体に集中することにした.sgRNA:DNAヘテロ2本鎖の長さや塩基配列が核酸との複合体の結晶化に大きく影響したため,さまざまなヘテロ2本鎖とCas9との複合体を調製し,複合体ごとに結晶化スクリーニングを行った.さらに,巨大かつ新規なCas9-sgRNA-標的DNA三者複合体の構造決定は簡単ではなかった.
筆者らの論文がオンライン出版されるちょうど1週間前,競争相手と予想していた米国の研究グループがCas9の結晶構造を報告した8)(PDB ID:4CMP).予想どおり,彼らもS. pyogenesに由来するCas9を結晶化しており,結晶化の条件も筆者らが最初に見い出したものと類似していた.彼らは,Cas9の単体の構造とクライオ電子顕微鏡により決定した構造とを組み合わせることにより,Cas9単体は閉じた不活性型の構造をとること,および,Cas9はガイド鎖RNAとの結合により開いた活性型へと構造変化することを示した.このCas9単体の構造と筆者らの構造決定したCas9-sgRNA-標的DNA三者複合体の構造との比較から,この2つの構造は大きく異なることがわかった(図1 c, d).ガイド鎖RNAの結合による構造変化は想定していたが,これほど大きな構造変化は想定外だった.Cas9単体の構造ではブリッジヘリックスが折れ曲がることによりRECローブとNUCローブとが近接し密に相互作用していた(図1d).その結果,ガイド領域:標的DNAヘテロ2本鎖を収容する溝は存在していなかった.さらに,Cas9-sgRNA-標的DNA三者複合体の構造においてガイド領域:標的DNAヘテロ2本鎖を認識していたREC1bドメインは,Cas9単体の構造では大きく移動していた(図1d).Cas9単体の構造解析にさきをこされることは想定していたが,その時期は想定よりも遅かった.その理由として,Cas9単体は核酸との複合体よりもフレキシブルだったため,その構造決定には複合体とは別のむずかしさがあったこと,また,Cas9単体の構造は予想外にも閉じた構造をとっていたため,構造機能相関を説明するにはクライオ電子顕微鏡により決定した構造などの追加のデータが必要だったこと,などが考えられた.Cas9単体の構造解析をあきらめ,核酸との複合体に集中したのは正しい選択だった.もし,Cas9単体の構造解析を継続していたら完敗だった.
この研究は米国Massachusetts Institute of TechnologyのFeng Zhang博士のグループとの共同研究として行った.CRISPR研究の若きスーパースターFeng Zhangとともにしたこの1年間は,これまでの人生でもっともエキサイティングな1年だった.
文 献
- Marraffini, L. A. & Sontheimer, E. J.: CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat. Rev. Genet., 11, 181-190 (2010)[PubMed]
- Jinek, M., Chylinski, K., Fonfara, I. et al.: A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 337, 816-821 (2012)[PubMed]
- Gasiunas, G., Barrangou, R., Horvath, P. et al.: Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc. Natl. Acad. Sci. USA, 109, E2579-E2586 (2012)[PubMed]
- Cong, L., Ran, F. A., Cox, D. et al.: Multiplex genome engineering using CRISPR/Cas systems. Science, 339, 819-823 (2013)[PubMed]
- Mali, P., Yang, L., Esvelt, K. M. et al.: RNA-guided human genome engineering via Cas9. Science, 339, 823-826 (2013)[PubMed]
- Wang, H., Yang, H., Shivalila, C. S. et al.: One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell, 153, 910-918 (2013)[PubMed]
- Niu, Y., Shen, B., Cui, Y. et al.: Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos. Cell, 156, 836-843 (2014)[PubMed]
- Jinek, M., Jiang, F., Taylor, D. W. et al.: Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. Science, 343, 1247997 (2014)[PubMed]
- Fonfara, I., Le Rhun, A., Chylinski, K. et al.: Phylogeny of Cas9 determines functional exchangeability of dual-RNA and Cas9 among orthologous type II CRISPR-Cas systems. Nucleic Acids Res., 42, 2577-2590 (2014)[PubMed]
- Hsu, P. D., Scott, D. A., Weinstein, J. A. et al.: DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol., 31, 827-832 (2013)[PubMed]
- Ran, F. A., Hsu, P. D., Lin, C. Y. et al.: Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 154, 1380-1389 (2013)[PubMed]
- Sternberg, S. H., Redding, S., Jinek, M. et al.: DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature, 507, 62-67 (2014)[PubMed]
著者プロフィール
略歴:2007年 東京大学大学院農学系研究科 修了,2013年より東京大学大学院理学系研究科 助教.2013年より科学技術振興機構 さきがけ研究者 兼任.
研究テーマ:RNAサイレンシング.
抱負:たった一度の人生,やりたいことを貫きたい.国際競争の激化のため研究にスピードが求められる今日だが,妥協せずにていねいな仕事をしていきたい.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究テーマ:遺伝暗号の翻訳,膜輸送,自然炎症.
抱負:細胞の生理機構を原子分解能で解明したい.
研究室URL:http://www.nurekilab.net/
© 2014 西増弘志・濡木 理 Licensed under CC 表示 2.1 日本