遺伝情報の拡張による高親和性のDNAアプタマーの創出
木本路子・平尾一郎
(理化学研究所ライフサイエンス技術基盤研究センター 合成分子生物学研究チーム)
email:平尾一郎
DOI: 10.7875/first.author.2013.055
Generation of high-affinity DNA aptamers using an expanded genetic alphabet.
Michiko Kimoto, Rie Yamashige, Ken-ichiro Matsunaga, Shigeyuki Yokoyama, Ichiro Hirao
Nature Biotechnology, 31, 453-457 (2013)
DNAアプタマーはタンパク質などの標的分子に特異的に結合するDNA断片であり,SELEX法という人工進化の手法により作製することが可能で,医薬への利用が期待されている.しかし,天然型あるいはその修飾ヌクレオチドにより作製される従来型のDNAアプタマーは,標的となるタンパク質との結合において親和性や特異性の低い場合が多い.今回,筆者らは,4種類の天然型塩基A,G,C,Tにくわえ,人工的に作り出した疎水性の塩基Dsを組み込んだDNAアプタマーの作製に成功した.天然型塩基からなるランダムな配列の1~3箇所にDs塩基を組み込んだDNA断片からなるライブラリーを作製し,これを用いたSELEX法により標的とした2種類のヒトのタンパク質,VEGF165およびインターフェロンγに対するDNAアプタマーを得た.従来型の天然型塩基のみからなるDNAアプタマーと比較して,それぞれのDNAアプタマーの標的に対する結合能は100倍以上に向上した.この結果から,人工塩基による遺伝情報の拡張は高機能な核酸を創出するための強力なツールになりうることがはじめて示された.今後,人工塩基からなるDNAアプタマーの医療への応用が期待される.
核酸は複製や遺伝子発現における遺伝情報分子としての機能にくわえ,リガンドや触媒などとしての機能もあわせもつ.この核酸のユニークな性質を利用した進化工学の手法により,用途にあわせた機能性核酸の創出が可能になっている.SELEX法(systematic evolution of ligands by exponential enrichment,in vitroセレクション法ともいう)による核酸アプタマーの作製もそのひとつである1,2).SELEX法では,ランダムな配列からなる核酸断片のライブラリーから標的となる分子に結合する断片を選別し,得られた断片をPCRなどにより増幅して再びライブラリーとする.この選別と増幅のラウンドを選択圧を高めながらくり返すことにより,最終的に,高い親和性で特異的に標的分子に結合する核酸断片(核酸アプタマー)が得られる.タンパク質からなる抗体とは異なり,核酸アプタマーは試験管の中で創出することができるので,毒性の高い分子を標的にすることもできる.また,得られた核酸アプタマーの塩基配列を特定することができれば,核酸合成により安定した品質をもって大量生産が可能である.
核酸アプタマーの問題点のひとつは,標的分子に対する結合能がタンパク質からなる抗体と比較して低い場合が多いことである.これは,20種類のアミノ酸からなるタンパク質に対し,核酸は4種類の塩基のみを側鎖とするヌクレオチドからなるためである.さらに,核酸は親水性が高いため疎水性の高い標的分子との親和性がとくに低くなる.これらの問題に対処するため,ヌクレオチドの塩基,糖,リン酸のそれぞれの部位を修飾したアプタマーの作製法が検討されるようになった.そして,既存の塩基部分を疎水性の置換基で修飾することにより,DNAアプタマー作製の成功率が高められるようになった.しかし,これらの改良法を用いても,核酸アプタマーの親和性の著しい向上にまではいたっていない.
以前より,核酸の機能の向上を図るための本質的な方法として,塩基の種類を人工的に増やす方法が注目されてきた.塩基の種類が増えれば,遺伝情報の拡張と同時に,リガンドや触媒としての核酸の高機能化も達成できるかもしれない.そのためには,新たな塩基を組み込んだDNAが複製により増幅され,転写によりRNAにコピーされなければならず,第3の塩基対として生物のもつシステムにおいて選択的に機能する人工の塩基対を創出しなければならない.
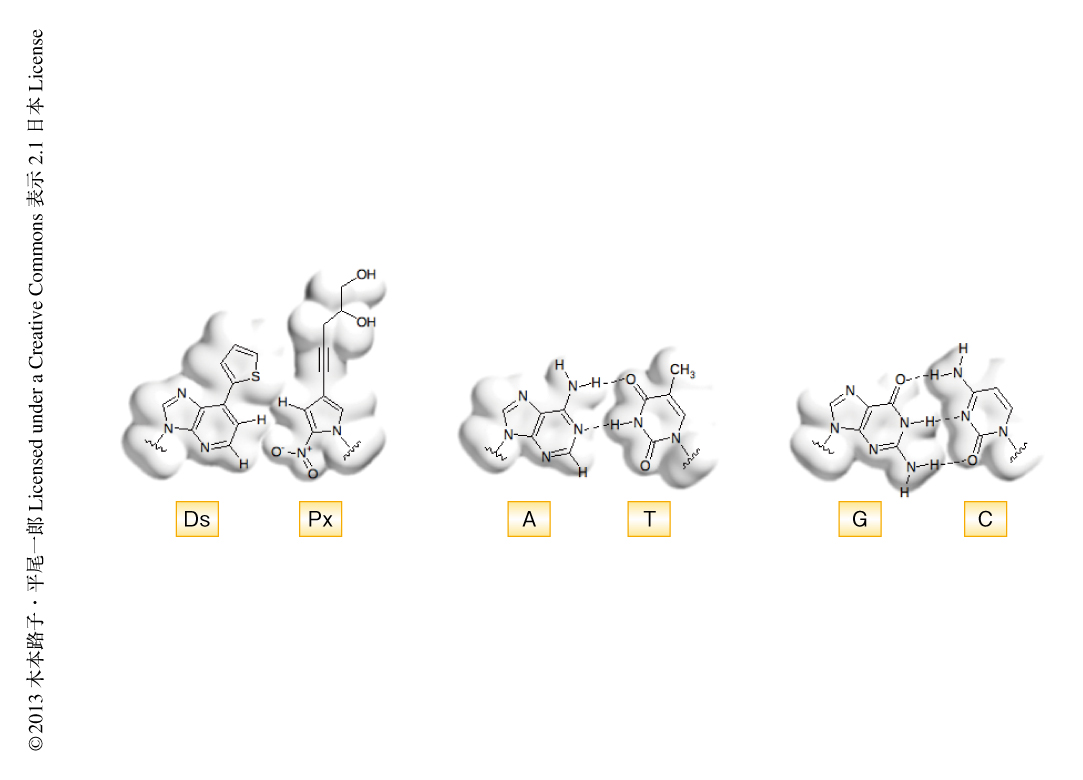
人工塩基対のアイデアは,すでに1962年に出版された総説において記されている3).1980年代の後半からは,それらの人工塩基対およびその類似塩基対が合成された4).残念ながら,これらの人工塩基対は複製や転写において実用化できるレベルにはいたらなかった.そののち,1995年以降,複製,転写,翻訳において機能する人工塩基対の開発がいくつかの研究チームにより競合的に進められている.筆者らは,2002年に転写と翻訳において機能する人工塩基対s-yを5),2006年には複製(PCR)において機能する人工塩基対Ds-Paを6),開発してきた.そして,2012年までに,PCRにおいて非常に高い選択性を示す人工塩基対Ds-Pxの開発に成功した7,8)(図1).
7-(2-チエニル)イミダゾ[4,5-b]ピリジン(Ds)と2-ニトロ-4-プロピニルピロール(Px)からなる人工塩基対Ds-Pxは,天然型の塩基との対合をさけるため,それぞれの人工塩基から塩基対の面にある水素結合性をもつ置換基や原子を取り除いた疎水性の塩基対である7).プリン類似体にチエニル基を結合したDs塩基はAやGよりも形状が大きく,逆に,Px塩基の母体は五員環であるピロールを用いているので六員環のTやCよりも小さい.そして,Dsの形状はジグソーパズルのようにPxとのみ適合するようになっている.PxにはDNAポリメラーゼとの芳香属性の相互作用を強めるためプロピニル基を付加している.さらに,PxとAとの塩基対の形成を抑えるため2位にニトロ基を導入している.このニトロ基の酸素原子とAの1位の窒素原子とは静電的に反発しあう.こうして,100サイクルのPCRにも耐えうる第3の塩基対Ds-Pxができあがった8).
そののち,米国の2つのチームもPCRにおいて機能する人工塩基対の開発に成功し,つぎの課題は,人工塩基を組み込んだDNAの機能が本当に向上するかどうかになった9).とくに,人工塩基対を用いたSELEX法による核酸アプタマーや核酸触媒の開発は,これらのチームのあいだで競われていた.
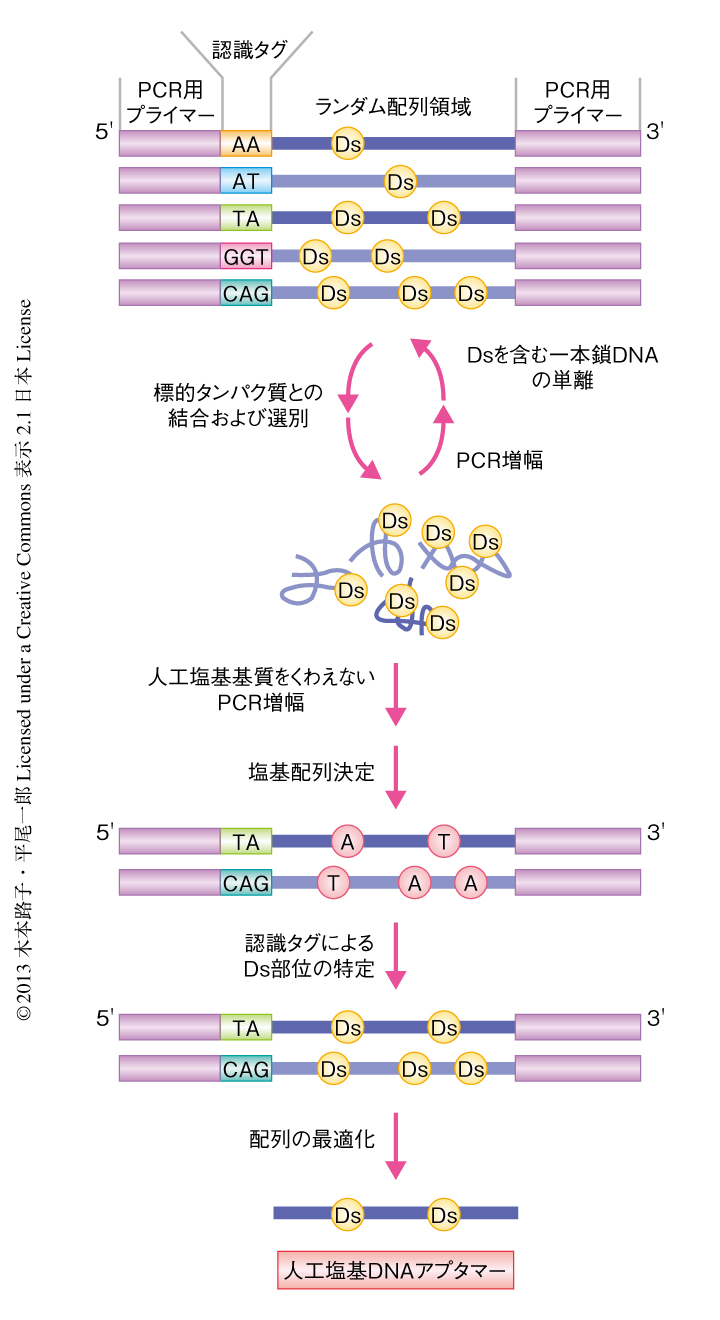
人工塩基を用いた核酸アプタマーに対するSELEX法において,DsをDNAライブラリーに導入した.DNAアプタマーはRNAアプタマーより大量合成が容易なため実用化には有利である.しかし,DNAはRNAほどその高次構造が柔軟ではなく,A-TとG-Cの塩基対形成による二本鎖直鎖構造をとりやすいのでアプタマーには適していない.そこで,Dsのみを第5の塩基として導入することにより,DNAライブラリーにおける高次構造の多様性を高めることにした.相補塩基であるPxを導入していないので,Dsは塩基対を形成できずDNAの高次構造は多様化される.さらに,Dsは疎水性が非常に高いので,標的となるタンパク質のもつ疎水性ポケットとの相互作用が期待できる.この5種類の塩基A,G,C,T,Dsからなる一本鎖DNA断片のライブラリーを用いて,標的タンパク質に結合するDNA断片の選別を行った.そして,選別されたライブラリーの増幅の際には基質としてDsとPxをくわえてPCRを行い二本鎖DNAとした.そののち,Dsを含む一本鎖DNAを単離してつぎのラウンドに進んだ(図2).
問題は,最終ラウンドののちライブラリーから得られたDNAアプタマーの塩基配列の決定法である.従来の方法では,最終ライブラリーをプラスミドに組み込み大腸菌に形質転換することにより,それぞれのコロニーからアプタマーの配列を決定する.あるいは最近では,次世代シークエンサーを用いてライブラリーから直接,塩基配列を決定する方法もある.しかし,どちらの方法も人工塩基を含むライブラリーには適用できない.現時点では,人工塩基を含むプラスミドDNAの大腸菌におけるクローニング法や,次世代シークエンサーによる第5の塩基を含む塩基配列の決定法は確立していない.
そこで,天然型塩基からなる43塩基長のランダムな配列において特定の部位に1つから3つのDsを導入した22種類のサブライブラリーを作製した.さらに,それぞれのサブライブラリーにはDsの位置を特定するため,2つから3つの天然型塩基からなる個別のタグ配列をくわえた.人工塩基の位置の異なる22種類のサブライブラリーを混合し,これをライブラリーとしてSELEX法に用いた.最終ラウンドののちのライブラリーは基質としてDsとPxを除いてPCRにより増幅し,人工塩基対を天然型塩基対に変異させて,そのあと次世代シークエンサーを用いて塩基配列を決定した.それぞれの配列のタグ配列から人工塩基の位置を特定し,Dsを含むDNAアプタマーの最終配列を決定した(図2).ただし,Ds-Px塩基対は選択性が高いため天然型の塩基対に変異しにくく,基質として人工塩基をくわえないとPCRが人工塩基対の位置で止まってしまう.そこで,以前に開発した,Pxよりも精度の低いPa塩基をくわえることにより人工塩基対から天然型塩基対への変異を容易にした.
人工塩基対を用いたSELEX法を確立するため,モデル標的タンパク質としてVEGF165とインターフェロンγを選んだ.VEGF165に対するアプタマーは,すでに,その修飾RNAアプタマーが加齢黄斑変性症の治療薬として用いられている10).その結合定数は49~130 pMである11).また,天然型塩基のみからなるDNAアプタマーも得られていて,その結合定数は370 pMである12).インターフェロンγはサイトカインの一種であり,ウイルス感染などのバイオマーカーとして有用である.しかし,これまでに得られたDNAアプタマーの結合定数は3~28 nMである13).
それぞれの標的タンパク質に対し7ラウンドの選別を行い,Dsを含むDNAアプタマーの配列を得た.さらに,それぞれのDNAアプタマーの配列に変異を導入してふたたびSELEX法を行い,DNAアプタマーの最適化を行った.その結果,VEGF165に結合する2つのDsを含む全長47塩基のDNAアプタマーが得られ,その結合定数は0.65 pMであった.また,インターフェロンγに結合する全長49塩基のDNAアプタマーには3つのDsが含まれ,その結合定数は38.4 pMであった.天然型塩基のみからなる従来のDNAアプタマーと比較して,標的に対する親和性は大幅に向上していた.それぞれのアプタマーにおいてDsをAに置換したところ親和性は100倍ほど低下し,人工塩基に依存したDNAアプタマーであることが確認された.また,それぞれのDNAアプタマーについて,ほか数種類のタンパク質に対する親和性も調べたが,それぞれの標的のみに高い選択性で結合し,従来のアプタマーに比べ選択性も向上していることがわかった.
人工塩基対の研究のおもな目的のひとつは,塩基の種類を増やすことにより遺伝情報を拡張し,タンパク質に非天然型アミノ酸を導入することである.しかし,核酸の多様な機能がわかってくると,人工塩基を核酸に導入することにより,リガンドや触媒などとしての機能の向上も期待されるようになってきた.今回の人工塩基対を導入したDNAアプタマーの研究から,天然型塩基とは性質の異なるわずか1種類の人工塩基を2~3箇所に組み込むことにより,DNAの高機能化が可能であることがはじめて実証された.今後,この手法を用いて得られる人工塩基DNAアプタマーの医療への応用が期待される.
Ds-Px塩基対をSELEX法に応用できたことは,人工塩基対が第3の塩基対として実用化レベルに達しつつあることを示した.このSELEX法では,それぞれのラウンドで20サイクル以上のPCRを行い,合計で7ラウンドの選別を行った.したがって,人工塩基は総計140サイクル以上のPCR増幅に生き残ったことになった.とくにこの手法では,あらかじめサブライブラリーにおいて人工塩基の位置を固定しているので,SELEX法において人工塩基が天然型塩基に置き換わったり,あるいは,天然型塩基が人工塩基に置き換わったりしてしまっては成り立たなくなってしまう.したがって,Ds-Px塩基対のPCRにおける選択性は天然型塩基対に匹敵するレベルに達していると考えられた.
今後,人工塩基対システムは次世代の遺伝子操作の基盤をなす技術として,SELEX法のみならず,幅広い分野に利用されるものと期待している.
略歴:2002年 東京大学大学院理学系研究科博士課程 修了,2013年より理化学研究所ライフサイエンス技術基盤研究センター 上級研究員.
平尾一郎(Ichiro Hirao)
略歴:1983年 東京工業大学理工学研究科後期博士課程 修了,2007年よりタグシクス・バイオ株式会社 代表取締役社長,2013年より理化学研究所ライフサイエンス技術基盤研究センター チームリーダー.
研究室URL:http://protein.gsc.riken.jp/hirao/index.html
© 2013 木本路子・平尾一郎 Licensed under CC 表示 2.1 日本
(理化学研究所ライフサイエンス技術基盤研究センター 合成分子生物学研究チーム)
email:平尾一郎
DOI: 10.7875/first.author.2013.055
Generation of high-affinity DNA aptamers using an expanded genetic alphabet.
Michiko Kimoto, Rie Yamashige, Ken-ichiro Matsunaga, Shigeyuki Yokoyama, Ichiro Hirao
Nature Biotechnology, 31, 453-457 (2013)
要 約
DNAアプタマーはタンパク質などの標的分子に特異的に結合するDNA断片であり,SELEX法という人工進化の手法により作製することが可能で,医薬への利用が期待されている.しかし,天然型あるいはその修飾ヌクレオチドにより作製される従来型のDNAアプタマーは,標的となるタンパク質との結合において親和性や特異性の低い場合が多い.今回,筆者らは,4種類の天然型塩基A,G,C,Tにくわえ,人工的に作り出した疎水性の塩基Dsを組み込んだDNAアプタマーの作製に成功した.天然型塩基からなるランダムな配列の1~3箇所にDs塩基を組み込んだDNA断片からなるライブラリーを作製し,これを用いたSELEX法により標的とした2種類のヒトのタンパク質,VEGF165およびインターフェロンγに対するDNAアプタマーを得た.従来型の天然型塩基のみからなるDNAアプタマーと比較して,それぞれのDNAアプタマーの標的に対する結合能は100倍以上に向上した.この結果から,人工塩基による遺伝情報の拡張は高機能な核酸を創出するための強力なツールになりうることがはじめて示された.今後,人工塩基からなるDNAアプタマーの医療への応用が期待される.
はじめに
核酸は複製や遺伝子発現における遺伝情報分子としての機能にくわえ,リガンドや触媒などとしての機能もあわせもつ.この核酸のユニークな性質を利用した進化工学の手法により,用途にあわせた機能性核酸の創出が可能になっている.SELEX法(systematic evolution of ligands by exponential enrichment,in vitroセレクション法ともいう)による核酸アプタマーの作製もそのひとつである1,2).SELEX法では,ランダムな配列からなる核酸断片のライブラリーから標的となる分子に結合する断片を選別し,得られた断片をPCRなどにより増幅して再びライブラリーとする.この選別と増幅のラウンドを選択圧を高めながらくり返すことにより,最終的に,高い親和性で特異的に標的分子に結合する核酸断片(核酸アプタマー)が得られる.タンパク質からなる抗体とは異なり,核酸アプタマーは試験管の中で創出することができるので,毒性の高い分子を標的にすることもできる.また,得られた核酸アプタマーの塩基配列を特定することができれば,核酸合成により安定した品質をもって大量生産が可能である.
核酸アプタマーの問題点のひとつは,標的分子に対する結合能がタンパク質からなる抗体と比較して低い場合が多いことである.これは,20種類のアミノ酸からなるタンパク質に対し,核酸は4種類の塩基のみを側鎖とするヌクレオチドからなるためである.さらに,核酸は親水性が高いため疎水性の高い標的分子との親和性がとくに低くなる.これらの問題に対処するため,ヌクレオチドの塩基,糖,リン酸のそれぞれの部位を修飾したアプタマーの作製法が検討されるようになった.そして,既存の塩基部分を疎水性の置換基で修飾することにより,DNAアプタマー作製の成功率が高められるようになった.しかし,これらの改良法を用いても,核酸アプタマーの親和性の著しい向上にまではいたっていない.
以前より,核酸の機能の向上を図るための本質的な方法として,塩基の種類を人工的に増やす方法が注目されてきた.塩基の種類が増えれば,遺伝情報の拡張と同時に,リガンドや触媒としての核酸の高機能化も達成できるかもしれない.そのためには,新たな塩基を組み込んだDNAが複製により増幅され,転写によりRNAにコピーされなければならず,第3の塩基対として生物のもつシステムにおいて選択的に機能する人工の塩基対を創出しなければならない.
1.人工塩基対の開発
人工塩基対のアイデアは,すでに1962年に出版された総説において記されている3).1980年代の後半からは,それらの人工塩基対およびその類似塩基対が合成された4).残念ながら,これらの人工塩基対は複製や転写において実用化できるレベルにはいたらなかった.そののち,1995年以降,複製,転写,翻訳において機能する人工塩基対の開発がいくつかの研究チームにより競合的に進められている.筆者らは,2002年に転写と翻訳において機能する人工塩基対s-yを5),2006年には複製(PCR)において機能する人工塩基対Ds-Paを6),開発してきた.そして,2012年までに,PCRにおいて非常に高い選択性を示す人工塩基対Ds-Pxの開発に成功した7,8)(図1).
7-(2-チエニル)イミダゾ[4,5-b]ピリジン(Ds)と2-ニトロ-4-プロピニルピロール(Px)からなる人工塩基対Ds-Pxは,天然型の塩基との対合をさけるため,それぞれの人工塩基から塩基対の面にある水素結合性をもつ置換基や原子を取り除いた疎水性の塩基対である7).プリン類似体にチエニル基を結合したDs塩基はAやGよりも形状が大きく,逆に,Px塩基の母体は五員環であるピロールを用いているので六員環のTやCよりも小さい.そして,Dsの形状はジグソーパズルのようにPxとのみ適合するようになっている.PxにはDNAポリメラーゼとの芳香属性の相互作用を強めるためプロピニル基を付加している.さらに,PxとAとの塩基対の形成を抑えるため2位にニトロ基を導入している.このニトロ基の酸素原子とAの1位の窒素原子とは静電的に反発しあう.こうして,100サイクルのPCRにも耐えうる第3の塩基対Ds-Pxができあがった8).
そののち,米国の2つのチームもPCRにおいて機能する人工塩基対の開発に成功し,つぎの課題は,人工塩基を組み込んだDNAの機能が本当に向上するかどうかになった9).とくに,人工塩基対を用いたSELEX法による核酸アプタマーや核酸触媒の開発は,これらのチームのあいだで競われていた.
2.人工塩基を用いた核酸アプタマーに対するSELEX法
人工塩基を用いた核酸アプタマーに対するSELEX法において,DsをDNAライブラリーに導入した.DNAアプタマーはRNAアプタマーより大量合成が容易なため実用化には有利である.しかし,DNAはRNAほどその高次構造が柔軟ではなく,A-TとG-Cの塩基対形成による二本鎖直鎖構造をとりやすいのでアプタマーには適していない.そこで,Dsのみを第5の塩基として導入することにより,DNAライブラリーにおける高次構造の多様性を高めることにした.相補塩基であるPxを導入していないので,Dsは塩基対を形成できずDNAの高次構造は多様化される.さらに,Dsは疎水性が非常に高いので,標的となるタンパク質のもつ疎水性ポケットとの相互作用が期待できる.この5種類の塩基A,G,C,T,Dsからなる一本鎖DNA断片のライブラリーを用いて,標的タンパク質に結合するDNA断片の選別を行った.そして,選別されたライブラリーの増幅の際には基質としてDsとPxをくわえてPCRを行い二本鎖DNAとした.そののち,Dsを含む一本鎖DNAを単離してつぎのラウンドに進んだ(図2).
問題は,最終ラウンドののちライブラリーから得られたDNAアプタマーの塩基配列の決定法である.従来の方法では,最終ライブラリーをプラスミドに組み込み大腸菌に形質転換することにより,それぞれのコロニーからアプタマーの配列を決定する.あるいは最近では,次世代シークエンサーを用いてライブラリーから直接,塩基配列を決定する方法もある.しかし,どちらの方法も人工塩基を含むライブラリーには適用できない.現時点では,人工塩基を含むプラスミドDNAの大腸菌におけるクローニング法や,次世代シークエンサーによる第5の塩基を含む塩基配列の決定法は確立していない.
そこで,天然型塩基からなる43塩基長のランダムな配列において特定の部位に1つから3つのDsを導入した22種類のサブライブラリーを作製した.さらに,それぞれのサブライブラリーにはDsの位置を特定するため,2つから3つの天然型塩基からなる個別のタグ配列をくわえた.人工塩基の位置の異なる22種類のサブライブラリーを混合し,これをライブラリーとしてSELEX法に用いた.最終ラウンドののちのライブラリーは基質としてDsとPxを除いてPCRにより増幅し,人工塩基対を天然型塩基対に変異させて,そのあと次世代シークエンサーを用いて塩基配列を決定した.それぞれの配列のタグ配列から人工塩基の位置を特定し,Dsを含むDNAアプタマーの最終配列を決定した(図2).ただし,Ds-Px塩基対は選択性が高いため天然型の塩基対に変異しにくく,基質として人工塩基をくわえないとPCRが人工塩基対の位置で止まってしまう.そこで,以前に開発した,Pxよりも精度の低いPa塩基をくわえることにより人工塩基対から天然型塩基対への変異を容易にした.
3.人工塩基を用いたDNAアプタマーの高い親和性と特異性
人工塩基対を用いたSELEX法を確立するため,モデル標的タンパク質としてVEGF165とインターフェロンγを選んだ.VEGF165に対するアプタマーは,すでに,その修飾RNAアプタマーが加齢黄斑変性症の治療薬として用いられている10).その結合定数は49~130 pMである11).また,天然型塩基のみからなるDNAアプタマーも得られていて,その結合定数は370 pMである12).インターフェロンγはサイトカインの一種であり,ウイルス感染などのバイオマーカーとして有用である.しかし,これまでに得られたDNAアプタマーの結合定数は3~28 nMである13).
それぞれの標的タンパク質に対し7ラウンドの選別を行い,Dsを含むDNAアプタマーの配列を得た.さらに,それぞれのDNAアプタマーの配列に変異を導入してふたたびSELEX法を行い,DNAアプタマーの最適化を行った.その結果,VEGF165に結合する2つのDsを含む全長47塩基のDNAアプタマーが得られ,その結合定数は0.65 pMであった.また,インターフェロンγに結合する全長49塩基のDNAアプタマーには3つのDsが含まれ,その結合定数は38.4 pMであった.天然型塩基のみからなる従来のDNAアプタマーと比較して,標的に対する親和性は大幅に向上していた.それぞれのアプタマーにおいてDsをAに置換したところ親和性は100倍ほど低下し,人工塩基に依存したDNAアプタマーであることが確認された.また,それぞれのDNAアプタマーについて,ほか数種類のタンパク質に対する親和性も調べたが,それぞれの標的のみに高い選択性で結合し,従来のアプタマーに比べ選択性も向上していることがわかった.
おわりに
人工塩基対の研究のおもな目的のひとつは,塩基の種類を増やすことにより遺伝情報を拡張し,タンパク質に非天然型アミノ酸を導入することである.しかし,核酸の多様な機能がわかってくると,人工塩基を核酸に導入することにより,リガンドや触媒などとしての機能の向上も期待されるようになってきた.今回の人工塩基対を導入したDNAアプタマーの研究から,天然型塩基とは性質の異なるわずか1種類の人工塩基を2~3箇所に組み込むことにより,DNAの高機能化が可能であることがはじめて実証された.今後,この手法を用いて得られる人工塩基DNAアプタマーの医療への応用が期待される.
Ds-Px塩基対をSELEX法に応用できたことは,人工塩基対が第3の塩基対として実用化レベルに達しつつあることを示した.このSELEX法では,それぞれのラウンドで20サイクル以上のPCRを行い,合計で7ラウンドの選別を行った.したがって,人工塩基は総計140サイクル以上のPCR増幅に生き残ったことになった.とくにこの手法では,あらかじめサブライブラリーにおいて人工塩基の位置を固定しているので,SELEX法において人工塩基が天然型塩基に置き換わったり,あるいは,天然型塩基が人工塩基に置き換わったりしてしまっては成り立たなくなってしまう.したがって,Ds-Px塩基対のPCRにおける選択性は天然型塩基対に匹敵するレベルに達していると考えられた.
今後,人工塩基対システムは次世代の遺伝子操作の基盤をなす技術として,SELEX法のみならず,幅広い分野に利用されるものと期待している.
文 献
- Ellington, A. D. & Szostak, J. W.: In vitro selection of RNA molecules that bind specific ligands. Nature, 346, 818-822 (1990)[PubMed]
- Tuerk, C. & Gold, L.: Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science, 249, 505-510 (1990)[PubMed]
- Rich, A.: Problems of evolution and biochemical information transfer. in Horizons in Biochemistry (Kasha, M. & Pullman, B. Eds.), pp.103-126, Academic Press, New York (1962)
- Switzer, C., Moroney, S. E. & Benner, S. A.: Enzymatic incorporation of a new base pair into DNA and RNA. J. Am. Chem. Soc., 111, 8322-8323 (1989)[PubMed]
- Hirao, I., Ohtsuki, T., Fujiwara, T. et al.: An unnatural base pair for incorporating amino acid analogs into proteins. Nat. Biotechnol., 20, 177-182 (2002)[PubMed]
- Hirao, I., Kimoto, M., Mitsui, T. et al.: An unnatural hydrophobic base pair system: site-specific incorporation of nucleotide analogs into DNA and RNA. Nat. Methods, 3, 729-735 (2006)[PubMed]
- Kimoto, M., Kawai, R., Mitsui, T. et al.: An unnatural base pair system for efficient PCR amplification and functionalization of DNA molecules. Nucleic Acids Res., 37, e14 (2009)[PubMed]
- Yamashige, R., Kimoto, M., Takezawa, Y. et al.: Highly specific unnatural base pair systems as a third base pair for PCR amplification. Nucleic Acids Res., 40, 2793-2806 (2012)[PubMed]
- Hirao, I. & Kimoto, M.: Unnatural base pair systems toward the expansion of the genetic alphabet in the central dogma. Proc. Jpn. Acad. Ser. B. Phys. Biol. Sci., 88, 345-367 (2012)[PubMed]
- Ng, E. W. M., Shima, D. T., Calias, P. et al.: Pegaptanib, a targeted anti-VEGF aptamer for ocular vascular disease. Nat. Rev. Drug Discov., 5, 123-132 (2006)[PubMed]
- Ruckman, J., Green, L. S., Beeson, J. et al.: 2'-Fluoropyrimidine RNA-based aptamers to the 165-amino acid form of vascular endothelial growth factor (VEGF165). Inhibition of receptor binding and VEGF-induced vascular permeability through interactions requiring the exon 7-encoded domain. J. Biol. Chem., 273, 20556-20567 (1998)[PubMed]
- Potty, A. S. R., Kourentzi, K., Fang, H. et al.: Biophysical characterization of DNA aptamer interactions with vascular endothelial growth factor. Biopolymers, 91, 145-156 (2009)[PubMed]
- Liu, Y., Tuleouva, N., Ramanculov, E. et al.: Aptamer-based electrochemical biosensor for interferon gamma detection. Anal. Chem., 82, 8131-8136 (2010)[PubMed]
著者プロフィール
略歴:2002年 東京大学大学院理学系研究科博士課程 修了,2013年より理化学研究所ライフサイエンス技術基盤研究センター 上級研究員.
平尾一郎(Ichiro Hirao)
略歴:1983年 東京工業大学理工学研究科後期博士課程 修了,2007年よりタグシクス・バイオ株式会社 代表取締役社長,2013年より理化学研究所ライフサイエンス技術基盤研究センター チームリーダー.
研究室URL:http://protein.gsc.riken.jp/hirao/index.html
© 2013 木本路子・平尾一郎 Licensed under CC 表示 2.1 日本