イネの栽培化の起源がゲノムの全域における変異比較解析により判明した
倉田のり・久保貴彦
(国立遺伝学研究所 系統生物研究センター植物遺伝研究室)
email:倉田のり
DOI: 10.7875/first.author.2012.139
A map of rice genome variation reveals the origin of cultivated rice.
Xuehui Huang, Nori Kurata, Xinghua Wei, Zi-Xuan Wang, Ahong Wang, Qiang Zhao, Yan Zhao, Kunyan Liu, Hengyun Lu, Wenjun Li, Yunli Guo, Yiqi Lu, Congcong Zhou, Danlin Fan, Qijun Weng, Chuanrang Zhu, Tao Huang, Lei Zhang, Yongchun Wang, Lei Feng, Hiroyasu Furuumi, Takahiko Kubo, Toshie Miyabayashi, Xiaoping Yuan, Qun Xu, Guojun Dong, Qilin Zhan, Canyang Li, Asao Fujiyama, Atsushi Toyoda, Tingting Lu, Qi Feng, Qian Qian, Jiayang Li, Bin Han
Nature, 490, 497-501 (2012)
イネの栽培化は人類の歴史においてもっとも重要な進歩のひとつであるが,その起源地や栽培化のプロセスについては長いあいだ論争がつづいていた.今回,筆者らは,世界各地から収集した栽培イネOryza sativa 1083品種と,その起源種とされる野生イネO. rufipogon 446系統のゲノムの解読を行い,包括的なゲノム変異マップを作成した.また,遺伝的な変異パターンにもとづき,栽培化の過程で強い選択的なスイープの起こった55箇所のゲノム領域を検出した.これらの領域には,脱粒性,草型,粒幅など,栽培化にかかわる重要な遺伝子が存在していた.選択的なスイープの起こったゲノム領域,および,全ゲノムの精密な解析から,イネの栽培化は中国の珠江中流域ではじまり,O. rufipogonのかぎられた集団からジャポニカ(O. sativa japonica)の生まれたことが明らかになった.また,ジャポニカの誕生につづいて,東南アジアや南アジアの野生イネ系統とジャポニカとの交配によりインディカ(O. sativa indica)の生まれたことが判明した.今回,得られた,遺伝的に多様なイネのゲノム情報と栽培化遺伝子に関する知見は今後の研究の発展と応用に重要な成果となる.
イネは主要な栄養源として世界中で栽培されている重要な作物であり,また,モデル植物のひとつでもある.栽培イネ(学名Oryza sativa L.)は数千年前に近縁の野生イネO. rufipogonから栽培化されたといわれている.この2つの種は遺伝的に近縁であるものの,生理的および形態的な特徴は大きく異なっている.これまで,遺伝学や考古学などさまざまな視点からイネ系統間の進化関係や栽培化に関する研究が進められてきたが,栽培イネの起源や具体的な栽培化のプロセスについては論争がつづいていた.争点の中心は,1)地理的な起源はどこであるか,2)遺伝的な変異の多様なO. rufipogonの集団のなかでどの系統が直接的な起源であるか,3)O. sativaの2つの亜種であるインディカ(O. sativa indica,タイ米などの長粒米)とジャポニカ(O. sativa japonica,日本米などの短粒米)はどのように分化したか,などである.分子系統解析はインディカとジャポニカは独立に栽培化の過程をへて成立したという“二元説”を支持している1,2).一方,すでに単離同定された栽培化関連遺伝子の解析は,いずれの遺伝子もインディカとジャポニカのあいだで同一の遺伝子型で固定していたことから,2つの亜種が同じ起源をもつこと(一元説)を示唆している3,4).最近,報告された630個の遺伝子におけるSNP(single nucleotide polymorphism,一塩基多型)を用いた集団構造の解析は,一元説をサポートする結果であった5).このように起源についてはさまざまな見解があり,直接的な起源系統や集団構造の変遷についてはいまだに明らかにされていない.このような研究背景のもとでは,遺伝的に幅広い多数の系統を用いて系統関係の解析を行うと同時に,栽培化遺伝子の周辺領域について遺伝子型構造(ハプロタイプ)の精密な比較解析を進めることが,重要なステップであると考えられる.筆者らの共同研究グループは,さきの報告において,栽培イネの保存品種のゲノム塩基配列の解読と,収量形質にかかわるGWAS(genome-wide association study,ゲノムワイド相関解析)を進めた3,6).この研究では,野生イネO. rufipogon 446系統のゲノム塩基配列の解読を行い,栽培イネとの系統関係を調べて栽培進化とその起源についての解析を行った.
栽培イネの分類されるイネ属Oryzaは23種からなり,そのなかでも栽培イネO. sativaの直接的な起源となる野生種とされているのがO. rufipogonである.中国および日本の遺伝子バンクにおいて保存されていた野生イネの系統保存品種より446系統のO. rufipogonを選びゲノム解析に用いた.この446の系統は1年で世代をおえる一年生の系統と栄養体で繁殖をつづける多年生の系統を含んでおり,地理的にはアジアからオセアニアと広範囲に分布する変異に富んだ系統であった.これらすべての系統について,ゲノム塩基配列の薄読みの解読(ゲノムカバー度は約2倍)を行い,ゲノム全長の塩基配列(398 Mb)の決定されている日本晴(ジャポニカ)を参照配列として解読した塩基配列を整理し,日本晴とのあいだで5,037,497個のSNPを同定した.これらのSNPから算出したO. rufipogonにおける塩基多様性は0.003以下と見積もられ,この値はO. sativaにおける塩基多様性(0.0024)よりも高かった.これは,栽培種では人為的な選抜において一部の個体のみが選択的に利用されるため起源種における遺伝的な変異は失われていくという見解7) とよく一致していた.
つぎに,検出したSNPをもとにO. rufipogonの遺伝的な集団構造の解析を行った.系統樹解析と主成分分析により,O. rufipogonは3つのサブグループOr-I,Or-II,Or-IIIに分類された.これら3つのサブグループは採集地の地理的な分布の位置関係とおおむね一致していた.また興味深いことに,中国南方から採集した系統の多くはOr-IIIグループに属していた.おのおののサブグループのあいだの遺伝的な分化の指標となるFst値は0.18であり,これはO. sativaにおける値(0.55以下)と比べると低い値であった.染色体における遺伝的な変異の分布は均一ではなく,染色体の部位により大小の差違がみられた.染色体において変異率の高い領域は68箇所あり,この領域には雑種不和合性にかかわるDPL2遺伝子や,出穂期遺伝子のOsSOC1遺伝子とGhd7遺伝子,耐冷性遺伝子のqCTS12遺伝子が含まれていた.これらの遺伝子はいずれもインディカとジャポニカとの分化に関係することが知られている.したがって,今回,検出された遺伝的な変異に富む68箇所の染色体領域は,地域適応性にかかわる遺伝子の探索において足がかりとなる情報を提供するものと考えられた.
野生イネO. rufipogonは他殖性であるため,自殖性の栽培イネと比較して連鎖不平衡ブロックは小さいと予測される.実際の解析結果によると,O. rufipogonでは平均して20 kbから急速な連鎖不平衡の減衰がみられ,栽培イネであるインディカの123 kb,ジャポニカの167 kbと比較するときわめて早い減衰が観察された.この結果をふまえて葉鞘色と草型の2つの形質に関してGWASを行ったところ,既知の出穂期遺伝子OsC1と草型遺伝子PROG1の領域に高い相関を検出した.O. sativaの集団のみでのGWASの結果と比較すると,平均で3倍以上もの高解像度でのマッピングが可能であることがわかった.この結果,野生イネの集団を用いた農業形質のGWASでは高い解像度での関与遺伝子の領域の同定が可能となることが実証された.
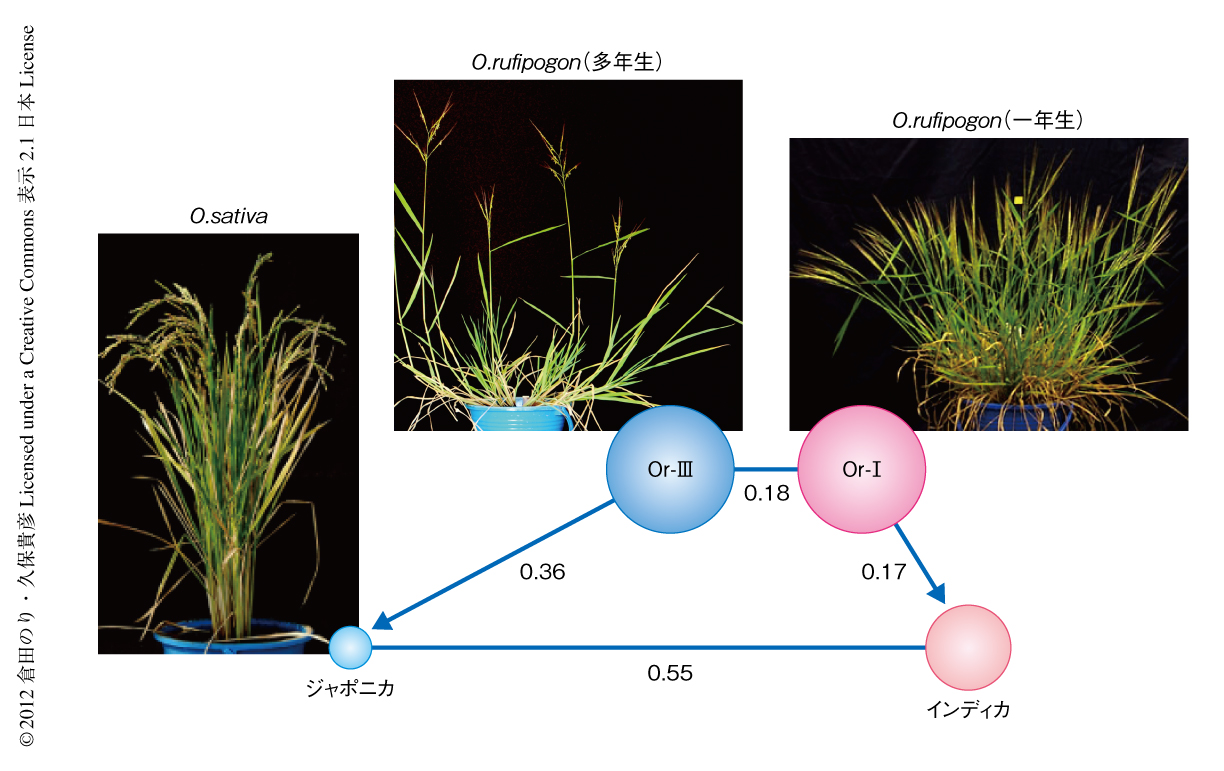
野生イネと栽培イネとの系統関係を詳細に調べるため,O. rufipogon 446系統とO. sativa 1083品種のゲノムワイドなSNP解析を行った.O. sativaの1083品種は,O. rufipogon 446系統と同様,世界各地から収集した保存品種である.O. rufipogonとO. sativaの合計1529系統から7,970,359個のSNPを同定した.さらに,これらSNPの起源を探る目的で,Aゲノム起源種である4種の野生イネO. glaberrima,O. barthii,O. longistaminata,O. meridionalisのゲノム塩基配列の解読と比較を行い,6,119,311個のSNPの起源を明らかにした.これらのSNPを利用して1529系統の系統関係について系統樹解析と主成分分析を行ったところ,インディカとジャポニカはそれぞれOr-IグループとOr-IIIグループを起源にもつことが判明した(図1).Or-Iグループとインディカ,および,Or-IIIグループとジャポニカのそれぞれ個別の関係について調べたところ,Or-Iグループに属するインディカの遺伝的な変異は小さく,インディカはOr-Iグループにおける変異の75%を含むことがわかった.また,一部のインディカとOr-Iグループの系統はこの2つのグループの中間型として位置づけられた.対照的に,ジャポニカはひとつのクラスターにまとまり,中国南方のO. rufipogon(サブクレードOr-IIIa)に起源をもつことが判明した.ジャポニカとサブクレードOr-IIIaとのあいだには明確な集団変異が含まれていた.驚いたことに,ジャポニカにおける変異はOr-IIIグループにおける変異のわずか33%しか含まれていなかった.この強いボトルネック効果はOr-IIIグループのごく一部の個体が栽培化に用いられたことを示すものであった.
ジャポニカとインディカとのあいだにみられたSNPはこの2つが数千年前に分化したことを示していたが,これらは起源種であるO. rufipogonにおいてすでに分化していたことがわかった.しかしながら,O. rufipogonの集団のあいだと比べ,ジャポニカとインディカとのあいだでは変異の広がりは強化されていた(図1).この栽培化による変異の広がりは,野生イネの集団のあいだですでに存在していた変異にくわえ,近代育種のボトルネック効果が組み合わさった結果であろうと考えられた.粒形遺伝子GS3を例としてあげると,GS3遺伝子の欠失型は野生イネの集団ではおもに中国南方の広西地区と広東省にみられたが,ジャポニカではそれが急速に広がり集団の98%をしめるにいたっていった.その結果,今日の丸い粒のジャポニカと細長い粒のインディカとの典型的な表現型の違いがもたらされたものと推察された.
さらに,インディカとジャポニカのそれぞれの集団で固定していた213,188個のSNPに着目した.これらSNPのO. rufipogonの集団のあいだでの分布の違いと,ジャポニカ,および,インディカでの栽培化のボトルネック効果の割合は,それぞれ50%,40%以下,10%以下であった.ジャポニカとインディカとのあいだにみられたSNPのAゲノム起源種に由来する遺伝子型の頻度は55%がインディカのSNP,45%がジャポニカのSNPであり,dN/dS比は0.34でSNP全体におけるdN/dS比と類似していた.一方,O. rufipogonとO. sativaとのあいだにみられたSNPに関しては93.3%のAゲノム起源種に由来するSNPがO. rufipogonと同じであり,このことはO. rufipogonがよりAゲノム起源種に近く,O. sativaの起源がO. rufipogonであることをサポートしていた.dN/dS比は1.04であり,栽培化における正の淘汰のあることが示唆された.
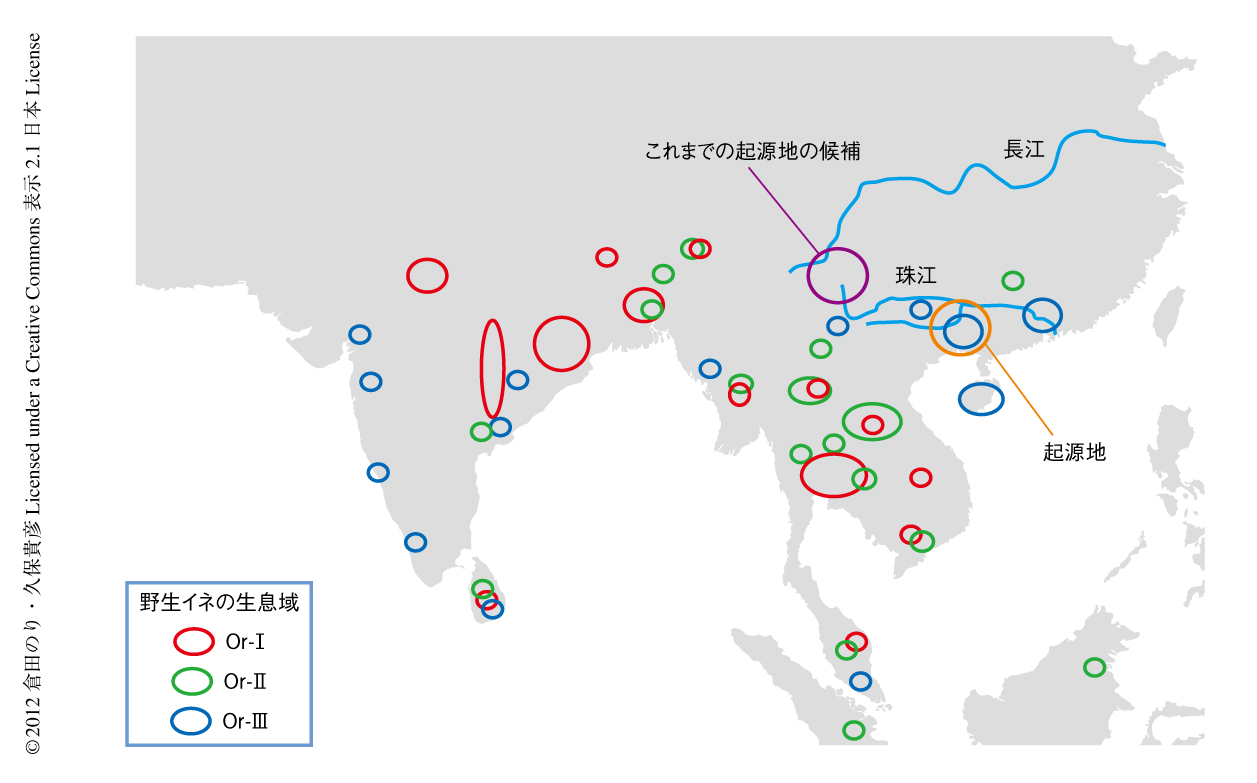
人為的な選抜による爪痕は遺伝子変異の減少と栽培化遺伝子における遺伝子頻度のかたよりとして表われる8,9).選択的なスイープの痕跡をゲノム全体にわたり検索し,インディカで60座,ジャポニカで62座,全体で55座を同定した.強い淘汰をうけたゲノム領域はジャポニカとインディカでほぼ同じであり,Fst値の低いことから多くのケースは交配による遺伝子導入により生じたともの推察された.この55座のなかには既知の遺伝子として,Bh4遺伝子(果皮色),PROG1遺伝子(分けつ角度),sh4遺伝子(脱粒性),qSW5遺伝子(粒幅),OsC1遺伝子(葉鞘色とふ先色)が含まれていた.既知の3つの遺伝子,qSH1遺伝子(脱粒性),wx遺伝子(糯性),Rc遺伝子(果皮色)は,ジャポニカでは強い選択をうけていたがインディカでは同様の選択はうけていなかった.これらの55座の栽培化遺伝子に焦点をあてSNPにより系統樹を作成したところ,ジャポニカとインディカは同じクラスターに分類され,また,中国南方に由来するOr-IIIのサブクレードはO. rufipogonのなかでもO. sativaにもっとも近い関係にあった.さきに述べたゲノム全体での系統解析ではインディカはOr-Iグループに近かったが,この55座におけるSNPの系統解析に着目した結果ではOr-IIIグループに近いという結果になった.さらに,この55座に関して野生イネ系統を採集地別に分類したうえでO. sativaとの遺伝距離を算出した結果,栽培化は中国広西地区の珠江中流域においてはじめられたことが明らかになった.以上の結果から,最初に中国南方のOr-IIIグループよりジャポニカが生まれ,つづいて,東南アジアや南アジア地域のO. rufipogonとジャポニカとの交配をへてインディカが誕生した,という栽培化の流れがもっとも矛盾の少ない説明であることが明らかになった(図2).
これらの遺伝子座が栽培化のなかでどのようにして選抜されたのかを知る目的で,Guangluai-4(インディカ)とW1943(Or-IIIaグループ)の交雑後代を用いて栽培化関連形質のQTL(quantitative trait locus,量的形質遺伝子座)解析を進めた.271系統からなる組換え自殖系統についてゲノム塩基配列の解読を行い,おのおのの系統の遺伝子型を決定した.15種類の栽培化関連形質にかかわる58個のQTLを検出し,そのうち10個は既知の遺伝子であることがわかった.これら既知遺伝子は約200 kbの精度で特定できたことから,この分離集団を用いて高解像度で遺伝子マッピングが可能であることがわかった.58個のうち32個のQTLは栽培化において選択的なスイープの起こった染色体領域に位置しており,両者の位置的な重なりは統計的に有意であった.検出されたQTLのうち,柱頭露出度(交配様式に関係)と粒サイズ(収量性)に関しては,sh4遺伝子(脱粒性)およびPROG1遺伝子(草型)と比較してもきわめて強い選択をうけていた.柱頭露出度のような交配様式の変化は作物育種における重要なターゲットであることが示唆されている.今回の解析から,柱頭露出にかかわる3つの主働QTLと微働QTLが同定され,これら5つのQTLはいずれも選択的なスイープの起こった染色体領域に位置することが見い出された.
栽培化関連遺伝子の詳細を調べるためには,栽培イネだけでなく野生イネについても精度の高いゲノム塩基配列情報が必要である.そこで,O. rufipogonの1系統であるW1943について,100倍のゲノムカバー度でゲノム塩基配列の詳細な解読を行った.W1943のゲノム全長は406 Mbで,2,621,077個のSNPと619,132箇所の挿入欠失変異,140,075箇所の微細染色体構造変異を見い出した.W1943のゲノム塩基配列情報にもとづいて予測タンパク質コード領域におけるSNPの影響を調べたところ,128,010個の非同義置換SNPと49,236個のタンパク質の大きな構造変異をもたらす変異アリルを見い出した.つぎに,すべての栽培イネ系統と野生イネ系統においてこれら変異アレルの頻度を求めた.すでに機能解析が進んでいる栽培化遺伝子について機能に影響を及ぼすと思われるSNPを絞り込むことができた.栽培化にかかわる204個の未同定遺伝子に関しては273個の機能的なSNPを同定した.くわえて,O. rufipogonとO. sativaとのあいだでプロモーター配列における305個の変異を,インディカとジャポニカとのあいだで1120個の機能変異を見い出した.
この研究は,イネがどこから,そして,どのように栽培化されてきたかという長年の疑問にひとつの答えを出した.また,栽培化において選択的なスイープの起こった染色体領域とその原因となる遺伝子を特定することができた.動物と比べ,植物では機能未知のタンパク質がたくさんあるため,今後,遺伝子の変異と機能特性をさらに詳細に調べることがイネの生理と栽培進化の成り立ちを深く理解することにつながると考えている.また,今回の結果で示されたように,栽培品種で利用されている遺伝的な変異は野生イネにみられる変異のごく一部にすぎない.野生資源の多様な変異を開発しこれらを積極的かつ効率的に利用することは,イネの育種の飛躍的な発展につながるものと期待される.
略歴:1981年 九州大学大学院農学研究科 修了,1983年 三菱化成生命科学研究所 ポスドク,1985年 藤田保健衛生大学 助手,1989年 同 講師,1991年 農業生物資源研究所 主任研究官,1996年 国立遺伝学研究所 助教授を経て,2003年より同 教授.
研究テーマ:植物のゲノム機能の進化と,生殖にはたす役割.
関心事:細胞遺伝学,細胞生物学,ゲノムマッピングと染色体の機能,植物の形態発生とゲノム機能との連関,などの分野を彷徨って今日にいたり,残りの年月で何ができるかを思案中.ゲノムの構造と機能の多様性がおもしろい.
研究室URL:http://www.nig.ac.jp/labs/PlantGen/japanese/home-j.html
久保 貴彦(Takahiko Kubo)
国立遺伝学研究所 助教.
© 2012 倉田のり・久保貴彦 Licensed under CC 表示 2.1 日本
(国立遺伝学研究所 系統生物研究センター植物遺伝研究室)
email:倉田のり
DOI: 10.7875/first.author.2012.139
A map of rice genome variation reveals the origin of cultivated rice.
Xuehui Huang, Nori Kurata, Xinghua Wei, Zi-Xuan Wang, Ahong Wang, Qiang Zhao, Yan Zhao, Kunyan Liu, Hengyun Lu, Wenjun Li, Yunli Guo, Yiqi Lu, Congcong Zhou, Danlin Fan, Qijun Weng, Chuanrang Zhu, Tao Huang, Lei Zhang, Yongchun Wang, Lei Feng, Hiroyasu Furuumi, Takahiko Kubo, Toshie Miyabayashi, Xiaoping Yuan, Qun Xu, Guojun Dong, Qilin Zhan, Canyang Li, Asao Fujiyama, Atsushi Toyoda, Tingting Lu, Qi Feng, Qian Qian, Jiayang Li, Bin Han
Nature, 490, 497-501 (2012)
この論文に出現する遺伝子・タンパク質のUniprot ID
DPL2(Q5SMP0), SOC1(Q9XJ60), Ghd7(E5RQA1), qCTS12(Q945X3), C1(Q69NN9), PROG1(Q69NY0), GS3(C6L686), Bh4, sh4(Q7XR48), qSW5(A0A1D8GZC0), qSH1(Q941S9), wx(Q0DEV5), Rc
要 約
イネの栽培化は人類の歴史においてもっとも重要な進歩のひとつであるが,その起源地や栽培化のプロセスについては長いあいだ論争がつづいていた.今回,筆者らは,世界各地から収集した栽培イネOryza sativa 1083品種と,その起源種とされる野生イネO. rufipogon 446系統のゲノムの解読を行い,包括的なゲノム変異マップを作成した.また,遺伝的な変異パターンにもとづき,栽培化の過程で強い選択的なスイープの起こった55箇所のゲノム領域を検出した.これらの領域には,脱粒性,草型,粒幅など,栽培化にかかわる重要な遺伝子が存在していた.選択的なスイープの起こったゲノム領域,および,全ゲノムの精密な解析から,イネの栽培化は中国の珠江中流域ではじまり,O. rufipogonのかぎられた集団からジャポニカ(O. sativa japonica)の生まれたことが明らかになった.また,ジャポニカの誕生につづいて,東南アジアや南アジアの野生イネ系統とジャポニカとの交配によりインディカ(O. sativa indica)の生まれたことが判明した.今回,得られた,遺伝的に多様なイネのゲノム情報と栽培化遺伝子に関する知見は今後の研究の発展と応用に重要な成果となる.
はじめに
イネは主要な栄養源として世界中で栽培されている重要な作物であり,また,モデル植物のひとつでもある.栽培イネ(学名Oryza sativa L.)は数千年前に近縁の野生イネO. rufipogonから栽培化されたといわれている.この2つの種は遺伝的に近縁であるものの,生理的および形態的な特徴は大きく異なっている.これまで,遺伝学や考古学などさまざまな視点からイネ系統間の進化関係や栽培化に関する研究が進められてきたが,栽培イネの起源や具体的な栽培化のプロセスについては論争がつづいていた.争点の中心は,1)地理的な起源はどこであるか,2)遺伝的な変異の多様なO. rufipogonの集団のなかでどの系統が直接的な起源であるか,3)O. sativaの2つの亜種であるインディカ(O. sativa indica,タイ米などの長粒米)とジャポニカ(O. sativa japonica,日本米などの短粒米)はどのように分化したか,などである.分子系統解析はインディカとジャポニカは独立に栽培化の過程をへて成立したという“二元説”を支持している1,2).一方,すでに単離同定された栽培化関連遺伝子の解析は,いずれの遺伝子もインディカとジャポニカのあいだで同一の遺伝子型で固定していたことから,2つの亜種が同じ起源をもつこと(一元説)を示唆している3,4).最近,報告された630個の遺伝子におけるSNP(single nucleotide polymorphism,一塩基多型)を用いた集団構造の解析は,一元説をサポートする結果であった5).このように起源についてはさまざまな見解があり,直接的な起源系統や集団構造の変遷についてはいまだに明らかにされていない.このような研究背景のもとでは,遺伝的に幅広い多数の系統を用いて系統関係の解析を行うと同時に,栽培化遺伝子の周辺領域について遺伝子型構造(ハプロタイプ)の精密な比較解析を進めることが,重要なステップであると考えられる.筆者らの共同研究グループは,さきの報告において,栽培イネの保存品種のゲノム塩基配列の解読と,収量形質にかかわるGWAS(genome-wide association study,ゲノムワイド相関解析)を進めた3,6).この研究では,野生イネO. rufipogon 446系統のゲノム塩基配列の解読を行い,栽培イネとの系統関係を調べて栽培進化とその起源についての解析を行った.
1.野生イネの集団のゲノム解析
栽培イネの分類されるイネ属Oryzaは23種からなり,そのなかでも栽培イネO. sativaの直接的な起源となる野生種とされているのがO. rufipogonである.中国および日本の遺伝子バンクにおいて保存されていた野生イネの系統保存品種より446系統のO. rufipogonを選びゲノム解析に用いた.この446の系統は1年で世代をおえる一年生の系統と栄養体で繁殖をつづける多年生の系統を含んでおり,地理的にはアジアからオセアニアと広範囲に分布する変異に富んだ系統であった.これらすべての系統について,ゲノム塩基配列の薄読みの解読(ゲノムカバー度は約2倍)を行い,ゲノム全長の塩基配列(398 Mb)の決定されている日本晴(ジャポニカ)を参照配列として解読した塩基配列を整理し,日本晴とのあいだで5,037,497個のSNPを同定した.これらのSNPから算出したO. rufipogonにおける塩基多様性は0.003以下と見積もられ,この値はO. sativaにおける塩基多様性(0.0024)よりも高かった.これは,栽培種では人為的な選抜において一部の個体のみが選択的に利用されるため起源種における遺伝的な変異は失われていくという見解7) とよく一致していた.
つぎに,検出したSNPをもとにO. rufipogonの遺伝的な集団構造の解析を行った.系統樹解析と主成分分析により,O. rufipogonは3つのサブグループOr-I,Or-II,Or-IIIに分類された.これら3つのサブグループは採集地の地理的な分布の位置関係とおおむね一致していた.また興味深いことに,中国南方から採集した系統の多くはOr-IIIグループに属していた.おのおののサブグループのあいだの遺伝的な分化の指標となるFst値は0.18であり,これはO. sativaにおける値(0.55以下)と比べると低い値であった.染色体における遺伝的な変異の分布は均一ではなく,染色体の部位により大小の差違がみられた.染色体において変異率の高い領域は68箇所あり,この領域には雑種不和合性にかかわるDPL2遺伝子や,出穂期遺伝子のOsSOC1遺伝子とGhd7遺伝子,耐冷性遺伝子のqCTS12遺伝子が含まれていた.これらの遺伝子はいずれもインディカとジャポニカとの分化に関係することが知られている.したがって,今回,検出された遺伝的な変異に富む68箇所の染色体領域は,地域適応性にかかわる遺伝子の探索において足がかりとなる情報を提供するものと考えられた.
野生イネO. rufipogonは他殖性であるため,自殖性の栽培イネと比較して連鎖不平衡ブロックは小さいと予測される.実際の解析結果によると,O. rufipogonでは平均して20 kbから急速な連鎖不平衡の減衰がみられ,栽培イネであるインディカの123 kb,ジャポニカの167 kbと比較するときわめて早い減衰が観察された.この結果をふまえて葉鞘色と草型の2つの形質に関してGWASを行ったところ,既知の出穂期遺伝子OsC1と草型遺伝子PROG1の領域に高い相関を検出した.O. sativaの集団のみでのGWASの結果と比較すると,平均で3倍以上もの高解像度でのマッピングが可能であることがわかった.この結果,野生イネの集団を用いた農業形質のGWASでは高い解像度での関与遺伝子の領域の同定が可能となることが実証された.
2.野生イネと栽培イネの系統関係
野生イネと栽培イネとの系統関係を詳細に調べるため,O. rufipogon 446系統とO. sativa 1083品種のゲノムワイドなSNP解析を行った.O. sativaの1083品種は,O. rufipogon 446系統と同様,世界各地から収集した保存品種である.O. rufipogonとO. sativaの合計1529系統から7,970,359個のSNPを同定した.さらに,これらSNPの起源を探る目的で,Aゲノム起源種である4種の野生イネO. glaberrima,O. barthii,O. longistaminata,O. meridionalisのゲノム塩基配列の解読と比較を行い,6,119,311個のSNPの起源を明らかにした.これらのSNPを利用して1529系統の系統関係について系統樹解析と主成分分析を行ったところ,インディカとジャポニカはそれぞれOr-IグループとOr-IIIグループを起源にもつことが判明した(図1).Or-Iグループとインディカ,および,Or-IIIグループとジャポニカのそれぞれ個別の関係について調べたところ,Or-Iグループに属するインディカの遺伝的な変異は小さく,インディカはOr-Iグループにおける変異の75%を含むことがわかった.また,一部のインディカとOr-Iグループの系統はこの2つのグループの中間型として位置づけられた.対照的に,ジャポニカはひとつのクラスターにまとまり,中国南方のO. rufipogon(サブクレードOr-IIIa)に起源をもつことが判明した.ジャポニカとサブクレードOr-IIIaとのあいだには明確な集団変異が含まれていた.驚いたことに,ジャポニカにおける変異はOr-IIIグループにおける変異のわずか33%しか含まれていなかった.この強いボトルネック効果はOr-IIIグループのごく一部の個体が栽培化に用いられたことを示すものであった.
ジャポニカとインディカとのあいだにみられたSNPはこの2つが数千年前に分化したことを示していたが,これらは起源種であるO. rufipogonにおいてすでに分化していたことがわかった.しかしながら,O. rufipogonの集団のあいだと比べ,ジャポニカとインディカとのあいだでは変異の広がりは強化されていた(図1).この栽培化による変異の広がりは,野生イネの集団のあいだですでに存在していた変異にくわえ,近代育種のボトルネック効果が組み合わさった結果であろうと考えられた.粒形遺伝子GS3を例としてあげると,GS3遺伝子の欠失型は野生イネの集団ではおもに中国南方の広西地区と広東省にみられたが,ジャポニカではそれが急速に広がり集団の98%をしめるにいたっていった.その結果,今日の丸い粒のジャポニカと細長い粒のインディカとの典型的な表現型の違いがもたらされたものと推察された.
さらに,インディカとジャポニカのそれぞれの集団で固定していた213,188個のSNPに着目した.これらSNPのO. rufipogonの集団のあいだでの分布の違いと,ジャポニカ,および,インディカでの栽培化のボトルネック効果の割合は,それぞれ50%,40%以下,10%以下であった.ジャポニカとインディカとのあいだにみられたSNPのAゲノム起源種に由来する遺伝子型の頻度は55%がインディカのSNP,45%がジャポニカのSNPであり,dN/dS比は0.34でSNP全体におけるdN/dS比と類似していた.一方,O. rufipogonとO. sativaとのあいだにみられたSNPに関しては93.3%のAゲノム起源種に由来するSNPがO. rufipogonと同じであり,このことはO. rufipogonがよりAゲノム起源種に近く,O. sativaの起源がO. rufipogonであることをサポートしていた.dN/dS比は1.04であり,栽培化における正の淘汰のあることが示唆された.
3.栽培化遺伝子のゲノムワイドな解析
人為的な選抜による爪痕は遺伝子変異の減少と栽培化遺伝子における遺伝子頻度のかたよりとして表われる8,9).選択的なスイープの痕跡をゲノム全体にわたり検索し,インディカで60座,ジャポニカで62座,全体で55座を同定した.強い淘汰をうけたゲノム領域はジャポニカとインディカでほぼ同じであり,Fst値の低いことから多くのケースは交配による遺伝子導入により生じたともの推察された.この55座のなかには既知の遺伝子として,Bh4遺伝子(果皮色),PROG1遺伝子(分けつ角度),sh4遺伝子(脱粒性),qSW5遺伝子(粒幅),OsC1遺伝子(葉鞘色とふ先色)が含まれていた.既知の3つの遺伝子,qSH1遺伝子(脱粒性),wx遺伝子(糯性),Rc遺伝子(果皮色)は,ジャポニカでは強い選択をうけていたがインディカでは同様の選択はうけていなかった.これらの55座の栽培化遺伝子に焦点をあてSNPにより系統樹を作成したところ,ジャポニカとインディカは同じクラスターに分類され,また,中国南方に由来するOr-IIIのサブクレードはO. rufipogonのなかでもO. sativaにもっとも近い関係にあった.さきに述べたゲノム全体での系統解析ではインディカはOr-Iグループに近かったが,この55座におけるSNPの系統解析に着目した結果ではOr-IIIグループに近いという結果になった.さらに,この55座に関して野生イネ系統を採集地別に分類したうえでO. sativaとの遺伝距離を算出した結果,栽培化は中国広西地区の珠江中流域においてはじめられたことが明らかになった.以上の結果から,最初に中国南方のOr-IIIグループよりジャポニカが生まれ,つづいて,東南アジアや南アジア地域のO. rufipogonとジャポニカとの交配をへてインディカが誕生した,という栽培化の流れがもっとも矛盾の少ない説明であることが明らかになった(図2).
これらの遺伝子座が栽培化のなかでどのようにして選抜されたのかを知る目的で,Guangluai-4(インディカ)とW1943(Or-IIIaグループ)の交雑後代を用いて栽培化関連形質のQTL(quantitative trait locus,量的形質遺伝子座)解析を進めた.271系統からなる組換え自殖系統についてゲノム塩基配列の解読を行い,おのおのの系統の遺伝子型を決定した.15種類の栽培化関連形質にかかわる58個のQTLを検出し,そのうち10個は既知の遺伝子であることがわかった.これら既知遺伝子は約200 kbの精度で特定できたことから,この分離集団を用いて高解像度で遺伝子マッピングが可能であることがわかった.58個のうち32個のQTLは栽培化において選択的なスイープの起こった染色体領域に位置しており,両者の位置的な重なりは統計的に有意であった.検出されたQTLのうち,柱頭露出度(交配様式に関係)と粒サイズ(収量性)に関しては,sh4遺伝子(脱粒性)およびPROG1遺伝子(草型)と比較してもきわめて強い選択をうけていた.柱頭露出度のような交配様式の変化は作物育種における重要なターゲットであることが示唆されている.今回の解析から,柱頭露出にかかわる3つの主働QTLと微働QTLが同定され,これら5つのQTLはいずれも選択的なスイープの起こった染色体領域に位置することが見い出された.
4.栽培化関連遺伝子の配列変異
栽培化関連遺伝子の詳細を調べるためには,栽培イネだけでなく野生イネについても精度の高いゲノム塩基配列情報が必要である.そこで,O. rufipogonの1系統であるW1943について,100倍のゲノムカバー度でゲノム塩基配列の詳細な解読を行った.W1943のゲノム全長は406 Mbで,2,621,077個のSNPと619,132箇所の挿入欠失変異,140,075箇所の微細染色体構造変異を見い出した.W1943のゲノム塩基配列情報にもとづいて予測タンパク質コード領域におけるSNPの影響を調べたところ,128,010個の非同義置換SNPと49,236個のタンパク質の大きな構造変異をもたらす変異アリルを見い出した.つぎに,すべての栽培イネ系統と野生イネ系統においてこれら変異アレルの頻度を求めた.すでに機能解析が進んでいる栽培化遺伝子について機能に影響を及ぼすと思われるSNPを絞り込むことができた.栽培化にかかわる204個の未同定遺伝子に関しては273個の機能的なSNPを同定した.くわえて,O. rufipogonとO. sativaとのあいだでプロモーター配列における305個の変異を,インディカとジャポニカとのあいだで1120個の機能変異を見い出した.
おわりに
この研究は,イネがどこから,そして,どのように栽培化されてきたかという長年の疑問にひとつの答えを出した.また,栽培化において選択的なスイープの起こった染色体領域とその原因となる遺伝子を特定することができた.動物と比べ,植物では機能未知のタンパク質がたくさんあるため,今後,遺伝子の変異と機能特性をさらに詳細に調べることがイネの生理と栽培進化の成り立ちを深く理解することにつながると考えている.また,今回の結果で示されたように,栽培品種で利用されている遺伝的な変異は野生イネにみられる変異のごく一部にすぎない.野生資源の多様な変異を開発しこれらを積極的かつ効率的に利用することは,イネの育種の飛躍的な発展につながるものと期待される.
文 献
- Londo, J. P., Chiang, Y. C., Hung, K. H. et al.: Phylogeography of Asian wild rice, Oryza rufipogon, reveals multiple independent domestications of cultivated rice, Oryza sativa. Proc. Natl. Acad. Sci. USA, 103, 9578-9583 (2006)[PubMed]
- Ma, J. & Bennetzen, J. L.: Rapid recent growth and divergence of rice nuclear genomes. Proc. Natl. Acad. Sci. USA, 101, 12404-12410 (2004)[PubMed]
- Li, C., Zhou, A. & Sang, T.: Rice domestication by reducing shattering. Science, 311, 1936-1939 (2006)[PubMed]
- Jin, J., Huang, W., Gao, J. P. et al.: Genetic control of rice plant architecture under domestication. Nat. Genet., 40, 1365-1369 (2008)[PubMed]
- Molina, J., Sikora, M., Garud, N. et al.: Molecular evidence for a single evolutionary origin of domesticated rice. Proc. Natl. Acad. Sci. USA, 108, 8351-8356 (2011)[PubMed]
- Huang, X., Zhao, Y., Wei, X. et al.: Genome-wide association study of flowering time and grain yield traits in a worldwide collection of rice germplasm. Nat. Genet., 44, 32-39 (2012)[PubMed]
- Novembre, J. & Stephens, M.: Interpreting principal component analyses of spatial population genetic variation. Nat. Genet., 40, 646-649 (2008)[PubMed]
- Xu, X., Liu, X., Ge, S. et al.: Resequencing 50 accessions of cultivated and wild rice yields markers for identifying agronomically important genes. Nat. Biotechnol., 30, 105-111 (2012)[PubMed]
- Doebley, J. F., Gaut, B. S. & Smith, B. D.: The molecular genetics of crop domestication. Cell, 127, 1309-1321 (2006)[PubMed]
著者プロフィール
略歴:1981年 九州大学大学院農学研究科 修了,1983年 三菱化成生命科学研究所 ポスドク,1985年 藤田保健衛生大学 助手,1989年 同 講師,1991年 農業生物資源研究所 主任研究官,1996年 国立遺伝学研究所 助教授を経て,2003年より同 教授.
研究テーマ:植物のゲノム機能の進化と,生殖にはたす役割.
関心事:細胞遺伝学,細胞生物学,ゲノムマッピングと染色体の機能,植物の形態発生とゲノム機能との連関,などの分野を彷徨って今日にいたり,残りの年月で何ができるかを思案中.ゲノムの構造と機能の多様性がおもしろい.
研究室URL:http://www.nig.ac.jp/labs/PlantGen/japanese/home-j.html
久保 貴彦(Takahiko Kubo)
国立遺伝学研究所 助教.
© 2012 倉田のり・久保貴彦 Licensed under CC 表示 2.1 日本