レトロウイルスに対する宿主防御タンパク質APOBEC3CのX線結晶構造の決定とHIV-1のウイルス遺伝子産物Vifとの結合部位の同定
北村紳悟・岩谷靖雅
(名古屋医療センター臨床研究センター 感染・免疫研究部)
email:岩谷靖雅
DOI: 10.7875/first.author.2012.126
The APOBEC3C crystal structure and the interface for HIV-1 Vif binding.
Shingo Kitamura, Hirotaka Ode, Masaaki Nakashima, Mayumi Imahashi, Yuriko Naganawa, Teppei Kurosawa, Yoshiyuki Yokomaku, Takashi Yamane, Nobuhisa Watanabe, Atsuo Suzuki, Wataru Sugiura, Yasumasa Iwatani
Nature Structural & Molecular Biology, 19, 1005-1010 (2012)
APOBEC3ファミリータンパク質はシチジン脱アミノ化酵素であり,細胞においてレトロウイルスの増殖を抑制する宿主防御タンパク質である.しかし,HIV-1はウイルス遺伝子産物Vifを発現しAPOBEC3の分解を促進することにより,その抑制機構を解除し増殖することが知られていた.しかし,APOBEC3およびVifの構造学的な知見は乏しく,APOBEC3とVifとの結合における構造学的な分子基盤は不明であった.今回,筆者らは,X線結晶構造解析法によりAPOBEC3ファミリータンパク質のひとつAPOBEC3Cの分子構造を決定した.さらに,この構造情報を利用して点変異解析を行い,Vifとの結合に重要な10個のアミノ酸残基を同定した.これらの残基は浅い“くぼみ”を形成していることが明らかになった.さらに,Vifとの結合領域はほかの2つのAPOBEC3ファミリータンパク質においても高度に保存されていることがわかった.以上の研究は,APOBEC3とVifとの結合における新たな理解と,APOBEC3を活用した新規の抗HIV薬の開発にむけた重要な情報につながると考えられた.
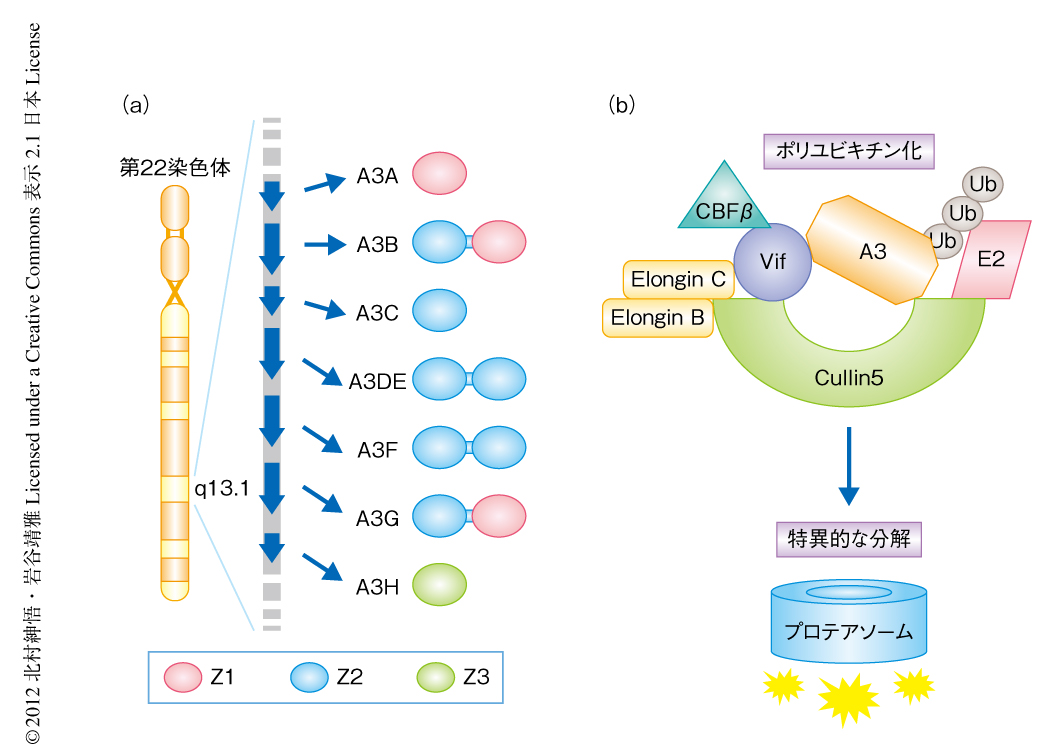
APOBEC3ファミリータンパク質はレトロウイルスやレトロトランスポゾンの複製を抑制する宿主防御タンパク質である1).ヒトでは,第22染色体に7種類(A,B,C,DE,F,G,H)がコードされ(図1a),リンパ球やマクロファージなどの細胞に発現している2).これらは共通してシチジン脱アミノ化酵素活性および核酸への結合能をもち,それぞれの抗レトロウイルス作用の機序に寄与している.
一方,レトロウイルスの仲間であるHIV-1(human immunodeficiency virus type-1,ヒト免疫不全ウイルス1型)は進化の過程でウイルス遺伝子vif(viral infectivity factor)を獲得し,感染細胞においてその遺伝子産物Vifを発現することにより,細胞に存在するAPOBEC3を分解し除去する.そのことにより,HIV-1はAPOBEC3による生体防御機構からのがれ生体において増殖することが可能となっている.VifによるAPOBEC3の抗レトロウイルス作用の解除の機序は,Vifがアダプタータンパク質としてAPOBEC3を特異的にユビキチンリガーゼ複合体にリクルートし,そのポリユビキチン化を介しプロテアソーム系による分解を促すことによる3).さらに,その分解はVifとAPOBEC3との結合の特異性により決定されることが知られていた(図1b).
APOBEC3は遺伝進化学的にZ1型ドメイン,Z2型ドメイン,Z3型ドメインのうち1つあるいは2つから構成され(図1a),このうちVifに結合する責任ドメインはZ2型ドメインあるいはZ3型ドメインであることは知られていたが4),APOBEC3とVifとの結合様式,とくに,その構造学的な情報は明らかになっていなかった.抗HIV薬の新規の作用点につながる可能性が高いことから世界的に精力的な研究がなされていたにもかかわらず,多くの研究グループは苦戦を強いられてきた.その一因は,VifおよびAPOBEC3は非常に難溶性のタンパク質であるためタンパク質の発現および精製が非常に困難なことによる.2008年,複数のグループにより,Z1型ドメインであるAPOBEC3GのC末端側ドメインの構造が決定された5-8).しかし,これはVifの結合する責任ドメインでないため,Vifの結合するドメインの構造の解明が求められていた.もちろん筆者らも,長年にわたりこの研究競争に参戦していた.
大腸菌を用いた発現系によりAPOBEC3CをGSTとの融合タンパク質として大量発現し精製した.GSTは可溶化を補助する目的でAPOBEC3CのN末端に融合させた.GSTを切断したのち再精製し,純度95%以上のAPOBEC3Cを得た.特筆すべきは,可溶化および濃縮方法の最条件を見い出すために多大な時間を費やしたことである.精製したAPOBEC3Cをハンギングドロップ蒸気拡散法により結晶化し,X線回折データを収集,解析し,分子構造を決定した.まず,3.00Åの分解能の構造(PDB ID:3VM8),ひきつづき,2.15Åの高分解能の構造(PDB ID:3VOW )を決定することに成功した.APOBEC3Cはすでに報告されていたAPOBEC3GのC末端側ドメイン8)(PDB ID:3IR2)と同様に,酵素活性中心にZn2+を配位した6つのαヘリックスと5つのβストランドからなるコア構造を形成していた(図2a).対照的に,それぞれのループ領域はAPOBEC3GのC末端側ドメインと異なっており,Vifへの結合能など生化学的な特性の違いに寄与していることが推測された.興味深いことに,このAPOBEC3Cのループ領域の構造はAPOBEC3GのC末端側ドメインとは異なっていたものの,核酸基質を認識するために重要なアミノ酸残基の位置は類似していた.
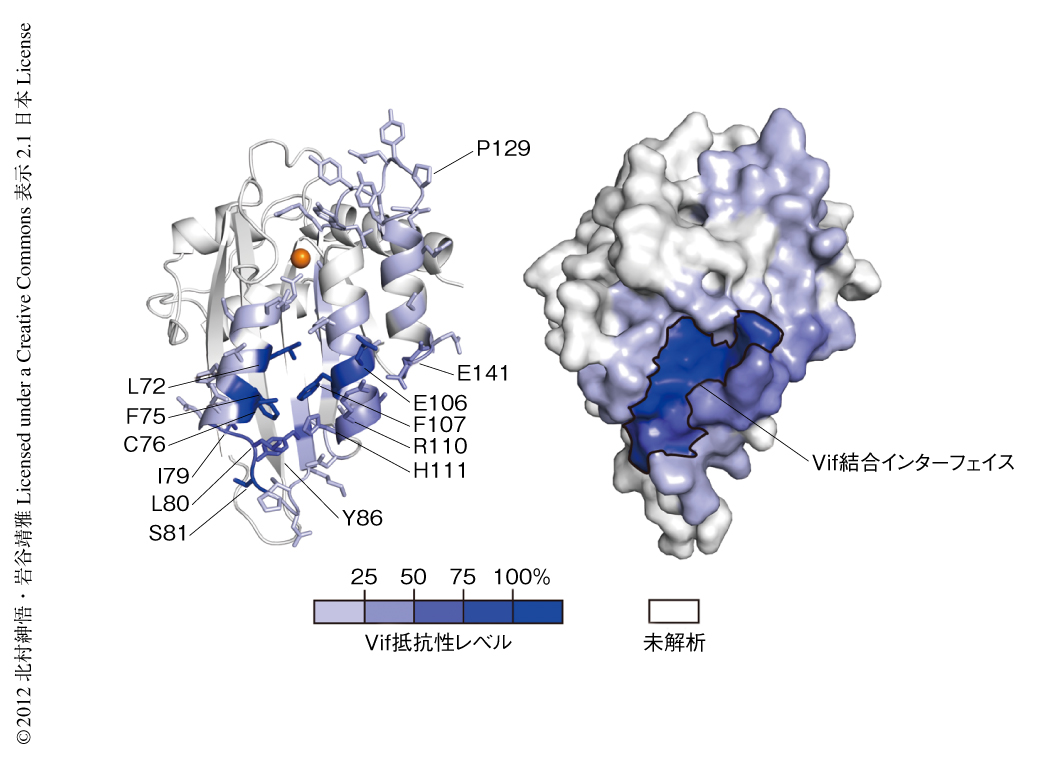
Vifに依存的なAPOBEC3Cの細胞内分解はAPOBEC3CとVifとの結合が前提になる.そこで,Vifとの結合に関与するAPOBEC3Cのアミノ酸残基を同定するため,Vifに依存的な分解の起こらない(Vif抵抗性)変異体を探索した.その際,アミノ酸配列のレベルではなくタンパク質の実構造情報をもとに変異体の解析を行う構造情報にもとづく変異解析法(structure-guided mutagenesis)を活用した.まず,決定されたAPOBEC3Cの分子構造をもとに分子表面に存在する残基を網羅的に抽出し点変異を導入した.作製したAPOBEC3C変異体をVifとともに293T細胞において共発現し,細胞におけるAPOBEC3Cの量をウェスタンブロット法により解析した.野生型のAPOBEC3CのようにVifに依存して完全に分解され消失するAPOBEC3C変異体をVif感受性と名づけた.これら一連の解析は,分子表面においてVif抵抗性を付与する残基が感受性を付与する残基によりおおわれるまでくり返し行った.最終的に99個のAPOBEC3C変異体を作製し,Vifに依存的な分解に関与する10個のアミノ酸残基を同定した.また,ウェスタンブロット法による解析の結果からAPOBEC3Cを定量しVifによる分解率を求めた.それぞれの残基への変異導入においてAPOBEC3Cの分解率を“Vif抵抗性レベル”とし,Vifの結合に関与するレベルに近似するものとして数値化した.ちなみに,共免疫沈降実験により,同定された10個の残基はVifとの結合において実質的にも重要であることが確認された.以上の結果から,これら10個の残基により構成される領域がAPOBEC3CにおけるVif結合インターフェイスであることが示された.また,これら10個の残基おのおののAPOBEC3C変異体は野生型のAPOBEC3Cと同様にウイルス粒子に取り込まれたことから,10個の残基は核酸との結合に関与する領域ではないと考えられた.
Vif抵抗性レベルを分子構造にマッピングし,Vif結合インターフェイスの構造学的な特性を詳細に解析した.その結果,Vif結合インターフェイスはタンパク質の内部の疎水性アミノ酸残基あるいは芳香族アミノ酸残基と,タンパク質の表面の負電荷アミノ酸残基からなる浅い“くぼみ”を形成していることが明らかになった(図2b).くぼみの底部には2つのπ結合が想定され,これらはVif結合インターフェイスの構造の維持に寄与していると考えられた.一方,くぼみのふちの一部の温度因子は高く,柔軟な構造をとる可能性のあることもわかった.このことは分子動力学的な構造解析によっても検証された.柔軟性のあるふちの構造は,Vifとの結合により変化し最終的により強い結合を補助する可能性もあり,一部に誘導適合(induced-fit)モデルがあてはまるのではないかと考えている.
APOBEC3Cと配列相同性が高いため類似したVif結合様式をもつと考えられているAPOBEC3FとAPOBEC3DEにおいて,APOBEC3Cで同定された10個のアミノ酸残基が実際にVif結合インターフェイスとして機能しているのかどうか検討した.その結果,APOBEC3Cと同様に,APOBEC3FおよびAPOBEC3DEにおいても10個の残基はVifに依存的な分解およびVifとの結合に重要であることが示された.APOBEC3Cの分子構造(PDB ID:3VOW )を鋳型としてAPOBEC3FのC末端側ドメインおよびAPOBEC3DEのC末端側ドメインのホモロジーモデル構造を構築し,同定した10個の残基をマッピングした結果,APOBEC3FとAPOBEC3DEにおいてもVif結合インターフェイスとしてAPOBEC3Cに類似した“くぼみ”を形成していることがわかった.以上のことから,APOBEC3C,APOBEC3F,APOBEC3DEにおけるVif結合インターフェイスは機能的にも構造的にも保存されていることが明らかになった.最後に,ヒトの細胞を用いてHIV-1に対する抗レトロウイルス作用を解析した結果,Vif抵抗性を示すすべてのAPOBEC3F変異体は細胞においてVifが存在しても分解されず,これらの変異体はHIV-1の感染を抑制することが示された.
今回,Vifと結合するZ2型ドメインとして,APOBEC3の分子構造をはじめて決定しただけでなく,構造情報にもとづく変異解析法によりAPOBEC3においてVifとの結合に重要な10個のアミノ酸残基を見い出すことができた.実は,一連の研究の途中,米国のグループが別のアプローチにより10個の残基のうち1つの残基を同定し報告した際には9),肝を冷やしたという裏話がある.さて,同定した10個の残基で構成されるVif結合インターフェイスは負電荷を帯びた浅い“くぼみ”を形成することも明らかになった.興味深いことに,Vifの側からAPOBEC3とVifとの結合を考えてみると,Vifにおける責任領域には正電荷を帯びたDRMRモチーフが必須であるという知見から10),APOBEC3C,APOBEC3F,APOBEC3DEとVifとの結合には静電的な力が重要であると思われた.
この研究は,APOBEC3とVifとの結合における構造学的な分子基盤の一翼を明らかにしただけでなく,APOBEC3とVifとのあいだの相互作用を阻害することで宿主防御機構を活用した薬剤の探索に道筋をつけ,新しいAIDS治療薬の開発にむけた動きをさらに加速するものと期待する.
略歴:名古屋大学大学院工学研究科博士課程 在学中.
研究テーマ:APOBEC3の分子構造にもとづく抗ウイルス分子機序の解明.
関心事:APOBEC3を活用した新規の薬剤を開発すること.APOBEC3Gの完全長の分子構造を決定すること.
岩谷 靖雅(Yasumasa Iwatani)
名古屋医療センター臨床研究センター 室長.
© 2012 北村紳悟・岩谷靖雅 Licensed under CC 表示 2.1 日本
(名古屋医療センター臨床研究センター 感染・免疫研究部)
email:岩谷靖雅
DOI: 10.7875/first.author.2012.126
The APOBEC3C crystal structure and the interface for HIV-1 Vif binding.
Shingo Kitamura, Hirotaka Ode, Masaaki Nakashima, Mayumi Imahashi, Yuriko Naganawa, Teppei Kurosawa, Yoshiyuki Yokomaku, Takashi Yamane, Nobuhisa Watanabe, Atsuo Suzuki, Wataru Sugiura, Yasumasa Iwatani
Nature Structural & Molecular Biology, 19, 1005-1010 (2012)
要 約
APOBEC3ファミリータンパク質はシチジン脱アミノ化酵素であり,細胞においてレトロウイルスの増殖を抑制する宿主防御タンパク質である.しかし,HIV-1はウイルス遺伝子産物Vifを発現しAPOBEC3の分解を促進することにより,その抑制機構を解除し増殖することが知られていた.しかし,APOBEC3およびVifの構造学的な知見は乏しく,APOBEC3とVifとの結合における構造学的な分子基盤は不明であった.今回,筆者らは,X線結晶構造解析法によりAPOBEC3ファミリータンパク質のひとつAPOBEC3Cの分子構造を決定した.さらに,この構造情報を利用して点変異解析を行い,Vifとの結合に重要な10個のアミノ酸残基を同定した.これらの残基は浅い“くぼみ”を形成していることが明らかになった.さらに,Vifとの結合領域はほかの2つのAPOBEC3ファミリータンパク質においても高度に保存されていることがわかった.以上の研究は,APOBEC3とVifとの結合における新たな理解と,APOBEC3を活用した新規の抗HIV薬の開発にむけた重要な情報につながると考えられた.
はじめに
APOBEC3ファミリータンパク質はレトロウイルスやレトロトランスポゾンの複製を抑制する宿主防御タンパク質である1).ヒトでは,第22染色体に7種類(A,B,C,DE,F,G,H)がコードされ(図1a),リンパ球やマクロファージなどの細胞に発現している2).これらは共通してシチジン脱アミノ化酵素活性および核酸への結合能をもち,それぞれの抗レトロウイルス作用の機序に寄与している.
一方,レトロウイルスの仲間であるHIV-1(human immunodeficiency virus type-1,ヒト免疫不全ウイルス1型)は進化の過程でウイルス遺伝子vif(viral infectivity factor)を獲得し,感染細胞においてその遺伝子産物Vifを発現することにより,細胞に存在するAPOBEC3を分解し除去する.そのことにより,HIV-1はAPOBEC3による生体防御機構からのがれ生体において増殖することが可能となっている.VifによるAPOBEC3の抗レトロウイルス作用の解除の機序は,Vifがアダプタータンパク質としてAPOBEC3を特異的にユビキチンリガーゼ複合体にリクルートし,そのポリユビキチン化を介しプロテアソーム系による分解を促すことによる3).さらに,その分解はVifとAPOBEC3との結合の特異性により決定されることが知られていた(図1b).
APOBEC3は遺伝進化学的にZ1型ドメイン,Z2型ドメイン,Z3型ドメインのうち1つあるいは2つから構成され(図1a),このうちVifに結合する責任ドメインはZ2型ドメインあるいはZ3型ドメインであることは知られていたが4),APOBEC3とVifとの結合様式,とくに,その構造学的な情報は明らかになっていなかった.抗HIV薬の新規の作用点につながる可能性が高いことから世界的に精力的な研究がなされていたにもかかわらず,多くの研究グループは苦戦を強いられてきた.その一因は,VifおよびAPOBEC3は非常に難溶性のタンパク質であるためタンパク質の発現および精製が非常に困難なことによる.2008年,複数のグループにより,Z1型ドメインであるAPOBEC3GのC末端側ドメインの構造が決定された5-8).しかし,これはVifの結合する責任ドメインでないため,Vifの結合するドメインの構造の解明が求められていた.もちろん筆者らも,長年にわたりこの研究競争に参戦していた.
1.APOBEC3Cの分子構造を決定した
大腸菌を用いた発現系によりAPOBEC3CをGSTとの融合タンパク質として大量発現し精製した.GSTは可溶化を補助する目的でAPOBEC3CのN末端に融合させた.GSTを切断したのち再精製し,純度95%以上のAPOBEC3Cを得た.特筆すべきは,可溶化および濃縮方法の最条件を見い出すために多大な時間を費やしたことである.精製したAPOBEC3Cをハンギングドロップ蒸気拡散法により結晶化し,X線回折データを収集,解析し,分子構造を決定した.まず,3.00Åの分解能の構造(PDB ID:3VM8),ひきつづき,2.15Åの高分解能の構造(PDB ID:3VOW )を決定することに成功した.APOBEC3Cはすでに報告されていたAPOBEC3GのC末端側ドメイン8)(PDB ID:3IR2)と同様に,酵素活性中心にZn2+を配位した6つのαヘリックスと5つのβストランドからなるコア構造を形成していた(図2a).対照的に,それぞれのループ領域はAPOBEC3GのC末端側ドメインと異なっており,Vifへの結合能など生化学的な特性の違いに寄与していることが推測された.興味深いことに,このAPOBEC3Cのループ領域の構造はAPOBEC3GのC末端側ドメインとは異なっていたものの,核酸基質を認識するために重要なアミノ酸残基の位置は類似していた.
2.Vifとの結合に重要なAPOBEC3Cのアミノ酸残基を同定した
Vifに依存的なAPOBEC3Cの細胞内分解はAPOBEC3CとVifとの結合が前提になる.そこで,Vifとの結合に関与するAPOBEC3Cのアミノ酸残基を同定するため,Vifに依存的な分解の起こらない(Vif抵抗性)変異体を探索した.その際,アミノ酸配列のレベルではなくタンパク質の実構造情報をもとに変異体の解析を行う構造情報にもとづく変異解析法(structure-guided mutagenesis)を活用した.まず,決定されたAPOBEC3Cの分子構造をもとに分子表面に存在する残基を網羅的に抽出し点変異を導入した.作製したAPOBEC3C変異体をVifとともに293T細胞において共発現し,細胞におけるAPOBEC3Cの量をウェスタンブロット法により解析した.野生型のAPOBEC3CのようにVifに依存して完全に分解され消失するAPOBEC3C変異体をVif感受性と名づけた.これら一連の解析は,分子表面においてVif抵抗性を付与する残基が感受性を付与する残基によりおおわれるまでくり返し行った.最終的に99個のAPOBEC3C変異体を作製し,Vifに依存的な分解に関与する10個のアミノ酸残基を同定した.また,ウェスタンブロット法による解析の結果からAPOBEC3Cを定量しVifによる分解率を求めた.それぞれの残基への変異導入においてAPOBEC3Cの分解率を“Vif抵抗性レベル”とし,Vifの結合に関与するレベルに近似するものとして数値化した.ちなみに,共免疫沈降実験により,同定された10個の残基はVifとの結合において実質的にも重要であることが確認された.以上の結果から,これら10個の残基により構成される領域がAPOBEC3CにおけるVif結合インターフェイスであることが示された.また,これら10個の残基おのおののAPOBEC3C変異体は野生型のAPOBEC3Cと同様にウイルス粒子に取り込まれたことから,10個の残基は核酸との結合に関与する領域ではないと考えられた.
Vif抵抗性レベルを分子構造にマッピングし,Vif結合インターフェイスの構造学的な特性を詳細に解析した.その結果,Vif結合インターフェイスはタンパク質の内部の疎水性アミノ酸残基あるいは芳香族アミノ酸残基と,タンパク質の表面の負電荷アミノ酸残基からなる浅い“くぼみ”を形成していることが明らかになった(図2b).くぼみの底部には2つのπ結合が想定され,これらはVif結合インターフェイスの構造の維持に寄与していると考えられた.一方,くぼみのふちの一部の温度因子は高く,柔軟な構造をとる可能性のあることもわかった.このことは分子動力学的な構造解析によっても検証された.柔軟性のあるふちの構造は,Vifとの結合により変化し最終的により強い結合を補助する可能性もあり,一部に誘導適合(induced-fit)モデルがあてはまるのではないかと考えている.
3.Vif結合インターフェイスはほかのAPOBEC3ファミリータンパク質でも保存されていた
APOBEC3Cと配列相同性が高いため類似したVif結合様式をもつと考えられているAPOBEC3FとAPOBEC3DEにおいて,APOBEC3Cで同定された10個のアミノ酸残基が実際にVif結合インターフェイスとして機能しているのかどうか検討した.その結果,APOBEC3Cと同様に,APOBEC3FおよびAPOBEC3DEにおいても10個の残基はVifに依存的な分解およびVifとの結合に重要であることが示された.APOBEC3Cの分子構造(PDB ID:3VOW )を鋳型としてAPOBEC3FのC末端側ドメインおよびAPOBEC3DEのC末端側ドメインのホモロジーモデル構造を構築し,同定した10個の残基をマッピングした結果,APOBEC3FとAPOBEC3DEにおいてもVif結合インターフェイスとしてAPOBEC3Cに類似した“くぼみ”を形成していることがわかった.以上のことから,APOBEC3C,APOBEC3F,APOBEC3DEにおけるVif結合インターフェイスは機能的にも構造的にも保存されていることが明らかになった.最後に,ヒトの細胞を用いてHIV-1に対する抗レトロウイルス作用を解析した結果,Vif抵抗性を示すすべてのAPOBEC3F変異体は細胞においてVifが存在しても分解されず,これらの変異体はHIV-1の感染を抑制することが示された.
おわりに
今回,Vifと結合するZ2型ドメインとして,APOBEC3の分子構造をはじめて決定しただけでなく,構造情報にもとづく変異解析法によりAPOBEC3においてVifとの結合に重要な10個のアミノ酸残基を見い出すことができた.実は,一連の研究の途中,米国のグループが別のアプローチにより10個の残基のうち1つの残基を同定し報告した際には9),肝を冷やしたという裏話がある.さて,同定した10個の残基で構成されるVif結合インターフェイスは負電荷を帯びた浅い“くぼみ”を形成することも明らかになった.興味深いことに,Vifの側からAPOBEC3とVifとの結合を考えてみると,Vifにおける責任領域には正電荷を帯びたDRMRモチーフが必須であるという知見から10),APOBEC3C,APOBEC3F,APOBEC3DEとVifとの結合には静電的な力が重要であると思われた.
この研究は,APOBEC3とVifとの結合における構造学的な分子基盤の一翼を明らかにしただけでなく,APOBEC3とVifとのあいだの相互作用を阻害することで宿主防御機構を活用した薬剤の探索に道筋をつけ,新しいAIDS治療薬の開発にむけた動きをさらに加速するものと期待する.
文 献
- Goila-Gaur, R. & Strebel, K.: HIV-1 Vif, APOBEC, and intrinsic immunity. Retrovirology, 5, 51 (2008)[PubMed]
- LaRue, R. S., Andresdottir, V., Blanchard, Y. et al.: Guidelines for naming nonprimate APOBEC3 genes and proteins. J. Virol., 83, 494-497 (2009)[PubMed]
- Yu, X., Yu, Y., Liu, B. et al.: Induction of APOBEC3G ubiquitination and degradation by an HIV-1 Vif-Cul5-SCF complex. Science, 302, 1056-1060 (2003)[PubMed]
- Kitamura, S., Ode, H. & Iwatani, Y.: Structural features of antiviral APOBEC3 proteins are linked to their functional activities. Front. Microbiol., 2, 258 (2011)[PubMed]
- Chen, K. M., Harjes, E., Gross, P. J. et al.: Structure of the DNA deaminase domain of the HIV-1 restriction factor APOBEC3G. Nature, 452, 116-119 (2008)[PubMed]
- Furukawa, A., Nagata, T., Matsugami, A. et al.: Structure, interaction and real-time monitoring of the enzymatic reaction of wild-type APOBEC3G. EMBO J., 28, 440-451 (2009)[PubMed]
- Holden, L. G., Prochnow, C., Chang, Y. P. et al.: Crystal structure of the anti-viral APOBEC3G catalytic domain and functional implications. Nature, 456, 121-124 (2008)[PubMed]
- Shandilya, S. M., Nalam, M. N., Nalivaika, E. A. et al.: Crystal structure of the APOBEC3G catalytic domain reveals potential oligomerization interfaces. Structure, 18, 28-38 (2010)[PubMed]
- Smith, J. L. & Pathak, V. K.: Identification of specific determinants of human APOBEC3F, APOBEC3C, and APOBEC3DE and African green monkey APOBEC3F that interact with HIV-1 Vif. J. Virol., 84, 12599-12608 (2010)[PubMed]
- Russell, R. A. & Pathak, V. K.: Identification of two distinct human immunodeficiency virus type 1 Vif determinants critical for interactions with human APOBEC3G and APOBEC3F. J. Virol., 81, 8201-8210 (2007)[PubMed]
著者プロフィール
略歴:名古屋大学大学院工学研究科博士課程 在学中.
研究テーマ:APOBEC3の分子構造にもとづく抗ウイルス分子機序の解明.
関心事:APOBEC3を活用した新規の薬剤を開発すること.APOBEC3Gの完全長の分子構造を決定すること.
岩谷 靖雅(Yasumasa Iwatani)
名古屋医療センター臨床研究センター 室長.
© 2012 北村紳悟・岩谷靖雅 Licensed under CC 表示 2.1 日本