リボソーム-tmRNA-SmpB-EF-G複合体におけるトランスロケーション反応のクライオ電子顕微鏡による構造解析
山本 紘・David J. F. Ramrath・Christian M. T. Spahn
(ドイツCharité - Universitätsmedizin Berlin,Institute of Medical Physics and Biophysics)
email:山本 紘
DOI: 10.7875/first.author.2012.075
The complex of tmRNA-SmpB and EF-G on translocating ribosomes.
David J. F. Ramrath, Hiroshi Yamamoto, Kristian Rother, Daniela Wittek, Markus Pech, Thorsten Mielke, Justus Loerke, Patrick Scheerer, Pavel Ivanov, Yoshika Teraoka, Olga Shpanchenko, Knud H. Nierhaus, Christian M. T. Spahn
Nature, 485, 526-529 (2012)
tmRNAはtRNA様ドメインとmRNA様ドメインをもつ多機能なRNAである.原核生物の翻訳において,リボソームが終止コドンをもたないmRNAの3’末端で停滞してしまう場合がある.tmRNAはこのようなリボソームをトランストランスレーション反応によりレスキューする.tmRNAのtRNA様ドメインはSmpBと複合体を形成してリボソームに入り込み,未成熟のタンパク質を受け取ったのち翻訳伸長因子EF-Gによるトランスロケーション反応をうけ,mRNAの3’末端からtmRNAのmRNA様ドメインへと翻訳はスイッチして再開する.tmRNAとリボソームとの複合体のクライオ電子顕微鏡による解析については,いくつかの研究室から報告がなされているものの,tRNA様ドメインがどのようにトランスロケーション反応を起こしmRNA様ドメインがどのようにリボソームに挿入されるかは,非常に興味深い問題にもかかわらず解像度の点から長く未解明であった.今回,筆者らは,リボソーム-tmRNA-SmpB-EF-G複合体に抗生物質フシジン酸を作用させることによりこの複合体をトランスロケーション反応中間体の状態で固定し,クライオ電子顕微鏡により8.3Åの解像度にて解析することに成功した.このことにより,停滞したリボソームにおいてmRNAからtmRNAのtRNA様ドメインへの翻訳のスイッチは,tmRNAのトランスロケーション反応のあいだに起こっていることが明らかになった.
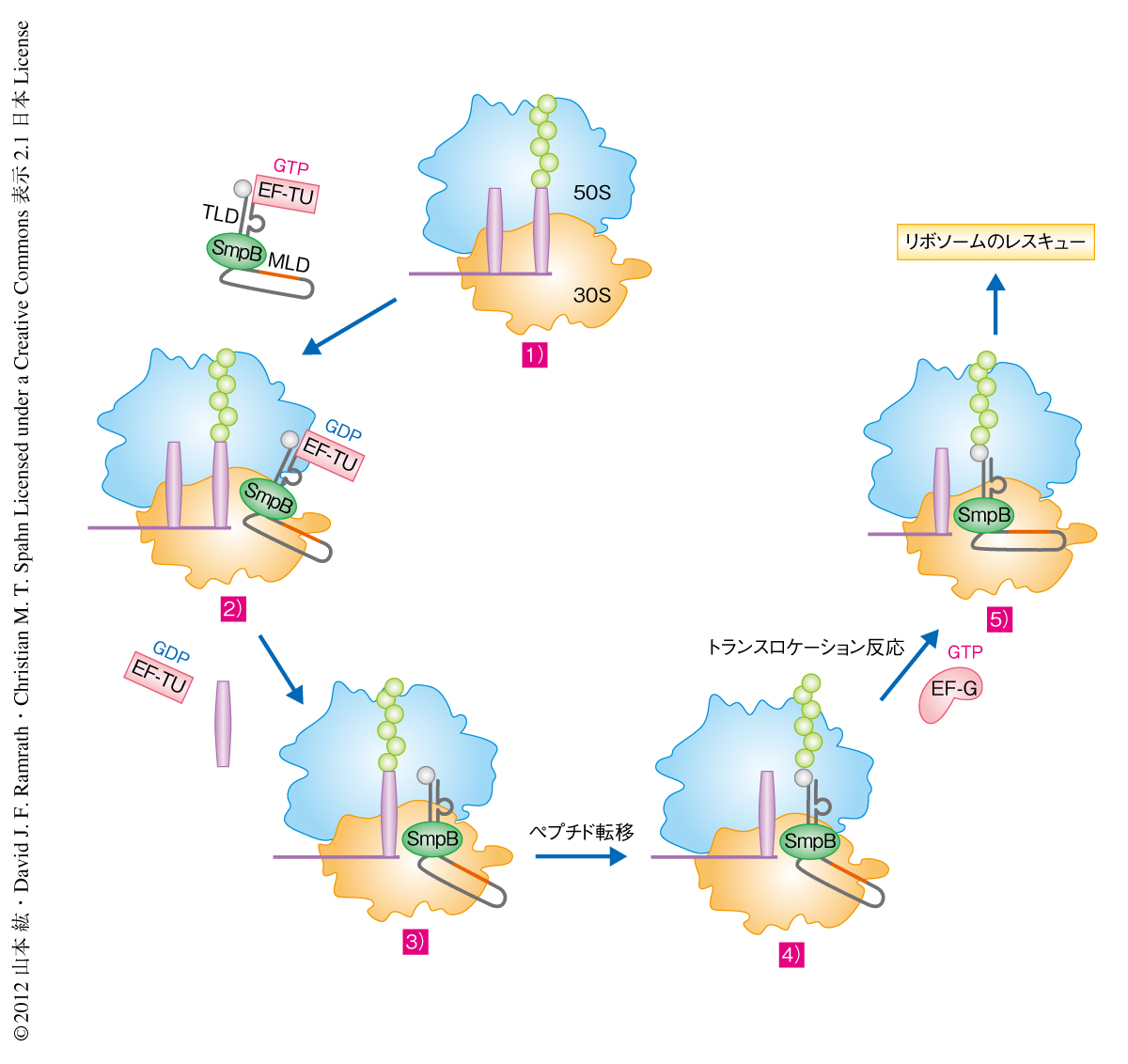
tmRNA(transfer-messenger RNA)とSmpB(small protein B)によるトランストランスレーション反応は,終止コドンをもたないmRNAの3’末端において停滞したリボソームをレスキューする反応である1)(図1).tmRNAは,tRNA様ドメイン,mRNA様ドメイン,そして,互いに離れた塩基との相互作用により形成される4個のシュードノットをもつ,非常に大きなRNAである2).tmRNAの3’末端にはアミノアシルtRNAのようにアラニル-tRNA合成酵素によりアラニンが付加される.SmpBはtmRNAのtRNA様ドメインに結合しtRNAのアンチコドンステムループを擬態している3,4).tmRNAのtRNA様ドメインはSmpBに結合し,翻訳伸長因子EF-Tu-GTPによりリボソームへと入り込む.そののち,未成熟なタンパク質はアコモデーション状態をへてAla-tmRNAにペプチド転移する.この反応につづき,tmRNAのtRNA様ドメインとSmpBとの複合体は,翻訳伸長因子EF-GのもつGTPase活性によりトランスロケーション反応へと進む.これにより,停滞していたリボソームに結合していたmRNAからtmRNAのmRNA様ドメインへと翻訳はスイッチし,新しいアミノアシルtRNAを受け入れるようになる.tmRNAのmRNA様ドメインはプロテアーゼの標的となるタグペプチドをコードしており未成熟なタンパク質の分解を助けている.tmRNAとリボソームとの複合体のクライオ電子顕微鏡による解析がいくつかなされており,tmRNAのtRNA様ドメインとSmpB,EF-Tu-GDPからなる複合体を抗生物質キロマイシンにより固定した構造5),アコモデーション反応ののちの構造6,7),EF-Gによるトランスロケーション反応ののちの構造7),などが示されてきた.しかしながら,クライオ電子顕微鏡による詳細な構造の解析にはナノメートル以下の解像度が必要であるがこれらの解析はそれには及んでいない.さらに,tRNAに比べ5倍も長いtmRNAがどのようにリボソームを通り抜けmRNA様ドメインがどのようにリボソームに導かれるかは未解明であった.
tmRNAがリボソームのうえをどのように移動するのかを解析するため,ペプチド転移反応とトランスロケーション反応との中間にあたる複合体に焦点をあてた.通常,リボソームのトランスロケーション反応はEF-G-GTPとの結合にともなうEF-GのGTPase活性とそののちのEF-G-GDPの解離により達成される.フシジン酸はEF-G-GDPをリボソームに固定しその解離を阻害する抗生物質である.フシジン酸の存在のもと,リボソーム-tmRNA-SmpB-EF-G複合体を調製し,マルチパーティクルクライオ電子顕微鏡法8) により解析したところ,トランスロケーション反応中間体の状態に固定した構造を8.3Åの解像度にて解くことができた(図2a,PDB ID:3J18,3J19).得られた構造はRNAおよびタンパク質の構造をはっきりともっており,これまでに報告されているtRNAfMet,EF-G-GDPとフシジン酸との複合体,tmRNAのtRNA様ドメインとSmpBとの複合体の結晶構造をフィッティングすることができた.また,いままで高い解像度にてモデリングされることのなかったtmRNAの大部分をモデリングすることにも成功した.
得られた構造を通常のリボソームの構造と比較したところ,大きな構造変化が観察された.すなわち,30Sサブユニットの5度の回転,および,30Sサブユニットのヘッド構造の19度の旋回がみられた.この構造変化は,以前に筆者らの研究室で確認された,EF-G-GDPとフシジン酸との複合体にみられたTIPOST(post-translocation-intermediate,トランスロケーション反応中間体)構造に似ていた9).しかしながら興味深いことに,得られた構造においては,新たに12度の傾きも確認された.これら3つの構造変化により,リボソーム30Sサブユニットのヘッド構造は50Åもの移動をひき起こしていた.
この50Åに及ぶリボソーム30Sサブユニットのヘッド構造の移動は,リボソーム50SサブユニットのA部位フィンガーと30Sサブユニットのタンパク質S13とからなるブリッジ1aを開いて20Åのギャップをつくり,その隙間にはtmRNAのシュードノット1が入り込んでいた(図2b).また,tmRNAのヘリックス5とシュードノット2がリボソームタンパク質S3に結合していることが確認された.この結合は,以前に報告されたアコモデーション状態においてすでに確認されているため7),アコモデーション状態からトランスロケーション反応中間体へのtmRNAのアーク構造の移行において,tmRNAはタンパク質S3を回転軸としていることが推測された.また,30SサブユニットのmRNAエントリーチャネルにある,ヘリックス34とG530領域からなるラッチ構造は,トランスロケーション反応中間体において20Åのギャップを生じていることがわかった.このことから,アコモデーション状態からtRNA様ドメインのトランスロケーション反応中間体への移行と同時に,mRNA様ドメインのつけ根にあたるシュードノット1とtmRNAのヘリックス2が,開いたラッチ構造の近傍へと引き込まれていることがわかった.ほかのシュードノットとは対照的に,シュードノット1の削除はtmRNAを不活性化させる10).このことから,シュードノット1のブリッジ1aへの挿入はtmRNAの活性において不可欠な役割を担っているものと考えられた.
tmRNAのmRNA様ドメインの3’側とリボソームタンパク質S3は,トランスロケーション反応ののちEF-Gが離れても結合を保っており,リボソーム30Sサブユニットのヘッド構造に回転はみられない7).このことから,トランスロケーション反応中間体からトランスロケーション反応ののちへの移行の際11),タンパク質S3との結合を保ちつつ,30Sサブユニットのヘッド構造の大きな逆回転,とくに,傾きにより得られるタンパク質S3の20Åの移動により,tmRNAのmRNA様ドメインは開いたラッチ構造をとおってmRNAの通り道へと導かれるものと考えられた.
今回の解析より,tmRNAのtRNA様ドメインのトランスロケーション反応にともなうリボソーム30Sサブユニットの回転,tmRNAのmRNA様ドメインの挿入,の2つの動きは連動していることが明らかになった.クライオ電子顕微鏡による解析技術の向上により,これまでの結晶構造解析によるスナップショットだけでは説明できなかったリボソームの構造変化の詳細をみることができるようになってきた.今後のさらなる技術の発展により,これまで議論できなかったリボソームの機能の詳細にせまれればと考えている.
略歴:2007年 新潟大学大学院自然科学研究科博士課程 修了,同年 新潟薬科大学 研究員,同年 ドイツMax Planck Institute for Molecular Genetics研究員を経て,2010年よりドイツCharité - Universitätsmedizin Berlin研究員.
研究テーマ:リボソームに結合するRNAおよびタンパク質の生化学的な手法を用いた機能解析と,その複合体のクライオ電子顕微鏡による構造解析.
抱負:仮説を説明するために必要なデータ収集の努力を惜しまず,技術をさらに磨いていきたい.
David J. F. Ramrath
ドイツCharité - Universitätsmedizin Berlin研究員.
Christian M. T. Spahn
ドイツCharité - Universitätsmedizin Berlin教授.
© 2012 山本 紘・David J. F. Ramrath・Christian M. T. Spahn Licensed under CC 表示 2.1 日本
(ドイツCharité - Universitätsmedizin Berlin,Institute of Medical Physics and Biophysics)
email:山本 紘
DOI: 10.7875/first.author.2012.075
The complex of tmRNA-SmpB and EF-G on translocating ribosomes.
David J. F. Ramrath, Hiroshi Yamamoto, Kristian Rother, Daniela Wittek, Markus Pech, Thorsten Mielke, Justus Loerke, Patrick Scheerer, Pavel Ivanov, Yoshika Teraoka, Olga Shpanchenko, Knud H. Nierhaus, Christian M. T. Spahn
Nature, 485, 526-529 (2012)
要 約
tmRNAはtRNA様ドメインとmRNA様ドメインをもつ多機能なRNAである.原核生物の翻訳において,リボソームが終止コドンをもたないmRNAの3’末端で停滞してしまう場合がある.tmRNAはこのようなリボソームをトランストランスレーション反応によりレスキューする.tmRNAのtRNA様ドメインはSmpBと複合体を形成してリボソームに入り込み,未成熟のタンパク質を受け取ったのち翻訳伸長因子EF-Gによるトランスロケーション反応をうけ,mRNAの3’末端からtmRNAのmRNA様ドメインへと翻訳はスイッチして再開する.tmRNAとリボソームとの複合体のクライオ電子顕微鏡による解析については,いくつかの研究室から報告がなされているものの,tRNA様ドメインがどのようにトランスロケーション反応を起こしmRNA様ドメインがどのようにリボソームに挿入されるかは,非常に興味深い問題にもかかわらず解像度の点から長く未解明であった.今回,筆者らは,リボソーム-tmRNA-SmpB-EF-G複合体に抗生物質フシジン酸を作用させることによりこの複合体をトランスロケーション反応中間体の状態で固定し,クライオ電子顕微鏡により8.3Åの解像度にて解析することに成功した.このことにより,停滞したリボソームにおいてmRNAからtmRNAのtRNA様ドメインへの翻訳のスイッチは,tmRNAのトランスロケーション反応のあいだに起こっていることが明らかになった.
はじめに
tmRNA(transfer-messenger RNA)とSmpB(small protein B)によるトランストランスレーション反応は,終止コドンをもたないmRNAの3’末端において停滞したリボソームをレスキューする反応である1)(図1).tmRNAは,tRNA様ドメイン,mRNA様ドメイン,そして,互いに離れた塩基との相互作用により形成される4個のシュードノットをもつ,非常に大きなRNAである2).tmRNAの3’末端にはアミノアシルtRNAのようにアラニル-tRNA合成酵素によりアラニンが付加される.SmpBはtmRNAのtRNA様ドメインに結合しtRNAのアンチコドンステムループを擬態している3,4).tmRNAのtRNA様ドメインはSmpBに結合し,翻訳伸長因子EF-Tu-GTPによりリボソームへと入り込む.そののち,未成熟なタンパク質はアコモデーション状態をへてAla-tmRNAにペプチド転移する.この反応につづき,tmRNAのtRNA様ドメインとSmpBとの複合体は,翻訳伸長因子EF-GのもつGTPase活性によりトランスロケーション反応へと進む.これにより,停滞していたリボソームに結合していたmRNAからtmRNAのmRNA様ドメインへと翻訳はスイッチし,新しいアミノアシルtRNAを受け入れるようになる.tmRNAのmRNA様ドメインはプロテアーゼの標的となるタグペプチドをコードしており未成熟なタンパク質の分解を助けている.tmRNAとリボソームとの複合体のクライオ電子顕微鏡による解析がいくつかなされており,tmRNAのtRNA様ドメインとSmpB,EF-Tu-GDPからなる複合体を抗生物質キロマイシンにより固定した構造5),アコモデーション反応ののちの構造6,7),EF-Gによるトランスロケーション反応ののちの構造7),などが示されてきた.しかしながら,クライオ電子顕微鏡による詳細な構造の解析にはナノメートル以下の解像度が必要であるがこれらの解析はそれには及んでいない.さらに,tRNAに比べ5倍も長いtmRNAがどのようにリボソームを通り抜けmRNA様ドメインがどのようにリボソームに導かれるかは未解明であった.
1.リボソーム-tmRNA-SmpB-EF-G複合体には30Sリボソームのヘッド構造に大きな回転が生じる
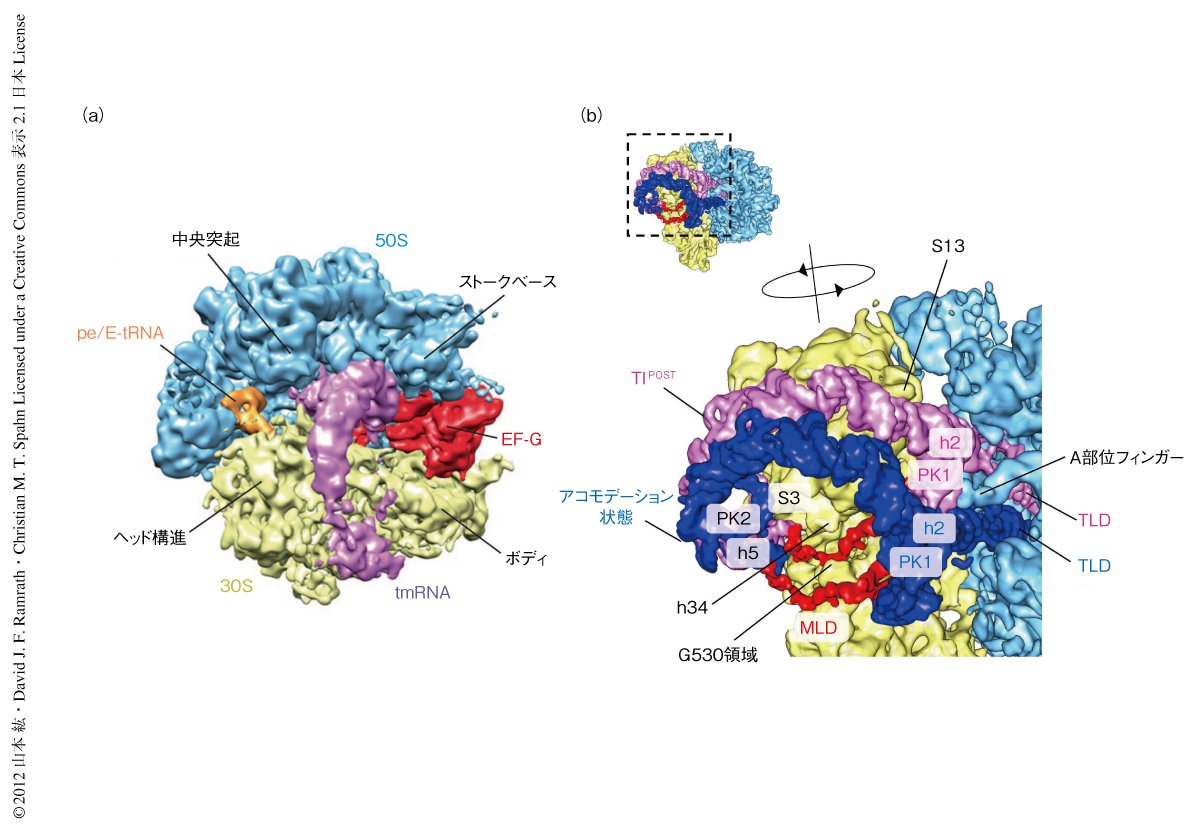
tmRNAがリボソームのうえをどのように移動するのかを解析するため,ペプチド転移反応とトランスロケーション反応との中間にあたる複合体に焦点をあてた.通常,リボソームのトランスロケーション反応はEF-G-GTPとの結合にともなうEF-GのGTPase活性とそののちのEF-G-GDPの解離により達成される.フシジン酸はEF-G-GDPをリボソームに固定しその解離を阻害する抗生物質である.フシジン酸の存在のもと,リボソーム-tmRNA-SmpB-EF-G複合体を調製し,マルチパーティクルクライオ電子顕微鏡法8) により解析したところ,トランスロケーション反応中間体の状態に固定した構造を8.3Åの解像度にて解くことができた(図2a,PDB ID:3J18,3J19).得られた構造はRNAおよびタンパク質の構造をはっきりともっており,これまでに報告されているtRNAfMet,EF-G-GDPとフシジン酸との複合体,tmRNAのtRNA様ドメインとSmpBとの複合体の結晶構造をフィッティングすることができた.また,いままで高い解像度にてモデリングされることのなかったtmRNAの大部分をモデリングすることにも成功した.
得られた構造を通常のリボソームの構造と比較したところ,大きな構造変化が観察された.すなわち,30Sサブユニットの5度の回転,および,30Sサブユニットのヘッド構造の19度の旋回がみられた.この構造変化は,以前に筆者らの研究室で確認された,EF-G-GDPとフシジン酸との複合体にみられたTIPOST(post-translocation-intermediate,トランスロケーション反応中間体)構造に似ていた9).しかしながら興味深いことに,得られた構造においては,新たに12度の傾きも確認された.これら3つの構造変化により,リボソーム30Sサブユニットのヘッド構造は50Åもの移動をひき起こしていた.
2.リボソームに生じた構造変化によりtmRNAのmRNA様ドメインがリボソームに挿入される
この50Åに及ぶリボソーム30Sサブユニットのヘッド構造の移動は,リボソーム50SサブユニットのA部位フィンガーと30Sサブユニットのタンパク質S13とからなるブリッジ1aを開いて20Åのギャップをつくり,その隙間にはtmRNAのシュードノット1が入り込んでいた(図2b).また,tmRNAのヘリックス5とシュードノット2がリボソームタンパク質S3に結合していることが確認された.この結合は,以前に報告されたアコモデーション状態においてすでに確認されているため7),アコモデーション状態からトランスロケーション反応中間体へのtmRNAのアーク構造の移行において,tmRNAはタンパク質S3を回転軸としていることが推測された.また,30SサブユニットのmRNAエントリーチャネルにある,ヘリックス34とG530領域からなるラッチ構造は,トランスロケーション反応中間体において20Åのギャップを生じていることがわかった.このことから,アコモデーション状態からtRNA様ドメインのトランスロケーション反応中間体への移行と同時に,mRNA様ドメインのつけ根にあたるシュードノット1とtmRNAのヘリックス2が,開いたラッチ構造の近傍へと引き込まれていることがわかった.ほかのシュードノットとは対照的に,シュードノット1の削除はtmRNAを不活性化させる10).このことから,シュードノット1のブリッジ1aへの挿入はtmRNAの活性において不可欠な役割を担っているものと考えられた.
tmRNAのmRNA様ドメインの3’側とリボソームタンパク質S3は,トランスロケーション反応ののちEF-Gが離れても結合を保っており,リボソーム30Sサブユニットのヘッド構造に回転はみられない7).このことから,トランスロケーション反応中間体からトランスロケーション反応ののちへの移行の際11),タンパク質S3との結合を保ちつつ,30Sサブユニットのヘッド構造の大きな逆回転,とくに,傾きにより得られるタンパク質S3の20Åの移動により,tmRNAのmRNA様ドメインは開いたラッチ構造をとおってmRNAの通り道へと導かれるものと考えられた.
おわりに
今回の解析より,tmRNAのtRNA様ドメインのトランスロケーション反応にともなうリボソーム30Sサブユニットの回転,tmRNAのmRNA様ドメインの挿入,の2つの動きは連動していることが明らかになった.クライオ電子顕微鏡による解析技術の向上により,これまでの結晶構造解析によるスナップショットだけでは説明できなかったリボソームの構造変化の詳細をみることができるようになってきた.今後のさらなる技術の発展により,これまで議論できなかったリボソームの機能の詳細にせまれればと考えている.
文 献
- Muto, A., Sato, M., Tadaki, T. et al.: Structure and function of 10Sa RNA: trans-translation system. Biochimie, 78, 985-991 (1996)[PubMed]
- Zwieb, C., Wower, I. & Wower, J.: Comparative sequence analysis of tmRNA. Nucleic Acids Res., 27, 2063-2071 (1999)[PubMed]
- Gutmann, S., Haebel, P. W., Metzinger, L. et al.: Crystal structure of the transfer-RNA domain of transfer-messenger RNA in complex with SmpB. Nature, 424, 699-703 (2003)[PubMed]
- Bessho, Y., Shibata, R., Sekine, S. et al.: Structural basis for functional mimicry of long-variable-arm tRNA by transfer-messenger RNA. Proc. Natl. Acad. Sci. USA, 104, 8293-8298 (2007)[PubMed]
- Valle, M., Gillet, R., Kaur, S. et al.: Visualizing tmRNA entry into a stalled ribosome. Science, 300, 127-130 (2003)[PubMed]
- Weis, F., Bron, P., Rolland, J. P. et al.: Accommodation of tmRNA-SmpB into stalled ribosomes: a cryo-EM study. RNA, 16, 299-306 (2010)[PubMed]
- Weis, F., Bron, P., Giudice, E. et al.: tmRNA-SmpB: a journey to the centre of the bacterial ribosome. EMBO J., 29, 3810-3818 (2010)[PubMed]
- Loerke, J., Giesebrecht, J. & Spahn, C. M.: Multiparticle cryo-EM of ribosomes. Methods Enzymol., 483, 161-177 (2010)[PubMed]
- Ratje, A. H., Loerke, J., Mikolajka, A. et al.: Head swivel on the ribosome facilitates translocation by means of intra-subunit tRNA hybrid sites. Nature, 468, 713-716 (2010)[PubMed]
- Nameki, N., Tadaki, T., Himeno, H. et al.: Three of four pseudoknots in tmRNA are interchangeable and are substitutable with single-stranded RNAs. FEBS Lett., 470, 345-349 (2000)[PubMed]
- Gao, Y. G., Selmer, M., Dunham, C. M. et al.: The structure of the ribosome with elongation factor G trapped in the posttranslocational state. Science, 326, 694-699 (2009)[PubMed]
著者プロフィール
略歴:2007年 新潟大学大学院自然科学研究科博士課程 修了,同年 新潟薬科大学 研究員,同年 ドイツMax Planck Institute for Molecular Genetics研究員を経て,2010年よりドイツCharité - Universitätsmedizin Berlin研究員.
研究テーマ:リボソームに結合するRNAおよびタンパク質の生化学的な手法を用いた機能解析と,その複合体のクライオ電子顕微鏡による構造解析.
抱負:仮説を説明するために必要なデータ収集の努力を惜しまず,技術をさらに磨いていきたい.
David J. F. Ramrath
ドイツCharité - Universitätsmedizin Berlin研究員.
Christian M. T. Spahn
ドイツCharité - Universitätsmedizin Berlin教授.
© 2012 山本 紘・David J. F. Ramrath・Christian M. T. Spahn Licensed under CC 表示 2.1 日本