ユビキチン-プロテアソーム系を介したタンパク質分解による翻訳制御の恒常性の維持
柳谷 朗子
(カナダMcGill大学Department of Biochemistry)
email:柳谷朗子
DOI: 10.7875/first.author.2012.062
Translational homeostasis via the mRNA cap-binding protein, eIF4E.
Akiko Yanagiya, Eigo Suyama, Hironori Adachi, Yuri V. Svitkin, Pedro Aza-Blanc, Hiroaki Imataka, Satoshi Mikami, Yvan Martineau, Ze'ev A. Ronai, Nahum Sonenberg
Molecular Cell, 46, 847-858 (2012)
さまざまな生命現象における遺伝子の発現に際し翻訳制御は重要な役割を担っており,その恒常性の維持は正常な細胞や個体の存続に必要不可欠である.mRNAのキャップ構造と結合するeIF4Eは翻訳の開始において主要な役割を担っており,その活性は低リン酸化型4E-BPが結合することにより阻害される.筆者らの研究により,翻訳の開始におけるeIF4Eの重要な役割にもかかわらず,HeLa細胞においてeIF4Eをノックダウンしても翻訳の効率は低下しないことが明らかになった.この意外な現象のもとでは,eIF4Eの抑制タンパク質である低リン酸化型4E-BP1がユビキチン-プロテアソーム系により迅速に分解されており,結果として,翻訳制御の恒常性が維持されていることが判明した.さらに,低リン酸化型4E-BP1に対するユビキチンリガーゼとしてKLHL25-CUL3複合体を同定した.
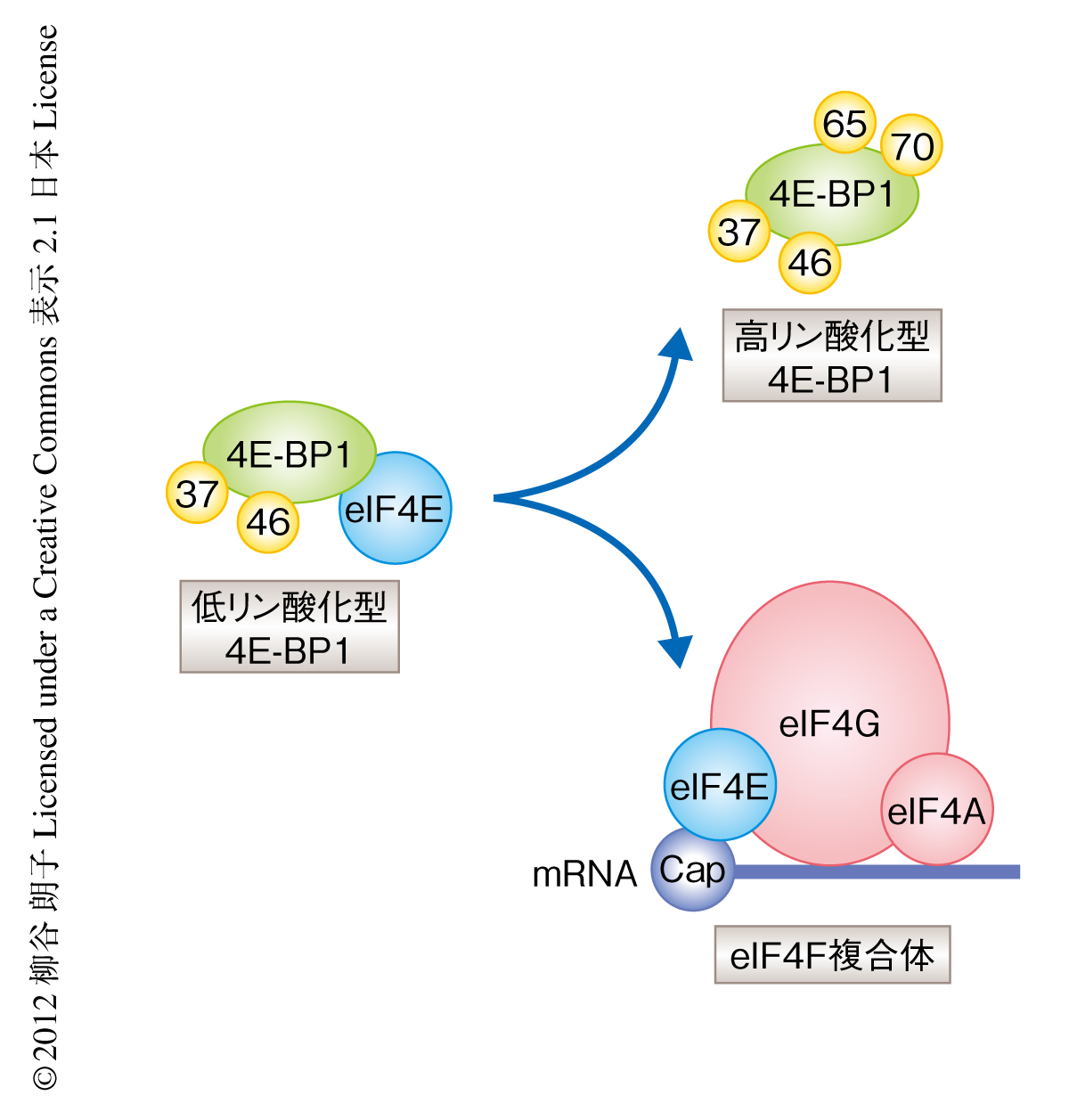
ユビキチン-プロテアソーム系によるタンパク質分解はさまざまな生命現象を制御しており,転写制御などによる遺伝子発現の制御に比べ,最終的な遺伝子産物であるタンパク質の迅速な制御が可能である.近年,リボソームの構成タンパク質1) や翻訳にかかわるタンパク質2,3) がユビキチン-プロテアソーム系により制御されていることが報告されつつあるが,この系が翻訳制御にどのように関与しているかについてはまだ解明されていない.翻訳開始因子のひとつeIF4E(eukaryotic translation initiation factor 4E)はmRNAの5’末端に存在するキャップ構造と結合し,ほかの翻訳開始因子であるeIF4G,eIF4AとともにeIF4F複合体を形成する.4E-BP(eIF4E-binding protein,eIF4E結合タンパク質)はeIF4Eと結合することによりeIF4F複合体の形成を阻害する.4E-BPのeIF4Eに対する結合能はプロテインキナーゼであるmTORによるリン酸化により負に制御されており,低リン酸化型4E-BPのみがeIF4Eと結合し,高リン酸化型4E-BPはeIF4Eから解離し翻訳は活性化される4)(図1).この研究では,翻訳開始因子において中心的な存在であるeIF4Eとその抑制タンパク質である低リン酸化型4E-BP1の発現量がユビキチン-プロテアソーム系を介するタンパク質分解により相対的に制御され,その結果として,翻訳制御の恒常性が保たれる分子機構を解明した.さらに,低リン酸化型4E-BP1に対するユビキチンリガーゼとしてKLHL25-CUL3複合体を同定した.
主要な翻訳開始因子であるeIF4EをノックダウンしたHeLa細胞を樹立し,翻訳の効率にあたえる影響を[35S]メチオニン標識により検討した.その結果,eIF4Eの発現量は減少しているのにもかかわらず,翻訳の効率は低下しないことが明らかになった.つぎに,eIF4Eノックダウン細胞におけるeIF4E以外の翻訳にかかわるタンパク質,eIF4G,eIF4A,4E-BP1,ポリA結合タンパク質(PABP)のタンパク質量をウェスタンブロット法により検討した.興味深いことに,eIF4G,eIF4A,PABP,高リン酸化型4E-BP1の発現量に変化はなかったが,低リン酸化型4E-BP1の発現量がeIF4Eノックダウン細胞において減少していることが明らかになった.
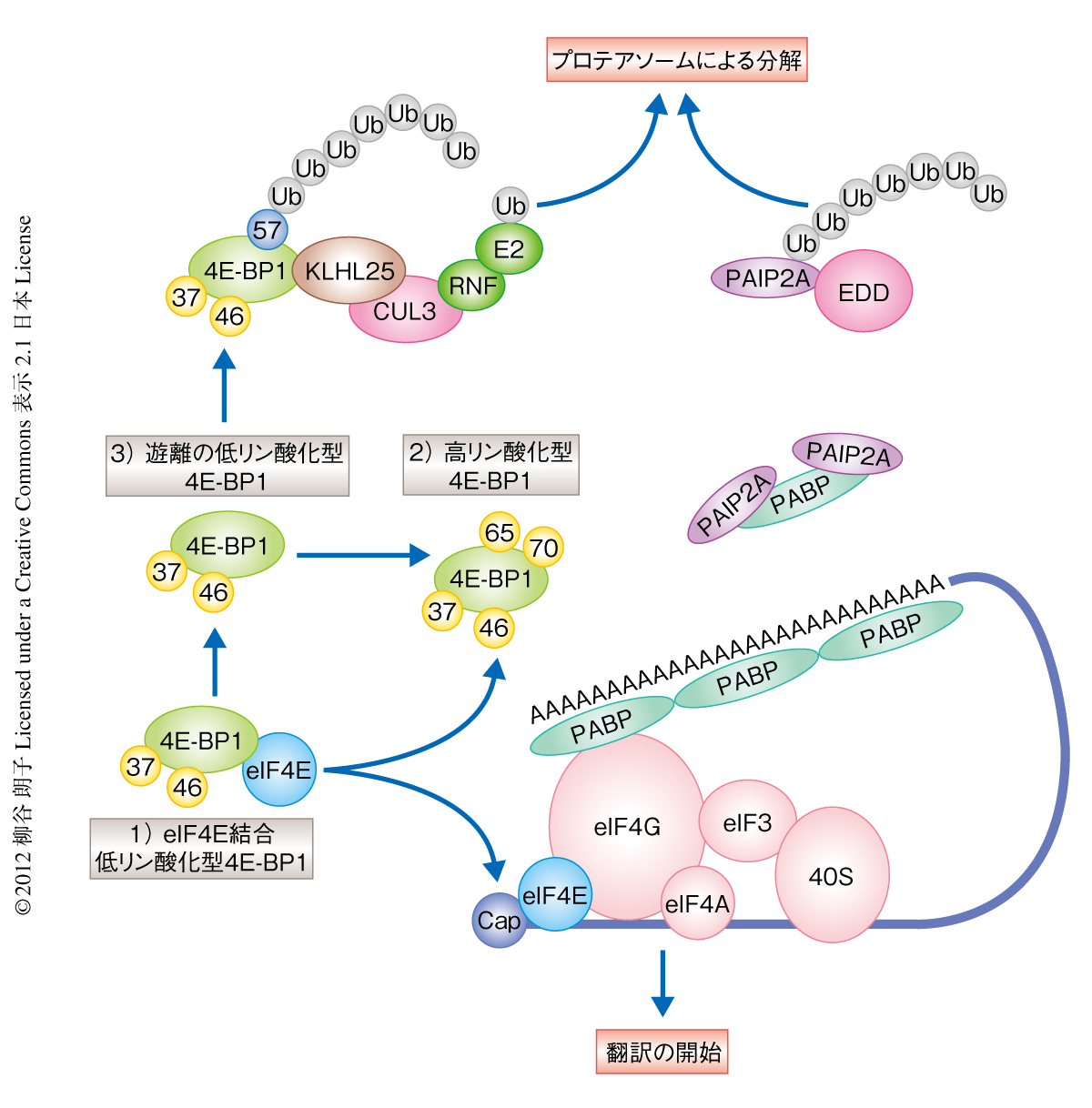
ノーザンブロット法によりeIF4Eノックダウン細胞における4E-BP1遺伝子のmRNAの発現量を検討したところ対照細胞と同じであったことから,eIF4Eノックダウン細胞における低リン酸化型4E-BP1の減少は転写後の制御によるものであることが示唆された.eIF4Eノックダウン細胞をMG132やラクタシスチンといったプロテアソーム阻害剤により処理すると低リン酸化型4E-BP1の発現量は回復した.このことから,eIF4Eの発現量の減少している条件では,低リン酸化型4E-BP1はプロテアソームにより分解されていることが明らかになった.また,細胞をmTORの選択的な阻害剤であるPP242で処理して4E-BP1を脱リン酸化するとそのユビキチン化が促進されたことから,4E-BP1のリン酸化の状態はそのユビキチン化に影響することが判明した.さらに,野生型eIF4Eまたは4E-BP1と結合できない変異型eIF4Eを過剰発現させた細胞をPP242で処理したところ,野生型eIF4Eは低リン酸化型4E-BP1のユビキチン化を阻害したのに対し,変異型eIF4Eはなんの影響もあたえなかった.これらの結果から,遊離の4E-BP1はユビキチン化されやすく,eIF4Eと結合した低リン酸化型4E-BP1はユビキチン化からまぬがれているものと考えられた(図2).
非リン酸化4E-BP1変異体を安定して発現するeIF4Eノックダウン細胞と,ユビキチンリガーゼの構成タンパク質に対するsiRNAライブラリーとを用いたスクリーニングにより,KLHL25とCUL3が同定された.CUL3はBTBドメインをもつタンパク質と複合体を形成しユビキチンリガーゼとしてはたらくことが知られており,このBTBドメインをもつタンパク質が基質を認識することでユビキチン化が起こる5-8).興味深いことに,CUL3とともに同定されたKLHL25はN末端にBTBドメインをもっていた.そこで,このKLHL25がユビキチンリガーゼ複合体の基質認識アダプターとして機能するかどうかを免疫沈降法により解析した.その結果,KLHL25は低リン酸化型4E-BP1とCUL3の両方と結合することが確かめられた.さらに,in vitroユビキチン化アッセイによりKLHL25とCUL3との共発現は低リン酸化型4E-BP1のユビキチン化を促進したことから,KLHL25とCUL3との複合体が低リン酸化型4E-BP1に対するユビキチンリガーゼとして機能することが明らかになった.さらに,eIF4EとKLHL25をともにノックダウンすると低リン酸化型4E-BP1の発現量が回復し,その結果として,翻訳の阻害が認められた.
4E-BP1には57番目,69番目,105番目の3つのリジン残基が存在し,これらのうち,57番目のリジン残基はeIF4Eとの結合部位に存在し,また,さまざまな種において保存されている.これら3つのリジン残基のどれにユビキチンが結合するのかを検討するため,リジン残基をアルギニン残基へと置換した変異体を作製しin vitroユビキチン化アッセイを行った.その結果,57番目のリジン残基の変異体においてユビキチン化が阻害されたことから,ユビキチンはKLHL25-CUL3複合体によりこのリジン残基に付加されることが明らかになった.さらに,57番目のリジン残基に変異をもつ4E-BP1を過剰発現させたeIF4Eノックダウン細胞で翻訳が阻害されたので,このリジン残基へのユビキチンの結合が4E-BP1の分解において重要であることが示唆された.
以上の実験結果から,遊離の低リン酸化型4E-BP1はユビキチンリガーゼKLHL25-CUL3複合体を介しユビキチン-プロテアソーム系により分解されることが明らかになった.eIF4Eと結合した低リン酸化型4E-BP1が安定であるのは,eIF4Eとの結合により4E-BP1の57番目のリジン残基がマスクされ9),KLHL25-CUL3複合体がこのリジン残基にユビキチンを結合できないことが原因であるものと予測された.また,4E-BP1の70番目のスレオニン残基と65番目のセリン残基におけるリン酸化はKLHL25-CUL3複合体によるユビキチン化を阻害したが,これらのリン酸化がeIF4Eと4E-BP1との結合をさまたげるのと同様に,高リン酸化型4E-BP1とKLHL25-CUL3複合体との結合を阻んでいる可能性が考えられた.
この研究では,翻訳開始において中心的な存在であるeIF4Eとその阻害タンパク質である低リン酸化型4E-BP1のタンパク質量がユビキチン-プロテアソーム系により制御されていることを実証した.同様に,mRNAの安定性や翻訳の効率を制御するPABPをノックダウンした場合にも,その抑制タンパク質であるPAIP2AがユビキチンリガーゼEDDを介しユビキチン-プロテアソーム系により分解され翻訳制御の恒常性は維持される2)(図2).翻訳にかかわるタンパク質とその阻害タンパク質としてeIF4Eと4E-BP1,または,PABPとPAIP2A以外の組合せでも,ユビキチン-プロテアソーム系を介しそれらのタンパク質量が相対的に制御されている可能性があり,翻訳制御におけるユビキチン-プロテアソーム系の役割を解明する今後の研究が期待される.
多くのがん組織または培養がん細胞においてeIF4Eは過剰発現しており,新たな抗がん剤として,現在,eIF4Eアンチセンスが臨床試験において検討されている.筆者らの研究により,eIF4Eをノックダウンしても,その抑制タンパク質である低リン酸化型4E-BP1がユビキチン-プロテアソーム系により分解されてしまい,がん細胞においてeIF4Eの活性を十分に阻害できない可能性が示唆された.現在,eIF4Eアンチセンスとプロテアソーム阻害剤であるVelcade(Bortezomib)との併用により,効果的にeIF4Eの活性を阻害し,より強い抗がん作用を発揮できるのか検討している.
略歴:東京大学大学院薬学系研究科 修了,カナダMcGill大学Research Associate.
関心事:さまざまな生命現象を翻訳制御のレベルにおいて解析すること.
© 2012 柳谷 朗子 Licensed under CC 表示 2.1 日本
(カナダMcGill大学Department of Biochemistry)
email:柳谷朗子
DOI: 10.7875/first.author.2012.062
Translational homeostasis via the mRNA cap-binding protein, eIF4E.
Akiko Yanagiya, Eigo Suyama, Hironori Adachi, Yuri V. Svitkin, Pedro Aza-Blanc, Hiroaki Imataka, Satoshi Mikami, Yvan Martineau, Ze'ev A. Ronai, Nahum Sonenberg
Molecular Cell, 46, 847-858 (2012)
この論文に出現する遺伝子・タンパク質のUniprot ID
ユビキチン, プロテアソーム, eIF4E(P06730), 4E-BP, 4E-BP1(Q13541), ユビキチンリガーゼ, KLHL25(Q9H0H3), CUL3(Q13618), eukaryotic translation initiation factor 4E(P06730), eIF4G, eIF4A, eIF4F, eIF4E-binding protein, eIF4E結合タンパク質, mTOR(P42345), ポリA結合タンパク質, PABP, PAIP2A(Q9BPZ3), ユビキチンリガーゼEDD(O95071), EDD(O95071), Ub
要 約
さまざまな生命現象における遺伝子の発現に際し翻訳制御は重要な役割を担っており,その恒常性の維持は正常な細胞や個体の存続に必要不可欠である.mRNAのキャップ構造と結合するeIF4Eは翻訳の開始において主要な役割を担っており,その活性は低リン酸化型4E-BPが結合することにより阻害される.筆者らの研究により,翻訳の開始におけるeIF4Eの重要な役割にもかかわらず,HeLa細胞においてeIF4Eをノックダウンしても翻訳の効率は低下しないことが明らかになった.この意外な現象のもとでは,eIF4Eの抑制タンパク質である低リン酸化型4E-BP1がユビキチン-プロテアソーム系により迅速に分解されており,結果として,翻訳制御の恒常性が維持されていることが判明した.さらに,低リン酸化型4E-BP1に対するユビキチンリガーゼとしてKLHL25-CUL3複合体を同定した.
はじめに
ユビキチン-プロテアソーム系によるタンパク質分解はさまざまな生命現象を制御しており,転写制御などによる遺伝子発現の制御に比べ,最終的な遺伝子産物であるタンパク質の迅速な制御が可能である.近年,リボソームの構成タンパク質1) や翻訳にかかわるタンパク質2,3) がユビキチン-プロテアソーム系により制御されていることが報告されつつあるが,この系が翻訳制御にどのように関与しているかについてはまだ解明されていない.翻訳開始因子のひとつeIF4E(eukaryotic translation initiation factor 4E)はmRNAの5’末端に存在するキャップ構造と結合し,ほかの翻訳開始因子であるeIF4G,eIF4AとともにeIF4F複合体を形成する.4E-BP(eIF4E-binding protein,eIF4E結合タンパク質)はeIF4Eと結合することによりeIF4F複合体の形成を阻害する.4E-BPのeIF4Eに対する結合能はプロテインキナーゼであるmTORによるリン酸化により負に制御されており,低リン酸化型4E-BPのみがeIF4Eと結合し,高リン酸化型4E-BPはeIF4Eから解離し翻訳は活性化される4)(図1).この研究では,翻訳開始因子において中心的な存在であるeIF4Eとその抑制タンパク質である低リン酸化型4E-BP1の発現量がユビキチン-プロテアソーム系を介するタンパク質分解により相対的に制御され,その結果として,翻訳制御の恒常性が保たれる分子機構を解明した.さらに,低リン酸化型4E-BP1に対するユビキチンリガーゼとしてKLHL25-CUL3複合体を同定した.
1.eIF4Eをノックダウンしても翻訳の効率は低下しない
主要な翻訳開始因子であるeIF4EをノックダウンしたHeLa細胞を樹立し,翻訳の効率にあたえる影響を[35S]メチオニン標識により検討した.その結果,eIF4Eの発現量は減少しているのにもかかわらず,翻訳の効率は低下しないことが明らかになった.つぎに,eIF4Eノックダウン細胞におけるeIF4E以外の翻訳にかかわるタンパク質,eIF4G,eIF4A,4E-BP1,ポリA結合タンパク質(PABP)のタンパク質量をウェスタンブロット法により検討した.興味深いことに,eIF4G,eIF4A,PABP,高リン酸化型4E-BP1の発現量に変化はなかったが,低リン酸化型4E-BP1の発現量がeIF4Eノックダウン細胞において減少していることが明らかになった.
2.低リン酸化型4E-BP1はeIF4Eノックダウン細胞において分解されている
ノーザンブロット法によりeIF4Eノックダウン細胞における4E-BP1遺伝子のmRNAの発現量を検討したところ対照細胞と同じであったことから,eIF4Eノックダウン細胞における低リン酸化型4E-BP1の減少は転写後の制御によるものであることが示唆された.eIF4Eノックダウン細胞をMG132やラクタシスチンといったプロテアソーム阻害剤により処理すると低リン酸化型4E-BP1の発現量は回復した.このことから,eIF4Eの発現量の減少している条件では,低リン酸化型4E-BP1はプロテアソームにより分解されていることが明らかになった.また,細胞をmTORの選択的な阻害剤であるPP242で処理して4E-BP1を脱リン酸化するとそのユビキチン化が促進されたことから,4E-BP1のリン酸化の状態はそのユビキチン化に影響することが判明した.さらに,野生型eIF4Eまたは4E-BP1と結合できない変異型eIF4Eを過剰発現させた細胞をPP242で処理したところ,野生型eIF4Eは低リン酸化型4E-BP1のユビキチン化を阻害したのに対し,変異型eIF4Eはなんの影響もあたえなかった.これらの結果から,遊離の4E-BP1はユビキチン化されやすく,eIF4Eと結合した低リン酸化型4E-BP1はユビキチン化からまぬがれているものと考えられた(図2).
3.KLHL25-CUL3複合体により遊離の低リン酸化型4E-BP1はユビキチン化される
非リン酸化4E-BP1変異体を安定して発現するeIF4Eノックダウン細胞と,ユビキチンリガーゼの構成タンパク質に対するsiRNAライブラリーとを用いたスクリーニングにより,KLHL25とCUL3が同定された.CUL3はBTBドメインをもつタンパク質と複合体を形成しユビキチンリガーゼとしてはたらくことが知られており,このBTBドメインをもつタンパク質が基質を認識することでユビキチン化が起こる5-8).興味深いことに,CUL3とともに同定されたKLHL25はN末端にBTBドメインをもっていた.そこで,このKLHL25がユビキチンリガーゼ複合体の基質認識アダプターとして機能するかどうかを免疫沈降法により解析した.その結果,KLHL25は低リン酸化型4E-BP1とCUL3の両方と結合することが確かめられた.さらに,in vitroユビキチン化アッセイによりKLHL25とCUL3との共発現は低リン酸化型4E-BP1のユビキチン化を促進したことから,KLHL25とCUL3との複合体が低リン酸化型4E-BP1に対するユビキチンリガーゼとして機能することが明らかになった.さらに,eIF4EとKLHL25をともにノックダウンすると低リン酸化型4E-BP1の発現量が回復し,その結果として,翻訳の阻害が認められた.
4.4E-BP1の57番目のリジン残基がKLHL25-CUL3複合体によりユビキチン化される
4E-BP1には57番目,69番目,105番目の3つのリジン残基が存在し,これらのうち,57番目のリジン残基はeIF4Eとの結合部位に存在し,また,さまざまな種において保存されている.これら3つのリジン残基のどれにユビキチンが結合するのかを検討するため,リジン残基をアルギニン残基へと置換した変異体を作製しin vitroユビキチン化アッセイを行った.その結果,57番目のリジン残基の変異体においてユビキチン化が阻害されたことから,ユビキチンはKLHL25-CUL3複合体によりこのリジン残基に付加されることが明らかになった.さらに,57番目のリジン残基に変異をもつ4E-BP1を過剰発現させたeIF4Eノックダウン細胞で翻訳が阻害されたので,このリジン残基へのユビキチンの結合が4E-BP1の分解において重要であることが示唆された.
おわりに
以上の実験結果から,遊離の低リン酸化型4E-BP1はユビキチンリガーゼKLHL25-CUL3複合体を介しユビキチン-プロテアソーム系により分解されることが明らかになった.eIF4Eと結合した低リン酸化型4E-BP1が安定であるのは,eIF4Eとの結合により4E-BP1の57番目のリジン残基がマスクされ9),KLHL25-CUL3複合体がこのリジン残基にユビキチンを結合できないことが原因であるものと予測された.また,4E-BP1の70番目のスレオニン残基と65番目のセリン残基におけるリン酸化はKLHL25-CUL3複合体によるユビキチン化を阻害したが,これらのリン酸化がeIF4Eと4E-BP1との結合をさまたげるのと同様に,高リン酸化型4E-BP1とKLHL25-CUL3複合体との結合を阻んでいる可能性が考えられた.
この研究では,翻訳開始において中心的な存在であるeIF4Eとその阻害タンパク質である低リン酸化型4E-BP1のタンパク質量がユビキチン-プロテアソーム系により制御されていることを実証した.同様に,mRNAの安定性や翻訳の効率を制御するPABPをノックダウンした場合にも,その抑制タンパク質であるPAIP2AがユビキチンリガーゼEDDを介しユビキチン-プロテアソーム系により分解され翻訳制御の恒常性は維持される2)(図2).翻訳にかかわるタンパク質とその阻害タンパク質としてeIF4Eと4E-BP1,または,PABPとPAIP2A以外の組合せでも,ユビキチン-プロテアソーム系を介しそれらのタンパク質量が相対的に制御されている可能性があり,翻訳制御におけるユビキチン-プロテアソーム系の役割を解明する今後の研究が期待される.
多くのがん組織または培養がん細胞においてeIF4Eは過剰発現しており,新たな抗がん剤として,現在,eIF4Eアンチセンスが臨床試験において検討されている.筆者らの研究により,eIF4Eをノックダウンしても,その抑制タンパク質である低リン酸化型4E-BP1がユビキチン-プロテアソーム系により分解されてしまい,がん細胞においてeIF4Eの活性を十分に阻害できない可能性が示唆された.現在,eIF4Eアンチセンスとプロテアソーム阻害剤であるVelcade(Bortezomib)との併用により,効果的にeIF4Eの活性を阻害し,より強い抗がん作用を発揮できるのか検討している.
文 献
- Spence, J., Gali, R. R., Dittmar, G. et al.: Cell cycle-regulated modification of the ribosome by a variant multiubiquitin chain. Cell, 102, 67-76 (2000)[PubMed]
- Yoshida, M., Yoshida, K., Kozlov, G. et al.: Poly(A) binding protein (PABP) homeostasis is mediated by the stability of its inhibitor, Paip2. EMBO J., 25, 1934-1944 (2006)[PubMed]
- Murata, T. & Shimotohno, K.: Ubiquitination and proteasome-dependent degradation of human eukaryotic translation initiation factor 4E. J. Biol. Chem., 281, 20788-20800 (2006)[PubMed]
- Gingras, A. C., Raught, B., Gygi, S. P. et al.: Hierarchical phosphorylation of the translation inhibitor 4E-BP1. Genes. Dev., 15, 2852-2864 (2001)[PubMed]
- Furukawa, M., He, Y. J., Borchers, C. et al.: Targeting of protein ubiquitination by BTB-Cullin 3-Roc1 ubiquitin ligases. Nat. Cell Biol., 5, 1001-1007 (2003)[PubMed]
- Geyer, R., Wee, S., Anderson, S. et al.: BTB/POZ domain proteins are putative substrate adaptors for cullin 3 ubiquitin ligases. Mol. Cell, 12, 783-790 (2003)[PubMed]
- Pintard, L., Willis, J. H., Willems, A. et al.: The BTB protein MEL-26 is a substrate-specific adaptor of the CUL-3 ubiquitin-ligase. Nature, 425, 311-316 (2003)[PubMed]
- Xu, L., Wei, Y., Reboul, J. et al.: BTB proteins are substrate-specific adaptors in an SCF-like modular ubiquitin ligase containing CUL-3. Nature, 425, 316-321 (2003)[PubMed]
- Gross, J. D., Moerke, N. J., von der Haar, T. et al.: Ribosome loading onto the mRNA cap is driven by conformational coupling between eIF4G and eIF4E. Cell, 115, 739-750 (2003)[PubMed]
著者プロフィール
略歴:東京大学大学院薬学系研究科 修了,カナダMcGill大学Research Associate.
関心事:さまざまな生命現象を翻訳制御のレベルにおいて解析すること.
© 2012 柳谷 朗子 Licensed under CC 表示 2.1 日本