tRNAをアグマチンで化学修飾するしくみ
大澤拓生・沼田倫征
(産業技術総合研究所バイオメディカル研究部門 RNAプロセシング研究グループ)
email:大澤拓生,沼田倫征
DOI: 10.7875/first.author.2011.165
Structural basis of tRNA agmatinylation essential for AUA codon decoding.
Takuo Osawa, Satoshi Kimura, Naohiro Terasaka, Hideko Inanaga, Tsutomu Suzuki, Tomoyuki Numata
Nature Structural & Molecular Biology, 18, 1275-1280 (2011)
タンパク質を正確に生合成するためには,リボソームにおいてtRNAが適正なコドンを選択しなければならない.tRNAのアンチコドン領域に存在する修飾ヌクレオシドは正しいコドン認識を可能にするという重要な役割を担っている.古細菌において,イソロイシンコドンのひとつであるAUAコドンを解読するtRNAIle2では,アンチコドン1文字目のシチジンがポリアミンの一種であるアグマチンにより修飾され2-アグマチニルシチジンへと変換されている.また,酵素TiaSはATP依存的に2-アグマチニルシチジンを合成することが知られている.今回,筆者らは,ATPもしくはAMPCPPおよびアグマチンと結合したTiaS-tRNAIle2複合体の結晶構造を解析した.その結果,アミノ酸配列の情報だけでは不明であった新規のATP結合ドメインを同定した.TiaS-tRNAIle2-ATP複合体において,アンチコドン1文字目のシチジンはATP結合部位から隔離されたポケットに結合していた.一方,TiaS-tRNAIle2-AMPCPP-アグマチン複合体ではそのポケットがアグマチンで占有され,アンチコドン1文字目のシチジンはAMPCPPのγ-リン酸の近傍に配置されていた.したがって,TiaSはATPを利用してアンチコドン1文字目のシチジンをリン酸化しいったん活性化すること,そののち,アグマチンがこのリン酸化反応中間体を求核攻撃することで2-アグマチニルシチジンが産生されることが示唆された.この反応機構は関連する生化学的な実験からも支持された.以上の結果は,TiaSへのアグマチンの結合がアンチコドン1文字目のシチジンのリン酸化反応に不可欠であることを示唆するとともに,TiaSには反応を効率よく触媒するしくみが存在することを示した.
遺伝情報の発現の過程においてmRNAのコドンはtRNAを介し対応するアミノ酸へと解読される.つまり,tRNAはコドンというヌクレオチド配列の情報をアミノ酸配列の情報へと変換するためのアダプターとして機能している.したがって,遺伝情報を正確に発現させるためには,tRNAのアンチコドンが対応するコドンのみを忠実に認識しなければならない.tRNAによるコドンの認識はコドン-アンチコドン塩基対の形成により遂行される.その際,tRNAのアンチコドン領域に存在する修飾ヌクレオシドが正確なコドンの認識に重要な役割をはたしている.とくに,アンチコドン1文字目に存在する修飾ヌクレオシドはコドンの第3塩基と対合することから,コドンの縮重(ゆらぎ)と密接にかかわっている.
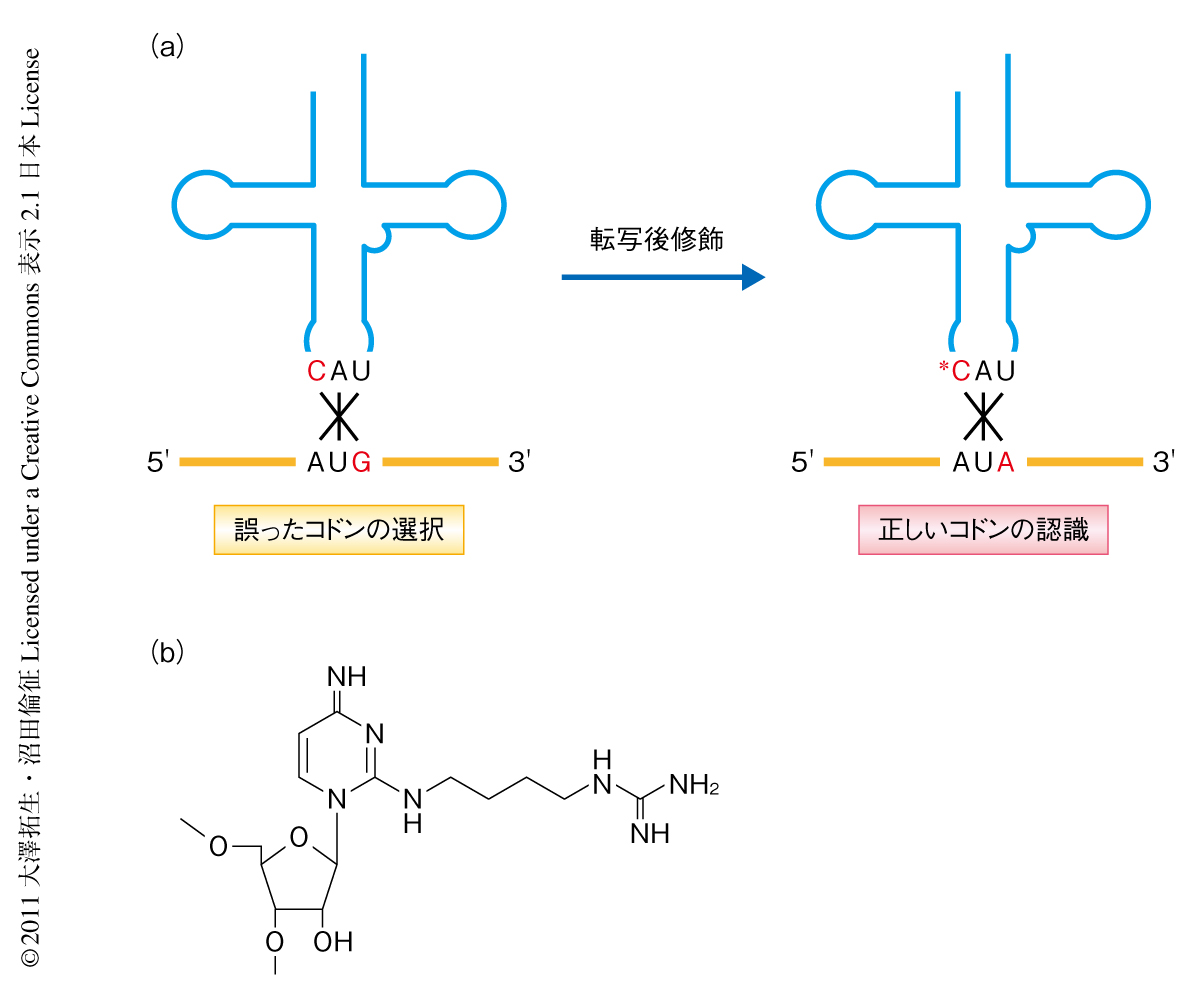
原核生物にはイソロイシンのコドン(AUU,AUC,AUA)に対応する2種類のtRNA,tRNAIle1とtRNAIle2が存在する.このうち,AUAコドンを解読するtRNAIle2のアンチコドン配列はCAUであり,メチオニンのコドン(AUG)を解読するtRNAMetのアンチコドン配列(CAU)と同じである.このため,修飾をうけていないtRNAIle2はメチオニンのコドンを誤って解読してしまうことが知られている(図1a).この誤翻訳を防止しtRNAIle2のアンチコドンとイソロイシンのAUAコドンとが正しい塩基対を形成することができるように,アンチコドン1文字目のシチジン(C34)が転写後修飾されている(図1a).真正細菌ではC34がアミノ酸のリジンで修飾されていることが20年以上もまえから明らかになっており1,2),その反応を触媒する酵素も同定され反応機構もすでに解明されている3-5).一方,古細菌のC34の修飾形態は長いあいだ謎のままであったが,最近になり,C34はポリアミンの一種であるアグマチンで修飾され2-アグマチニルシチジン(agm2C)に変換されていることが明らかになった6)(図1b).2-アグマチニルシチジンはtRNAIle2のAUAコドンの認識に不可欠であるのと同時に6,7),イソロイシルtRNA合成酵素がtRNAIle2を認識しアミノアシル化(イソロイシンを付加)する際に重要であることも報告されている7,8).修飾をうけていないtRNAIle2はメチオニルtRNA合成酵素により誤ってアミノアシル化(メチオニンを付加)されることから,2-アグマチニルシチジンはtRNAIle2のイソロイシン受容能およびAUAコドン特異性を決定する重要な修飾ヌクレオシドであり,正しいタンパク質の生合成に欠かすことができない.
2-アグマチニルシチジンの生合成はTiaS(tRNAIle-2-アグマチニルシチジン合成酵素)とよばれる酵素によりATP依存的に行われる6).しかしながら,TiaSの一次構造には既知のATP結合モチーフは存在せず,修飾反応を触媒するしくみは不明であった.また,tRNAIle2とtRNAMetはともに同じアンチコドン配列をもっているが,TiaSはtRNAIle2のみを特異的に認識する.しかしながら,TiaSによるこれら2種類のtRNAの識別機構は明らかではない.そこで,筆者らは,TiaSの構造機能解析をつうじtRNAIle2に特異的にアグマチンが導入されるしくみの解明を試みた.
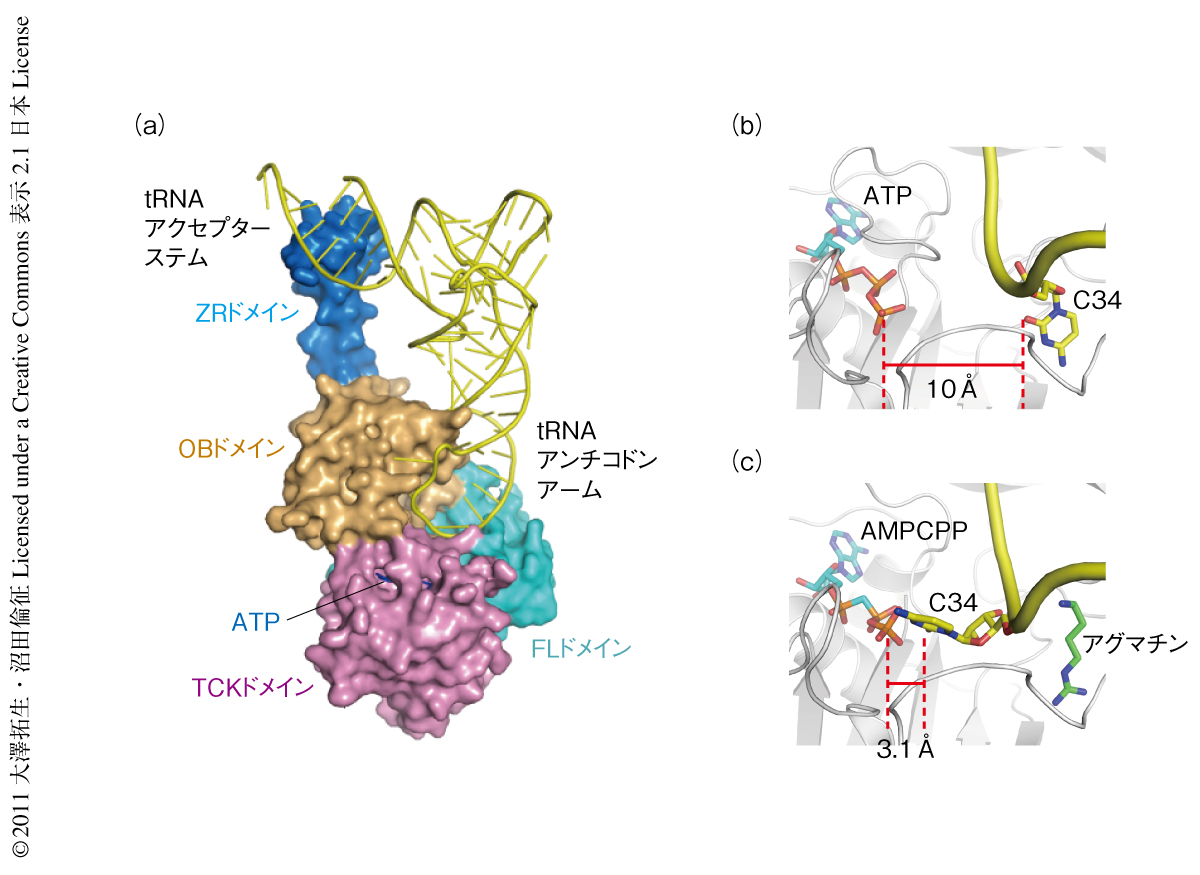
TiaSは古細菌に特異的に存在する酵素である.一般的に,古細菌に由来するタンパク質は大量調製が容易であり結晶構造解析に適したサンプルであるとのイメージが強い.しかしながら,TiaSは凝集しやすいタンパク質であり,大腸菌BL21-CodonPlus(DE3)-RIL株を用いた発現系での収量は非常に少ないものであった6).結晶構造解析を行うためには少しでも収量をあげる必要があったため,まず,さまざまな宿主大腸菌株を用いて,古細菌Archaeoglobus fulgidus,Pyrococcus horikoshii,Sulfolobus tokodaii,Aeropyrum pernix,Methanococcus jannaschiiに由来するTiaSの発現スクリーニングを行った.その結果,C41(DE3)株を宿主とした際にA. fulgidusに由来するTiaSの発現量が上昇することが明らかになった.しかし,精製して得られたTiaSは溶解度が低く,タンパク質濃度として最大でも1.9 mg/ml程度にしか濃縮できなかった.このように調製したサンプルを用いてタンパク質単独での結晶化を試みたが,結晶を再現性よく得ることはできなかった.そこで,tRNAIle2を含んだ複合体の結晶化へと切り替えたところ,TiaS-tRNAIle2-ATP三者複合体の結晶を再現性よく得ることに成功した.大型放射光施設フォトンファクトリーにてX線回折データを収集し,セレノメチオニン置換タンパク質を用いた単波長異常分散法によりTiaS-tRNAIle2-ATP三者複合体の結晶構造を2.9Å分解能で決定した(PDB ID:3AMT).TiaSはTCK(Thr18-Cyt34キナーゼ)ドメイン,フェレドキシン様ドメイン,OBフォールドドメイン,ジンクリボン様ドメインと名づけた4つのドメインから構成されていた(図2a).tRNAIle2のアンチコドンアームはTCKドメイン,フェレドキシン様ドメイン,および,OBフォールドドメインと相互作用していた.一方,tRNAIle2のアクセプターステムはジンクリボン様ドメインと相互作用していることが明らかになった.
TCKドメイン,フェレドキシン様ドメイン,および,OBフォールドドメインは互いに相互作用しながらゆりかご状の構造を形成していた.一方,ジンクリボン様ドメインはそのゆりかご状の構造から突出し,tRNAIle2のアクセプターステムの主溝と相補的な形状をとっていた(図2a).ジンクリボン様ドメインはおもに水素結合を介してtRNAIle2のアクセプターステムと相互作用していた.とくに,Arg369は1位および2位のグアニン塩基(G1およびG2)と水素結合しており,塩基配列に特異的にtRNAIle2のアクセプターステムを認識していることが示唆された.Arg369をアラニン残基に置換した変異体では2-アグマチニルシチジンの合成活性が消失しておりこの相互作用の重要性が確認された.また,Ser361の主鎖と71位のシトシン塩基(C71)とのあいだに水素結合が形成されていた.C71はG2と塩基対合しており,これらの結果から,TiaSはG2-C71塩基対およびG1-C72塩基対を特異的に認識していることが示唆された.
古細菌には2種類のtRNAMetが存在しており,それらはいずれもtRNAIle2と同じアンチコドンループ配列をもっている.リボソームがタンパク質を正しく合成するためには,TiaSが同じアンチコドンループ配列をもつこれらtRNAのなかからtRNAIle2のみを選択してC34を修飾しなければならない.では,いったいTiaSはどうやってtRNAMetとtRNAIle2とを識別しているのであろうか? TiaS-tRNAIle2-ATP三者複合体の結晶構造解析から,TiaSはtRNAIle2のアクセプターステムおよびアンチコドンアームと相互作用していることが明らかになった(図2a).そこで,tRNAIle2のこれらの領域をtRNAMetと同じものに置換したところ,アクセプターステムを入れ替えた変異型tRNAIle2でのみアグマチンによる修飾がほぼ消失することが明らかになった.興味深いことに,古細菌のtRNAIle2ではG1-C72塩基対およびG2-C71塩基対は完全に保存されている.一方,tRNAMetにおいては相当する領域がA1-U72塩基対またはC2-G71塩基対に置換している.tRNAIle2のG1-C72塩基対とG2-C71塩基対をそれぞれA1-U72塩基対とC2-G71塩基対に置換した結果,変異型tRNAIle2のアグマチン受容活性はほぼ消失していた.これは,TiaSがG1-C72塩基対およびG2-C71塩基対を配列特異的に認識しているという結晶構造解析の結果とも一致しており,以上の結果から,TiaSがこれら2つの塩基対を認識することによりtRNA特異性を決定していることが明らかになった.
TiaSはATP依存的に2-アグマチニルシチジンを合成する6).しかしながら,そのアミノ酸配列に既知のATP結合モチーフは存在せず,これまで,修飾反応を触媒するしくみは不明であった.今回,ATPを含んだTiaS-tRNAIle2複合体の結晶構造解析から,これまで不明であったATP結合部位をはじめて同定した(図2a).ATPはTiaSのN末端に存在するTCKドメインに結合しており,その結合にはTiaSファミリーで保存されている数多くの残基が関与していた.TiaSはその主鎖を利用してATPのアデニン塩基と特異的な水素結合を形成しており,これは,筆者らを含む共同研究者によりNature Structural & Molecular Biology誌に同時に発表された,TiaSのATP特異性を示した生化学的な解析の結果9)(新着論文レビュー でも掲載)とも合致するものであった.また,ATPの三リン酸部分はタンパク質の主鎖およびスレオニン残基の側鎖により強固に認識されており,さらに,3つのアスパラギン酸残基にとりかこまれていた.一方,関連する生化学的な実験から,TiaSはまずATPをAMPとピロリン酸に加水分解すること,ついで,生じたピロリン酸を利用してC34をリン酸化し活性化することが報告されている9).ATPの三リン酸部分をとりかこむように配置されていた3つのアスパラギン酸残基をそれぞれアラニン残基に置換した変異体では,ATPの加水分解活性および2-アグマチニルシチジンの合成活性がともに消失していたことから,このドメインがC34のリン酸化に関連する一連の反応にかかわることが示唆された.
結晶構造解析からATP結合部位を構成するThr18はリン酸化されていることが明らかになった.Thr18のリン酸化は質量分析によっても確認されており,TiaSがATPを用いてThr18を自己リン酸化していることが示唆された9).Thr18に付加したリン酸基はC34のリン酸化反応には“利用されない”ことが生化学的な実験から証明されており,Thr18のリン酸化は翻訳後修飾のひとつの形態であると考えられている9).このように,TiaSのN末端側のドメインはC34およびThr18をともにリン酸化する活性をもっていたことから,さきに述べたように,TCK(Thr18-Cyt34キナーゼ)ドメインと命名した.Thr18はTiaSファミリーにおいて高度に保存されており,変異体の解析の結果,ATPの加水分解活性および2-アグマチニルシチジンの合成活性に不可欠であることがわかった.今回の結晶構造解析ではMg2+結合部位を特定することはできなかったが,TiaSはMg2+依存的に2-アグマチニルシチジンを合成することから9),ATPの三リン酸部分をとりかこむように配置されていた3つのアスパラギン酸残基およびリン酸化Thr18により構成されたアニオンクラスターにMg2+が配位し触媒に関与しているものと考えられた.
フェレドキシン様ドメインと同様に,TCKドメインの立体構造はフェレドキシンと類似していた.フェレドキシン様の構造はさまざまなタンパク質にみられる普遍的な基本構造である.この構造はDNAポリメラーゼおよびRNAポリメラーゼの触媒ドメイン10),ヌクレオチド二リン酸キナーゼ11) などにも存在し,それらはいずれもATPなどヌクレオチド三リン酸を基質とする.しかしながら,TCKドメインにおける触媒残基の配向やATPの結合様式はこれまで知られているものとまったく異なっていたことから,TCKドメインは新規なATP利用モジュールであることが明らかになった.結晶構造解析によりこれまでに知られていなかった新たなATP利用モジュールを同定できたことは,タンパク質の構造と機能を分子進化の側面から考察するうえでたいへん興味深いものであった.
TiaS-tRNAIle2-ATP三者複合体において,tRNAIle2のアンチコドンアームはTCKドメイン,フェレドキシン様ドメイン,および,OBフォールドドメインが形成するゆりかご状の構造にそって結合していた(図2a).その際,アンチコドンループはTiaSとの結合にともない大きく構造変化していた.とくに,修飾をうけるC34はフリップアウトしフェレドキシン様ドメインに形成されたポケット(フェレドキシン様ドメインポケット)の奥深くに押し込められ(図2b),Gly215の主鎖と水素結合を形成し特異的に認識されていた.
2-アグマチニルシチジンの合成の過程において,C34はリン酸化によりいったん活性化され,その際,ATPのγ位に由来するリン酸基がC34に付加することが知られている9).C34が結合していたフェレドキシン様ドメインポケットはTCKドメインに結合しているATPのγ-リン酸と10Åも離れていた(図2b).これは,C34がATPと反応するには遠すぎる距離に配置されていることを意味しており,TiaS-tRNAIle2-ATP三者複合体はC34のリン酸化反応を触媒できない構造をとっているものと考えられた.
では,TiaSはC34をいったいどのようにリン酸化しているのであろうか? つぎに,アグマチンとATPアナログ(AMPCPP)を含むTiaS-tRNAIle2複合体(四者複合体)の結晶構造を決定した(PDB ID:3AMU,図2c).アグマチンはフェレドキシン様ドメインポケットに結合しており,Gly215の主鎖と水素結合していた.TiaS-tRNAIle2-ATP三者複合体において,このGly215はC34の認識にかかわる重要なアミノ酸残基である.したがって,TiaS-tRNAIle2-AMPCPP-アグマチン四者複合体におけるアグマチンの結合部位は,三者複合体におけるC34の結合部位と完全にオーバーラップしていることが明らかになった.その結果,TiaS-tRNAIle2-AMPCPP-アグマチン四者複合体ではC34がフェレドキシン様ドメインポケットに結合できず,AMPCPPのγ-リン酸の近傍(3.1Å)に配置されていることがわかり(図2c),この部位でTiaSはC34をリン酸化するものと結論づけた.以上のように,アグマチンの有無によりC34の結合部位が異なることが明らかになった.
アグマチンの存在下でC34がAMPCPPのγ-リン酸の近傍に配置されていることを考慮すると,TiaS-tRNAIle2-AMPCPP-アグマチン四者複合体の構造はTiaS-tRNAIle2-ATP三者複合体の構造と比較して,より反応ステージの進んだ(リン酸化に適した)構造であるものと考えられた.では,TiaS-tRNAIle2-ATP三者複合体の構造は反応ステージにおいてどのような“状態”を反映しているのであろうか? また,C34がリン酸化されたのち,どのようにしてリン酸基は遊離しアグマチンと置換するのであろうか?
TiaSのアグマチンおよびtRNAIle2に対するKm値はそれぞれ0.87μMと0.29μMであり,同じ程度の親和性を示すことが報告されている9).古細菌におけるアグマチンおよびtRNAIle2の正確な細胞内濃度は知られていない.しかし,古細菌に多量に存在するプトレシンやスペルミジンといったポリアミンの生合成の材料としてアグマチンが利用されていることを考えると12),アグマチンの細胞内濃度はtRNAIle2の濃度よりも高いことが強く示唆される.TiaS-tRNAIle2-ATP三者複合体におけるC34の結合部位とTiaS-tRNAIle2-AMPCPP-アグマチン四者複合体におけるアグマチン結合部位は完全に一致しているが,基質に対する親和性と推定される細胞内濃度を考慮すると,TiaSはtRNAIle2と相互作用するまえにすでにアグマチンと結合しているものと考えられた.そして,tRNAとの結合にともないC34はATPのγ-リン酸の近傍に直接的に配置されるものと予想された(図3,1と4).一方,万が一,細胞内のアグマチン濃度が低い場合,TiaSはC34をフェレドキシン様ドメインポケットにとらえATP結合部位から隔離していると考えられた(図3,2と3).TiaSはtRNAやアグマチンの有無にかかわらずATPを加水分解することが報告されている9).よって,アグマチンが不足する条件においてC34をATPから隔離することは,むだなC34のリン酸化とその反応中間体の過剰な蓄積を防止するのに役立っているものと思われた.この場合,アグマチンの供給にともないtRNAアンチコドン領域の構造が変化し,C34はATPのγ-リン酸の近傍に配置されると考えられた(図3,3と4).いずれの場合においても,アグマチンとTiaSとが結合することによりC34が活性部位に適切に配置されることが示唆された.C34はリン酸化によりいったん活性化されたのち,リン酸化されたThr18とのあいだの静電的な反発などによりアグマチンの近傍に再配置されることが予想され,その場において,アグマチンが活性化されたC34を求核攻撃し2-アグマチニルシチジンが生成するものと考えられた(図3,5から7).これらを総合すると,今回,決定したTiaS-tRNAIle2-ATP三者複合体およびTiaS-tRNAIle2-AMPCPP-アグマチン四者複合体の構造は,それぞれ“C34捕捉状態”および“リン酸化反応前状態”であると考えられ,タンパク質とRNAが協同しながら互いの構造を変化させ反応を促進していることが明らかになった.
真正細菌ではtRNAIle2のC34はアミノ酸のリジンで修飾されてライシジンとよばれる修飾ヌクレオシドとして存在している1).ライシジンは酵素TilS(tRNAIle-ライシジン合成酵素)によりATP依存的に産生される3-5).TilSにはN型ATPピロホスファターゼモチーフが存在し,ATPを基質としてC34をアデニル化し活性化する.そののち,リジンがアデニル化反応中間体を求核攻撃することでライシジンが産生される.このようにTiaSとTilSは,C34をそれぞれリン酸化反応中間体またはアデニル化反応中間体として活性化するなど,まったく異なる機構で修飾反応を触媒している.しかしながら,結果的に産生される2-アグマチニルシチジンとライシジンの化学構造は類似しており,また,それら修飾ヌクレオシドの機能もイソロイシンのAUAコドンの特異的な認識にかかわるなど共通する点が多い.さらに特筆すべき点として,TiaSとTilSにはアミノ酸配列および立体構造における相同性はないにもかかわらず,ともにtRNAのアクセプターステムの配列を特異的に認識しtRNAIle2を選択するという共通性をもっている.このように,これら2つの酵素は構造においてまったく異なるファミリーに属しているがその機能は類似しており,これはイソロイシンのAUAコドンの解読を目的とした収斂進化の結果であると考えられる.
略歴:2008年 九州大学大学院システム生命科学府 修了,同年 産業技術総合研究所生物機能工学研究部門 博士研究員を経て,2010年より産業技術総合研究所バイオメディカル研究部門 博士研究員.
研究テーマ:生体高分子複合体の構造と機能.
抱負:新規の分子機構をみつけだしたい.
沼田 倫征(Tomoyuki Numata)
産業技術総合研究所バイオメディカル研究部門 研究員.
© 2011 大澤拓生・沼田倫征 Licensed under CC 表示 2.1 日本
(産業技術総合研究所バイオメディカル研究部門 RNAプロセシング研究グループ)
email:大澤拓生,沼田倫征
DOI: 10.7875/first.author.2011.165
Structural basis of tRNA agmatinylation essential for AUA codon decoding.
Takuo Osawa, Satoshi Kimura, Naohiro Terasaka, Hideko Inanaga, Tsutomu Suzuki, Tomoyuki Numata
Nature Structural & Molecular Biology, 18, 1275-1280 (2011)
要 約
タンパク質を正確に生合成するためには,リボソームにおいてtRNAが適正なコドンを選択しなければならない.tRNAのアンチコドン領域に存在する修飾ヌクレオシドは正しいコドン認識を可能にするという重要な役割を担っている.古細菌において,イソロイシンコドンのひとつであるAUAコドンを解読するtRNAIle2では,アンチコドン1文字目のシチジンがポリアミンの一種であるアグマチンにより修飾され2-アグマチニルシチジンへと変換されている.また,酵素TiaSはATP依存的に2-アグマチニルシチジンを合成することが知られている.今回,筆者らは,ATPもしくはAMPCPPおよびアグマチンと結合したTiaS-tRNAIle2複合体の結晶構造を解析した.その結果,アミノ酸配列の情報だけでは不明であった新規のATP結合ドメインを同定した.TiaS-tRNAIle2-ATP複合体において,アンチコドン1文字目のシチジンはATP結合部位から隔離されたポケットに結合していた.一方,TiaS-tRNAIle2-AMPCPP-アグマチン複合体ではそのポケットがアグマチンで占有され,アンチコドン1文字目のシチジンはAMPCPPのγ-リン酸の近傍に配置されていた.したがって,TiaSはATPを利用してアンチコドン1文字目のシチジンをリン酸化しいったん活性化すること,そののち,アグマチンがこのリン酸化反応中間体を求核攻撃することで2-アグマチニルシチジンが産生されることが示唆された.この反応機構は関連する生化学的な実験からも支持された.以上の結果は,TiaSへのアグマチンの結合がアンチコドン1文字目のシチジンのリン酸化反応に不可欠であることを示唆するとともに,TiaSには反応を効率よく触媒するしくみが存在することを示した.
はじめに
遺伝情報の発現の過程においてmRNAのコドンはtRNAを介し対応するアミノ酸へと解読される.つまり,tRNAはコドンというヌクレオチド配列の情報をアミノ酸配列の情報へと変換するためのアダプターとして機能している.したがって,遺伝情報を正確に発現させるためには,tRNAのアンチコドンが対応するコドンのみを忠実に認識しなければならない.tRNAによるコドンの認識はコドン-アンチコドン塩基対の形成により遂行される.その際,tRNAのアンチコドン領域に存在する修飾ヌクレオシドが正確なコドンの認識に重要な役割をはたしている.とくに,アンチコドン1文字目に存在する修飾ヌクレオシドはコドンの第3塩基と対合することから,コドンの縮重(ゆらぎ)と密接にかかわっている.
原核生物にはイソロイシンのコドン(AUU,AUC,AUA)に対応する2種類のtRNA,tRNAIle1とtRNAIle2が存在する.このうち,AUAコドンを解読するtRNAIle2のアンチコドン配列はCAUであり,メチオニンのコドン(AUG)を解読するtRNAMetのアンチコドン配列(CAU)と同じである.このため,修飾をうけていないtRNAIle2はメチオニンのコドンを誤って解読してしまうことが知られている(図1a).この誤翻訳を防止しtRNAIle2のアンチコドンとイソロイシンのAUAコドンとが正しい塩基対を形成することができるように,アンチコドン1文字目のシチジン(C34)が転写後修飾されている(図1a).真正細菌ではC34がアミノ酸のリジンで修飾されていることが20年以上もまえから明らかになっており1,2),その反応を触媒する酵素も同定され反応機構もすでに解明されている3-5).一方,古細菌のC34の修飾形態は長いあいだ謎のままであったが,最近になり,C34はポリアミンの一種であるアグマチンで修飾され2-アグマチニルシチジン(agm2C)に変換されていることが明らかになった6)(図1b).2-アグマチニルシチジンはtRNAIle2のAUAコドンの認識に不可欠であるのと同時に6,7),イソロイシルtRNA合成酵素がtRNAIle2を認識しアミノアシル化(イソロイシンを付加)する際に重要であることも報告されている7,8).修飾をうけていないtRNAIle2はメチオニルtRNA合成酵素により誤ってアミノアシル化(メチオニンを付加)されることから,2-アグマチニルシチジンはtRNAIle2のイソロイシン受容能およびAUAコドン特異性を決定する重要な修飾ヌクレオシドであり,正しいタンパク質の生合成に欠かすことができない.
2-アグマチニルシチジンの生合成はTiaS(tRNAIle-2-アグマチニルシチジン合成酵素)とよばれる酵素によりATP依存的に行われる6).しかしながら,TiaSの一次構造には既知のATP結合モチーフは存在せず,修飾反応を触媒するしくみは不明であった.また,tRNAIle2とtRNAMetはともに同じアンチコドン配列をもっているが,TiaSはtRNAIle2のみを特異的に認識する.しかしながら,TiaSによるこれら2種類のtRNAの識別機構は明らかではない.そこで,筆者らは,TiaSの構造機能解析をつうじtRNAIle2に特異的にアグマチンが導入されるしくみの解明を試みた.
1.TiaS-tRNAIle2-ATP三者複合体の結晶構造解析
TiaSは古細菌に特異的に存在する酵素である.一般的に,古細菌に由来するタンパク質は大量調製が容易であり結晶構造解析に適したサンプルであるとのイメージが強い.しかしながら,TiaSは凝集しやすいタンパク質であり,大腸菌BL21-CodonPlus(DE3)-RIL株を用いた発現系での収量は非常に少ないものであった6).結晶構造解析を行うためには少しでも収量をあげる必要があったため,まず,さまざまな宿主大腸菌株を用いて,古細菌Archaeoglobus fulgidus,Pyrococcus horikoshii,Sulfolobus tokodaii,Aeropyrum pernix,Methanococcus jannaschiiに由来するTiaSの発現スクリーニングを行った.その結果,C41(DE3)株を宿主とした際にA. fulgidusに由来するTiaSの発現量が上昇することが明らかになった.しかし,精製して得られたTiaSは溶解度が低く,タンパク質濃度として最大でも1.9 mg/ml程度にしか濃縮できなかった.このように調製したサンプルを用いてタンパク質単独での結晶化を試みたが,結晶を再現性よく得ることはできなかった.そこで,tRNAIle2を含んだ複合体の結晶化へと切り替えたところ,TiaS-tRNAIle2-ATP三者複合体の結晶を再現性よく得ることに成功した.大型放射光施設フォトンファクトリーにてX線回折データを収集し,セレノメチオニン置換タンパク質を用いた単波長異常分散法によりTiaS-tRNAIle2-ATP三者複合体の結晶構造を2.9Å分解能で決定した(PDB ID:3AMT).TiaSはTCK(Thr18-Cyt34キナーゼ)ドメイン,フェレドキシン様ドメイン,OBフォールドドメイン,ジンクリボン様ドメインと名づけた4つのドメインから構成されていた(図2a).tRNAIle2のアンチコドンアームはTCKドメイン,フェレドキシン様ドメイン,および,OBフォールドドメインと相互作用していた.一方,tRNAIle2のアクセプターステムはジンクリボン様ドメインと相互作用していることが明らかになった.
2.tRNAIle2のアクセプターステムの認識とtRNA特異性
TCKドメイン,フェレドキシン様ドメイン,および,OBフォールドドメインは互いに相互作用しながらゆりかご状の構造を形成していた.一方,ジンクリボン様ドメインはそのゆりかご状の構造から突出し,tRNAIle2のアクセプターステムの主溝と相補的な形状をとっていた(図2a).ジンクリボン様ドメインはおもに水素結合を介してtRNAIle2のアクセプターステムと相互作用していた.とくに,Arg369は1位および2位のグアニン塩基(G1およびG2)と水素結合しており,塩基配列に特異的にtRNAIle2のアクセプターステムを認識していることが示唆された.Arg369をアラニン残基に置換した変異体では2-アグマチニルシチジンの合成活性が消失しておりこの相互作用の重要性が確認された.また,Ser361の主鎖と71位のシトシン塩基(C71)とのあいだに水素結合が形成されていた.C71はG2と塩基対合しており,これらの結果から,TiaSはG2-C71塩基対およびG1-C72塩基対を特異的に認識していることが示唆された.
古細菌には2種類のtRNAMetが存在しており,それらはいずれもtRNAIle2と同じアンチコドンループ配列をもっている.リボソームがタンパク質を正しく合成するためには,TiaSが同じアンチコドンループ配列をもつこれらtRNAのなかからtRNAIle2のみを選択してC34を修飾しなければならない.では,いったいTiaSはどうやってtRNAMetとtRNAIle2とを識別しているのであろうか? TiaS-tRNAIle2-ATP三者複合体の結晶構造解析から,TiaSはtRNAIle2のアクセプターステムおよびアンチコドンアームと相互作用していることが明らかになった(図2a).そこで,tRNAIle2のこれらの領域をtRNAMetと同じものに置換したところ,アクセプターステムを入れ替えた変異型tRNAIle2でのみアグマチンによる修飾がほぼ消失することが明らかになった.興味深いことに,古細菌のtRNAIle2ではG1-C72塩基対およびG2-C71塩基対は完全に保存されている.一方,tRNAMetにおいては相当する領域がA1-U72塩基対またはC2-G71塩基対に置換している.tRNAIle2のG1-C72塩基対とG2-C71塩基対をそれぞれA1-U72塩基対とC2-G71塩基対に置換した結果,変異型tRNAIle2のアグマチン受容活性はほぼ消失していた.これは,TiaSがG1-C72塩基対およびG2-C71塩基対を配列特異的に認識しているという結晶構造解析の結果とも一致しており,以上の結果から,TiaSがこれら2つの塩基対を認識することによりtRNA特異性を決定していることが明らかになった.
3.結晶構造解析による触媒ドメインの同定
TiaSはATP依存的に2-アグマチニルシチジンを合成する6).しかしながら,そのアミノ酸配列に既知のATP結合モチーフは存在せず,これまで,修飾反応を触媒するしくみは不明であった.今回,ATPを含んだTiaS-tRNAIle2複合体の結晶構造解析から,これまで不明であったATP結合部位をはじめて同定した(図2a).ATPはTiaSのN末端に存在するTCKドメインに結合しており,その結合にはTiaSファミリーで保存されている数多くの残基が関与していた.TiaSはその主鎖を利用してATPのアデニン塩基と特異的な水素結合を形成しており,これは,筆者らを含む共同研究者によりNature Structural & Molecular Biology誌に同時に発表された,TiaSのATP特異性を示した生化学的な解析の結果9)(新着論文レビュー でも掲載)とも合致するものであった.また,ATPの三リン酸部分はタンパク質の主鎖およびスレオニン残基の側鎖により強固に認識されており,さらに,3つのアスパラギン酸残基にとりかこまれていた.一方,関連する生化学的な実験から,TiaSはまずATPをAMPとピロリン酸に加水分解すること,ついで,生じたピロリン酸を利用してC34をリン酸化し活性化することが報告されている9).ATPの三リン酸部分をとりかこむように配置されていた3つのアスパラギン酸残基をそれぞれアラニン残基に置換した変異体では,ATPの加水分解活性および2-アグマチニルシチジンの合成活性がともに消失していたことから,このドメインがC34のリン酸化に関連する一連の反応にかかわることが示唆された.
結晶構造解析からATP結合部位を構成するThr18はリン酸化されていることが明らかになった.Thr18のリン酸化は質量分析によっても確認されており,TiaSがATPを用いてThr18を自己リン酸化していることが示唆された9).Thr18に付加したリン酸基はC34のリン酸化反応には“利用されない”ことが生化学的な実験から証明されており,Thr18のリン酸化は翻訳後修飾のひとつの形態であると考えられている9).このように,TiaSのN末端側のドメインはC34およびThr18をともにリン酸化する活性をもっていたことから,さきに述べたように,TCK(Thr18-Cyt34キナーゼ)ドメインと命名した.Thr18はTiaSファミリーにおいて高度に保存されており,変異体の解析の結果,ATPの加水分解活性および2-アグマチニルシチジンの合成活性に不可欠であることがわかった.今回の結晶構造解析ではMg2+結合部位を特定することはできなかったが,TiaSはMg2+依存的に2-アグマチニルシチジンを合成することから9),ATPの三リン酸部分をとりかこむように配置されていた3つのアスパラギン酸残基およびリン酸化Thr18により構成されたアニオンクラスターにMg2+が配位し触媒に関与しているものと考えられた.
フェレドキシン様ドメインと同様に,TCKドメインの立体構造はフェレドキシンと類似していた.フェレドキシン様の構造はさまざまなタンパク質にみられる普遍的な基本構造である.この構造はDNAポリメラーゼおよびRNAポリメラーゼの触媒ドメイン10),ヌクレオチド二リン酸キナーゼ11) などにも存在し,それらはいずれもATPなどヌクレオチド三リン酸を基質とする.しかしながら,TCKドメインにおける触媒残基の配向やATPの結合様式はこれまで知られているものとまったく異なっていたことから,TCKドメインは新規なATP利用モジュールであることが明らかになった.結晶構造解析によりこれまでに知られていなかった新たなATP利用モジュールを同定できたことは,タンパク質の構造と機能を分子進化の側面から考察するうえでたいへん興味深いものであった.
4.TiaS-tRNAIle2-ATP三者複合体におけるC34の結合部位
TiaS-tRNAIle2-ATP三者複合体において,tRNAIle2のアンチコドンアームはTCKドメイン,フェレドキシン様ドメイン,および,OBフォールドドメインが形成するゆりかご状の構造にそって結合していた(図2a).その際,アンチコドンループはTiaSとの結合にともない大きく構造変化していた.とくに,修飾をうけるC34はフリップアウトしフェレドキシン様ドメインに形成されたポケット(フェレドキシン様ドメインポケット)の奥深くに押し込められ(図2b),Gly215の主鎖と水素結合を形成し特異的に認識されていた.
2-アグマチニルシチジンの合成の過程において,C34はリン酸化によりいったん活性化され,その際,ATPのγ位に由来するリン酸基がC34に付加することが知られている9).C34が結合していたフェレドキシン様ドメインポケットはTCKドメインに結合しているATPのγ-リン酸と10Åも離れていた(図2b).これは,C34がATPと反応するには遠すぎる距離に配置されていることを意味しており,TiaS-tRNAIle2-ATP三者複合体はC34のリン酸化反応を触媒できない構造をとっているものと考えられた.
5.アグマチンの有無によるC34結合部位の違い
では,TiaSはC34をいったいどのようにリン酸化しているのであろうか? つぎに,アグマチンとATPアナログ(AMPCPP)を含むTiaS-tRNAIle2複合体(四者複合体)の結晶構造を決定した(PDB ID:3AMU,図2c).アグマチンはフェレドキシン様ドメインポケットに結合しており,Gly215の主鎖と水素結合していた.TiaS-tRNAIle2-ATP三者複合体において,このGly215はC34の認識にかかわる重要なアミノ酸残基である.したがって,TiaS-tRNAIle2-AMPCPP-アグマチン四者複合体におけるアグマチンの結合部位は,三者複合体におけるC34の結合部位と完全にオーバーラップしていることが明らかになった.その結果,TiaS-tRNAIle2-AMPCPP-アグマチン四者複合体ではC34がフェレドキシン様ドメインポケットに結合できず,AMPCPPのγ-リン酸の近傍(3.1Å)に配置されていることがわかり(図2c),この部位でTiaSはC34をリン酸化するものと結論づけた.以上のように,アグマチンの有無によりC34の結合部位が異なることが明らかになった.
6.2-アグマチニルシチジンの合成反応のスキーム
アグマチンの存在下でC34がAMPCPPのγ-リン酸の近傍に配置されていることを考慮すると,TiaS-tRNAIle2-AMPCPP-アグマチン四者複合体の構造はTiaS-tRNAIle2-ATP三者複合体の構造と比較して,より反応ステージの進んだ(リン酸化に適した)構造であるものと考えられた.では,TiaS-tRNAIle2-ATP三者複合体の構造は反応ステージにおいてどのような“状態”を反映しているのであろうか? また,C34がリン酸化されたのち,どのようにしてリン酸基は遊離しアグマチンと置換するのであろうか?
TiaSのアグマチンおよびtRNAIle2に対するKm値はそれぞれ0.87μMと0.29μMであり,同じ程度の親和性を示すことが報告されている9).古細菌におけるアグマチンおよびtRNAIle2の正確な細胞内濃度は知られていない.しかし,古細菌に多量に存在するプトレシンやスペルミジンといったポリアミンの生合成の材料としてアグマチンが利用されていることを考えると12),アグマチンの細胞内濃度はtRNAIle2の濃度よりも高いことが強く示唆される.TiaS-tRNAIle2-ATP三者複合体におけるC34の結合部位とTiaS-tRNAIle2-AMPCPP-アグマチン四者複合体におけるアグマチン結合部位は完全に一致しているが,基質に対する親和性と推定される細胞内濃度を考慮すると,TiaSはtRNAIle2と相互作用するまえにすでにアグマチンと結合しているものと考えられた.そして,tRNAとの結合にともないC34はATPのγ-リン酸の近傍に直接的に配置されるものと予想された(図3,1と4).一方,万が一,細胞内のアグマチン濃度が低い場合,TiaSはC34をフェレドキシン様ドメインポケットにとらえATP結合部位から隔離していると考えられた(図3,2と3).TiaSはtRNAやアグマチンの有無にかかわらずATPを加水分解することが報告されている9).よって,アグマチンが不足する条件においてC34をATPから隔離することは,むだなC34のリン酸化とその反応中間体の過剰な蓄積を防止するのに役立っているものと思われた.この場合,アグマチンの供給にともないtRNAアンチコドン領域の構造が変化し,C34はATPのγ-リン酸の近傍に配置されると考えられた(図3,3と4).いずれの場合においても,アグマチンとTiaSとが結合することによりC34が活性部位に適切に配置されることが示唆された.C34はリン酸化によりいったん活性化されたのち,リン酸化されたThr18とのあいだの静電的な反発などによりアグマチンの近傍に再配置されることが予想され,その場において,アグマチンが活性化されたC34を求核攻撃し2-アグマチニルシチジンが生成するものと考えられた(図3,5から7).これらを総合すると,今回,決定したTiaS-tRNAIle2-ATP三者複合体およびTiaS-tRNAIle2-AMPCPP-アグマチン四者複合体の構造は,それぞれ“C34捕捉状態”および“リン酸化反応前状態”であると考えられ,タンパク質とRNAが協同しながら互いの構造を変化させ反応を促進していることが明らかになった.
おわりに
真正細菌ではtRNAIle2のC34はアミノ酸のリジンで修飾されてライシジンとよばれる修飾ヌクレオシドとして存在している1).ライシジンは酵素TilS(tRNAIle-ライシジン合成酵素)によりATP依存的に産生される3-5).TilSにはN型ATPピロホスファターゼモチーフが存在し,ATPを基質としてC34をアデニル化し活性化する.そののち,リジンがアデニル化反応中間体を求核攻撃することでライシジンが産生される.このようにTiaSとTilSは,C34をそれぞれリン酸化反応中間体またはアデニル化反応中間体として活性化するなど,まったく異なる機構で修飾反応を触媒している.しかしながら,結果的に産生される2-アグマチニルシチジンとライシジンの化学構造は類似しており,また,それら修飾ヌクレオシドの機能もイソロイシンのAUAコドンの特異的な認識にかかわるなど共通する点が多い.さらに特筆すべき点として,TiaSとTilSにはアミノ酸配列および立体構造における相同性はないにもかかわらず,ともにtRNAのアクセプターステムの配列を特異的に認識しtRNAIle2を選択するという共通性をもっている.このように,これら2つの酵素は構造においてまったく異なるファミリーに属しているがその機能は類似しており,これはイソロイシンのAUAコドンの解読を目的とした収斂進化の結果であると考えられる.
文 献
- Muramatsu, T., Yokoyama, S., Horie, N. et al.: A novel lysine-substituted nucleoside in the first position of the anticodon of minor isoleucine tRNA from Escherichia coli. J. Biol. Chem., 263, 9261-9267 (1988)[PubMed]
- Muramatsu, T., Nishikawa, K., Nemoto, F. et al.: Codon and amino-acid specificities of a transfer RNA are both converted by a single post-transcriptional modification. Nature, 336, 179-181 (1988)[PubMed]
- Soma, A., Ikeuchi, Y., Kanemasa, S. et al.: An RNA-modifying enzyme that governs both the codon and amino acid specificities of isoleucine tRNA. Mol. Cell, 12, 689-698 (2003)[PubMed]
- Ikeuchi, Y., Soma, A., Ote, T. et al.: Molecular mechanism of lysidine synthesis that determines tRNA identity and codon recognition. Mol. Cell, 19, 235-246 (2005)[PubMed]
- Nakanishi, K., Bonnefond, L., Kimura, S. et al.: Structural basis for translational fidelity ensured by transfer RNA lysidine synthetase. Nature, 461, 1144-1148 (2009)[PubMed]
- Ikeuchi, Y., Kimura, S., Numata, T. et al.: Agmatine-conjugated cytidine in a tRNA anticodon is essential for AUA decoding in archaea. Nat. Chem. Biol., 6, 277-282 (2010)[PubMed]
- Mandal, D., Kohrer, C., Su, D. et al.: Agmatidine, a modified cytidine in the anticodon of archaeal tRNAIle, base pairs with adenosine but not with guanosine. Proc. Natl. Acad. Sci. USA, 107, 2872-2877 (2010)[PubMed]
- Kohrer, C., Srinivasan, G., Mandal, D. et al.: Identification and characterization of a tRNA decoding the rare AUA codon in Haloarcula marismortui. RNA, 14, 117-126 (2008)[PubMed]
- Terasaka, N., Kimura, S., Osawa, T. et al.: Biogenesis of 2-agmatinylcytidine catalyzed by the dual protein and RNA kinase TiaS. Nat. Struct. Mol. Biol., 18, 1268-1274 (2011)[PubMed] [新着論文レビュー]
- Doublie, S., Tabor, S., Long, A. M. et al.: Crystal structure of a bacteriophage T7 DNA replication complex at 2.2Å resolution. Nature, 391, 251-258 (1998)[PubMed]
- Janin, J., Dumas, C., Morera, S. et al.: Three-dimensional structure of nucleoside diphosphate kinase. J. Bioenerg. Biomembr., 32, 215-225 (2000)[PubMed]
- Fukuda, W., Morimoto, N., Imanaka, T. et al.: Agmatine is essential for the cell growth of Thermococcus kodakaraensis. FEMS Microbiol. Lett., 287, 113-120 (2008)[PubMed]
著者プロフィール
略歴:2008年 九州大学大学院システム生命科学府 修了,同年 産業技術総合研究所生物機能工学研究部門 博士研究員を経て,2010年より産業技術総合研究所バイオメディカル研究部門 博士研究員.
研究テーマ:生体高分子複合体の構造と機能.
抱負:新規の分子機構をみつけだしたい.
沼田 倫征(Tomoyuki Numata)
産業技術総合研究所バイオメディカル研究部門 研究員.
© 2011 大澤拓生・沼田倫征 Licensed under CC 表示 2.1 日本