DNA複製装置クランプの枯草菌の細胞内における動的な集合
末次 正幸
(英国Newcastle大学Institute for Cell and Molecular Biosciences,Centre for Bacterial Cell Biology)
email:末次正幸
DOI: 10.7875/first.author.2011.065
The replicase sliding clamp dynamically accumulates behind progressing replication forks in Bacillus subtilis cells.
Masayuki Su'etsugu & Jeff Errington
Molecular Cell, 41, 720-732 (2011)
クランプはDNA複製装置の構成因子であるとともに,DNA修復や岡崎フラグメントの成熟化などDNA代謝にかかわる多様な反応に機能している.筆者らは,枯草菌のクランプの細胞内における動態を顕微鏡によりとらえることで,複製開始ののち細胞内に散らばっていたクランプが毎秒1分子の速度で集合し約200分子からなる“クランプゾーン”を形成することを見い出した.この集合は岡崎フラグメントの合成ごとに装着されたクランプがDNAに残留することにより導かれていた.一定量のクランプが蓄積するとクランプの解離反応が進行する.そして,クランプゾーンはクランプ分子の装着と解離をくり返しながら複製フォークとともに染色体を移動する.この分子動態を数理モデル化してシミュレーションを行うと,その結果は実際に細胞において検出されたクランプの動的なふるまいをうまく再現した.筆者らはさらに,クランプゾーンがタンパク質を複製焦点へと局在化させる足場として機能していること示した.
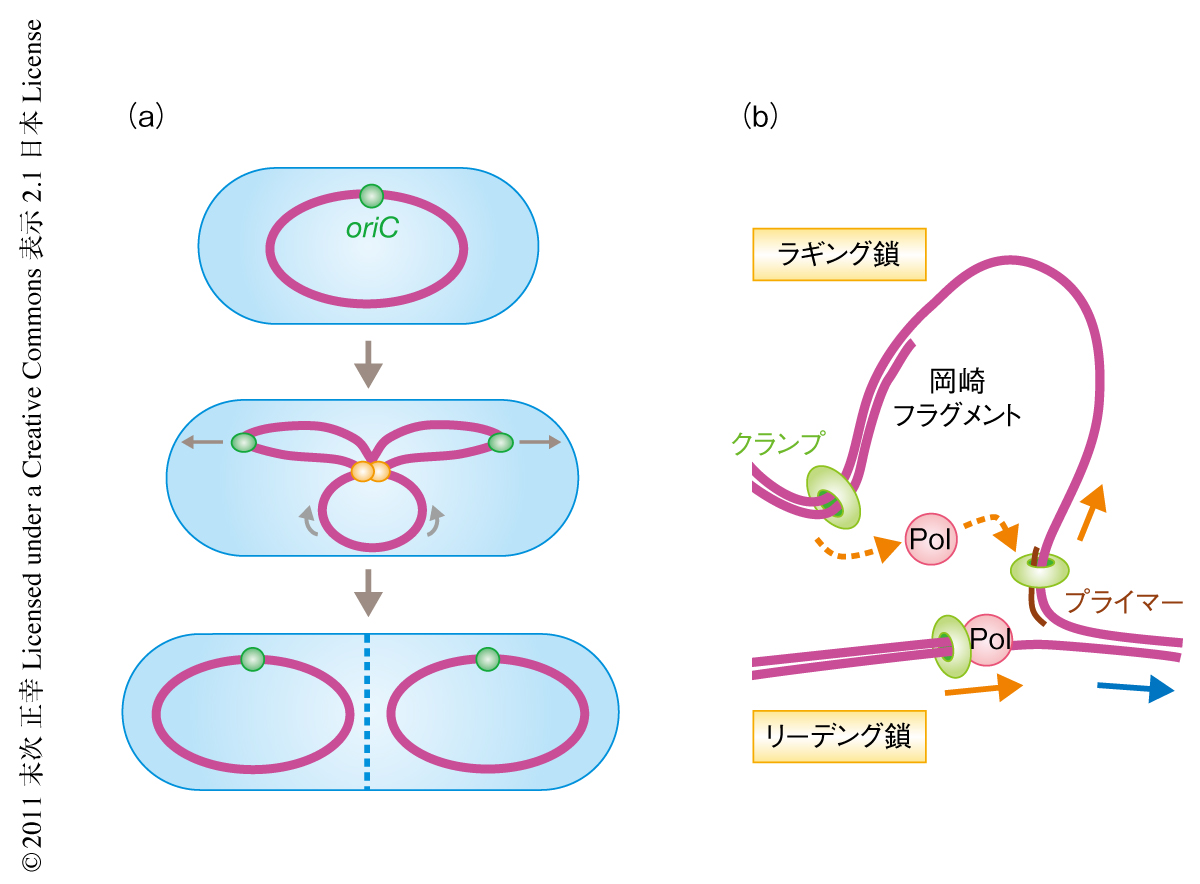
DNAを複製している細胞では複製に機能するタンパク質がいくつも集まって細胞において焦点をなしている1).この焦点は複製焦点とよばれ,細菌から哺乳動物にいたるまで広く観察されている.しかしながら,複製に機能するタンパク質がどのようにして複製焦点を形成するのかはよくわかっていない.大腸菌や枯草菌といった細菌は基本的に環状の染色体を1つだけもちその複製起点(oriC)も1つだけである.よって,通常,1つもしくは2つの複製焦点が形成されている2)(図1a).このシンプルな構造は複製に機能するタンパク質の細胞での挙動を調べるうえで都合がよい.
細菌の染色体の複製はその多くのステップが精製タンパク質を用いてin vitro再構成されており詳細な解析がなされている3,4).複製開始ののち,複製起点の2本鎖DNAの開裂によって生じた1本鎖DNAにプライマーが形成され,そこにクランプが装着される.クランプはDnaNホモ2量体からなり,そのリングのなかに2本鎖DNAを通すので,構造上,安定にDNAに維持される5).クランプは複製反応においてDNAポリメラーゼをDNAにつなぎとめておく役割をはたす.複製フォークの進行は互いに逆向きの鋳型鎖を生じる.このためリーディング鎖は継続的に合成される一方,ラギング鎖は岡崎フラグメントとして知られる1~2 kbの断片ごとに不連続合成される6).岡崎フラグメントの合成はプライマーの形成とそこへのクランプの装着により開始する.岡崎フラグメントの合成ののちDNAポリメラーゼはクランプから外れるので,in vitroではクランプは単独でDNAに残留する7,8)(図1b).では,in vivoにおいてもクランプはDNAに残留しつづけるのだろうか? 細胞は数百個しかないクランプ分子により染色体の複製あたり2000~4000もの岡崎フラグメントを合成しなければならない.だとすると,残留したクランプを積極的に解離してリサイクルする機構があると考えたほうがよい.しかしながら,in vivoにおいてクランプを解離する機構があるのかどうか知られていない.
今回,筆者らは,クランプが岡崎フラグメントの合成ごとに装着→残留→解離のサイクルをくり返しながら,複製フォークの近傍に超高次な集合体(焦点)をなしているようすをin vivoにおいてとらえることに成功した.
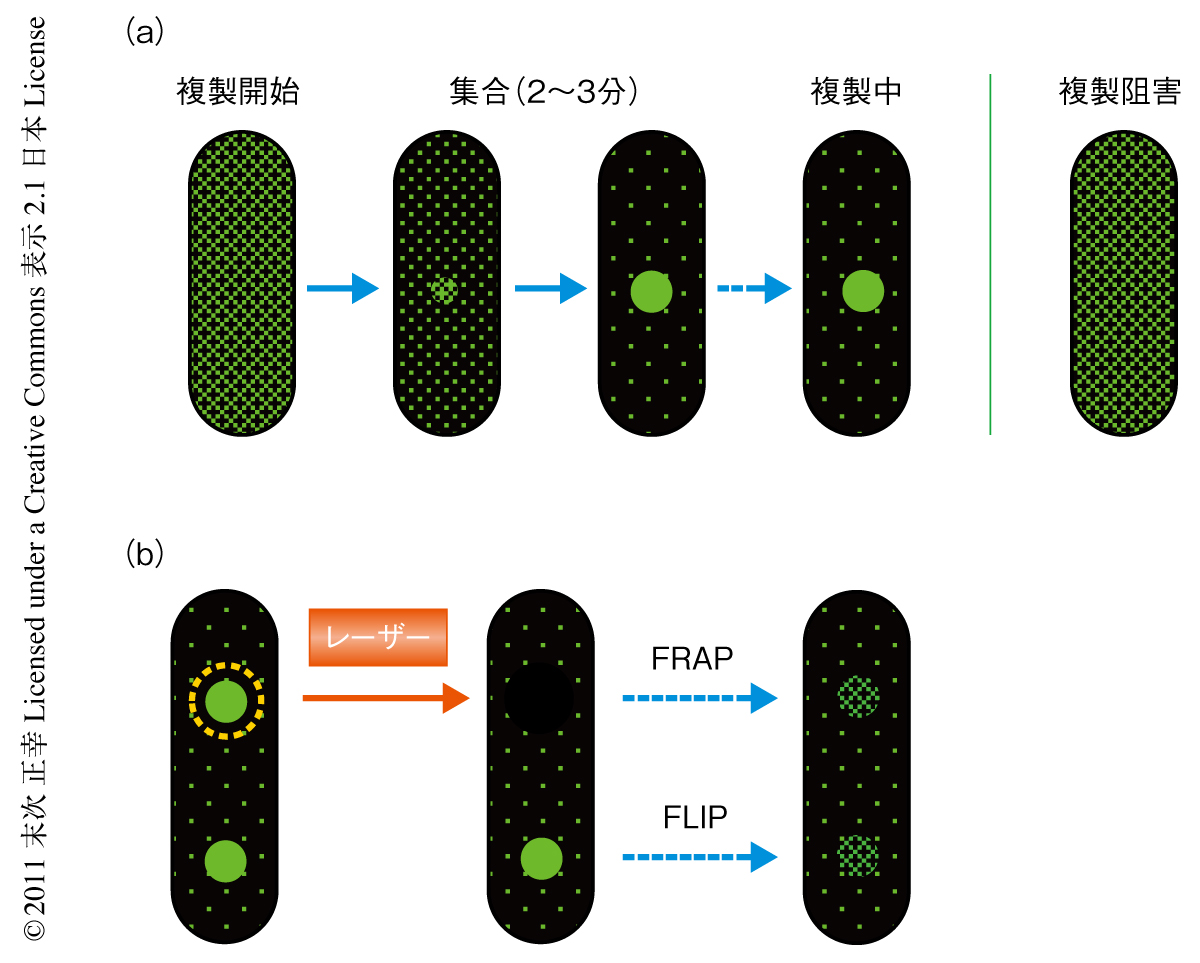
クランプがどのようにして焦点を形成するのかをGFP融合型クランプを用いて調べた.複製開始まえの細胞では予想どおりクランプは細胞全体に拡散していた.複製が開始されると細胞内に散らばっていたクランプ分子はごく小さな焦点にはじまりしだいにその焦点に集まっていった(図2a).これを定量化したところ,焦点は2~3分かけて直線性をもって蛍光強度を増し,そののち定常状態に達することがわかった.定常状態では細胞全体の蛍光シグナルの30~40%が焦点に集まっていた.1細胞あたりのGFP融合型クランプの数は約600分子であったので,これが細胞全体の蛍光シグナルをなしていると想定するとクランプは毎秒1分子の速度で焦点に集まり,最終的に約200分子からなる超高次な集合体を形成していると算出された.
岡崎フラグメントのサイズは1~2 kbなので,複製フォークが両方向に進んでいるときには(片方あたりの進行速度は毎秒0.5 kb),毎秒0.5~1フラグメントの速度で岡崎フラグメントが合成されていく.この速度はクランプの集合速度(毎秒1分子)に近い.もしかすると,岡崎フラグメントごとに1分子のクランプがDNAに装着され,これが蓄積することによって焦点の形成が導かれているのかもしれない.実際に,リングをつくれずDNAにとどまることができなくなったクランプ変異体では焦点を形成できなくなっていた.
そこで,岡崎フラグメント合成の開始頻度を低下させることによって,クランプの集合速度を低下させることができるかどうか確かめた.in vitro複製反応系ではDnaG(プライマーゼ)の濃度を低下させることにより岡崎フラグメント合成の開始頻度を低下させることができる9)(ひとつひとつの岡崎フラグメントが長くなる).そこで,プロモーターをIPTG誘導性のものに置換し細胞におけるDnaG濃度を操作できるような株を構築した.この株を用いてDnaG濃度を減少させると,期待どおりクランプの集合速度が約1/3に低下した.また,この速度低下に応じて集合にかかる時間も3倍に延びていた.
クランプが染色体のどの領域に集合しているかを調べた.複製を同調的に開始し経時的にサンプリングした細胞についてHisタグ化クランプを利用したクロマチンアフィニティー沈降実験を行った.その結果,クランプの集合した領域が複製起点の近傍からその左右へと複製フォークをおうようにして染色体を移動していくようすが検出された.
集合したクランプはどのようにして染色体を移動するのだろうか.進行している複製フォークには岡崎フラグメントの合成のため新しいクランプがつぎつぎと導入されているはずである.だとすると,複製フォークの下流に残留した古いクランプはどんどん解離していっているのではないだろうか.実際に,DNAポリメラーゼ阻害剤を利用して複製フォークの進行を停止させたところ,GFP融合型クランプの焦点からの解離が検出された(図2a).この解離は定数0.004 sec-1(半減期3分)で表わされる1次反応にしたがうものであった.in vitroではクランプはDNAに安定に維持されるので(半減期2時間),この結果はクランプを積極的に解離する機構の存在を示唆した.
焦点においてたえずクランプ分子の入れ替えがなされているのならば,焦点の蛍光をレーザー照射により光退色させるとそののちの蛍光の回復(fluorescence recovery after photobleaching:FRAP,光退色後蛍光回復)を焦点において観察することができると思われた.まず,焦点を1つだけもつ細胞について検討したところ,確かに焦点において蛍光の回復(FRAP)が認められた.枯草菌では2つの姉妹染色体が細胞分裂にさきだってつぎのラウンドの複製を開始することができるが,このとき細胞は2つの焦点をもつことになる.そこで,この一方の焦点だけで蛍光を退色させたところ,この焦点の蛍光の回復(FRAP)だけでなく,この回復と共役してもう一方の焦点の蛍光が減少していくようす(fluorescence loss in photobleaching:FLIP,光退色による蛍光減少)を観察することができた(図2b).
このFRAP/FLIP解析で得られた分子動態は速度論的にクランプの装着/解離サイクルによって説明できるのだろうか.これを知るため,細胞内で拡散している遊離クランプ分子と,DNAに装着され焦点をなしているクランプ分子とをコンピューターにおいてモデル化し,その動態の2次元シミュレーションを行った.クランプの装着速度は新規のプライマー形成により制限される0次反応式で表わされる.一方,クランプの解離速度は1次反応にしたがう.この想定のもと構築した数理モデルは,1つの焦点をもつ細胞におけるFRAPの挙動,および,2つの焦点をもつ細胞でみられたFRAPおよびFLIPの挙動をうまく再現した.
一方,複製の開始ののちにクランプが集合していく挙動をシミュレーションしたところ,定常期に達するまでのあいだはクランプの解離反応の制御されていることが示唆された.おそらく,クランプの解離反応は細胞における遊離のクランプ濃度の減少と共役して起こり,とくに複数の焦点を生じる場合などに遊離のクランプの枯渇することを防いでいるものと考えられた.実際,2つの焦点を生じる細胞において,2番目の焦点の形成にともない,さきに生じた焦点からのクランプの解離の促進されるようすが検出された.
これまでクランプと結合する数多くのタンパク質が知られており,その多くは共通して5アミノ酸残基からなるクランプ結合配列(コンセンサス配列:Gln-Leu-Ser/Asp-Leu-Phe)をもっている10).この配列を付加したGFPはN末端あるいはC末端への付加にかかわらずクランプの集合部位に焦点を形成できるようになった.また,この焦点の形成にはクランプの集合が必要であった.これらのことから,集合したクランプはクランプ結合タンパク質を複製焦点に局在化させる役割を担っていることが示された.
進行中の複製フォークの下流ではたえず大量のクランプが装着および解離をくり返しながら動的に蓄積しクランプゾーンをなしていることがわかった.また,in vivoにおいてクランプの解離機構が存在し,それが制御的に発動されていることが示唆された.クランプの解離とその制御の分子機構ついて今後の解明が期待される.
細胞内において染色体は小さく折りたたまれているので,クランプゾーンは焦点として検出される.この焦点はまさに細胞におけるDNA複製の場である.クランプゾーンはそこに多様なタンパク質をよび込むプラットフォームとして機能し,複製や複製と共役して起こるさまざまな反応をオーガナイズしているのであろう.
略歴:2003年 九州大学大学院薬学研究科博士課程 中退,同年 同 助手,2005年 博士(薬学)取得,2008年より英国Newcastle大学 ポスドク.
研究テーマ:細菌の染色体の複製(生化学・細胞生物学).
関心事:生化学反応をin vivoでみる.シンプルな生命体である細菌をモデルとして,自己複製という生命の根幹にかかわる現象にアプローチする.
© 2011 末次 正幸 Licensed under CC 表示 2.1 日本
(英国Newcastle大学Institute for Cell and Molecular Biosciences,Centre for Bacterial Cell Biology)
email:末次正幸
DOI: 10.7875/first.author.2011.065
The replicase sliding clamp dynamically accumulates behind progressing replication forks in Bacillus subtilis cells.
Masayuki Su'etsugu & Jeff Errington
Molecular Cell, 41, 720-732 (2011)
要 約
クランプはDNA複製装置の構成因子であるとともに,DNA修復や岡崎フラグメントの成熟化などDNA代謝にかかわる多様な反応に機能している.筆者らは,枯草菌のクランプの細胞内における動態を顕微鏡によりとらえることで,複製開始ののち細胞内に散らばっていたクランプが毎秒1分子の速度で集合し約200分子からなる“クランプゾーン”を形成することを見い出した.この集合は岡崎フラグメントの合成ごとに装着されたクランプがDNAに残留することにより導かれていた.一定量のクランプが蓄積するとクランプの解離反応が進行する.そして,クランプゾーンはクランプ分子の装着と解離をくり返しながら複製フォークとともに染色体を移動する.この分子動態を数理モデル化してシミュレーションを行うと,その結果は実際に細胞において検出されたクランプの動的なふるまいをうまく再現した.筆者らはさらに,クランプゾーンがタンパク質を複製焦点へと局在化させる足場として機能していること示した.
はじめに
DNAを複製している細胞では複製に機能するタンパク質がいくつも集まって細胞において焦点をなしている1).この焦点は複製焦点とよばれ,細菌から哺乳動物にいたるまで広く観察されている.しかしながら,複製に機能するタンパク質がどのようにして複製焦点を形成するのかはよくわかっていない.大腸菌や枯草菌といった細菌は基本的に環状の染色体を1つだけもちその複製起点(oriC)も1つだけである.よって,通常,1つもしくは2つの複製焦点が形成されている2)(図1a).このシンプルな構造は複製に機能するタンパク質の細胞での挙動を調べるうえで都合がよい.
細菌の染色体の複製はその多くのステップが精製タンパク質を用いてin vitro再構成されており詳細な解析がなされている3,4).複製開始ののち,複製起点の2本鎖DNAの開裂によって生じた1本鎖DNAにプライマーが形成され,そこにクランプが装着される.クランプはDnaNホモ2量体からなり,そのリングのなかに2本鎖DNAを通すので,構造上,安定にDNAに維持される5).クランプは複製反応においてDNAポリメラーゼをDNAにつなぎとめておく役割をはたす.複製フォークの進行は互いに逆向きの鋳型鎖を生じる.このためリーディング鎖は継続的に合成される一方,ラギング鎖は岡崎フラグメントとして知られる1~2 kbの断片ごとに不連続合成される6).岡崎フラグメントの合成はプライマーの形成とそこへのクランプの装着により開始する.岡崎フラグメントの合成ののちDNAポリメラーゼはクランプから外れるので,in vitroではクランプは単独でDNAに残留する7,8)(図1b).では,in vivoにおいてもクランプはDNAに残留しつづけるのだろうか? 細胞は数百個しかないクランプ分子により染色体の複製あたり2000~4000もの岡崎フラグメントを合成しなければならない.だとすると,残留したクランプを積極的に解離してリサイクルする機構があると考えたほうがよい.しかしながら,in vivoにおいてクランプを解離する機構があるのかどうか知られていない.
今回,筆者らは,クランプが岡崎フラグメントの合成ごとに装着→残留→解離のサイクルをくり返しながら,複製フォークの近傍に超高次な集合体(焦点)をなしているようすをin vivoにおいてとらえることに成功した.
1.複製開始ののちクランプは徐々に焦点へと集まっていく
クランプがどのようにして焦点を形成するのかをGFP融合型クランプを用いて調べた.複製開始まえの細胞では予想どおりクランプは細胞全体に拡散していた.複製が開始されると細胞内に散らばっていたクランプ分子はごく小さな焦点にはじまりしだいにその焦点に集まっていった(図2a).これを定量化したところ,焦点は2~3分かけて直線性をもって蛍光強度を増し,そののち定常状態に達することがわかった.定常状態では細胞全体の蛍光シグナルの30~40%が焦点に集まっていた.1細胞あたりのGFP融合型クランプの数は約600分子であったので,これが細胞全体の蛍光シグナルをなしていると想定するとクランプは毎秒1分子の速度で焦点に集まり,最終的に約200分子からなる超高次な集合体を形成していると算出された.
2.クランプの集合速度と岡崎フラグメント合成の開始頻度との相関
岡崎フラグメントのサイズは1~2 kbなので,複製フォークが両方向に進んでいるときには(片方あたりの進行速度は毎秒0.5 kb),毎秒0.5~1フラグメントの速度で岡崎フラグメントが合成されていく.この速度はクランプの集合速度(毎秒1分子)に近い.もしかすると,岡崎フラグメントごとに1分子のクランプがDNAに装着され,これが蓄積することによって焦点の形成が導かれているのかもしれない.実際に,リングをつくれずDNAにとどまることができなくなったクランプ変異体では焦点を形成できなくなっていた.
そこで,岡崎フラグメント合成の開始頻度を低下させることによって,クランプの集合速度を低下させることができるかどうか確かめた.in vitro複製反応系ではDnaG(プライマーゼ)の濃度を低下させることにより岡崎フラグメント合成の開始頻度を低下させることができる9)(ひとつひとつの岡崎フラグメントが長くなる).そこで,プロモーターをIPTG誘導性のものに置換し細胞におけるDnaG濃度を操作できるような株を構築した.この株を用いてDnaG濃度を減少させると,期待どおりクランプの集合速度が約1/3に低下した.また,この速度低下に応じて集合にかかる時間も3倍に延びていた.
3.クランプの集合は複製フォークの進行と共役している
クランプが染色体のどの領域に集合しているかを調べた.複製を同調的に開始し経時的にサンプリングした細胞についてHisタグ化クランプを利用したクロマチンアフィニティー沈降実験を行った.その結果,クランプの集合した領域が複製起点の近傍からその左右へと複製フォークをおうようにして染色体を移動していくようすが検出された.
集合したクランプはどのようにして染色体を移動するのだろうか.進行している複製フォークには岡崎フラグメントの合成のため新しいクランプがつぎつぎと導入されているはずである.だとすると,複製フォークの下流に残留した古いクランプはどんどん解離していっているのではないだろうか.実際に,DNAポリメラーゼ阻害剤を利用して複製フォークの進行を停止させたところ,GFP融合型クランプの焦点からの解離が検出された(図2a).この解離は定数0.004 sec-1(半減期3分)で表わされる1次反応にしたがうものであった.in vitroではクランプはDNAに安定に維持されるので(半減期2時間),この結果はクランプを積極的に解離する機構の存在を示唆した.
4.クランプの動的なふるまいと数理モデルによるシミュレーション
焦点においてたえずクランプ分子の入れ替えがなされているのならば,焦点の蛍光をレーザー照射により光退色させるとそののちの蛍光の回復(fluorescence recovery after photobleaching:FRAP,光退色後蛍光回復)を焦点において観察することができると思われた.まず,焦点を1つだけもつ細胞について検討したところ,確かに焦点において蛍光の回復(FRAP)が認められた.枯草菌では2つの姉妹染色体が細胞分裂にさきだってつぎのラウンドの複製を開始することができるが,このとき細胞は2つの焦点をもつことになる.そこで,この一方の焦点だけで蛍光を退色させたところ,この焦点の蛍光の回復(FRAP)だけでなく,この回復と共役してもう一方の焦点の蛍光が減少していくようす(fluorescence loss in photobleaching:FLIP,光退色による蛍光減少)を観察することができた(図2b).
このFRAP/FLIP解析で得られた分子動態は速度論的にクランプの装着/解離サイクルによって説明できるのだろうか.これを知るため,細胞内で拡散している遊離クランプ分子と,DNAに装着され焦点をなしているクランプ分子とをコンピューターにおいてモデル化し,その動態の2次元シミュレーションを行った.クランプの装着速度は新規のプライマー形成により制限される0次反応式で表わされる.一方,クランプの解離速度は1次反応にしたがう.この想定のもと構築した数理モデルは,1つの焦点をもつ細胞におけるFRAPの挙動,および,2つの焦点をもつ細胞でみられたFRAPおよびFLIPの挙動をうまく再現した.
一方,複製の開始ののちにクランプが集合していく挙動をシミュレーションしたところ,定常期に達するまでのあいだはクランプの解離反応の制御されていることが示唆された.おそらく,クランプの解離反応は細胞における遊離のクランプ濃度の減少と共役して起こり,とくに複数の焦点を生じる場合などに遊離のクランプの枯渇することを防いでいるものと考えられた.実際,2つの焦点を生じる細胞において,2番目の焦点の形成にともない,さきに生じた焦点からのクランプの解離の促進されるようすが検出された.
5.クランプの集合の役割
これまでクランプと結合する数多くのタンパク質が知られており,その多くは共通して5アミノ酸残基からなるクランプ結合配列(コンセンサス配列:Gln-Leu-Ser/Asp-Leu-Phe)をもっている10).この配列を付加したGFPはN末端あるいはC末端への付加にかかわらずクランプの集合部位に焦点を形成できるようになった.また,この焦点の形成にはクランプの集合が必要であった.これらのことから,集合したクランプはクランプ結合タンパク質を複製焦点に局在化させる役割を担っていることが示された.
おわりに
進行中の複製フォークの下流ではたえず大量のクランプが装着および解離をくり返しながら動的に蓄積しクランプゾーンをなしていることがわかった.また,in vivoにおいてクランプの解離機構が存在し,それが制御的に発動されていることが示唆された.クランプの解離とその制御の分子機構ついて今後の解明が期待される.
細胞内において染色体は小さく折りたたまれているので,クランプゾーンは焦点として検出される.この焦点はまさに細胞におけるDNA複製の場である.クランプゾーンはそこに多様なタンパク質をよび込むプラットフォームとして機能し,複製や複製と共役して起こるさまざまな反応をオーガナイズしているのであろう.
文 献
- Lemon, K. P. & Grossman, A. D.: Localization of bacterial DNA polymerase: evidence for a factory model of replication. Science, 282, 1516-1519 (1998)[PubMed]
- Berkmen, M. B. & Grossman, A. D.: Spatial and temporal organization of the Bacillus subtilis replication cycle. Mol. Microbiol., 62, 57-71(2006)[PubMed]
- Kornberg, A. & Baker, T. A.: DNA Replication, 2nd ed. W. H. Freeman, New York (1992)
- Sanders, G. M., Dallmann, H. G. & McHenry, C. S.: Reconstitution of the B. subtilis replisome with 13 proteins including two distinct replicases. Mol. Cell, 37, 273-281 (2010)[PubMed]
- Johnson, A. & O'Donnell, M.: Cellular DNA replicases: components and dynamics at the replication fork. Annu. Rev. Biochem., 74, 283-315 (2005)[PubMed]
- Ogawa, T. & Okazaki, T.: Discontinuous DNA replication. Annu. Rev. Biochem., 49, 421-457 (1980)[PubMed]
- Stukenberg, P. T., Turner, J. & O'Donnell, M.: An explanation for lagging strand replication: polymerase hopping among DNA sliding clamps. Cell, 78, 877-887 (1994)[PubMed]
- Yuzhakov, A., Turner, J. & O'Donnell, M.: Replisome assembly reveals the basis for asymmetric function in leading and lagging strand replication. Cell, 86, 877-886 (1996)[PubMed]
- Wu, C. A., Zechner, E. L. & Marians, K. J.: Coordinated leading- and lagging-strand synthesis at the Escherichia coli DNA replication fork. I. Multiple effectors act to modulate Okazaki fragment size. J. Biol. Chem., 267, 4030-4044 (1992)[PubMed]
- Dalrymple, B. P., Kongsuwan, K., Wijffels, G. et al.: A universal protein-protein interaction motif in the eubacterial DNA replication and repair systems. Proc. Natl. Acad. Sci. USA, 98, 11627-11632 (2001)[PubMed]
著者プロフィール
略歴:2003年 九州大学大学院薬学研究科博士課程 中退,同年 同 助手,2005年 博士(薬学)取得,2008年より英国Newcastle大学 ポスドク.
研究テーマ:細菌の染色体の複製(生化学・細胞生物学).
関心事:生化学反応をin vivoでみる.シンプルな生命体である細菌をモデルとして,自己複製という生命の根幹にかかわる現象にアプローチする.
© 2011 末次 正幸 Licensed under CC 表示 2.1 日本