リボソームのリン酸化を介した翻訳の制御
今見 考志
(ドイツMax Delbrück Center for Molecular Medicine)
email:今見考志
DOI: 10.7875/first.author.2018.099
Phosphorylation of the ribosomal protein RPL12/uL11 affects translation during mitosis.
Koshi Imami, Miha Milek, Boris Bogdanow, Tomoharu Yasuda, Nicolai Kastelic, Henrik Zauber, Yasushi Ishihama, Markus Landthaler, Matthias Selbach
Molecular Cell, 72, 84-98.e9 (2018)
リボソームはタンパク質を合成するだけの静的な装置であると考えられていたが,近年,リボソームそれ自体が結合タンパク質や化学修飾を介して翻訳の制御にかかわることが報告されつつある.この研究において,筆者らは,リボソームによる翻訳の制御について理解するため,ポリソームプロテオームプロファイリング法を開発した.この方法を用いることにより,リボソームの構成サブユニットやポリソームに結合するタンパク質を網羅的かつ正確に同定することが可能になった.さらに,この方法とリン酸化ペプチドを濃縮する技術とを組み合わせることによりリボソームのリン酸化をとらえることも可能になり,リボソームタンパク質RPL12のリン酸化が翻訳にかかわることが明らかにされた.このことから,リボソームのリン酸化を介した新しい遺伝子発現の制御機構の存在しうることが示唆された.
リボソームはタンパク質を合成する装置として広く認知されているが,翻訳の制御における役割についてはこれまでほとんど認識されていない.しかし,2002年,リボソームそれ自体が翻訳の制御にはたらくという“リボソームフィルター”仮説が提唱された1).これは,特定のリボソームが特定のmRNAを選択的に認識し翻訳することにより,リボソームのレベルにおいて翻訳が制御されることを示唆するものであった.実際,これまでに,“特殊化リボソーム”として,リボソームRNAやリボソームタンパク質に化学修飾をもつリボソーム,構成タンパク質の異なるリボソーム,特定の結合タンパク質をもつリボソームが発見されており,これらを介して翻訳が選択的に制御されることが報告されつつある2).一方,これまでの研究はリボソームの構成タンパク質や結合タンパク質に焦点をあてたものがほとんどであり,リボソームの翻訳後修飾による翻訳への影響について系統的に調べた研究はなかった.この研究において,筆者らは,リボソームのリン酸化に着目し,リボソームにおけるリン酸化部位を系統的に同定しその機能を解析した.
細胞には小サブユニット,大サブユニット,翻訳活性をもつモノソームやポリソームなど,異なるリボソーム複合体が存在する.そこで,翻訳活性をもつモノソームやポリソームに結合するタンパク質を系統的に同定することにより,翻訳の制御にかかわるタンパク質を同定できるのではないかと考えた.従来の免疫沈降法や酵母ツーハイブリッド法では,どのリボソーム複合体にどのタンパク質が結合するかは不明であった.そこで,筆者が過去に在籍していた研究室において開発された,カラムクロマトグラフィーから得られたタンパク質の共溶出プロファイルの類似性をもとに複合体の構成タンパク質を同定する技術から着想を得て3),そのコンセプトを応用することによりこの問題を解決できるのではないかと考えた.
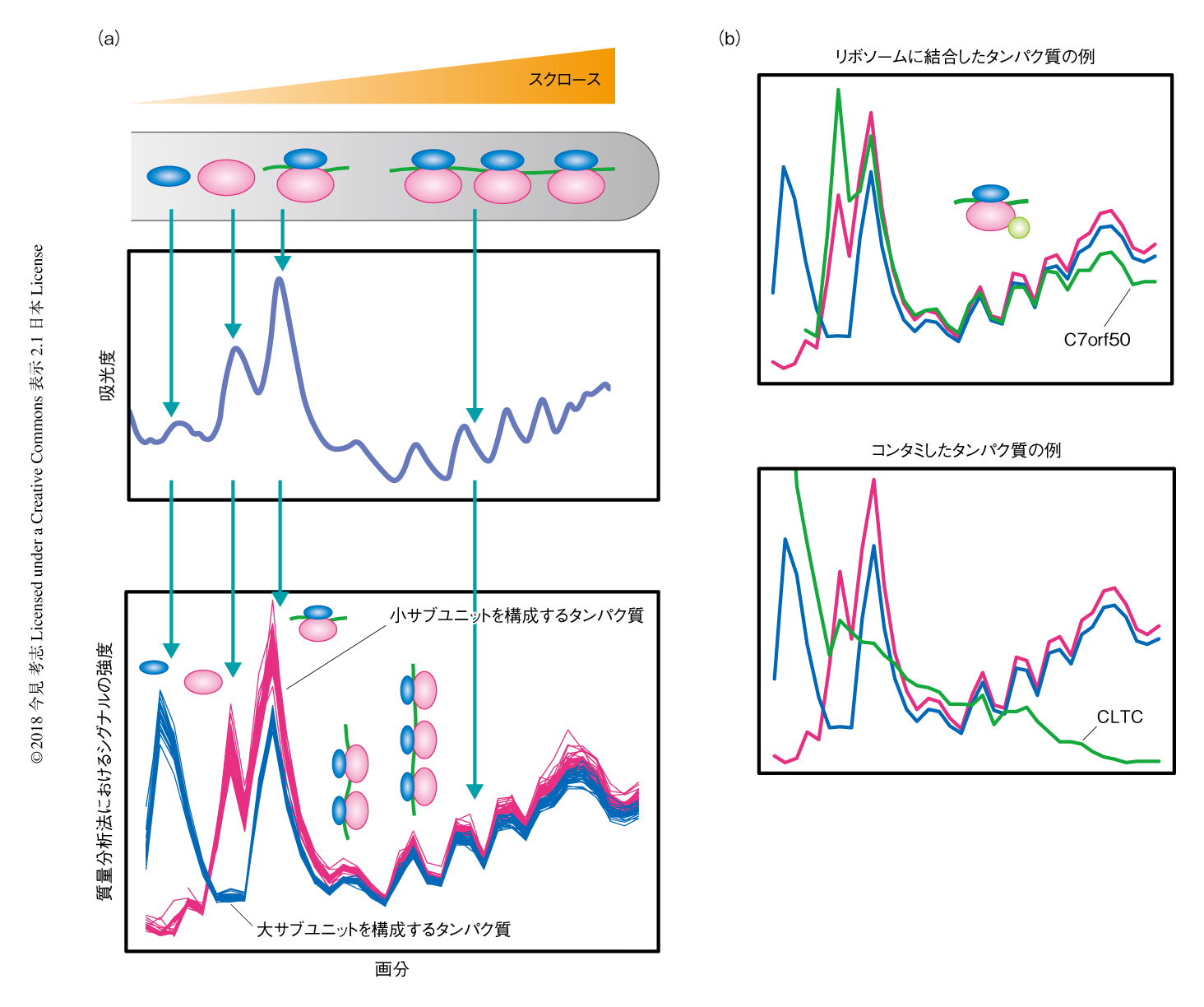
方法としては,おのおののリボソーム複合体を密度勾配遠心法により生化学的に分画し,質量分析計を用いて密度勾配にそったタンパク質の定量プロファイルを取得する.リボソームタンパク質の定量プロファイルとの相関から,どのタンパク質がどのリボソーム複合体に結合しているかをプロテオームワイドに同定することが可能になる(図1a).この方法のポイントは,密度勾配にそったコンセンサスの定量プロファイルをもとに,真の結合タンパク質とコンタミタンパク質とを区別し,さらに,どのリボソーム複合体と結合しているかを同定できる点である(図1b).この方法をポリソームプロテオームプロファイリング法と命名し,最終的には,約150種のポリソーム結合タンパク質を同定することに成功した.
この手法の利点は,タグの付加や過剰発現が必要ないため,組織やモデル生物に応用の可能な点があげられる.一方,分画によるサンプル数の増加のため測定に長い時間を要すること,アフィニティ精製と同様に一過的なタンパク質相互作用の検出が困難であり,細胞の破砕ののちに人工的な相互作用の起こる可能性があるといった短所もある.
このポリソームプロテオームプロファイリング法と,筆者らが開発してきたリン酸化ペプチドを濃縮する技術とを組み合わせることにより,リボソーム複合体におけるリン酸化をマッピングし,さらに,特定のリボソーム複合体に特異的なリン酸化をとらえることができるのではないかと考えた.そこで,ポリソームプロテオームプロファイリング法から得られた画分に対しリン酸化ペプチドの濃縮を実施し,密度勾配にそったリン酸化の定量プロファイルを取得した.すると,リボソームタンパク質RPL12のSer38におけるリン酸化はモノソームに特異的にみられ,ポリソームにおいてほとんど検出されないことを発見した.さらに,RPL12のSer38におけるリン酸化模倣変異体を用いた解析においても同様にモノソームのみに観察されることが確認され,ポリソームプロテオームプロファイリング法の結果が裏づけられた.したがって,以上の結果から,RPL12のSer38におけるリン酸化はモノソームに特異的に観察され,このリン酸化は翻訳になんらかの影響をおよぼす可能性が示唆された.実際に,RPL12のSer38はA部位の近傍に存在したことからも,翻訳に影響するとの仮説をたてた.
RPL12のSer38におけるリン酸化の機能について理解するため,どのキナーゼによりどのようなときにリン酸化されるのか明らかにすることを試みた.RPL12のSer38の周辺のアミノ酸配列に着目したところ,CDK1およびCDK2の基質モチーフと一致した.この情報にもとづき文献を調査したところ,1)ヒトのCDK1およびCDK2はRPL12をin vitroにおいてリン酸化すること,2)酵母においてCDK1はM期にRPL12のSer38をリン酸化すること,3)ヒトの細胞においてCDK1-サイクリンB複合体はM期にRPL12と結合すること,4)ヒトの細胞においてRPL12のSer38におけるリン酸化はS期と比べM期は数十倍も高い,といった報告がなされていた.これらの独立した報告から,RPL12のSer38はM期においてCDK1によりリン酸化されることが強く支持された.さらに,酵母においても同様のことが観察されていたことから,この細胞周期に依存的なリン酸化は真核生物において保存されていることが示唆された.
RPL12のSer38におけるリン酸化がM期において翻訳に影響をおよぼすとの仮説をたてた.この仮説を検証するため,RPL12のSer38におけるリン酸化模倣変異体あるいは非リン酸化変異体を過剰に発現したHEK293細胞を用いて,RPL12のSer38におけるリン酸化のグローバルな翻訳に対する影響について検証した.メチオニンの類似体であるアジドホモアラニンにより新生タンパク質をパルス標識し,フローサイトメトリー法を用いて新生タンパク質への取り込みを1細胞のレベルでモニターした.また,M期のマーカータンパク質であるリン酸化ヒストンH3も同時にモニターすることにより,M期および間期におけるタンパク質の合成量を定量した.その結果,細胞周期にかかわらず,タンパク質の合成量は野生型とリン酸化模倣変異体あるいは非リン酸化変異体とで有意な差はみられなかった.このことから,RPL12のSer38におけるリン酸化はグローバルな翻訳には影響しないことが示唆された.一方,リン酸化模倣変異体および非リン酸化変異体は野生型と比べM期の長さがわずかではあるが減少した.なお,最近の報告において,モノソームそれ自体も特定のmRNAの翻訳に活発にかかわることが示されており4),モノソームだけでも十分にタンパク質を合成する装置として機能することをつけくわえる.
RPL12のSer38におけるリン酸化が特定のmRNA,ここでは,M期に関連するmRNAの翻訳に影響するとの仮説をたてた.まず,パルスSILAC(stable isotope labeling by amino acids in cell culture)法を用いてこの仮説について検証した.この方法では,RPL12のSer38におけるリン酸化模倣変異体あるいは非リン酸化変異体を過剰に発現した細胞に対し,安定同位体で標識したアミノ酸により新生タンパク質をパルス標識し,質量分析によりプロテオームワイドに個々のタンパク質の合成量を定量することができる.すでにリボソームプロファイリング法を用いた研究により,どのようなmRNAがM期やS期において活発に翻訳されるかは報告されている5).そこで,それらの情報をもとに解析した結果,リン酸化模倣変異体はM期に関連するmRNAをより選択的に翻訳すること,また逆に,非リン酸化変異体はS期に関連するmRNAをより選択的に翻訳することが見い出された.
この結果を別の手法により検証するため,RPL12のSer38におけるリン酸化模倣変異体を含むモノソームは非リン酸化変異体を含むモノソームよりもM期に関連するmRNAとより選択的に結合するとの仮説をたてた.そこで,野生型のモノソームあるいは変異型のモノソームに結合したmRNAを免疫沈降しRNA-seq法により定量した.その結果,リン酸化模倣変異体は非リン酸化変異体よりもM期に関連するmRNAとより選択的に結合することが明らかにされた.このことから仮説は支持され,また,パルスSILAC法から得られた結果も独立に支持された.
では,どういった配列の特徴をもつmRNAがRPL12のSer38においてリン酸化されたリボソームにより選択的に翻訳されるのであろうか? この問題を解明するため,RPL12のSer38におけるリン酸化模倣変異体との選択的な結合と相関するようなmRNAの配列の特徴をRNA-seq法の結果から得ることを試みた.その結果,コドンの3番目の塩基がAあるいはUであるmRNAはリン酸模倣変異体と,GあるいはCであるmRNAは非リン酸化変異体と,選択的に結合することが明らかにされた.これは,M期に関連するmRNAのコドンの3番目の塩基はAあるいはUであるという特徴をもつという報告と一致した6).したがって,RPL12のSer38においてリン酸化されたリボソームはコドンの3番目の塩基がAあるいはUであるmRNAをより選択的に翻訳することが示唆された.
さらに最近の報告から,M期のクロマチンには転写活性がないと考えられてきたが実際には転写の起こっていることがわかってきた7).この報告と筆者らのデータとを照らしあわせたところ,M期において転写されるmRNAはRPL12のSer38におけるリン酸化模倣変異体により選択的に翻訳されることが明らかにされた.このことから,RPL12のSer38におけるリン酸化と有糸分裂との関係がさらに関連づけられた.また,M期において選択的に転写されるmRNAはRPL12のSer38におけるリン酸化模倣変異体により選択的に翻訳されるわけではなかった.つまり,M期におけるタンパク質の合成は少なくとも2つの機構,転写レベルおよびRPL12のリン酸化を介した翻訳レベルにおいて制御されることが示唆された.
これまでの実験はヒトのHEK293細胞を用いたものであり,ほかの系においても同様の翻訳の制御が起こっているのかマウスのB細胞を用いて検証した.CRISPR-Cas9を利用したゲノム編集によるリン酸化変異体のノックイン細胞を用いたところ,HEK293細胞と一致する結果が得られた.さらに,リボソームプロファイリング法により,これまでのデータを支持する結果が得られた.以上をまとめると,パルスSILAC法,RNA-seq法,リボソームプロファイリング法の3つの独立した手法,および,ヒトのHEK293細胞とマウスのB細胞の2つの異なるモデル系を用いた実験から得られた結論は一致し,RPL12のSer38におけるリン酸化はM期における翻訳に影響をおよぼすと結論づけられた.また,酵母,マウス,ヒトにおいてM期におけるRPL12のSer38におけるリン酸化が保存されていることも考慮すると,このリン酸化されたリボソームによる翻訳の制御は真核生物において保存されている可能性が考えられた.
この研究において,筆者らは,これまでに確立された古典的な生化学的な手法とプロテオミクスの技術とを組み合わせることにより,リボソーム複合体におけるリン酸化をマッピングし,リボソームタンパク質RPL12のリン酸化が翻訳の制御にかかわることを明らかにした.さまざまな手法やモデル系を用いて解析したが,いずれもRPL12のSer38におけるリン酸化がM期における翻訳に影響をおよぼすという結論が導かれた.当然ながら,未解決なこともたくさんある.まず,リン酸化されたリボソームがどのように特定のmRNAを選択に翻訳するのか,その機序は不明である.また,このリン酸化はなぜモノソームにのみ観察され,ポリソームには観察されないのか? リン酸化されたモノソームと翻訳の制御との関係性は何か? さらに,RPL12のSer38におけるリン酸化模倣変異体あるいは非リン酸化変異体を過剰に発現させても,M期の長さがわずかに減少する程度で顕著な表現型はみられなかった.したがって,マイクロRNAのように,多数の標的に対しわずかなタンパク質量の変化をひき起こすことによりプロテオームを精密にチューニングしている可能性も考えられる.
また,同じ時期にサイズ排除クロマトグラフィーを用いたリボソームの分離法も報告されており8),超遠心法に依存しない新しい分離法の登場がこの分野をさらに進展させることを期待する.
最後に,特殊化リボソームによる翻訳の制御が最近のトレンドになりつつあるが,その意義については依然として不明な点が多い.さまざまなリボソームが存在することは確かに報告されているが,このようなリボソームの不均一性が細胞におけるイベントにどれぐらいの意味をもって寄与するのかは不明である.また,特殊化リボソームではなく,単純にmRNAに特異的な翻訳開始速度および細胞におけるリボソームの濃度が翻訳レベルでの遺伝子の発現制御を説明するのに十分であるという意見もある9).今回,偶然にもリボソームのリン酸化を介した翻訳の制御を見い出すにいたったが,客観的な目で,これからでてくるデータをみていきたい.
略歴:2010年 慶應義塾大学大学院政策・メディア研究科後期博士課程 修了,同年 カナダBritish Columbia大学 博士研究員,2015年 ドイツMax Delbrück Center for Molecular Medicine博士研究員,2017年 京都大学大学院薬学研究科 特任助教を経て,2018年より科学技術振興機構 さきがけ研究員.
研究テーマ:シグナル伝達と遺伝子発現のクロストーク.
© 2018 今見 考志 Licensed under CC 表示 2.1 日本
(ドイツMax Delbrück Center for Molecular Medicine)
email:今見考志
DOI: 10.7875/first.author.2018.099
Phosphorylation of the ribosomal protein RPL12/uL11 affects translation during mitosis.
Koshi Imami, Miha Milek, Boris Bogdanow, Tomoharu Yasuda, Nicolai Kastelic, Henrik Zauber, Yasushi Ishihama, Markus Landthaler, Matthias Selbach
Molecular Cell, 72, 84-98.e9 (2018)
要 約
リボソームはタンパク質を合成するだけの静的な装置であると考えられていたが,近年,リボソームそれ自体が結合タンパク質や化学修飾を介して翻訳の制御にかかわることが報告されつつある.この研究において,筆者らは,リボソームによる翻訳の制御について理解するため,ポリソームプロテオームプロファイリング法を開発した.この方法を用いることにより,リボソームの構成サブユニットやポリソームに結合するタンパク質を網羅的かつ正確に同定することが可能になった.さらに,この方法とリン酸化ペプチドを濃縮する技術とを組み合わせることによりリボソームのリン酸化をとらえることも可能になり,リボソームタンパク質RPL12のリン酸化が翻訳にかかわることが明らかにされた.このことから,リボソームのリン酸化を介した新しい遺伝子発現の制御機構の存在しうることが示唆された.
はじめに
リボソームはタンパク質を合成する装置として広く認知されているが,翻訳の制御における役割についてはこれまでほとんど認識されていない.しかし,2002年,リボソームそれ自体が翻訳の制御にはたらくという“リボソームフィルター”仮説が提唱された1).これは,特定のリボソームが特定のmRNAを選択的に認識し翻訳することにより,リボソームのレベルにおいて翻訳が制御されることを示唆するものであった.実際,これまでに,“特殊化リボソーム”として,リボソームRNAやリボソームタンパク質に化学修飾をもつリボソーム,構成タンパク質の異なるリボソーム,特定の結合タンパク質をもつリボソームが発見されており,これらを介して翻訳が選択的に制御されることが報告されつつある2).一方,これまでの研究はリボソームの構成タンパク質や結合タンパク質に焦点をあてたものがほとんどであり,リボソームの翻訳後修飾による翻訳への影響について系統的に調べた研究はなかった.この研究において,筆者らは,リボソームのリン酸化に着目し,リボソームにおけるリン酸化部位を系統的に同定しその機能を解析した.
1.ポリソームプロテオームプロファイリング法はポリソームの構成タンパク質を正確に同定する
細胞には小サブユニット,大サブユニット,翻訳活性をもつモノソームやポリソームなど,異なるリボソーム複合体が存在する.そこで,翻訳活性をもつモノソームやポリソームに結合するタンパク質を系統的に同定することにより,翻訳の制御にかかわるタンパク質を同定できるのではないかと考えた.従来の免疫沈降法や酵母ツーハイブリッド法では,どのリボソーム複合体にどのタンパク質が結合するかは不明であった.そこで,筆者が過去に在籍していた研究室において開発された,カラムクロマトグラフィーから得られたタンパク質の共溶出プロファイルの類似性をもとに複合体の構成タンパク質を同定する技術から着想を得て3),そのコンセプトを応用することによりこの問題を解決できるのではないかと考えた.
方法としては,おのおののリボソーム複合体を密度勾配遠心法により生化学的に分画し,質量分析計を用いて密度勾配にそったタンパク質の定量プロファイルを取得する.リボソームタンパク質の定量プロファイルとの相関から,どのタンパク質がどのリボソーム複合体に結合しているかをプロテオームワイドに同定することが可能になる(図1a).この方法のポイントは,密度勾配にそったコンセンサスの定量プロファイルをもとに,真の結合タンパク質とコンタミタンパク質とを区別し,さらに,どのリボソーム複合体と結合しているかを同定できる点である(図1b).この方法をポリソームプロテオームプロファイリング法と命名し,最終的には,約150種のポリソーム結合タンパク質を同定することに成功した.
この手法の利点は,タグの付加や過剰発現が必要ないため,組織やモデル生物に応用の可能な点があげられる.一方,分画によるサンプル数の増加のため測定に長い時間を要すること,アフィニティ精製と同様に一過的なタンパク質相互作用の検出が困難であり,細胞の破砕ののちに人工的な相互作用の起こる可能性があるといった短所もある.
2.リボソームタンパク質RPL12のリン酸化はモノソームにおいて顕著に観察される
このポリソームプロテオームプロファイリング法と,筆者らが開発してきたリン酸化ペプチドを濃縮する技術とを組み合わせることにより,リボソーム複合体におけるリン酸化をマッピングし,さらに,特定のリボソーム複合体に特異的なリン酸化をとらえることができるのではないかと考えた.そこで,ポリソームプロテオームプロファイリング法から得られた画分に対しリン酸化ペプチドの濃縮を実施し,密度勾配にそったリン酸化の定量プロファイルを取得した.すると,リボソームタンパク質RPL12のSer38におけるリン酸化はモノソームに特異的にみられ,ポリソームにおいてほとんど検出されないことを発見した.さらに,RPL12のSer38におけるリン酸化模倣変異体を用いた解析においても同様にモノソームのみに観察されることが確認され,ポリソームプロテオームプロファイリング法の結果が裏づけられた.したがって,以上の結果から,RPL12のSer38におけるリン酸化はモノソームに特異的に観察され,このリン酸化は翻訳になんらかの影響をおよぼす可能性が示唆された.実際に,RPL12のSer38はA部位の近傍に存在したことからも,翻訳に影響するとの仮説をたてた.
3.RPL12はM期においてCDK1によりリン酸化される
RPL12のSer38におけるリン酸化の機能について理解するため,どのキナーゼによりどのようなときにリン酸化されるのか明らかにすることを試みた.RPL12のSer38の周辺のアミノ酸配列に着目したところ,CDK1およびCDK2の基質モチーフと一致した.この情報にもとづき文献を調査したところ,1)ヒトのCDK1およびCDK2はRPL12をin vitroにおいてリン酸化すること,2)酵母においてCDK1はM期にRPL12のSer38をリン酸化すること,3)ヒトの細胞においてCDK1-サイクリンB複合体はM期にRPL12と結合すること,4)ヒトの細胞においてRPL12のSer38におけるリン酸化はS期と比べM期は数十倍も高い,といった報告がなされていた.これらの独立した報告から,RPL12のSer38はM期においてCDK1によりリン酸化されることが強く支持された.さらに,酵母においても同様のことが観察されていたことから,この細胞周期に依存的なリン酸化は真核生物において保存されていることが示唆された.
4.RPL12のリン酸化はグローバルな翻訳には影響しない
RPL12のSer38におけるリン酸化がM期において翻訳に影響をおよぼすとの仮説をたてた.この仮説を検証するため,RPL12のSer38におけるリン酸化模倣変異体あるいは非リン酸化変異体を過剰に発現したHEK293細胞を用いて,RPL12のSer38におけるリン酸化のグローバルな翻訳に対する影響について検証した.メチオニンの類似体であるアジドホモアラニンにより新生タンパク質をパルス標識し,フローサイトメトリー法を用いて新生タンパク質への取り込みを1細胞のレベルでモニターした.また,M期のマーカータンパク質であるリン酸化ヒストンH3も同時にモニターすることにより,M期および間期におけるタンパク質の合成量を定量した.その結果,細胞周期にかかわらず,タンパク質の合成量は野生型とリン酸化模倣変異体あるいは非リン酸化変異体とで有意な差はみられなかった.このことから,RPL12のSer38におけるリン酸化はグローバルな翻訳には影響しないことが示唆された.一方,リン酸化模倣変異体および非リン酸化変異体は野生型と比べM期の長さがわずかではあるが減少した.なお,最近の報告において,モノソームそれ自体も特定のmRNAの翻訳に活発にかかわることが示されており4),モノソームだけでも十分にタンパク質を合成する装置として機能することをつけくわえる.
5.RPL12のリン酸化はM期に関連するmRNAの翻訳に影響をおよぼす
RPL12のSer38におけるリン酸化が特定のmRNA,ここでは,M期に関連するmRNAの翻訳に影響するとの仮説をたてた.まず,パルスSILAC(stable isotope labeling by amino acids in cell culture)法を用いてこの仮説について検証した.この方法では,RPL12のSer38におけるリン酸化模倣変異体あるいは非リン酸化変異体を過剰に発現した細胞に対し,安定同位体で標識したアミノ酸により新生タンパク質をパルス標識し,質量分析によりプロテオームワイドに個々のタンパク質の合成量を定量することができる.すでにリボソームプロファイリング法を用いた研究により,どのようなmRNAがM期やS期において活発に翻訳されるかは報告されている5).そこで,それらの情報をもとに解析した結果,リン酸化模倣変異体はM期に関連するmRNAをより選択的に翻訳すること,また逆に,非リン酸化変異体はS期に関連するmRNAをより選択的に翻訳することが見い出された.
この結果を別の手法により検証するため,RPL12のSer38におけるリン酸化模倣変異体を含むモノソームは非リン酸化変異体を含むモノソームよりもM期に関連するmRNAとより選択的に結合するとの仮説をたてた.そこで,野生型のモノソームあるいは変異型のモノソームに結合したmRNAを免疫沈降しRNA-seq法により定量した.その結果,リン酸化模倣変異体は非リン酸化変異体よりもM期に関連するmRNAとより選択的に結合することが明らかにされた.このことから仮説は支持され,また,パルスSILAC法から得られた結果も独立に支持された.
6.RPL12のリン酸化により選択的に翻訳されるmRNAの配列の特徴
では,どういった配列の特徴をもつmRNAがRPL12のSer38においてリン酸化されたリボソームにより選択的に翻訳されるのであろうか? この問題を解明するため,RPL12のSer38におけるリン酸化模倣変異体との選択的な結合と相関するようなmRNAの配列の特徴をRNA-seq法の結果から得ることを試みた.その結果,コドンの3番目の塩基がAあるいはUであるmRNAはリン酸模倣変異体と,GあるいはCであるmRNAは非リン酸化変異体と,選択的に結合することが明らかにされた.これは,M期に関連するmRNAのコドンの3番目の塩基はAあるいはUであるという特徴をもつという報告と一致した6).したがって,RPL12のSer38においてリン酸化されたリボソームはコドンの3番目の塩基がAあるいはUであるmRNAをより選択的に翻訳することが示唆された.
さらに最近の報告から,M期のクロマチンには転写活性がないと考えられてきたが実際には転写の起こっていることがわかってきた7).この報告と筆者らのデータとを照らしあわせたところ,M期において転写されるmRNAはRPL12のSer38におけるリン酸化模倣変異体により選択的に翻訳されることが明らかにされた.このことから,RPL12のSer38におけるリン酸化と有糸分裂との関係がさらに関連づけられた.また,M期において選択的に転写されるmRNAはRPL12のSer38におけるリン酸化模倣変異体により選択的に翻訳されるわけではなかった.つまり,M期におけるタンパク質の合成は少なくとも2つの機構,転写レベルおよびRPL12のリン酸化を介した翻訳レベルにおいて制御されることが示唆された.
7.RPL12のリン酸化を介した翻訳の制御はマウスにおいて保存されている
これまでの実験はヒトのHEK293細胞を用いたものであり,ほかの系においても同様の翻訳の制御が起こっているのかマウスのB細胞を用いて検証した.CRISPR-Cas9を利用したゲノム編集によるリン酸化変異体のノックイン細胞を用いたところ,HEK293細胞と一致する結果が得られた.さらに,リボソームプロファイリング法により,これまでのデータを支持する結果が得られた.以上をまとめると,パルスSILAC法,RNA-seq法,リボソームプロファイリング法の3つの独立した手法,および,ヒトのHEK293細胞とマウスのB細胞の2つの異なるモデル系を用いた実験から得られた結論は一致し,RPL12のSer38におけるリン酸化はM期における翻訳に影響をおよぼすと結論づけられた.また,酵母,マウス,ヒトにおいてM期におけるRPL12のSer38におけるリン酸化が保存されていることも考慮すると,このリン酸化されたリボソームによる翻訳の制御は真核生物において保存されている可能性が考えられた.
おわりに
この研究において,筆者らは,これまでに確立された古典的な生化学的な手法とプロテオミクスの技術とを組み合わせることにより,リボソーム複合体におけるリン酸化をマッピングし,リボソームタンパク質RPL12のリン酸化が翻訳の制御にかかわることを明らかにした.さまざまな手法やモデル系を用いて解析したが,いずれもRPL12のSer38におけるリン酸化がM期における翻訳に影響をおよぼすという結論が導かれた.当然ながら,未解決なこともたくさんある.まず,リン酸化されたリボソームがどのように特定のmRNAを選択に翻訳するのか,その機序は不明である.また,このリン酸化はなぜモノソームにのみ観察され,ポリソームには観察されないのか? リン酸化されたモノソームと翻訳の制御との関係性は何か? さらに,RPL12のSer38におけるリン酸化模倣変異体あるいは非リン酸化変異体を過剰に発現させても,M期の長さがわずかに減少する程度で顕著な表現型はみられなかった.したがって,マイクロRNAのように,多数の標的に対しわずかなタンパク質量の変化をひき起こすことによりプロテオームを精密にチューニングしている可能性も考えられる.
また,同じ時期にサイズ排除クロマトグラフィーを用いたリボソームの分離法も報告されており8),超遠心法に依存しない新しい分離法の登場がこの分野をさらに進展させることを期待する.
最後に,特殊化リボソームによる翻訳の制御が最近のトレンドになりつつあるが,その意義については依然として不明な点が多い.さまざまなリボソームが存在することは確かに報告されているが,このようなリボソームの不均一性が細胞におけるイベントにどれぐらいの意味をもって寄与するのかは不明である.また,特殊化リボソームではなく,単純にmRNAに特異的な翻訳開始速度および細胞におけるリボソームの濃度が翻訳レベルでの遺伝子の発現制御を説明するのに十分であるという意見もある9).今回,偶然にもリボソームのリン酸化を介した翻訳の制御を見い出すにいたったが,客観的な目で,これからでてくるデータをみていきたい.
文 献
- Mauro, V. P. & Edelman, G. M.: The ribosome filter hypothesis. Proc. Natl. Acad. Sci. USA, 99, 12031-12036 (2002)[PubMed]
- Shi, Z. & Barna, M.: Translating the genome in time and space: specialized ribosomes, RNA regulons, and RNA-binding proteins. Annu. Rev. Cell Dev. Biol., 31, 31-54 (2015)[PubMed]
- Kristensen, A. R., Gsponer, J. & Foster, L. J.: A high-throughput approach for measuring temporal changes in the interactome. Nat. Methods, 9, 907-909 (2012)[PubMed]
- Heyer, E. E. & Moore, M. J.: Redefining the translational status of 80S monosomes. Cell, 164, 757-769 (2016)[PubMed]
- Stumpf, C. R., Moreno, M. V., Olshen, A. B. et al.: The translational landscape of the mammalian cell cycle. Mol. Cell, 52, 574-582 (2013)[PubMed]
- Gingold, H., Tehler, D., Christoffersen, N. R. et al.: A dual program for translation regulation in cellular proliferation and differentiation. Cell, 158, 1281-1292 (2014)[PubMed]
- Palozola, K. C., Donahue, G., Liu, H. et al.: Mitotic transcription and waves of gene reactivation during mitotic exit. Science, 358, 119-122 (2017)[PubMed]
- Yoshikawa, H., Larance, M., Harney, D. J.: Efficient analysis of mammalian polysomes in cells and tissues using Ribo Mega-SEC. Elife, 7, e36530 (2018)[PubMed]
- Mills, E. W. & Green, R.: Ribosomopathies: there's strength in numbers. Science, 358, eaan2755 (2017)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2010年 慶應義塾大学大学院政策・メディア研究科後期博士課程 修了,同年 カナダBritish Columbia大学 博士研究員,2015年 ドイツMax Delbrück Center for Molecular Medicine博士研究員,2017年 京都大学大学院薬学研究科 特任助教を経て,2018年より科学技術振興機構 さきがけ研究員.
研究テーマ:シグナル伝達と遺伝子発現のクロストーク.
© 2018 今見 考志 Licensed under CC 表示 2.1 日本