ゲノム編集において標的の範囲の拡張されたCas9改変体の開発
西増弘志・濡木 理
(東京大学大学院理学系研究科 生物科学専攻生物化学講座構造生命科学研究室)
email:西増弘志,濡木 理
DOI: 10.7875/first.author.2018.094
Engineered CRISPR-Cas9 nuclease with expanded targeting space.
Hiroshi Nishimasu, Xi Shi, Soh Ishiguro, Linyi Gao, Seiichi Hirano, Sanae Okazaki, Taichi Noda, Omar O. Abudayyeh, Jonathan S. Gootenberg, Hideto Mori, Seiya Oura, Benjamin Holmes, Mamoru Tanaka, Motoaki Seki, Hisato Hirano, Hiroyuki Aburatani, Ryuichiro Ishitani, Masahito Ikawa, Nozomu Yachie, Feng Zhang, Osamu Nureki
Science, 361, 1259-1262 (2018)
RNA依存性DNAエンドヌクレアーゼであるCas9はゲノム編集のツールとして利用されている.しかし,Cas9がDNAを認識するには標的とする配列の近傍にPAMとよばれる特定の塩基配列が必要であるため,その適用の範囲には制限がある.この研究において,筆者らは,Cas9に7つのアミノ酸置換を導入し,1つのグアニン塩基をPAMとして認識するSpCas9-NGを開発した.SpCas9-NGはこれまで標的とすることのできなかった領域のゲノム編集を可能にし,さまざまな分野において有用なゲノム編集のツールとして利用されることが期待される.
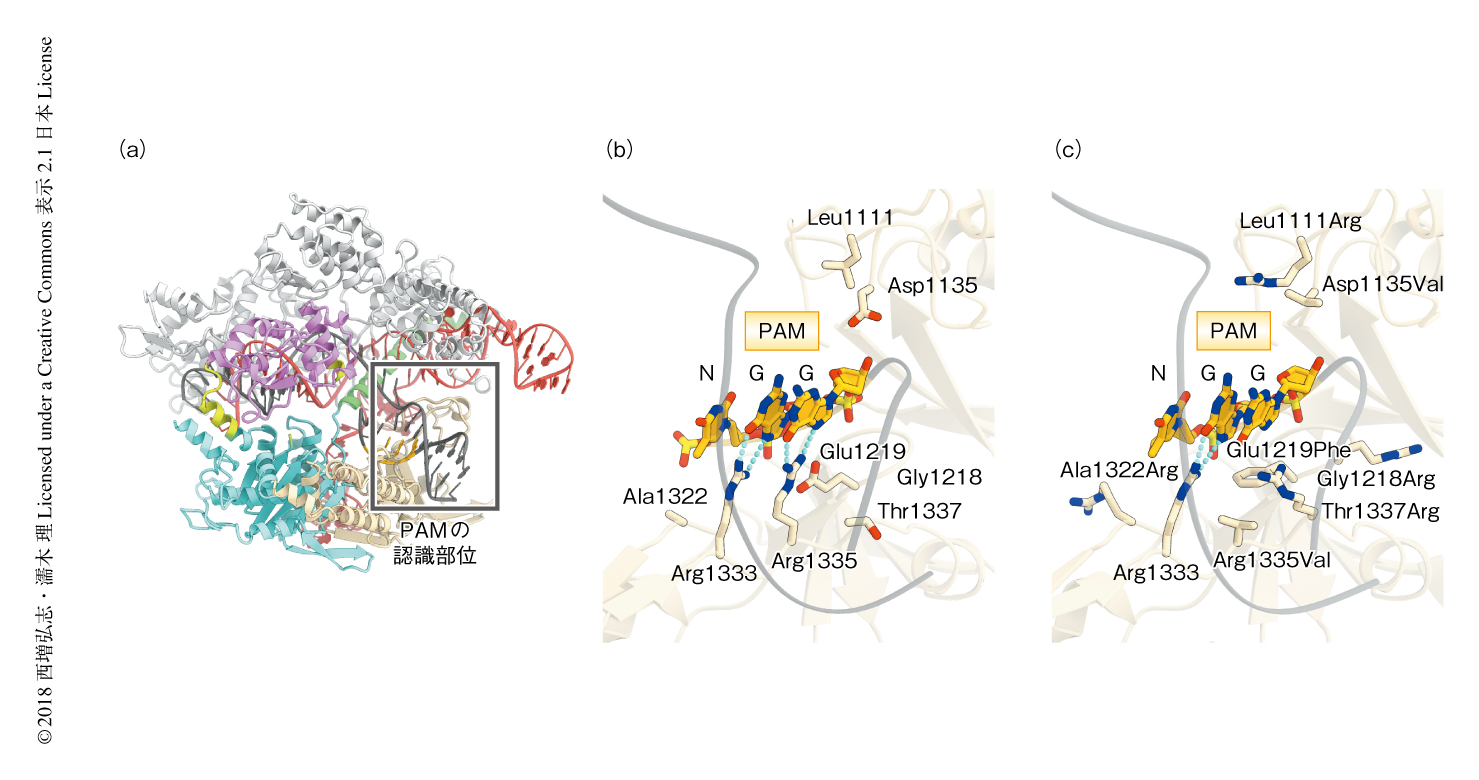
原核生物のCRISPR-Cas(CRISPR:clustered regularly interspaced short palindromic repeat,Cas:CRISPR-associated protein)獲得免疫機構に関与するRNA依存性DNAエンドヌクレアーゼであるCas9は,ガイドRNAと複合体を形成しガイド配列と相補的な2本鎖DNAを特異的に切断する1).20塩基のガイド配列は自由に設計できるため,Cas9およびガイドRNAを用いることにより2本鎖DNAをねらった位置で切断することができる.したがって,近年,Cas9を用いたゲノム編集はさまざまな分野において広く利用されている2).Cas9が標的となる2本鎖DNAを認識するためには,ガイド配列と標的配列とのあいだの相補性にくわえ,標的配列のとなりにPAM(protospacer adjacent motif)とよばれる特定の塩基配列が必要である.PAMは生物種により異なり,ゲノム編集に広く利用されているStreptococcus pyogenesに由来するCas9はNGG(N:任意の塩基)という配列をPAMとして認識する(図1a).したがって,Cas9を用いたゲノム編集には適応の範囲に制限があるという弱点が残されていた.
S. pyogenesに由来するCas9の結晶構造(PDB ID:4UN3)から,PAMの2塩基目および3塩基目のGはそれぞれArg1333およびArg1335により認識されることが明らかにされていた3)(図1b).そこで,以下の戦略により,PAMに対する特異性をNGGからNGに改変することを試みた.まず,3塩基目のGを認識するArg1335をAlaに置換し,3塩基目のGとの塩基特異的な相互作用を削除する.つぎに,DNAの糖-リン酸骨格と新たな相互作用を形成するようなアミノ酸置換を導入し,Arg1335のAlaへの置換による塩基特異的な相互作用の損失を補填する.
この仮説を確かめるため,まず,野生型Cas9およびArg1335Ala変異体の組換えタンパク質を精製し,TGGという配列をPAMとしてもつ標的DNAに対する切断活性をin vitroにおいて測定した.野生型Cas9はTGGをPAMとしてもつ標的DNAを効率よく切断した一方,Arg1335Ala変異体は標的DNAを切断しなかった.つぎに,結晶構造を参考に,Arg1335Ala置換にくわえ,DNAのリン酸骨格の近くに存在するアミノ酸残基をArgなどに置換した複数の二重変異体を設計した.これらの変異体の組換えタンパク質を精製しDNA切断活性を測定したところ,Arg1335Ala置換に,Leu1111Arg置換,Gly1218Arg置換,Ala1322Arg置換,Thr1337Arg置換のいずれかを追加した二重変異体は微弱なDNA切断活性を示した.さらに,これら4つの変異を組み合わせたところDNA切断活性が上昇した.しかし,DNA切断反応の経時変化を測定したところ,Arg1335Ala置換,Leu1111Arg置換,Gly1218Arg置換,Ala1322Arg置換,Thr1337Arg置換の5つの変異をもつARRRR変異体のDNA切断活性は,野生型Cas9よりもずっと低かった.したがって,Arg1335Ala置換による相互作用の損失を補填するには,DNAのリン酸基との相互作用を強化するだけでは不十分であると考えられた.
そこで,DNAのリボース部分と新たな相互作用を形成するようなアミノ酸変異の導入について検討した.過去に報告されたVQR改変体は,Asp1135Val置換,Arg1335Gln置換,Thr1337Arg置換の3つの変異をもち,NGAをPAMとして認識する4).結晶構造(PDB ID:5B2R)から,Val1135はDNAのリボース部分と相互作用することが明らかにされていた5,6)(文献5) は 新着論文レビュー でも掲載).また,結晶構造をもとに考察した結果,Val1335およびPhe1219はそれぞれArg1333およびDNAのリボース部分と相互作用を形成する可能性が示唆された.実際,ARRRR変異体にAsp1135Val置換,Glu1219Phe置換,Arg1335Val置換の3つの変異を追加したところ,TGGをPAMとしてもつ標的DNAに対する切断活性が大きく上昇した.
つぎに,TGA,TGT,TGG,TGCのいずれかをPAMとしてもつ4種類の標的DNAを用いて,野生型Cas9,および,Arg1335Val置換,Leu1111Arg置換,Asp1135Val置換,Gly1218Arg置換,Glu1219Phe置換,Ala1322Arg置換,Thr1337Arg置換の7つの変異をもつVRVRFRR変異体のDNA切断活性を測定した.野生型Cas9はTGGをPAMとしてもつ標的DNAを効率よく切断し,TGAをPAMとしてもつ標的DNAをわずかに切断したが,TGTあるいはTGCをPAMとしてもつ標的DNAはほとんど切断しなかった.一方,VRVRFRR変異体はTGGだけでなくTGH(H:A,T,C)をPAMとしてもつ標的DNAを効率的に切断した.さらに,標的配列のとなりに65,536(4の8乗)通りの配列をもつDNAライブラリーをin vitroにおいて切断し次世代シークエンサーを用いて解析した結果,VRVRFRR変異体はNGという配列をPAMとして認識することが確かめられた.以上の結果から,VRVRFRR変異体をSpCas9-NGと命名した.
SpCas9-NGによるPAMの認識機構を解明するため,SpCas9-NG,ガイドRNA,標的DNAからなる複合体の結晶構造を決定した(PDB ID:6AI6,図1c).PAMの2塩基目のGはArg1333と2本の水素結合を形成していた一方,3塩基目のGは特異的な相互作用を形成していなかった.Arg1111,Val1135,Phe1219はDNAの糖-リン酸骨格と相互作用していた.Arg1218およびArg1322もDNAの近傍に位置しており,DNAの糖-リン酸骨格と相互作用することが示唆された.
SpCas9-NGがゲノム編集に利用できるかどうかを検証するため,野生型Cas9あるいはSpCas9-NGおよびガイドRNAをヒト培養細胞に発現させ,NGA,NGT,NGG,NGCのいずれかをPAMとしてもつ標的部位への変異の導入について調べた.野生型Cas9はNGGをPAMとしてもつ標的部位に効率よく変異をひき起こした一方,NGTあるいはNGCをPAMとしてもつ標的部位には変異をひき起こさなかった.in vitroにおけるDNA切断活性と一致して,NGAをPAMとしてもつ標的部位には中程度の効率で変異が導入された.一方,SpCas9-NGはNGGだけでなくNGHをPAMとしてもつ標的部位にも変異をひき起こした.したがって,SpCas9-NGはゲノム編集のツールとして利用できることが示唆された.
Cas9はガイド配列と完全に相補的なDNAだけでなく,数塩基のミスマッチをもつDNAもわずかに切断する.そのため,Cas9を用いたゲノム編集においては,標的配列と類似した配列にも変異が導入されるオフターゲット効果の生じることがある.SpCas9-NGはPAMによる制約が緩和されているため,野生型Cas9と比較して,多くのオフターゲットの候補をもつ可能性が高い.そこで,EMX1遺伝子を標的とするガイドRNA,および,野生型Cas9あるいはSpCas9-NGをヒト培養細胞に発現させ,ゲノムに生じた変異をGUIDE-seq法7) を用いて網羅的に検出した.その結果,野生型Cas9およびSpCas9-NGは同じ程度のオフターゲットの変異をひき起こすことが明らかにされた.PAMに対する特異性と一致して,SpCas9-NGはNGGのほかにNGHをPAMとしてもつオフターゲット部位に変異をひき起こした.これまで,eSpCas9などオフターゲット効果の低減されたCas9改変体が報告されている8).そこで,eSpCas9のもつLys848Ala置換,Lys1003Ala置換,Arg1060Ala置換の3つの変異をSpCas9-NGに追加し,GUIDE-seq法を用いてオフターゲット効果を評価した.その結果,これら3つのアミノ酸置換の導入によりSpCas9-NGの特異性は向上することが明らかにされた.
Asp10Ala置換をもつCas9ニッカーゼとシチジンデアミナーゼとの融合タンパク質Target-AIDは,標的配列に含まれるCをTに変換する“塩基エディター”としてはたらく9).そこで,Asp10Ala置換を導入したSpCas9-NGとシチジンデアミナーゼとの融合タンパク質Target-AID-NGを作製しヒト培養細胞に発現させ,NGA,NGT,NGG,NGCのいずれかをPAMとしてもつ標的部位における塩基の置換について調べた.その結果,Target-AIDとは異なり,Target-AID-NGはNGA,NGT,NGG,NGCのいずれかをPAMとしてもつ標的部位に塩基の置換をひき起こした.したがって,DNA切断活性を欠損させたSpCas9-NGは標的の範囲の拡張したRNA依存的なDNA結合プラットフォームとして有用であることが示された.
野生型Cas9はNGGをPAMとして要求するため,確率的にゲノム領域の1/16しか標的にすることができない.一方,SpCas9-NGはNGをPAMとして認識するため,その4倍の領域を標的とすることができる.したがって,SpCas9-NGは,より広い領域を標的とすることのできる有用なゲノム編集のツールとして利用されることが期待される.
この研究は2015年6月に開始し,その4カ月後にはSpCas9-NGはほぼ完成していた.しかし,ほかのプロジェクトとの兼ね合いやゲノム編集のツールとしての性能を慎重に評価したこともあり,論文の投稿までに2年以上の歳月を要した.2018年1月4日,論文をScience誌に投稿したところ,1月29日に返信があった.2人のレビュアーのうち,1人は非常に好意的でマイナーなコメントがたったひとつ.もう1人の評価もおおむね良好であり,必要な追加実験はGUIDE-seq法を用いたオフターゲット効果の評価のみであった.筆者らにとりGUIDE-seq法ははじめての実験手法であったが,プロトコールは確立されているため問題なく結果がでるだろうと考えた.しかし,GUIDE-seq法は簡単ではなかった.細胞培養や次世代シークエンス解析などの複数のステップからなるため,実験がうまく進んだとしても結果が得られるまでに2週間はかかる.実験条件を検討しているうちに1カ月が経過した.
2月28日,事態は急転した.SpCas9-NGと同様にNGをPAMとして認識するCas9改変体であるxCas9についての論文が“Accelerated Article Preview”としてNature誌に掲載されたのだ10).この論文は筆者らの論文の約2週間後に投稿されており,わずか1カ月で受理されていた.SpCas9-NGは構造情報にもとづき合理的に設計された改変体であったのに対し,xCas9は人工進化的な手法により開発されていた.xCas9はSpCas9-NGとは異なる7つのアミノ酸置換をもっていた.Science誌のエディターに連絡したところ,要求されている追加実験を行えば問題ないとの返信があり,ひとまず胸をなでおろした.
xCas9の論文には不可解な点がいくつかあった.まず,立体構造を考慮すると,xCas9はNGをPAMとして認識するようには思えなかった.PAMの3塩基目のGを認識するArg1335は置換されておらず,また,7つのアミノ酸置換のほとんどはPAMから離れた位置に存在した.精製したxCas9のin vitroにおけるDNA切断活性は調べられていなかった.さらに,培養細胞の実験には過剰量の発現ベクターが用いられており,野生型Cas9を発現させた場合においても,NGG以外のPAMをもつ標的部位にも変異が生じていた.そこで,純粋な好奇心から,すぐさまxCas9のもつ変異を導入するためのオリゴヌクレオチドを発注し,xCas9の組換えタンパク質を精製してそのDNA切断活性を測定してみた.これが,のちに大きな意味をもつことになった.最初の実験結果をみたとき,実験条件をまちがえたのではないかと思った.NGGをPAMとしてもつ標的DNAはわずかに切断されていたが,それ以外のDNAはまったく切断されていなかったからだ.そののち,注意深く実験をくり返したところ,in vitroにおいてxCas9はNGGをPAMとして認識するが,NGHはほとんど認識しないことがわかった.NGGの認識もSpCas9-NGに比べてずっと弱かった.
追加実験を開始してから2カ月が経過したが,GUIDE-seq法の結果はまだでていなかった.そのような状況のなか,Science誌のエディターから1通のメールが届いた.“4月5日にxCas9の論文が“Article”としてNature誌に掲載された.4月9日までに改定原稿が投稿されなければ,論文の掲載を正当化するのは困難になるだろう”.残された時間は3日.残念ながら,最後の実験も失敗に終わった.万事休すと思われたが,xCas9のDNA切断実験の結果をエディターに伝えたところ,SpCas9-NGおよびxCas9のDNA切断活性をin vitroおよび培養細胞において比較したデータを追加するという条件で,改訂の期限を1カ月延長してもらえることになった.
実験の結果,SpCas9-NGは培養細胞においてもxCas9より高いDNA切断活性を示すことがわかった.こうなったら,できるだけよい論文にしようと考え,標的部位を当初の32個から69個に増やし,野生型Cas9,SpCas9-NG,xCas9によりひき起こされる変異を調べ直した.さらに,別の論文にまとめる予定だった“塩基エディター”に関するデータもこの論文にくわえることにした.また,当初,Arg1335Ala置換をもつ変異体をSpCas9-NGとしていたが,改訂の途中でArg1335をValに置換したほうがDNA切断活性の高いことを発見していたので,Arg1335Val置換をもつ変異体をSpCas9-NGとして,in vitroにおけるDNA切断実験,PAMの網羅的な解析,結晶構造解析をやり直した.志願して追加実験を増やしたこともあり,延長された改訂の期限から2カ月が過ぎてしまったが,8月11日には改訂原稿を投稿し,8月16日,ついに論文は受理された.苦しい期間が半年以上もつづくことになったが,論文の質を向上させる機会をあたえてくれたxCas9に感謝したい.
略歴:東京大学大学院理学系研究科 助教.
研究テーマ:CRISPR-Cas.RNAサイレンシング.
関心事:つぎの10年.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究室URL:http://www.nurekilab.net/
© 2018 西増弘志・濡木 理 Licensed under CC 表示 2.1 日本
(東京大学大学院理学系研究科 生物科学専攻生物化学講座構造生命科学研究室)
email:西増弘志,濡木 理
DOI: 10.7875/first.author.2018.094
Engineered CRISPR-Cas9 nuclease with expanded targeting space.
Hiroshi Nishimasu, Xi Shi, Soh Ishiguro, Linyi Gao, Seiichi Hirano, Sanae Okazaki, Taichi Noda, Omar O. Abudayyeh, Jonathan S. Gootenberg, Hideto Mori, Seiya Oura, Benjamin Holmes, Mamoru Tanaka, Motoaki Seki, Hisato Hirano, Hiroyuki Aburatani, Ryuichiro Ishitani, Masahito Ikawa, Nozomu Yachie, Feng Zhang, Osamu Nureki
Science, 361, 1259-1262 (2018)
要 約
RNA依存性DNAエンドヌクレアーゼであるCas9はゲノム編集のツールとして利用されている.しかし,Cas9がDNAを認識するには標的とする配列の近傍にPAMとよばれる特定の塩基配列が必要であるため,その適用の範囲には制限がある.この研究において,筆者らは,Cas9に7つのアミノ酸置換を導入し,1つのグアニン塩基をPAMとして認識するSpCas9-NGを開発した.SpCas9-NGはこれまで標的とすることのできなかった領域のゲノム編集を可能にし,さまざまな分野において有用なゲノム編集のツールとして利用されることが期待される.
はじめに
原核生物のCRISPR-Cas(CRISPR:clustered regularly interspaced short palindromic repeat,Cas:CRISPR-associated protein)獲得免疫機構に関与するRNA依存性DNAエンドヌクレアーゼであるCas9は,ガイドRNAと複合体を形成しガイド配列と相補的な2本鎖DNAを特異的に切断する1).20塩基のガイド配列は自由に設計できるため,Cas9およびガイドRNAを用いることにより2本鎖DNAをねらった位置で切断することができる.したがって,近年,Cas9を用いたゲノム編集はさまざまな分野において広く利用されている2).Cas9が標的となる2本鎖DNAを認識するためには,ガイド配列と標的配列とのあいだの相補性にくわえ,標的配列のとなりにPAM(protospacer adjacent motif)とよばれる特定の塩基配列が必要である.PAMは生物種により異なり,ゲノム編集に広く利用されているStreptococcus pyogenesに由来するCas9はNGG(N:任意の塩基)という配列をPAMとして認識する(図1a).したがって,Cas9を用いたゲノム編集には適応の範囲に制限があるという弱点が残されていた.
1.SpCas9-NGの開発
S. pyogenesに由来するCas9の結晶構造(PDB ID:4UN3)から,PAMの2塩基目および3塩基目のGはそれぞれArg1333およびArg1335により認識されることが明らかにされていた3)(図1b).そこで,以下の戦略により,PAMに対する特異性をNGGからNGに改変することを試みた.まず,3塩基目のGを認識するArg1335をAlaに置換し,3塩基目のGとの塩基特異的な相互作用を削除する.つぎに,DNAの糖-リン酸骨格と新たな相互作用を形成するようなアミノ酸置換を導入し,Arg1335のAlaへの置換による塩基特異的な相互作用の損失を補填する.
この仮説を確かめるため,まず,野生型Cas9およびArg1335Ala変異体の組換えタンパク質を精製し,TGGという配列をPAMとしてもつ標的DNAに対する切断活性をin vitroにおいて測定した.野生型Cas9はTGGをPAMとしてもつ標的DNAを効率よく切断した一方,Arg1335Ala変異体は標的DNAを切断しなかった.つぎに,結晶構造を参考に,Arg1335Ala置換にくわえ,DNAのリン酸骨格の近くに存在するアミノ酸残基をArgなどに置換した複数の二重変異体を設計した.これらの変異体の組換えタンパク質を精製しDNA切断活性を測定したところ,Arg1335Ala置換に,Leu1111Arg置換,Gly1218Arg置換,Ala1322Arg置換,Thr1337Arg置換のいずれかを追加した二重変異体は微弱なDNA切断活性を示した.さらに,これら4つの変異を組み合わせたところDNA切断活性が上昇した.しかし,DNA切断反応の経時変化を測定したところ,Arg1335Ala置換,Leu1111Arg置換,Gly1218Arg置換,Ala1322Arg置換,Thr1337Arg置換の5つの変異をもつARRRR変異体のDNA切断活性は,野生型Cas9よりもずっと低かった.したがって,Arg1335Ala置換による相互作用の損失を補填するには,DNAのリン酸基との相互作用を強化するだけでは不十分であると考えられた.
そこで,DNAのリボース部分と新たな相互作用を形成するようなアミノ酸変異の導入について検討した.過去に報告されたVQR改変体は,Asp1135Val置換,Arg1335Gln置換,Thr1337Arg置換の3つの変異をもち,NGAをPAMとして認識する4).結晶構造(PDB ID:5B2R)から,Val1135はDNAのリボース部分と相互作用することが明らかにされていた5,6)(文献5) は 新着論文レビュー でも掲載).また,結晶構造をもとに考察した結果,Val1335およびPhe1219はそれぞれArg1333およびDNAのリボース部分と相互作用を形成する可能性が示唆された.実際,ARRRR変異体にAsp1135Val置換,Glu1219Phe置換,Arg1335Val置換の3つの変異を追加したところ,TGGをPAMとしてもつ標的DNAに対する切断活性が大きく上昇した.
つぎに,TGA,TGT,TGG,TGCのいずれかをPAMとしてもつ4種類の標的DNAを用いて,野生型Cas9,および,Arg1335Val置換,Leu1111Arg置換,Asp1135Val置換,Gly1218Arg置換,Glu1219Phe置換,Ala1322Arg置換,Thr1337Arg置換の7つの変異をもつVRVRFRR変異体のDNA切断活性を測定した.野生型Cas9はTGGをPAMとしてもつ標的DNAを効率よく切断し,TGAをPAMとしてもつ標的DNAをわずかに切断したが,TGTあるいはTGCをPAMとしてもつ標的DNAはほとんど切断しなかった.一方,VRVRFRR変異体はTGGだけでなくTGH(H:A,T,C)をPAMとしてもつ標的DNAを効率的に切断した.さらに,標的配列のとなりに65,536(4の8乗)通りの配列をもつDNAライブラリーをin vitroにおいて切断し次世代シークエンサーを用いて解析した結果,VRVRFRR変異体はNGという配列をPAMとして認識することが確かめられた.以上の結果から,VRVRFRR変異体をSpCas9-NGと命名した.
2.SpCas9-NGによるPAMの認識機構
SpCas9-NGによるPAMの認識機構を解明するため,SpCas9-NG,ガイドRNA,標的DNAからなる複合体の結晶構造を決定した(PDB ID:6AI6,図1c).PAMの2塩基目のGはArg1333と2本の水素結合を形成していた一方,3塩基目のGは特異的な相互作用を形成していなかった.Arg1111,Val1135,Phe1219はDNAの糖-リン酸骨格と相互作用していた.Arg1218およびArg1322もDNAの近傍に位置しており,DNAの糖-リン酸骨格と相互作用することが示唆された.
3.SpCas9-NGを用いたゲノム編集
SpCas9-NGがゲノム編集に利用できるかどうかを検証するため,野生型Cas9あるいはSpCas9-NGおよびガイドRNAをヒト培養細胞に発現させ,NGA,NGT,NGG,NGCのいずれかをPAMとしてもつ標的部位への変異の導入について調べた.野生型Cas9はNGGをPAMとしてもつ標的部位に効率よく変異をひき起こした一方,NGTあるいはNGCをPAMとしてもつ標的部位には変異をひき起こさなかった.in vitroにおけるDNA切断活性と一致して,NGAをPAMとしてもつ標的部位には中程度の効率で変異が導入された.一方,SpCas9-NGはNGGだけでなくNGHをPAMとしてもつ標的部位にも変異をひき起こした.したがって,SpCas9-NGはゲノム編集のツールとして利用できることが示唆された.
4.SpCas9-NGのオフターゲット効果
Cas9はガイド配列と完全に相補的なDNAだけでなく,数塩基のミスマッチをもつDNAもわずかに切断する.そのため,Cas9を用いたゲノム編集においては,標的配列と類似した配列にも変異が導入されるオフターゲット効果の生じることがある.SpCas9-NGはPAMによる制約が緩和されているため,野生型Cas9と比較して,多くのオフターゲットの候補をもつ可能性が高い.そこで,EMX1遺伝子を標的とするガイドRNA,および,野生型Cas9あるいはSpCas9-NGをヒト培養細胞に発現させ,ゲノムに生じた変異をGUIDE-seq法7) を用いて網羅的に検出した.その結果,野生型Cas9およびSpCas9-NGは同じ程度のオフターゲットの変異をひき起こすことが明らかにされた.PAMに対する特異性と一致して,SpCas9-NGはNGGのほかにNGHをPAMとしてもつオフターゲット部位に変異をひき起こした.これまで,eSpCas9などオフターゲット効果の低減されたCas9改変体が報告されている8).そこで,eSpCas9のもつLys848Ala置換,Lys1003Ala置換,Arg1060Ala置換の3つの変異をSpCas9-NGに追加し,GUIDE-seq法を用いてオフターゲット効果を評価した.その結果,これら3つのアミノ酸置換の導入によりSpCas9-NGの特異性は向上することが明らかにされた.
5.SpCas9-NGを応用した塩基の編集
Asp10Ala置換をもつCas9ニッカーゼとシチジンデアミナーゼとの融合タンパク質Target-AIDは,標的配列に含まれるCをTに変換する“塩基エディター”としてはたらく9).そこで,Asp10Ala置換を導入したSpCas9-NGとシチジンデアミナーゼとの融合タンパク質Target-AID-NGを作製しヒト培養細胞に発現させ,NGA,NGT,NGG,NGCのいずれかをPAMとしてもつ標的部位における塩基の置換について調べた.その結果,Target-AIDとは異なり,Target-AID-NGはNGA,NGT,NGG,NGCのいずれかをPAMとしてもつ標的部位に塩基の置換をひき起こした.したがって,DNA切断活性を欠損させたSpCas9-NGは標的の範囲の拡張したRNA依存的なDNA結合プラットフォームとして有用であることが示された.
おわりに
野生型Cas9はNGGをPAMとして要求するため,確率的にゲノム領域の1/16しか標的にすることができない.一方,SpCas9-NGはNGをPAMとして認識するため,その4倍の領域を標的とすることができる.したがって,SpCas9-NGは,より広い領域を標的とすることのできる有用なゲノム編集のツールとして利用されることが期待される.
この研究は2015年6月に開始し,その4カ月後にはSpCas9-NGはほぼ完成していた.しかし,ほかのプロジェクトとの兼ね合いやゲノム編集のツールとしての性能を慎重に評価したこともあり,論文の投稿までに2年以上の歳月を要した.2018年1月4日,論文をScience誌に投稿したところ,1月29日に返信があった.2人のレビュアーのうち,1人は非常に好意的でマイナーなコメントがたったひとつ.もう1人の評価もおおむね良好であり,必要な追加実験はGUIDE-seq法を用いたオフターゲット効果の評価のみであった.筆者らにとりGUIDE-seq法ははじめての実験手法であったが,プロトコールは確立されているため問題なく結果がでるだろうと考えた.しかし,GUIDE-seq法は簡単ではなかった.細胞培養や次世代シークエンス解析などの複数のステップからなるため,実験がうまく進んだとしても結果が得られるまでに2週間はかかる.実験条件を検討しているうちに1カ月が経過した.
2月28日,事態は急転した.SpCas9-NGと同様にNGをPAMとして認識するCas9改変体であるxCas9についての論文が“Accelerated Article Preview”としてNature誌に掲載されたのだ10).この論文は筆者らの論文の約2週間後に投稿されており,わずか1カ月で受理されていた.SpCas9-NGは構造情報にもとづき合理的に設計された改変体であったのに対し,xCas9は人工進化的な手法により開発されていた.xCas9はSpCas9-NGとは異なる7つのアミノ酸置換をもっていた.Science誌のエディターに連絡したところ,要求されている追加実験を行えば問題ないとの返信があり,ひとまず胸をなでおろした.
xCas9の論文には不可解な点がいくつかあった.まず,立体構造を考慮すると,xCas9はNGをPAMとして認識するようには思えなかった.PAMの3塩基目のGを認識するArg1335は置換されておらず,また,7つのアミノ酸置換のほとんどはPAMから離れた位置に存在した.精製したxCas9のin vitroにおけるDNA切断活性は調べられていなかった.さらに,培養細胞の実験には過剰量の発現ベクターが用いられており,野生型Cas9を発現させた場合においても,NGG以外のPAMをもつ標的部位にも変異が生じていた.そこで,純粋な好奇心から,すぐさまxCas9のもつ変異を導入するためのオリゴヌクレオチドを発注し,xCas9の組換えタンパク質を精製してそのDNA切断活性を測定してみた.これが,のちに大きな意味をもつことになった.最初の実験結果をみたとき,実験条件をまちがえたのではないかと思った.NGGをPAMとしてもつ標的DNAはわずかに切断されていたが,それ以外のDNAはまったく切断されていなかったからだ.そののち,注意深く実験をくり返したところ,in vitroにおいてxCas9はNGGをPAMとして認識するが,NGHはほとんど認識しないことがわかった.NGGの認識もSpCas9-NGに比べてずっと弱かった.
追加実験を開始してから2カ月が経過したが,GUIDE-seq法の結果はまだでていなかった.そのような状況のなか,Science誌のエディターから1通のメールが届いた.“4月5日にxCas9の論文が“Article”としてNature誌に掲載された.4月9日までに改定原稿が投稿されなければ,論文の掲載を正当化するのは困難になるだろう”.残された時間は3日.残念ながら,最後の実験も失敗に終わった.万事休すと思われたが,xCas9のDNA切断実験の結果をエディターに伝えたところ,SpCas9-NGおよびxCas9のDNA切断活性をin vitroおよび培養細胞において比較したデータを追加するという条件で,改訂の期限を1カ月延長してもらえることになった.
実験の結果,SpCas9-NGは培養細胞においてもxCas9より高いDNA切断活性を示すことがわかった.こうなったら,できるだけよい論文にしようと考え,標的部位を当初の32個から69個に増やし,野生型Cas9,SpCas9-NG,xCas9によりひき起こされる変異を調べ直した.さらに,別の論文にまとめる予定だった“塩基エディター”に関するデータもこの論文にくわえることにした.また,当初,Arg1335Ala置換をもつ変異体をSpCas9-NGとしていたが,改訂の途中でArg1335をValに置換したほうがDNA切断活性の高いことを発見していたので,Arg1335Val置換をもつ変異体をSpCas9-NGとして,in vitroにおけるDNA切断実験,PAMの網羅的な解析,結晶構造解析をやり直した.志願して追加実験を増やしたこともあり,延長された改訂の期限から2カ月が過ぎてしまったが,8月11日には改訂原稿を投稿し,8月16日,ついに論文は受理された.苦しい期間が半年以上もつづくことになったが,論文の質を向上させる機会をあたえてくれたxCas9に感謝したい.
文 献
- Jinek, M., Chylinski, K., Fonfara, I. et al.: A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 337, 816-821 (2012)[PubMed]
- Cong, L., Ran, F. A., Cox, D. et al.: Multiplex genome engineering using CRISPR/Cas systems. Science, 339, 819-823 (2013)[PubMed]
- Anders, C., Niewoehner, O., Duerst, A. et al.: Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature, 513, 569-573 (2014)[PubMed]
- Kleinstiver, B. P., Prew, M. S., Tsai, S. Q. et al.: Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature, 523, 481-485 (2015)[PubMed]
- Hirano, S., Nishimasu, H., Ishitani, R. et al.: Structural basis for the altered PAM specificities of engineered CRISPR-Cas9. Mol. Cell, 61, 886-894 (2016)[PubMed] [新着論文レビュー]
- Anders, C., Niewoehner, O., Duerst, A. et al.: Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature, 513, 569-573 (2014)[PubMed]
- Tsai, S. Q., Zheng, Z., Nguyen, N. T. et al.: GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat. Biotechnol., 33, 187-197 (2015)[PubMed]
- Slaymaker, I. M., Gao, L., Zetsche, B. et al.: Rationally engineered Cas9 nucleases with improved specificity. Science. 351, 84-88 (2016)[PubMed]
- Nishida, K., Arazoe, T., Yachie, N. et al.: Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems. Science, 353, aaf8729 (2016)[PubMed]
- Hu, J. H., Miller, S. M., Geurts, M. H. et al.: Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature, 556, 57-63 (2018)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:東京大学大学院理学系研究科 助教.
研究テーマ:CRISPR-Cas.RNAサイレンシング.
関心事:つぎの10年.
濡木 理(Osamu Nureki)
東京大学大学院理学系研究科 教授.
研究室URL:http://www.nurekilab.net/
© 2018 西増弘志・濡木 理 Licensed under CC 表示 2.1 日本