がん遺伝子の大規模なスクリーニングにおけるRNA結合タンパク質を介したオフターゲット効果
鈴木 洋・Phillip A. Sharp
(米国Massachusetts Institute of Technology,David H. Koch Institute for Integrative Cancer Research)
email:鈴木 洋
DOI: 10.7875/first.author.2018.047
Deconvolution of seed and RNA-binding protein crosstalk in RNAi-based functional genomics.
Hiroshi I. Suzuki, Ryan M. Spengler, Giedre Grigelioniene, Tatsuya Kobayashi, Phillip A. Sharp
Nature Genetics, 50, 657-661 (2018)
RNAi法やCRISPR-Cas9系はゲノムワイドな遺伝子のスクリーニングにおいて主要かつ強力な解析プラットフォームであるが,それぞれに特有のオフターゲット効果が問題になる.この研究において,筆者らは,RNA結合タンパク質の結合モチーフとオーバーラップしたシード配列をもつsiRNAやmiRNAが内在性のRNA結合タンパク質の機能に干渉することにより新たな機能を獲得するというクロストークを見い出した.siRNAによるオフターゲット効果のデータ,および,RNAi法によるゲノムワイドながん遺伝子のスクリーニングのビッグデータを統合的に解析することにより,このシード配列とRNA結合タンパク質とのクロストークが広範囲に起こっており,RNAiにおけるオフターゲット効果に寄与することが明らかにされた.とくに,RNAi法によるゲノムワイドながん遺伝子のスクリーニングにおいて,オンターゲット効果とオフターゲット効果を分離することにより,シード配列とRNA結合タンパク質とのクロストークが表現型の修飾に関与することが明らかにされた.この研究により,miRNAの新しい機能的な側面,および,RNAi法にもとづく機能ゲノミクスの性能を向上させるための方向性が明らかにされた.
RNAi法やCRISPR-Cas9系はゲノムワイドな遺伝子のスクリーニングにおいて主要な解析プラットフォームであるが,それぞれに特有のオフターゲット効果が問題になる.RNAiは内在性のmiRNA経路を利用するため1)(新着論文レビュー でも掲載),siRNAやshRNAのシード配列と相補的な配列をもつmRNAが抑制されるというオフターゲット効果が問題になり,このオフターゲット効果に対し,目的とする遺伝子をノックダウンしたオンターゲット効果をin silicoにおいて分離しようとする試みがなされている2-5).細胞においては,RNA,小分子RNA,RNA結合タンパク質のあいだで多様な相互作用が起こっており,miRNAとの関係においては,同じmiRNAが結合する標的RNAがmiRNAをとりあうことにより発現のレベルにおいて間接的に正の相関を示すという競合的内在性RNA(competing endogenous RNA:ceRNA)の効果や,さまざまなRNA結合タンパク質がmiRNAの機能を制御することなどが知られている6,7).一方で,これらのクロストークがRNAi法にもとづく機能ゲノミクスの結果あるいはその解釈にどのように影響するかについては検討されていない.
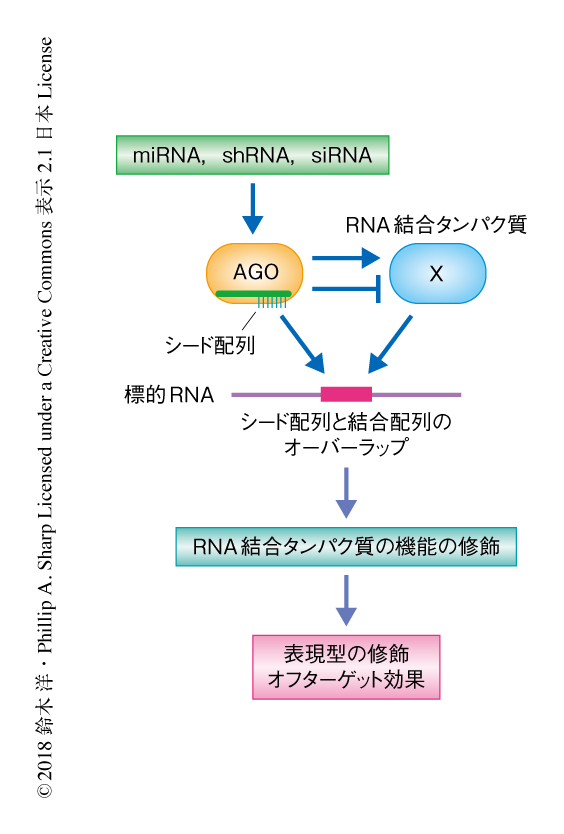
これまでに筆者らは,スーパーエンハンサーが細胞種に特異的なmiRNAの発現パターンを規定するうえで非常に重要であることを報告したが8)(新着論文レビュー でも掲載),さらに最近,スーパーエンハンサーに関連したmiRNAの変異がヒトの疾患をひき起こすことを見い出した.この過程において,変異したmiRNAが,変異により変化したシード配列とオーバーラップする結合モチーフをもつRNA結合タンパク質の活性を制御する可能性が見い出された.この結果から,RNA結合タンパク質の結合モチーフとオーバーラップしたシード配列をもつsiRNAやmiRNAが,内在性のRNA結合タンパク質の機能に干渉することにより新たな機能を獲得する,という新しいクロストーク(crosstalk with endogenous RNA-binding protein:ceRBP)の存在が示唆された(図1).そこで,このシード配列とRNA結合タンパク質とのクロストークがRNAiの機能的な側面として重要であり,また,RNAi法にもとづく機能ゲノミクスの解釈においても重要ではないかと考え,その意義について検証した.
シード配列とRNA結合タンパク質とのクロストークとRNAiのオフターゲット効果との関係について解析するため,まず,93種類のsiRNAがトランスクリプトームにおよぼす影響との関係について解析した.その結果,siRNAのシード配列がRNA結合タンパク質の結合モチーフとオーバーラップする場合,より強いオフターゲット効果が生じることが明らかにされた.また,この場合には,2次的なトランスクリプトームの変動も強くなった.一方で,オフターゲット効果の低いsiRNAの多くは,シード配列とRNA結合タンパク質の結合モチーフとのオーバーラップが少なく,RNA結合タンパク質の結合モチーフとのオーバーラップをさけることによりオフターゲット効果は軽減されることが示唆された.
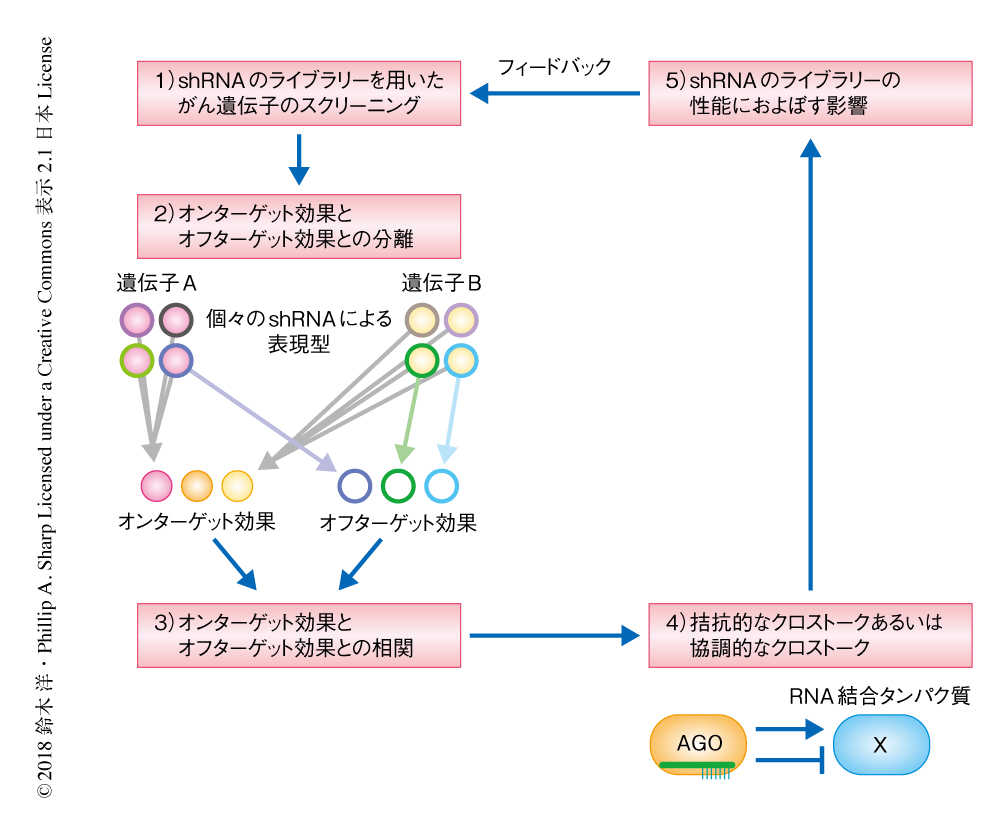
2017年に報告された,501種類のがん細胞株において約17,000個の遺伝子を標的として10万個のshRNAのライブラリーを用いたゲノムワイドながん遺伝子のスクリーニング(cancer dependency map)の結果を解析した4)(図2).この解析においては,同じ遺伝子を標的としたshRNAのペアの変動の相関よりも,同じシード配列をもつshRNAのペアの変動の相関のほうが大きいことが示されており,シード配列を介したオフターゲット効果が表現型に大きく影響することが示唆された.そのため,この報告においては,個々のshRNAについて,目的の遺伝子をノックダウンしたオンターゲット効果とシード配列によるオフターゲット効果とを分離するための新規のin silicoにおける解析の手法が開発された.この手法により,501種類のがん細胞株について,17,000個の遺伝子に対するオンターゲット効果と,15,000種類のシード配列に対するオフターゲット効果が得られた.ここで,シード配列とRNA結合タンパク質とのクロストークについて検証するため,遺伝子のレベルでの依存性のスコアとシード配列のレベルでの依存性のスコアとを比較することにより,シード配列を強制発現させた場合の表現型とRNA結合タンパク質をノックダウンした場合の表現型とがどのように相関するかを解析した.この解析においては,RNA結合タンパク質とその結合モチーフにオーバーラップするシード配列をもつshRNAとが機能的に拮抗する場合,RNA結合タンパク質をノックダウンした場合の表現型とシード配列を強制発現させた場合の表現型とのあいだに正の相関がみられることが予想される.一方で,RNA結合タンパク質とその結合モチーフにオーバーラップするシード配列をもつshRNAとのあいだに機能的な協調性あるいは冗長性がある場合には,負の相関がみられることが予想される.
166種類のRNA結合タンパク質について解析したところ,約70個のRNA結合タンパク質について拮抗的なクロストークあるいは協調的なクロストークの存在が示唆された.同じファミリーに属するRNA結合タンパク質については同じ方向性のクロストークがみられた.RNAを安定化し,また,miRNAと拮抗することが知られているIGF2BPやELAVL1といったRNA結合タンパク質については拮抗的なクロストークが確認された一方,ポリA鎖結合タンパク質やRBM47といったRNA結合タンパク質については協調的なクロストークがみられた.ポリA鎖結合タンパク質はmiRNAによる標的RNAの抑制を促進することが知られており,解析の結果はこの知見と合致した.これらの解析の結果は,標的RNAを安定化させるあるいは不安定化させるという対象としたRNA結合タンパク質の機能的な特徴とおおむね合致し,また,シード配列とRNA結合タンパク質とのクロストークが広範囲に起こっていることが明らかにされた.さらに,がんにおいて異常のみられる多くのRNA結合タンパク質について,潜在的なクロストークが示唆された.
RNA結合タンパク質であるIGF2BPとその結合モチーフとオーバーラップするシード配列をもつshRNAとの関係を実験的に検証したところ,解析の結果と合致して,IGF2BPの結合モチーフとオーバーラップするシード配列をもつshRNAが,IGF2BPの標的であるHMGA2遺伝子の発現を抑制することが確認された.さらに,IGF2BPのトリプルノックアウト細胞を用いて1細胞レポーター解析を施行したところ,IGF2BPの結合モチーフとオーバーラップするシード配列をもつshRNAに対するHMGA2遺伝子の3’側非翻訳領域の応答性は,IGF2BPの存在により増強されることも明らかにされた.
さきと同じRNAi法によるゲノムワイドながん遺伝子のスクリーニングのデータを用い,シード配列とRNA結合タンパク質とのクロストークがshRNAのライブラリーの性能におよぼす影響について調べたところ,RNA結合タンパク質との拮抗的なクロストークのあるシード配列をもつ場合には,オンターゲット効果の強い効率のよいshRNAの頻度が低下することが明らかにされた.さらに,内在性のmiRNAのシード配列とのオーバーラップについて調べたところ,幅広く保存されたmiRNAのシード配列をもつ場合にも,オンターゲット効果の強い効率のよいshRNAの頻度は低下することが確認された.
この研究により,siRNAやmiRNAのシード配列とRNA結合タンパク質の結合モチーフのオーバーラップを介したクロストークが広範囲に起こり,RNAiにおけるオフターゲット効果や表現型の修飾に寄与することが明らかにされた.これらの知見は,RNAiのデザイン,miRNAの機能,機能ゲノミクスの解釈について,3つの方向性を示唆した.第1に,RNAiのデザインにおいて,RNA結合タンパク質の結合モチーフとのオーバーラップをさけることが効率のよいshRNAあるいはsiRNAをデザインするうえで重要である.第2に,これまで内在性のmiRNAとRNA結合タンパク質とのあいだの機能的なクロストークについてはいくつかの機構が提案されていたが,シード配列とRNA結合タンパク質とのクロストークは新しいシナリオである.第3に,RNAi法によるゲノムワイドながん遺伝子のスクリーニングのデータを用いた解析から,データのデコンボリューションのプロセスを改善することにより機能ゲノミクスのデータの解釈が向上することが示唆された.CRISPR-Cas9系を用いた遺伝子のスクリーニングにおいてもオフターゲット効果を除く試みがなされているが9),RNAi法あるいはCRISPR-Cas9系を用いた遺伝子のスクリーニングのそれぞれの長所を考慮すると4),さらなるデータのデコンボリューションのプロセスの改善が両者のプラットフォームの相互的な補完を可能にすると考えられる.
略歴:2010年 東京大学大学院医学研究科 早期修了,同年 同 特任助教を経て,2014年より米国Massachusetts Institute of Technology客員研究員.
関心事:ゲノム相分離.
Phillip A. Sharp
米国Massachusetts Institute of TechnologyにてInstitute Professor.
研究室URL:http://web.mit.edu/sharplab/
© 2018 鈴木 洋・Phillip A. Sharp Licensed under CC 表示 2.1 日本
(米国Massachusetts Institute of Technology,David H. Koch Institute for Integrative Cancer Research)
email:鈴木 洋
DOI: 10.7875/first.author.2018.047
Deconvolution of seed and RNA-binding protein crosstalk in RNAi-based functional genomics.
Hiroshi I. Suzuki, Ryan M. Spengler, Giedre Grigelioniene, Tatsuya Kobayashi, Phillip A. Sharp
Nature Genetics, 50, 657-661 (2018)
要 約
RNAi法やCRISPR-Cas9系はゲノムワイドな遺伝子のスクリーニングにおいて主要かつ強力な解析プラットフォームであるが,それぞれに特有のオフターゲット効果が問題になる.この研究において,筆者らは,RNA結合タンパク質の結合モチーフとオーバーラップしたシード配列をもつsiRNAやmiRNAが内在性のRNA結合タンパク質の機能に干渉することにより新たな機能を獲得するというクロストークを見い出した.siRNAによるオフターゲット効果のデータ,および,RNAi法によるゲノムワイドながん遺伝子のスクリーニングのビッグデータを統合的に解析することにより,このシード配列とRNA結合タンパク質とのクロストークが広範囲に起こっており,RNAiにおけるオフターゲット効果に寄与することが明らかにされた.とくに,RNAi法によるゲノムワイドながん遺伝子のスクリーニングにおいて,オンターゲット効果とオフターゲット効果を分離することにより,シード配列とRNA結合タンパク質とのクロストークが表現型の修飾に関与することが明らかにされた.この研究により,miRNAの新しい機能的な側面,および,RNAi法にもとづく機能ゲノミクスの性能を向上させるための方向性が明らかにされた.
はじめに
RNAi法やCRISPR-Cas9系はゲノムワイドな遺伝子のスクリーニングにおいて主要な解析プラットフォームであるが,それぞれに特有のオフターゲット効果が問題になる.RNAiは内在性のmiRNA経路を利用するため1)(新着論文レビュー でも掲載),siRNAやshRNAのシード配列と相補的な配列をもつmRNAが抑制されるというオフターゲット効果が問題になり,このオフターゲット効果に対し,目的とする遺伝子をノックダウンしたオンターゲット効果をin silicoにおいて分離しようとする試みがなされている2-5).細胞においては,RNA,小分子RNA,RNA結合タンパク質のあいだで多様な相互作用が起こっており,miRNAとの関係においては,同じmiRNAが結合する標的RNAがmiRNAをとりあうことにより発現のレベルにおいて間接的に正の相関を示すという競合的内在性RNA(competing endogenous RNA:ceRNA)の効果や,さまざまなRNA結合タンパク質がmiRNAの機能を制御することなどが知られている6,7).一方で,これらのクロストークがRNAi法にもとづく機能ゲノミクスの結果あるいはその解釈にどのように影響するかについては検討されていない.
1.シード配列と内在性のRNA結合タンパク質のクロストーク
これまでに筆者らは,スーパーエンハンサーが細胞種に特異的なmiRNAの発現パターンを規定するうえで非常に重要であることを報告したが8)(新着論文レビュー でも掲載),さらに最近,スーパーエンハンサーに関連したmiRNAの変異がヒトの疾患をひき起こすことを見い出した.この過程において,変異したmiRNAが,変異により変化したシード配列とオーバーラップする結合モチーフをもつRNA結合タンパク質の活性を制御する可能性が見い出された.この結果から,RNA結合タンパク質の結合モチーフとオーバーラップしたシード配列をもつsiRNAやmiRNAが,内在性のRNA結合タンパク質の機能に干渉することにより新たな機能を獲得する,という新しいクロストーク(crosstalk with endogenous RNA-binding protein:ceRBP)の存在が示唆された(図1).そこで,このシード配列とRNA結合タンパク質とのクロストークがRNAiの機能的な側面として重要であり,また,RNAi法にもとづく機能ゲノミクスの解釈においても重要ではないかと考え,その意義について検証した.
2.シード配列とRNA結合タンパク質とのクロストークとRNAiにおけるオフターゲット効果との関係
シード配列とRNA結合タンパク質とのクロストークとRNAiのオフターゲット効果との関係について解析するため,まず,93種類のsiRNAがトランスクリプトームにおよぼす影響との関係について解析した.その結果,siRNAのシード配列がRNA結合タンパク質の結合モチーフとオーバーラップする場合,より強いオフターゲット効果が生じることが明らかにされた.また,この場合には,2次的なトランスクリプトームの変動も強くなった.一方で,オフターゲット効果の低いsiRNAの多くは,シード配列とRNA結合タンパク質の結合モチーフとのオーバーラップが少なく,RNA結合タンパク質の結合モチーフとのオーバーラップをさけることによりオフターゲット効果は軽減されることが示唆された.
3.がん遺伝子の大規模なスクリーニングにおけるシード配列とRNA結合タンパク質とのクロストークの意義
2017年に報告された,501種類のがん細胞株において約17,000個の遺伝子を標的として10万個のshRNAのライブラリーを用いたゲノムワイドながん遺伝子のスクリーニング(cancer dependency map)の結果を解析した4)(図2).この解析においては,同じ遺伝子を標的としたshRNAのペアの変動の相関よりも,同じシード配列をもつshRNAのペアの変動の相関のほうが大きいことが示されており,シード配列を介したオフターゲット効果が表現型に大きく影響することが示唆された.そのため,この報告においては,個々のshRNAについて,目的の遺伝子をノックダウンしたオンターゲット効果とシード配列によるオフターゲット効果とを分離するための新規のin silicoにおける解析の手法が開発された.この手法により,501種類のがん細胞株について,17,000個の遺伝子に対するオンターゲット効果と,15,000種類のシード配列に対するオフターゲット効果が得られた.ここで,シード配列とRNA結合タンパク質とのクロストークについて検証するため,遺伝子のレベルでの依存性のスコアとシード配列のレベルでの依存性のスコアとを比較することにより,シード配列を強制発現させた場合の表現型とRNA結合タンパク質をノックダウンした場合の表現型とがどのように相関するかを解析した.この解析においては,RNA結合タンパク質とその結合モチーフにオーバーラップするシード配列をもつshRNAとが機能的に拮抗する場合,RNA結合タンパク質をノックダウンした場合の表現型とシード配列を強制発現させた場合の表現型とのあいだに正の相関がみられることが予想される.一方で,RNA結合タンパク質とその結合モチーフにオーバーラップするシード配列をもつshRNAとのあいだに機能的な協調性あるいは冗長性がある場合には,負の相関がみられることが予想される.
166種類のRNA結合タンパク質について解析したところ,約70個のRNA結合タンパク質について拮抗的なクロストークあるいは協調的なクロストークの存在が示唆された.同じファミリーに属するRNA結合タンパク質については同じ方向性のクロストークがみられた.RNAを安定化し,また,miRNAと拮抗することが知られているIGF2BPやELAVL1といったRNA結合タンパク質については拮抗的なクロストークが確認された一方,ポリA鎖結合タンパク質やRBM47といったRNA結合タンパク質については協調的なクロストークがみられた.ポリA鎖結合タンパク質はmiRNAによる標的RNAの抑制を促進することが知られており,解析の結果はこの知見と合致した.これらの解析の結果は,標的RNAを安定化させるあるいは不安定化させるという対象としたRNA結合タンパク質の機能的な特徴とおおむね合致し,また,シード配列とRNA結合タンパク質とのクロストークが広範囲に起こっていることが明らかにされた.さらに,がんにおいて異常のみられる多くのRNA結合タンパク質について,潜在的なクロストークが示唆された.
4.シード配列とRNA結合タンパク質とのクロストークの検証およびshRNAのライブラリーの性能におよぼす影響
RNA結合タンパク質であるIGF2BPとその結合モチーフとオーバーラップするシード配列をもつshRNAとの関係を実験的に検証したところ,解析の結果と合致して,IGF2BPの結合モチーフとオーバーラップするシード配列をもつshRNAが,IGF2BPの標的であるHMGA2遺伝子の発現を抑制することが確認された.さらに,IGF2BPのトリプルノックアウト細胞を用いて1細胞レポーター解析を施行したところ,IGF2BPの結合モチーフとオーバーラップするシード配列をもつshRNAに対するHMGA2遺伝子の3’側非翻訳領域の応答性は,IGF2BPの存在により増強されることも明らかにされた.
さきと同じRNAi法によるゲノムワイドながん遺伝子のスクリーニングのデータを用い,シード配列とRNA結合タンパク質とのクロストークがshRNAのライブラリーの性能におよぼす影響について調べたところ,RNA結合タンパク質との拮抗的なクロストークのあるシード配列をもつ場合には,オンターゲット効果の強い効率のよいshRNAの頻度が低下することが明らかにされた.さらに,内在性のmiRNAのシード配列とのオーバーラップについて調べたところ,幅広く保存されたmiRNAのシード配列をもつ場合にも,オンターゲット効果の強い効率のよいshRNAの頻度は低下することが確認された.
おわりに
この研究により,siRNAやmiRNAのシード配列とRNA結合タンパク質の結合モチーフのオーバーラップを介したクロストークが広範囲に起こり,RNAiにおけるオフターゲット効果や表現型の修飾に寄与することが明らかにされた.これらの知見は,RNAiのデザイン,miRNAの機能,機能ゲノミクスの解釈について,3つの方向性を示唆した.第1に,RNAiのデザインにおいて,RNA結合タンパク質の結合モチーフとのオーバーラップをさけることが効率のよいshRNAあるいはsiRNAをデザインするうえで重要である.第2に,これまで内在性のmiRNAとRNA結合タンパク質とのあいだの機能的なクロストークについてはいくつかの機構が提案されていたが,シード配列とRNA結合タンパク質とのクロストークは新しいシナリオである.第3に,RNAi法によるゲノムワイドながん遺伝子のスクリーニングのデータを用いた解析から,データのデコンボリューションのプロセスを改善することにより機能ゲノミクスのデータの解釈が向上することが示唆された.CRISPR-Cas9系を用いた遺伝子のスクリーニングにおいてもオフターゲット効果を除く試みがなされているが9),RNAi法あるいはCRISPR-Cas9系を用いた遺伝子のスクリーニングのそれぞれの長所を考慮すると4),さらなるデータのデコンボリューションのプロセスの改善が両者のプラットフォームの相互的な補完を可能にすると考えられる.
文 献
- Suzuki, H. I., Katsura, A., Yasuda, T. et al.: Small-RNA asymmetry is directly driven by mammalian Argonautes. Nat. Struct. Mol. Biol., 22, 512-521 (2015)[PubMed] [新着論文レビュー]
- Konig, R., Chiang, C. Y., Tu, B. P. et al.: A probability-based approach for the analysis of large-scale RNAi screens. Nat. Methods, 4, 847-849 (2007)[PubMed]
- Shao, D. D., Tsherniak, A., Gopal, S. et al.: ATARiS: computational quantification of gene suppression phenotypes from multisample RNAi screens. Genome Res., 23, 665-678 (2013)[PubMed]
- Tsherniak, A., Vazquez, F., Montgomery, P. G. et al.: Defining a cancer dependency map. Cell, 170, 564-576.e516 (2017)[PubMed]
- McDonald, E. R. 3rd, de Weck, A., Schlabach, M. R. et al.: Project DRIVE: a compendium of cancer dependencies and synthetic lethal relationships uncovered by large-scale, deep RNAi screening. Cell, 170, 577-592.e510 (2017)[PubMed]
- Bosson, A. D., Zamudio, J. R. & Sharp, P. A.: Endogenous miRNA and target concentrations determine susceptibility to potential ceRNA competition. Mol. Cell, 56, 347-359 (2014)[PubMed]
- van Kouwenhove, M., Kedde, M. & Agami, R.: MicroRNA regulation by RNA-binding proteins and its implications for cancer. Nat. Rev. Cancer, 11, 644-656 (2011)[PubMed]
- Suzuki, H. I., Young, R. A. & Sharp, P. A.: Super-enhancer-mediated RNA processing revealed by integrative microRNA network analysis. Cell, 168, 1000-1014.e1015 (2017)[PubMed] [新着論文レビュー]
- Aguirre, A. J., Meyers, R. M., Weir, B. A. et al.: Genomic copy number dictates a gene-independent cell response to CRISPR/Cas9 targeting. Cancer Discov., 6, 914-929 (2016)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2010年 東京大学大学院医学研究科 早期修了,同年 同 特任助教を経て,2014年より米国Massachusetts Institute of Technology客員研究員.
関心事:ゲノム相分離.
Phillip A. Sharp
米国Massachusetts Institute of TechnologyにてInstitute Professor.
研究室URL:http://web.mit.edu/sharplab/
© 2018 鈴木 洋・Phillip A. Sharp Licensed under CC 表示 2.1 日本