細菌RNAポリメラーゼと転写因子Gfh1との複合体のX線結晶解析
田上俊輔1・関根俊一2・横山茂之1
(1理化学研究所生命分子システム基盤研究領域,2東京大学大学院理学系研究科 生物化学専攻)
email:横山茂之
DOI: 10.7875/first.author.2010.072
Crystal structure of bacterial RNA polymerase bound with a transcription inhibitor protein.
Shunsuke Tagami, Shun-ichi Sekine, Thirumananseri Kumarevel, Nobumasa Hino, Yuko Murayama, Syunsuke Kamegamori, Masaki Yamamoto, Kensaku Sakamoto, Shigeyuki Yokoyama
Nature, 468, 978-982 (2010)
RNAポリメラーゼは細胞における正確な遺伝子発現を実現するためにさまざまな転写因子による制御をうけている.この研究では,細菌のRNAポリメラーゼのはたらきを阻害するGfh1という転写因子に注目し,RNAポリメラーゼとGfh1との複合体のX線結晶構造解析を行った.その結果,Gfh1は基質であるヌクレオチド三リン酸を取り込むためのチャネルを完全にふさぐことで転写反応を阻害していることが明らかになった.さらに,RNAポリメラーゼ自体の大きな構造変化も観察された.一般に,タンパク質の構造変化はその機能においてなんらかの役割を担っている.この研究で観察されたRNAポリメラーゼの構造変化もその多様な機能を実現するために必要なのかもしれない.
RNAポリメラーゼはヌクレオチド三リン酸をつなぎあわせることでRNAを合成して転写反応を行う.細菌のRNAポリメラーゼ,古細菌のRNAポリメラーゼ,真核生物のRNAポリメラーゼであるPol I,Pol II,Pol IIIは配列および構造に高い相同性があり,共通の祖先タンパク質から進化してきたものと考えられる.したがって,RNAポリメラーゼによる転写反応の機構は基本的に細菌,古細菌,真核生物で共通であるものと考えられる.
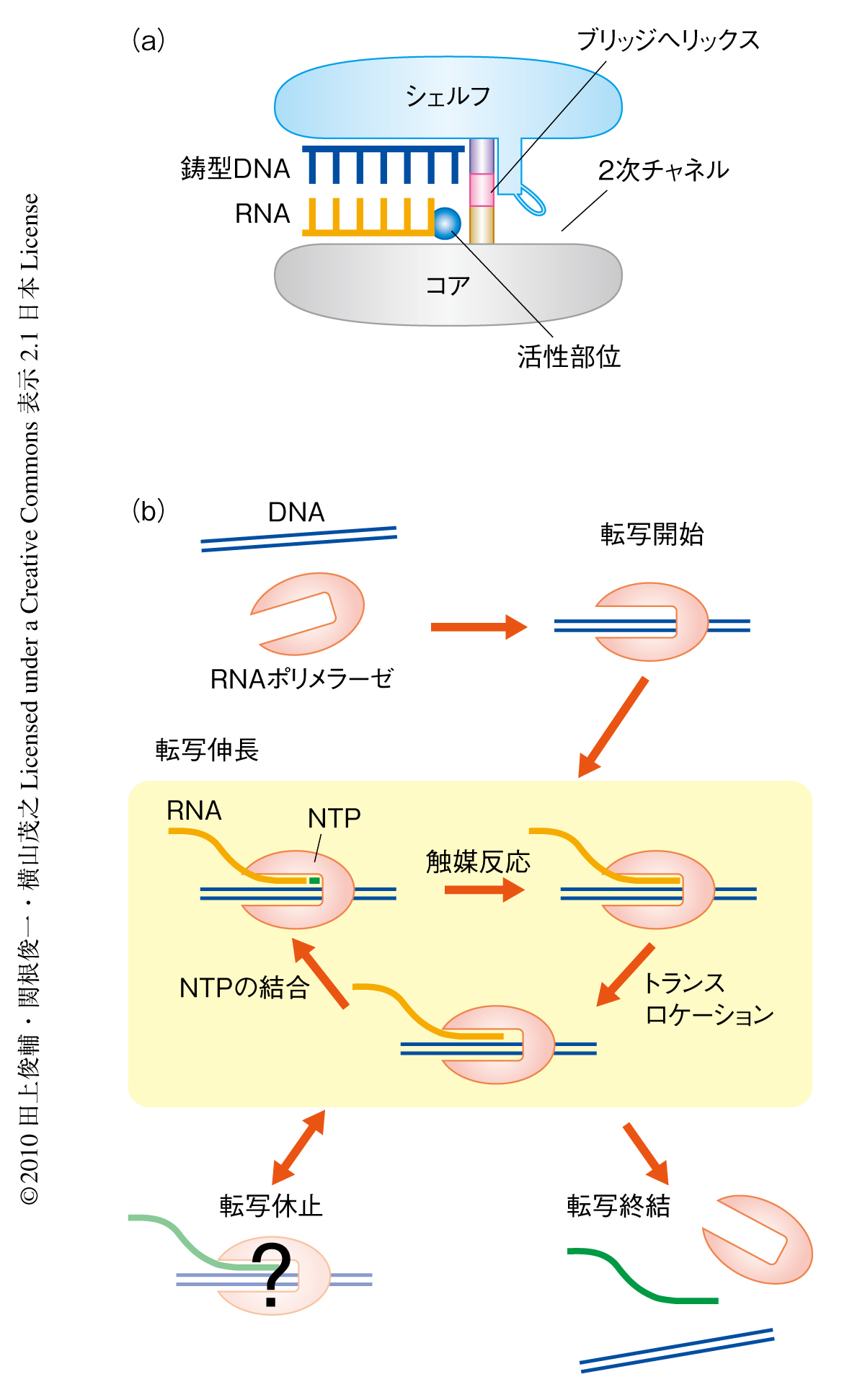
RNAポリメラーゼは約400 kDaの巨大なタンパク質複合体であり,その構造解析は一般のタンパク質と比較して困難である.これまでに,RNAポリメラーゼのX線結晶構造解析は細菌のRNAポリメラーゼと出芽酵母のPol IIを中心に行われてきており,RNAポリメラーゼと核酸,ヌクレオチド三リン酸などとの複合体の構造解析によりRNA合成反応の触媒機構が明らかになってきている1-4).RNAポリメラーゼはカニのハサミのようなかたちをしており,そのハサミの付け根にRNA合成反応の活性部位がある(図1a).RNAポリメラーゼにおいて合成反応中のRNAは鋳型DNAと塩基対を保持しており,8~9塩基対からなるDNA-RNAハイブリッド2本鎖を形成している.このDNA-RNAハイブリッド2本鎖はRNAポリメラーゼのハサミにはさまれている.ハサミの後ろ側には活性部位に通じる穴があいており2次チャネルとよばれている.この2次チャネルはRNA合成の際のヌクレオチド三リン酸の取り込み口であると考えられている.活性部位のとなりにはブリッジヘリックスとよばれる長いαヘリックスがありハサミの上下をつないでいる.
このように,RNAポリメラーゼの基本的な立体構造は報告されているが,転写の分子機構の大部分は未解明のままである.それは,転写が“さまざまな制御”をうけていること,そして,“複雑な多段階反応”であることによる.転写は遺伝子発現の第1段階であり,RNAポリメラーゼは遺伝子発現において中心的な役割を担う酵素のひとつである.細胞ではそのときどきの状況にあわせて必要な遺伝子を必要な量だけ転写する.そのため,RNAポリメラーゼによる転写反応は“正確に制御”されていなければならない.
また,転写反応はさまざまな段階を含む複雑なプロセスである(図1b).まず,RNAポリメラーゼはDNAのプロモーター領域と結合し,DNAの2本鎖をほどいて転写反応を開始させる.つぎに,DNAや合成途中のRNAとの結合を安定に保ちつつ数千塩基にもおよぶRNAを合成する.このとき,RNAポリメラーゼはRNAの3’末端にヌクレオチドを付加したのち,つぎのヌクレオチド三リン酸と鋳型DNAの塩基が活性部位に入ってくるようDNAにそって1塩基分だけトランスロケーションすることで連続的なポリメラーゼ反応を行っている.RNAポリメラーゼはヌクレオチド付加反応と同じ活性部位を用いてRNAの校正反応を行うことも知られている5,6).さらに,転写速度の調節や転写因子が結合するための時間の確保のため転写休止というプロセスが用いられているとも考えられている7).最後に,RNAポリメラーゼはDNAやRNAから解離し転写反応は終結する.RNAポリメラーゼはこのような複雑な反応を行うため“多機能性”をもつ必要がある.
RNAポリメラーゼにかぎらず,一般に,ある酵素に“正確な制御能”や“多機能性”をもたせるには2つの方法が考えられる.ひとつは“酵素自体の構造変化”であり,もうひとつは“制御因子の結合”である.そこで,転写の分子機構を明らかにするには,“RNAポリメラーゼの構造変化を観察すること”,“転写因子がRNAポリメラーゼにどのように作用しているかを明らかにすること”,が必須であり,そのため構造生物学的な研究が重要になる.しかし,RNAポリメラーゼは複数のサブユニットをもつ非常に大きな酵素であるためその構造解析は非常に困難であり,これまでに報告されている“RNAポリメラーゼの構造状態”や“RNAポリメラーゼと転写因子との複合体の構造”はごくかぎられていた.
高度好熱菌Thermus属でみつかった転写因子Gfh1はRNAポリメラーゼへのヌクレオチド三リン酸の結合に競合阻害的にはたらくことが報告されていた8-11).このことから,細胞が不利な環境におかれた際にはGfh1が転写を阻害することで細胞の活動を休止させるのではないかと提唱されている11).この研究では,Gfh1がRNAポリメラーゼに対してどのようにはたらくのかを解明するため,Thermus thermophilusのRNAポリメラーゼ-Gfh1複合体のX線結晶構造解析を行った(PDB ID:3AOH,3AOI).その結果,RNAポリメラーゼ-Gfh1複合体の結晶構造から“Gfh1による転写阻害機構”の詳細を説明することができた.また,この結晶構造ではRNAポリメラーゼがこれまでに報告されていた構造とは異なる状態にあることが観察された.この“RNAポリメラーゼの構造変化”はこれまでまったく予測されていなかったもので,転写の複雑な分子機構を理解するための基礎となると考えられた.
RNAポリメラーゼ-Gfh1複合体の結晶構造はGfh1による転写の阻害機構を明確に説明した.RNAポリメラーゼ-Gfh1複合体においてGfh1はRNAポリメラーゼのヌクレオチド三リン酸の取り込み口である2次チャネルに結合していた(図2a).Gfh1は2次チャネルを貫通しており,その先端はRNAポリメラーゼの活性部位の付近にまで達していた.Gfh1は2次チャネルを完全にふさいでおり,活性部位へのヌクレオチド三リン酸の取り込みを阻害することが示された.さらに,Gfh1の先端はRNA合成反応におけるヌクレオチド三リン酸のβリン酸基~γリン酸基の結合部位を占有しており,直接的にヌクレオチド三リン酸の結合を阻害していた.これらの観察結果は,Gfh1がヌクレオチド三リン酸の結合に対して競合阻害的にはたらくという生化学的な実験の結果を十分に説明した.
RNAポリメラーゼ-Gfh1複合体の構造を過去に報告されていたRNAポリメラーゼの結晶構造2)(PDB ID:2O5I)と比較したところ,この複合体のRNAポリメラーゼは構造が大きく変化していた.構造変化は2か所で観察されており,それらの構造変化をそれぞれ“ラチェット”と“スイング”と名づけた(図2a).
RNAポリメラーゼ-Gfh1複合体のRNAポリメラーゼはラチェットによってRNAポリメラーゼのDNA-RNA結合チャネルを形成する2つの構成モジュール(コアとシェルフ)が相互に約7度回転していた(図2b).ラチェット状態ではDNA-RNA結合チャネルが広がっており,その結果,RNAポリメラーゼとDNA-RNAハイブリッド2本鎖との結合が弱められているものと考えられた.また,構成モジュールのあいだの回転により2つの構成モジュールをつなぐブリッジヘリックスが中央で折れ曲がりDNA-RNA結合チャネルにちょうど1塩基分だけ突き出ていた.ラチェットによって2次チャネルも広がっていたが,ラチェット状態でないと2次チャネルが狭すぎてGfh1が結合できないことも示された.RNAポリメラーゼは通常の状態とラチェット状態を行き来しており,ラチェット状態になったときにGfh1が結合するものと考えられた.結晶構造ではGfh1がRNAポリメラーゼの2次チャネルにおさまることでラチェット状態がトラップされていたともいえた.
では,RNAポリメラーゼがラチェット状態をとることは転写の分子機構においてどのような意味をもつのだろうか.ここで,筆者らは,ラチェット構造を経由してRNAポリメラーゼのトランスロケーションが行われるというモデルを考案した.さきに述べたとおり,ラチェット状態では活性部位のとなりのブリッジヘリックスが折れ曲がってDNA-RNA結合チャネルに突き出ていた.その結果,DNA-RNAハイブリッド2本鎖はトランスロケーション後の位置に結合していた.しかしながら,突き出したブリッジヘリックスが鋳型DNAのつぎの塩基が活性部位に入ってくることをさまたげており,RNAポリメラーゼは完全にはトランスロケーション後の状態にはなっていなかった.そこで,RNAポリメラーゼがラチェット状態をとり,一時的にDNA-RNAハイブリッド2本鎖との結合を弱めつつ,同時にブリッジヘリックスを折り曲げてDNA-RNAハイブリッド2本鎖を1塩基分だけ押し込むという分子機構によってトランスロケーションが行われているのではないかと考えた(図2b).ラチェット状態ではRNAポリメラーゼとDNAあるいはRNAとの相互作用が大きく変わるので,トランスロケーションのほかにも転写開始や転写休止といった複数の過程でラチェット状態が現われる可能性もある.
RNAポリメラーゼ-Gfh1複合体ではラチェットでの構造変化にくわえて,RNAポリメラーゼの中心部(シェルフ)から飛び出したクランプとよばれる部位が15度回転していて,この構造変化をスイングと名づけた.クランプはGfh1の結合部位から離れた位置にあり,スイングはGfh1の結合とは直接の関係のないものと考えられる.結晶化したRNAポリメラーゼ-Gfh1複合体には上流にヘアピン構造を含むRNAが含まれているが,クランプのスイングはこのヘアピン構造の影響ではないかと考えられた.通常状態のRNAポリメラーゼの上流のRNA結合部位はヘアピン構造を含むRNAを収納するには狭いと考えられたが,RNAポリメラーゼ-Gfh1複合体ではスイングによってこの上流のRNA結合部位が大きく広がっていた.その結果,RNA結合部位からRNAがはずれるのに十分な割れ目もできていた.ただし,上流のヘアピン構造は結晶では固定されていないのか,その電子密度を観察することはできなかった.
細菌の転写の終結機構のひとつとしてRho因子に依存しない転写終結(intrinsic termination)があり,転写反応中のRNAに含まれるヘアピン構造とそれにつづくUリッチ配列が転写終結シグナルとなることが知られている12,13).今回のRNAポリメラーゼ-Gfh1複合体ではRNAのヘアピン構造により転写終結の初期段階に類似した状態が結晶化された可能性もあった.このことから,転写終結の際にはRNAヘアピン構造の形成によってクランプがスイングし,上流のRNA結合部位が大きく開くことで転写反応中のRNAがRNAポリメラーゼからはずれるのではないかと考えられた.
この研究では,RNAポリメラーゼ-Gfh1複合体のX線結晶構造解析によってGfh1の作用機構を明らかにした.この構造はGfh1のホモログであるGreAやGreBといった転写因子の作用機構の解明の基礎にもなると考えられた.今回,観察されたRNAポリメラーゼの構造変化はトランスロケーションや転写終結といった過程で重要な役割を演じているものと予想された.近年,リボソームについてもラチェットのような構造変化をすることが報告されている14,15).このような構造変化の情報によって,複雑な多段階反応を行いながら移動していく酵素の作用機構の理解の進むことが期待される.
略歴:2010年 東京大学大学院理学系研究科博士課程 修了,同年より理化学研究所生命分子システム基盤研究領域 特別研究員.
研究テーマ:RNAポリメラーゼのダイナミックな構造変化による転写の分子機構の解明.
抱負:生体高分子のメカニカルな作用機構を理解する.冒険的研究人生.健康.
関根 俊一(Shun-ichi Sekine)
東京大学大学院理学系研究科 特任准教授.
横山 茂之(Shigeyuki Yokoyama)
理化学研究所生命分子システム基盤研究領域 領域長,東京大学大学院理学系研究科 兼任教授.
© 2010 田上俊輔・関根俊一・横山茂之 Licensed under CC 表示 2.1 日本
(1理化学研究所生命分子システム基盤研究領域,2東京大学大学院理学系研究科 生物化学専攻)
email:横山茂之
DOI: 10.7875/first.author.2010.072
Crystal structure of bacterial RNA polymerase bound with a transcription inhibitor protein.
Shunsuke Tagami, Shun-ichi Sekine, Thirumananseri Kumarevel, Nobumasa Hino, Yuko Murayama, Syunsuke Kamegamori, Masaki Yamamoto, Kensaku Sakamoto, Shigeyuki Yokoyama
Nature, 468, 978-982 (2010)
要 約
RNAポリメラーゼは細胞における正確な遺伝子発現を実現するためにさまざまな転写因子による制御をうけている.この研究では,細菌のRNAポリメラーゼのはたらきを阻害するGfh1という転写因子に注目し,RNAポリメラーゼとGfh1との複合体のX線結晶構造解析を行った.その結果,Gfh1は基質であるヌクレオチド三リン酸を取り込むためのチャネルを完全にふさぐことで転写反応を阻害していることが明らかになった.さらに,RNAポリメラーゼ自体の大きな構造変化も観察された.一般に,タンパク質の構造変化はその機能においてなんらかの役割を担っている.この研究で観察されたRNAポリメラーゼの構造変化もその多様な機能を実現するために必要なのかもしれない.
はじめに
RNAポリメラーゼはヌクレオチド三リン酸をつなぎあわせることでRNAを合成して転写反応を行う.細菌のRNAポリメラーゼ,古細菌のRNAポリメラーゼ,真核生物のRNAポリメラーゼであるPol I,Pol II,Pol IIIは配列および構造に高い相同性があり,共通の祖先タンパク質から進化してきたものと考えられる.したがって,RNAポリメラーゼによる転写反応の機構は基本的に細菌,古細菌,真核生物で共通であるものと考えられる.
RNAポリメラーゼは約400 kDaの巨大なタンパク質複合体であり,その構造解析は一般のタンパク質と比較して困難である.これまでに,RNAポリメラーゼのX線結晶構造解析は細菌のRNAポリメラーゼと出芽酵母のPol IIを中心に行われてきており,RNAポリメラーゼと核酸,ヌクレオチド三リン酸などとの複合体の構造解析によりRNA合成反応の触媒機構が明らかになってきている1-4).RNAポリメラーゼはカニのハサミのようなかたちをしており,そのハサミの付け根にRNA合成反応の活性部位がある(図1a).RNAポリメラーゼにおいて合成反応中のRNAは鋳型DNAと塩基対を保持しており,8~9塩基対からなるDNA-RNAハイブリッド2本鎖を形成している.このDNA-RNAハイブリッド2本鎖はRNAポリメラーゼのハサミにはさまれている.ハサミの後ろ側には活性部位に通じる穴があいており2次チャネルとよばれている.この2次チャネルはRNA合成の際のヌクレオチド三リン酸の取り込み口であると考えられている.活性部位のとなりにはブリッジヘリックスとよばれる長いαヘリックスがありハサミの上下をつないでいる.
このように,RNAポリメラーゼの基本的な立体構造は報告されているが,転写の分子機構の大部分は未解明のままである.それは,転写が“さまざまな制御”をうけていること,そして,“複雑な多段階反応”であることによる.転写は遺伝子発現の第1段階であり,RNAポリメラーゼは遺伝子発現において中心的な役割を担う酵素のひとつである.細胞ではそのときどきの状況にあわせて必要な遺伝子を必要な量だけ転写する.そのため,RNAポリメラーゼによる転写反応は“正確に制御”されていなければならない.
また,転写反応はさまざまな段階を含む複雑なプロセスである(図1b).まず,RNAポリメラーゼはDNAのプロモーター領域と結合し,DNAの2本鎖をほどいて転写反応を開始させる.つぎに,DNAや合成途中のRNAとの結合を安定に保ちつつ数千塩基にもおよぶRNAを合成する.このとき,RNAポリメラーゼはRNAの3’末端にヌクレオチドを付加したのち,つぎのヌクレオチド三リン酸と鋳型DNAの塩基が活性部位に入ってくるようDNAにそって1塩基分だけトランスロケーションすることで連続的なポリメラーゼ反応を行っている.RNAポリメラーゼはヌクレオチド付加反応と同じ活性部位を用いてRNAの校正反応を行うことも知られている5,6).さらに,転写速度の調節や転写因子が結合するための時間の確保のため転写休止というプロセスが用いられているとも考えられている7).最後に,RNAポリメラーゼはDNAやRNAから解離し転写反応は終結する.RNAポリメラーゼはこのような複雑な反応を行うため“多機能性”をもつ必要がある.
RNAポリメラーゼにかぎらず,一般に,ある酵素に“正確な制御能”や“多機能性”をもたせるには2つの方法が考えられる.ひとつは“酵素自体の構造変化”であり,もうひとつは“制御因子の結合”である.そこで,転写の分子機構を明らかにするには,“RNAポリメラーゼの構造変化を観察すること”,“転写因子がRNAポリメラーゼにどのように作用しているかを明らかにすること”,が必須であり,そのため構造生物学的な研究が重要になる.しかし,RNAポリメラーゼは複数のサブユニットをもつ非常に大きな酵素であるためその構造解析は非常に困難であり,これまでに報告されている“RNAポリメラーゼの構造状態”や“RNAポリメラーゼと転写因子との複合体の構造”はごくかぎられていた.
高度好熱菌Thermus属でみつかった転写因子Gfh1はRNAポリメラーゼへのヌクレオチド三リン酸の結合に競合阻害的にはたらくことが報告されていた8-11).このことから,細胞が不利な環境におかれた際にはGfh1が転写を阻害することで細胞の活動を休止させるのではないかと提唱されている11).この研究では,Gfh1がRNAポリメラーゼに対してどのようにはたらくのかを解明するため,Thermus thermophilusのRNAポリメラーゼ-Gfh1複合体のX線結晶構造解析を行った(PDB ID:3AOH,3AOI).その結果,RNAポリメラーゼ-Gfh1複合体の結晶構造から“Gfh1による転写阻害機構”の詳細を説明することができた.また,この結晶構造ではRNAポリメラーゼがこれまでに報告されていた構造とは異なる状態にあることが観察された.この“RNAポリメラーゼの構造変化”はこれまでまったく予測されていなかったもので,転写の複雑な分子機構を理解するための基礎となると考えられた.
1.Gfh1による転写の阻害機構
RNAポリメラーゼ-Gfh1複合体の結晶構造はGfh1による転写の阻害機構を明確に説明した.RNAポリメラーゼ-Gfh1複合体においてGfh1はRNAポリメラーゼのヌクレオチド三リン酸の取り込み口である2次チャネルに結合していた(図2a).Gfh1は2次チャネルを貫通しており,その先端はRNAポリメラーゼの活性部位の付近にまで達していた.Gfh1は2次チャネルを完全にふさいでおり,活性部位へのヌクレオチド三リン酸の取り込みを阻害することが示された.さらに,Gfh1の先端はRNA合成反応におけるヌクレオチド三リン酸のβリン酸基~γリン酸基の結合部位を占有しており,直接的にヌクレオチド三リン酸の結合を阻害していた.これらの観察結果は,Gfh1がヌクレオチド三リン酸の結合に対して競合阻害的にはたらくという生化学的な実験の結果を十分に説明した.
2.RNAポリメラーゼの構造変化と機能の関係:ラチェット
RNAポリメラーゼ-Gfh1複合体の構造を過去に報告されていたRNAポリメラーゼの結晶構造2)(PDB ID:2O5I)と比較したところ,この複合体のRNAポリメラーゼは構造が大きく変化していた.構造変化は2か所で観察されており,それらの構造変化をそれぞれ“ラチェット”と“スイング”と名づけた(図2a).
RNAポリメラーゼ-Gfh1複合体のRNAポリメラーゼはラチェットによってRNAポリメラーゼのDNA-RNA結合チャネルを形成する2つの構成モジュール(コアとシェルフ)が相互に約7度回転していた(図2b).ラチェット状態ではDNA-RNA結合チャネルが広がっており,その結果,RNAポリメラーゼとDNA-RNAハイブリッド2本鎖との結合が弱められているものと考えられた.また,構成モジュールのあいだの回転により2つの構成モジュールをつなぐブリッジヘリックスが中央で折れ曲がりDNA-RNA結合チャネルにちょうど1塩基分だけ突き出ていた.ラチェットによって2次チャネルも広がっていたが,ラチェット状態でないと2次チャネルが狭すぎてGfh1が結合できないことも示された.RNAポリメラーゼは通常の状態とラチェット状態を行き来しており,ラチェット状態になったときにGfh1が結合するものと考えられた.結晶構造ではGfh1がRNAポリメラーゼの2次チャネルにおさまることでラチェット状態がトラップされていたともいえた.
では,RNAポリメラーゼがラチェット状態をとることは転写の分子機構においてどのような意味をもつのだろうか.ここで,筆者らは,ラチェット構造を経由してRNAポリメラーゼのトランスロケーションが行われるというモデルを考案した.さきに述べたとおり,ラチェット状態では活性部位のとなりのブリッジヘリックスが折れ曲がってDNA-RNA結合チャネルに突き出ていた.その結果,DNA-RNAハイブリッド2本鎖はトランスロケーション後の位置に結合していた.しかしながら,突き出したブリッジヘリックスが鋳型DNAのつぎの塩基が活性部位に入ってくることをさまたげており,RNAポリメラーゼは完全にはトランスロケーション後の状態にはなっていなかった.そこで,RNAポリメラーゼがラチェット状態をとり,一時的にDNA-RNAハイブリッド2本鎖との結合を弱めつつ,同時にブリッジヘリックスを折り曲げてDNA-RNAハイブリッド2本鎖を1塩基分だけ押し込むという分子機構によってトランスロケーションが行われているのではないかと考えた(図2b).ラチェット状態ではRNAポリメラーゼとDNAあるいはRNAとの相互作用が大きく変わるので,トランスロケーションのほかにも転写開始や転写休止といった複数の過程でラチェット状態が現われる可能性もある.
3.RNAポリメラーゼの構造変化と機能との関係:スイング
RNAポリメラーゼ-Gfh1複合体ではラチェットでの構造変化にくわえて,RNAポリメラーゼの中心部(シェルフ)から飛び出したクランプとよばれる部位が15度回転していて,この構造変化をスイングと名づけた.クランプはGfh1の結合部位から離れた位置にあり,スイングはGfh1の結合とは直接の関係のないものと考えられる.結晶化したRNAポリメラーゼ-Gfh1複合体には上流にヘアピン構造を含むRNAが含まれているが,クランプのスイングはこのヘアピン構造の影響ではないかと考えられた.通常状態のRNAポリメラーゼの上流のRNA結合部位はヘアピン構造を含むRNAを収納するには狭いと考えられたが,RNAポリメラーゼ-Gfh1複合体ではスイングによってこの上流のRNA結合部位が大きく広がっていた.その結果,RNA結合部位からRNAがはずれるのに十分な割れ目もできていた.ただし,上流のヘアピン構造は結晶では固定されていないのか,その電子密度を観察することはできなかった.
細菌の転写の終結機構のひとつとしてRho因子に依存しない転写終結(intrinsic termination)があり,転写反応中のRNAに含まれるヘアピン構造とそれにつづくUリッチ配列が転写終結シグナルとなることが知られている12,13).今回のRNAポリメラーゼ-Gfh1複合体ではRNAのヘアピン構造により転写終結の初期段階に類似した状態が結晶化された可能性もあった.このことから,転写終結の際にはRNAヘアピン構造の形成によってクランプがスイングし,上流のRNA結合部位が大きく開くことで転写反応中のRNAがRNAポリメラーゼからはずれるのではないかと考えられた.
おわりに
この研究では,RNAポリメラーゼ-Gfh1複合体のX線結晶構造解析によってGfh1の作用機構を明らかにした.この構造はGfh1のホモログであるGreAやGreBといった転写因子の作用機構の解明の基礎にもなると考えられた.今回,観察されたRNAポリメラーゼの構造変化はトランスロケーションや転写終結といった過程で重要な役割を演じているものと予想された.近年,リボソームについてもラチェットのような構造変化をすることが報告されている14,15).このような構造変化の情報によって,複雑な多段階反応を行いながら移動していく酵素の作用機構の理解の進むことが期待される.
文 献
- Gnatt, A. L., Cramer, P., Fu, J. et al.: Structural basis of transcription: an RNA polymerase II elongation complex at 3.3Å resolution. Science, 292, 1876-1882 (2001)[PubMed]
- Vassylyev, D. G., Vassylyeva, M. N., Perederina, A. et al.: Structural basis for transcription elongation by bacterial RNA polymerase. Nature, 448, 157-162 (2007)[PubMed]
- Vassylyev, D. G., Vassylyeva, M. N., Zhang, J. et al.: Structural basis for substrate loading in bacterial RNA polymerase. Nature, 448, 163-168 (2007)[PubMed]
- Wang, D., Bushnell, D. A., Westover, K. D. et al.: Structural basis of transcription: role of the trigger loop in substrate specificity and catalysis. Cell, 127, 941-954 (2006)[PubMed]
- Erie, D. A., Hajiseyedjavadi, O., Young, M. C. et al.: Multiple RNA polymerase conformations and GreA: control of the fidelity of transcription. Science, 262, 867-873 (1993)[PubMed]
- Jeon, C. & Agarwal, K.: Fidelity of RNA polymerase II transcription controlled by elongation factor TFIIS. Proc. Natl. Acad. Sci. USA, 93, 13677-13682 (1996)[PubMed]
- Artsimovitch, I. & Landick, R.: Pausing by bacterial RNA polymerase is mediated by mechanistically distinct classes of signals. Proc. Natl. Acad. Sci. USA, 97, 7090-7095 (2000)[PubMed]
- Hogan, B. P., Hartsch, T. & Erie, D. A.: Transcript cleavage by Thermus thermophilus RNA polymerase. Effects of GreA and anti-GreA factors. J. Biol. Chem., 277, 967-975 (2002)[PubMed]
- Lamour, V., Hogan, B. P., Erie, D. A. et al.: Crystal structure of Thermus aquaticus Gfh1, a Gre-factor paralog that inhibits rather than stimulates transcript cleavage. J. Mol. Biol., 356, 179-188 (2006)[PubMed]
- Laptenko, O. & Borukhov, S.: Biochemical assays of Gre factors of Thermus thermophilus. Methods Enzymol., 371, 219-232 (2003)[PubMed]
- Laptenko, O., Kim, S. S., Lee, J. et al.: pH-dependent conformational switch activates the inhibitor of transcription elongation. EMBO J., 25, 2131-2141 (2006)[PubMed]
- Brendel, V., Hamm, G. H. & Trifonov, E. N.: Terminators of transcription with RNA polymerase from Escherichia coli: what they look like and how to find them. J. Biomol. Struct. Dyn., 3, 705-723 (1986)[PubMed]
- Platt, T.: Transcription termination and the regulation of gene expression. Annu. Rev. Biochem., 55, 339-372 (1986)[PubMed]
- Zhang, W., Dunkle, J. A. & Cate, J. H.: Structures of the ribosome in intermediate states of ratcheting. Science, 325, 1014-1017 (2009)[PubMed]
- Julian, P., Konevega, A. L., Scheres, S. H. et al.: Structure of ratcheted ribosomes with tRNAs in hybrid states. Proc. Natl. Acad. Sci. USA, 105, 16924-16927 (2008)[PubMed]
著者プロフィール
略歴:2010年 東京大学大学院理学系研究科博士課程 修了,同年より理化学研究所生命分子システム基盤研究領域 特別研究員.
研究テーマ:RNAポリメラーゼのダイナミックな構造変化による転写の分子機構の解明.
抱負:生体高分子のメカニカルな作用機構を理解する.冒険的研究人生.健康.
関根 俊一(Shun-ichi Sekine)
東京大学大学院理学系研究科 特任准教授.
横山 茂之(Shigeyuki Yokoyama)
理化学研究所生命分子システム基盤研究領域 領域長,東京大学大学院理学系研究科 兼任教授.
© 2010 田上俊輔・関根俊一・横山茂之 Licensed under CC 表示 2.1 日本