ピロリジルtRNA合成酵素の結晶構造解析および進化分子工学的な手法による機能の改変
鈴木干城・Dieter Söll
(米国Yale大学Department of Molecular Biophysics and Biochemistry)
email:鈴木干城
DOI: 10.7875/first.author.2017.121
Crystal structures reveal an elusive functional domain of pyrrolysyl-tRNA synthetase.
Tateki Suzuki, Corwin Miller, Li-Tao Guo, Joanne M L Ho, David I Bryson, Yane-Shih Wang, David R Liu, Dieter Söll
Nature Chemical Biology, 13, 1261-1266 (2017)
ピロリジルtRNA合成酵素はタンパク質へのピロリジンの導入に関与する酵素であり,非天然アミノ酸を導入するためのツールとして細菌からヒトまで多くの系で使われている.この研究において,筆者らは,アミノ酸の導入に必須であるピロリジルtRNA合成酵素のN末端ドメインとtRNAPylとの複合体の結晶構造を決定した.その結果,ピロリジルtRNA合成酵素は通常のアミノアシルtRNA合成酵素において重要な認識部位であるアンチコドンを認識せず,可変ループの大きさを認識することによりtRNAPylに対する特異性を獲得したことが示された.さらに,ファージを用いた進化分子工学的な手法により,非天然アミノ酸の高い導入効率をもつピロリジルtRNA合成酵素の複数の変異体を取得した.アミノ酸配列を解析した結果,N末端ドメインへの変異はおのおのの変異体のあいだで保存されていた.また,変異を導入したN末端ドメインとtRNAPylとの複合体の結晶構造解析および速度論的な解析により,ピロリジルtRNA合成酵素のN末端ドメインにおける変異がどのように非天然アミノ酸の導入効率を上昇させるかについて考察した.
ピロリジンはメタン産生古細菌Methanosarciana barkeriのもつモノメチルアミンメチルトランスフェラーゼの活性部位にて発見された22番目のタンパク質性のアミノ酸である1).M. barkeriはメチルアミンをエネルギー源として利用し,その代謝によりメタンを産生する.この代謝経路において,モノメチルアミンメチルトランスフェラーゼはメチルアミンと結合しメチル基転移反応を行う.ピロリジンはモノメチルアミンメチルトランスフェラーゼの活性部位にメチルアミンを適切に配置するのに必要であると考えられている.モノメチルアミンメチルトランスフェラーゼへのピロリジンの導入はピロリジルtRNA合成酵素という特殊なアミノアシルtRNA合成酵素により行われる.ピロリジルtRNA合成酵素は対応するtRNAPylとピロリジンとを結合してPyl-tRNAPylを合成する.合成されたPyl-tRNAPylはリボソームへと輸送され,mRNAにおいて終止コドンUAGを解読することによりタンパク質にピロリジンを導入する.ピロリジルtRNA合成酵素はtRNAと結合するN末端ドメインと触媒活性をもつC末端ドメインから構成され,現在までに,C末端ドメインの単独の構造2) およびC末端ドメインとtRNAPylとの複合体の構造3) が解析されている.N末端ドメインはタンパク質へのアミノ酸の導入に必須であるが4),構造は未知でありtRNAPylの認識様式は不明であった.
ピロリジルtRNA合成酵素は比較的大きなアミノ酸結合ポケットをもちアミノ酸の側鎖をゆるく認識する.この性質を利用して,C末端ドメインの立体構造の情報をもとにアミノ酸結合ポケットを改変することにより,100種類以上の非天然アミノ酸を対象とするタンパク質に導入できることが報告されている5).非天然アミノ酸のタンパク質への導入は翻訳後修飾の再現やタンパク質の蛍光標識などに利用される6,7).しかしながら,改変により作製されたピロリジルtRNA合成酵素の変異体の非天然アミノ酸に対する触媒効率は,野生型のピロリジルtRNA合成酵素のピロリジンに対する触媒効率と比べ100倍ほど低いことから8),非天然アミノ酸を含むタンパク質を大量に生産するためには触媒効率の改善が必要とされていた.
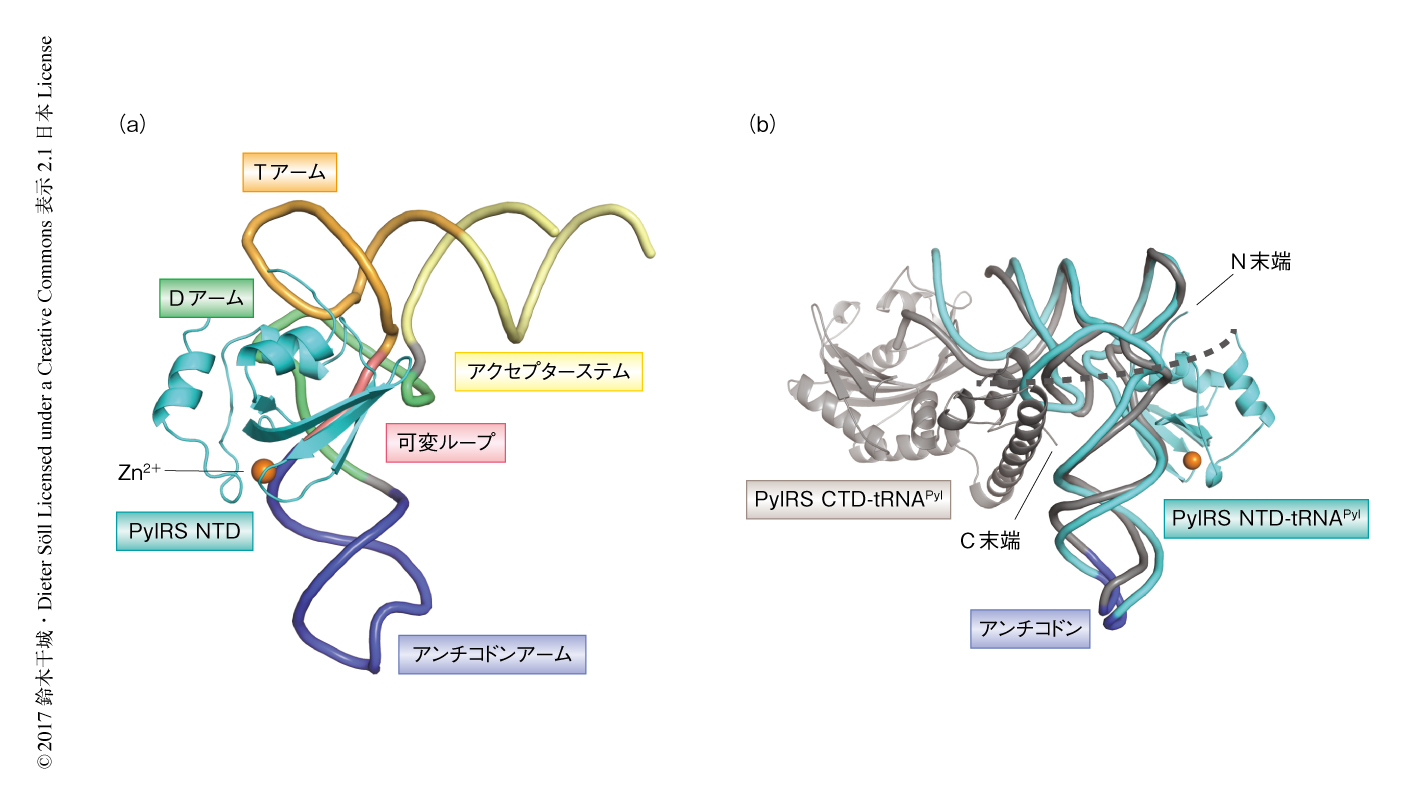
M. barkeriの近縁種であるMethanosarciana mazeiに由来するピロリジルtRNA合成酵素のN末端ドメインとtRNAPylとの複合体の結晶構造を2.4Å分解能で決定した(PDB ID:5UD5,図1a).N末端ドメインはコンパクトな球状をとり,Zn2+の配位により構造を安定化させていた.類似する構造を検索したところ,既知のアミノアシルtRNA合成酵素とは相同性をもたない新規のフォールドであった.N末端ドメインはtRNAPylのTアームと可変ループから構成されるくぼみにぴったりと結合していた.tRNAPylはもっともコンパクトな構造をもつtRNAとして知られており,より大きな可変ループをもつほかのtRNAは立体障害が生じるためピロリジルtRNA合成酵素に認識されないと考えられた.
ピロリジルtRNA合成酵素のN末端ドメインとtRNAPylとの複合体の構造と,C末端ドメインとtRNAPylとの複合体の構造とを重ね合わせ,全長のピロリジルtRNA合成酵素とtRNAPylとの複合体のモデルを作製した(図1b).その結果,N末端ドメインとC末端ドメインはそれぞれtRNAPylの反対側の面に結合し,tRNAに巻きつくように結合することがわかった.また,ピロリジルtRNA合成酵素とtRNAPylとの複合体はアミノアシルtRNA合成酵素とtRNAとの既知の複合体のなかで最大の相互作用面積をもっていた.さらに,N末端ドメインおよびC末端ドメインは通常のアミノアシルtRNA合成酵素において重要な認識部位であるアンチコドンを認識しないことが示された.これらの認識機構により,ピロリジルtRNA合成酵素は翻訳の直交性,すなわち,さまざまな生物種においてピロリジルtRNA合成酵素およびtRNAPylがほかのアミノアシルtRNA合成酵素およびtRNAと交差反応しないこと,を維持すると考えられた.
PACE(phage-assisted continuous evolution)法はファージを利用した進化分子工学的な手法で9),立体構造の情報なしにタンパク質を人工進化させることができ,これまで,ポリメラーゼ,受容体結合タンパク質,プロテアーゼ,アミノアシルtRNA合成酵素などの機能の改変に利用されている.この方法は培養液を循環させる特殊な機器を必要とするため,フラスコを使った連続希釈法を用いることによりこれを簡略化したPANCE(phage-assisted noncontinuous evolution)法を開発し,ピロリジルtRNA合成酵素の人工進化を試みた.この方法においては,大腸菌の培養液にピロリジルtRNA合成酵素の遺伝子をコードするファージ,アラビノース,非天然アミノ酸をくわえて培養したのち,培養液の一部を新鮮な大腸菌の培養液に植え継ぐ.アラビノースはファージが増殖する際にピロリジルtRNA合成酵素の遺伝子への変異の導入を促進する.また,高い活性をもつピロリジルtRNA合成酵素の遺伝子をもつものだけが増殖できるようファージは加工してあり,この過程をくり返すことにより結果的に高い活性をもつピロリジルtRNA合成酵素の遺伝子をもつファージだけが蓄積する.
非天然アミノ酸のひとつであるBoc-リジンに対し活性の高いピロリジルtRNA合成酵素を取得するのにPANCE法を適用し,その結果,野生型のピロリジルtRNA合成酵素よりも高い活性をもつ3つの変異体を取得した.これらの変異体のアミノ酸配列を確認したところ,変異はN末端ドメインおよびC末端ドメインにわたっていたが,N末端ドメインの変異は3つの変異体のあいだで比較的保存されていた.
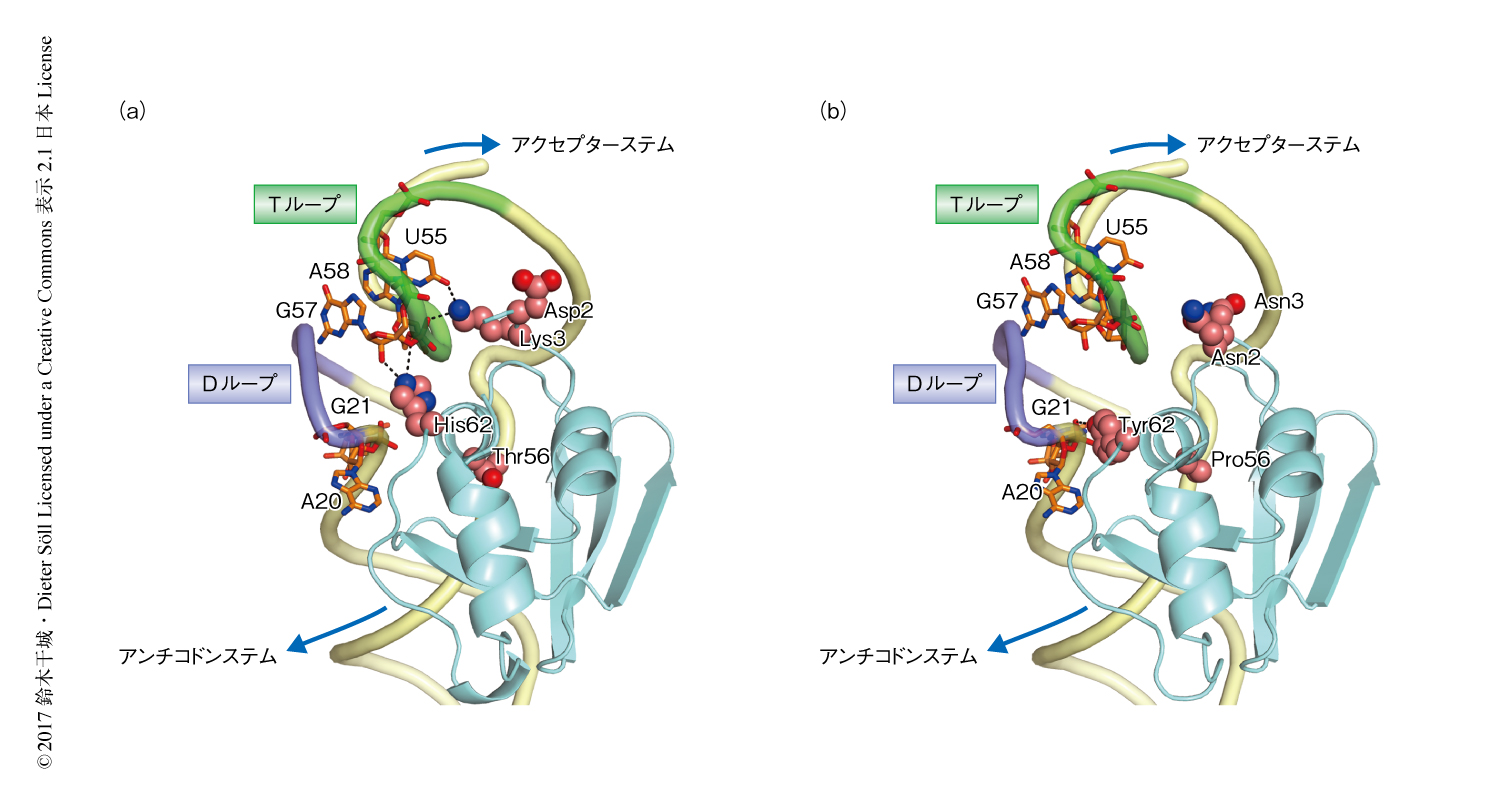
PANCE法により取得したピロリジルtRNA合成酵素の変異体はN末端ドメインに保存された変異をもっていたことから,取得した変異体のうちのひとつ32A変異体のN末端ドメインとtRNAPylとの複合体の結晶構造を2.8Å分解能で決定し(PDB ID:5V6X),導入された変異がtRNAPylとの相互作用にどのような影響をおよぼすのか調べた.その結果,32A変異体のN末端ドメインに導入された4つの変異のうち,2つはtRNAPylとの相互作用を弱めることが示唆された(図2).
PANCE法により導入された変異がピロリジルtRNA合成酵素の活性の向上にどのように寄与するのかさらに明らかにするため,速度論的な解析によりtRNAPyl,ピロリジン,Boc-リジンへの親和性を比較した.解析には野生型のピロリジルtRNA合成酵素,N末端ドメインおよびC末端ドメインに変異をもつ32A変異体にくわえ,N末端ドメインにのみ変異をもつ32A-Nter変異体を用いた.解析の結果,野生型のピロリジルtRNA合成酵素はBoc-リジンよりもピロリジンに対しきわめて高い親和性を示したが,32-Nter変異体はBoc-リジンに対する親和性を上昇させピロリジンに対する親和性を低下させていた.すなわち,N末端ドメインへの変異の導入はBoc-リジンに対する特異性を向上させた.一方,Boc-リジンの存在下において32A-Nter変異体は,野生型のピロリジルtRNA合成酵素に比べtRNAPylへの親和性が低くなった.これは,結晶構造において32A変異体のN末端ドメインとtRNAPylとの相互作用が低下していたことに相当すると考えられた.また,32A変異体は32A-Nter変異体と比較してtRNAPylに対する親和性が高かった.すなわち,C末端ドメインへの変異の導入はtRNAPylへの親和性を上昇させた.
以上の結果から,それぞれの変異がピロリジルtRNA合成酵素の活性におよぼす影響は以下のように考察された.N末端ドメインへの変異の導入はtRNAPylへの親和性を低下させ,その認識をフレキシブルにする.これにより,活性部位においてBoc-リジンと結合するのに最適なtRNAPylの配置が可能になり,Boc-リジンに対する特異性が向上する.また,C末端ドメインへの変異の導入はtRNAPylへの親和性を上昇させることによりN末端ドメインへの変異の導入による親和性の低下をおぎなうと考えられた.
近年,人工的なゲノムの合成により同義コドンの数を減らした大腸菌株が作製されつつある10,11).これらの大腸菌株は複数の空のコドンをもっており,複数の非天然アミノ酸を目的のタンパク質に導入するポテンシャルをもつ.ピロリジルtRNA合成酵素は触媒活性をもつC末端ドメインにあるアミノ酸結合ポケットの改変により100種類以上の非天然アミノ酸を導入できるようになったものの,すべてのピロリジルtRNA合成酵素の変異体は同一のtRNAPylしか認識できないため,目的のタンパク質に対し1種類の非天然アミノ酸しか導入できない.しかしながら,今回,明らかにされたピロリジルtRNA合成酵素のN末端ドメインとtRNAPylとの複合体の立体構造の情報をもとにN末端ドメインとtRNAPylとのあいだの相互作用を改変することにより,互いに直交なピロリジルtRNA合成酵素とtRNAPylのペアの作製が可能になる.そして,複数のピロリジルtRNA合成酵素とtRNAPylのペアを同時に用いることにより,複数の非天然アミノ酸を対象のタンパク質に導入できるようになると考えられる.導入できる非天然アミノ酸のバラエティが増えることにより,さらに高機能な生体高分子の開発が期待される.
略歴:2014年 北海道大学大学院生命科学院博士課程 修了,同年より米国Yale大学Postdoctoral Associate.
研究テーマ:翻訳に関連するタンパク質の構造生物学.
関心事:立体構造の情報を利用したタンパク質の改変.合成生物学.
Dieter Söll
米国Yale大学Sterling Professor.
研究室URL:http://trna.research.yale.edu/
© 2017 鈴木干城・Dieter Söll Licensed under CC 表示 2.1 日本
(米国Yale大学Department of Molecular Biophysics and Biochemistry)
email:鈴木干城
DOI: 10.7875/first.author.2017.121
Crystal structures reveal an elusive functional domain of pyrrolysyl-tRNA synthetase.
Tateki Suzuki, Corwin Miller, Li-Tao Guo, Joanne M L Ho, David I Bryson, Yane-Shih Wang, David R Liu, Dieter Söll
Nature Chemical Biology, 13, 1261-1266 (2017)
要 約
ピロリジルtRNA合成酵素はタンパク質へのピロリジンの導入に関与する酵素であり,非天然アミノ酸を導入するためのツールとして細菌からヒトまで多くの系で使われている.この研究において,筆者らは,アミノ酸の導入に必須であるピロリジルtRNA合成酵素のN末端ドメインとtRNAPylとの複合体の結晶構造を決定した.その結果,ピロリジルtRNA合成酵素は通常のアミノアシルtRNA合成酵素において重要な認識部位であるアンチコドンを認識せず,可変ループの大きさを認識することによりtRNAPylに対する特異性を獲得したことが示された.さらに,ファージを用いた進化分子工学的な手法により,非天然アミノ酸の高い導入効率をもつピロリジルtRNA合成酵素の複数の変異体を取得した.アミノ酸配列を解析した結果,N末端ドメインへの変異はおのおのの変異体のあいだで保存されていた.また,変異を導入したN末端ドメインとtRNAPylとの複合体の結晶構造解析および速度論的な解析により,ピロリジルtRNA合成酵素のN末端ドメインにおける変異がどのように非天然アミノ酸の導入効率を上昇させるかについて考察した.
はじめに
ピロリジンはメタン産生古細菌Methanosarciana barkeriのもつモノメチルアミンメチルトランスフェラーゼの活性部位にて発見された22番目のタンパク質性のアミノ酸である1).M. barkeriはメチルアミンをエネルギー源として利用し,その代謝によりメタンを産生する.この代謝経路において,モノメチルアミンメチルトランスフェラーゼはメチルアミンと結合しメチル基転移反応を行う.ピロリジンはモノメチルアミンメチルトランスフェラーゼの活性部位にメチルアミンを適切に配置するのに必要であると考えられている.モノメチルアミンメチルトランスフェラーゼへのピロリジンの導入はピロリジルtRNA合成酵素という特殊なアミノアシルtRNA合成酵素により行われる.ピロリジルtRNA合成酵素は対応するtRNAPylとピロリジンとを結合してPyl-tRNAPylを合成する.合成されたPyl-tRNAPylはリボソームへと輸送され,mRNAにおいて終止コドンUAGを解読することによりタンパク質にピロリジンを導入する.ピロリジルtRNA合成酵素はtRNAと結合するN末端ドメインと触媒活性をもつC末端ドメインから構成され,現在までに,C末端ドメインの単独の構造2) およびC末端ドメインとtRNAPylとの複合体の構造3) が解析されている.N末端ドメインはタンパク質へのアミノ酸の導入に必須であるが4),構造は未知でありtRNAPylの認識様式は不明であった.
ピロリジルtRNA合成酵素は比較的大きなアミノ酸結合ポケットをもちアミノ酸の側鎖をゆるく認識する.この性質を利用して,C末端ドメインの立体構造の情報をもとにアミノ酸結合ポケットを改変することにより,100種類以上の非天然アミノ酸を対象とするタンパク質に導入できることが報告されている5).非天然アミノ酸のタンパク質への導入は翻訳後修飾の再現やタンパク質の蛍光標識などに利用される6,7).しかしながら,改変により作製されたピロリジルtRNA合成酵素の変異体の非天然アミノ酸に対する触媒効率は,野生型のピロリジルtRNA合成酵素のピロリジンに対する触媒効率と比べ100倍ほど低いことから8),非天然アミノ酸を含むタンパク質を大量に生産するためには触媒効率の改善が必要とされていた.
1.野生型のピロリジルtRNA合成酵素のN末端ドメインとtRNAPylとの複合体の結晶構造解析
M. barkeriの近縁種であるMethanosarciana mazeiに由来するピロリジルtRNA合成酵素のN末端ドメインとtRNAPylとの複合体の結晶構造を2.4Å分解能で決定した(PDB ID:5UD5,図1a).N末端ドメインはコンパクトな球状をとり,Zn2+の配位により構造を安定化させていた.類似する構造を検索したところ,既知のアミノアシルtRNA合成酵素とは相同性をもたない新規のフォールドであった.N末端ドメインはtRNAPylのTアームと可変ループから構成されるくぼみにぴったりと結合していた.tRNAPylはもっともコンパクトな構造をもつtRNAとして知られており,より大きな可変ループをもつほかのtRNAは立体障害が生じるためピロリジルtRNA合成酵素に認識されないと考えられた.
ピロリジルtRNA合成酵素のN末端ドメインとtRNAPylとの複合体の構造と,C末端ドメインとtRNAPylとの複合体の構造とを重ね合わせ,全長のピロリジルtRNA合成酵素とtRNAPylとの複合体のモデルを作製した(図1b).その結果,N末端ドメインとC末端ドメインはそれぞれtRNAPylの反対側の面に結合し,tRNAに巻きつくように結合することがわかった.また,ピロリジルtRNA合成酵素とtRNAPylとの複合体はアミノアシルtRNA合成酵素とtRNAとの既知の複合体のなかで最大の相互作用面積をもっていた.さらに,N末端ドメインおよびC末端ドメインは通常のアミノアシルtRNA合成酵素において重要な認識部位であるアンチコドンを認識しないことが示された.これらの認識機構により,ピロリジルtRNA合成酵素は翻訳の直交性,すなわち,さまざまな生物種においてピロリジルtRNA合成酵素およびtRNAPylがほかのアミノアシルtRNA合成酵素およびtRNAと交差反応しないこと,を維持すると考えられた.
2.進化分子工学的な手法によるピロリジルtRNA合成酵素の活性の向上
PACE(phage-assisted continuous evolution)法はファージを利用した進化分子工学的な手法で9),立体構造の情報なしにタンパク質を人工進化させることができ,これまで,ポリメラーゼ,受容体結合タンパク質,プロテアーゼ,アミノアシルtRNA合成酵素などの機能の改変に利用されている.この方法は培養液を循環させる特殊な機器を必要とするため,フラスコを使った連続希釈法を用いることによりこれを簡略化したPANCE(phage-assisted noncontinuous evolution)法を開発し,ピロリジルtRNA合成酵素の人工進化を試みた.この方法においては,大腸菌の培養液にピロリジルtRNA合成酵素の遺伝子をコードするファージ,アラビノース,非天然アミノ酸をくわえて培養したのち,培養液の一部を新鮮な大腸菌の培養液に植え継ぐ.アラビノースはファージが増殖する際にピロリジルtRNA合成酵素の遺伝子への変異の導入を促進する.また,高い活性をもつピロリジルtRNA合成酵素の遺伝子をもつものだけが増殖できるようファージは加工してあり,この過程をくり返すことにより結果的に高い活性をもつピロリジルtRNA合成酵素の遺伝子をもつファージだけが蓄積する.
非天然アミノ酸のひとつであるBoc-リジンに対し活性の高いピロリジルtRNA合成酵素を取得するのにPANCE法を適用し,その結果,野生型のピロリジルtRNA合成酵素よりも高い活性をもつ3つの変異体を取得した.これらの変異体のアミノ酸配列を確認したところ,変異はN末端ドメインおよびC末端ドメインにわたっていたが,N末端ドメインの変異は3つの変異体のあいだで比較的保存されていた.
3.ピロリジルtRNA合成酵素のN末端ドメインに導入された変異が活性におよぼす影響
PANCE法により取得したピロリジルtRNA合成酵素の変異体はN末端ドメインに保存された変異をもっていたことから,取得した変異体のうちのひとつ32A変異体のN末端ドメインとtRNAPylとの複合体の結晶構造を2.8Å分解能で決定し(PDB ID:5V6X),導入された変異がtRNAPylとの相互作用にどのような影響をおよぼすのか調べた.その結果,32A変異体のN末端ドメインに導入された4つの変異のうち,2つはtRNAPylとの相互作用を弱めることが示唆された(図2).
PANCE法により導入された変異がピロリジルtRNA合成酵素の活性の向上にどのように寄与するのかさらに明らかにするため,速度論的な解析によりtRNAPyl,ピロリジン,Boc-リジンへの親和性を比較した.解析には野生型のピロリジルtRNA合成酵素,N末端ドメインおよびC末端ドメインに変異をもつ32A変異体にくわえ,N末端ドメインにのみ変異をもつ32A-Nter変異体を用いた.解析の結果,野生型のピロリジルtRNA合成酵素はBoc-リジンよりもピロリジンに対しきわめて高い親和性を示したが,32-Nter変異体はBoc-リジンに対する親和性を上昇させピロリジンに対する親和性を低下させていた.すなわち,N末端ドメインへの変異の導入はBoc-リジンに対する特異性を向上させた.一方,Boc-リジンの存在下において32A-Nter変異体は,野生型のピロリジルtRNA合成酵素に比べtRNAPylへの親和性が低くなった.これは,結晶構造において32A変異体のN末端ドメインとtRNAPylとの相互作用が低下していたことに相当すると考えられた.また,32A変異体は32A-Nter変異体と比較してtRNAPylに対する親和性が高かった.すなわち,C末端ドメインへの変異の導入はtRNAPylへの親和性を上昇させた.
以上の結果から,それぞれの変異がピロリジルtRNA合成酵素の活性におよぼす影響は以下のように考察された.N末端ドメインへの変異の導入はtRNAPylへの親和性を低下させ,その認識をフレキシブルにする.これにより,活性部位においてBoc-リジンと結合するのに最適なtRNAPylの配置が可能になり,Boc-リジンに対する特異性が向上する.また,C末端ドメインへの変異の導入はtRNAPylへの親和性を上昇させることによりN末端ドメインへの変異の導入による親和性の低下をおぎなうと考えられた.
おわりに
近年,人工的なゲノムの合成により同義コドンの数を減らした大腸菌株が作製されつつある10,11).これらの大腸菌株は複数の空のコドンをもっており,複数の非天然アミノ酸を目的のタンパク質に導入するポテンシャルをもつ.ピロリジルtRNA合成酵素は触媒活性をもつC末端ドメインにあるアミノ酸結合ポケットの改変により100種類以上の非天然アミノ酸を導入できるようになったものの,すべてのピロリジルtRNA合成酵素の変異体は同一のtRNAPylしか認識できないため,目的のタンパク質に対し1種類の非天然アミノ酸しか導入できない.しかしながら,今回,明らかにされたピロリジルtRNA合成酵素のN末端ドメインとtRNAPylとの複合体の立体構造の情報をもとにN末端ドメインとtRNAPylとのあいだの相互作用を改変することにより,互いに直交なピロリジルtRNA合成酵素とtRNAPylのペアの作製が可能になる.そして,複数のピロリジルtRNA合成酵素とtRNAPylのペアを同時に用いることにより,複数の非天然アミノ酸を対象のタンパク質に導入できるようになると考えられる.導入できる非天然アミノ酸のバラエティが増えることにより,さらに高機能な生体高分子の開発が期待される.
文 献
- Hao, B., Gong, W., Ferguson, T. K. et al.: A new UAG-encoded residue in the structure of a methanogen methyltransferase. Science, 296, 1462-1466 (2002)[PubMed]
- Kavran, J. M., Gundllapalli, S., O'Donoghue, P. et al.: Structure of pyrrolysyl-tRNA synthetase, an archaeal enzyme for genetic code innovation. Proc. Natl. Acad. Sci. USA, 104, 11268-11273 (2007)[PubMed]
- Nozawa, K., O'Donoghue, P., Gundllapalli, S. et al.: Pyrrolysyl-tRNA synthetase-tRNAPyl structure reveals the molecular basis of orthogonality. Nature, 457, 1163-1167 (2009)[PubMed]
- Herring, S., Ambrogelly, A., Gundllapalli, S. et al. .: The amino-terminal domain of pyrrolysyl-tRNA synthetase is dispensable in vitro but required for in vivo activity. FEBS Lett., 581, 3197-3203 (2007)[PubMed]
- Wan, W., Tharp, J. M. & Liu, W. R.: Pyrrolysyl-tRNA synthetase: an ordinary enzyme but an outstanding genetic code expansion tool. Biochim. Biophys. Acta, 1844, 1059-1070 (2014)[PubMed]
- Neumann, H., Peak-Chew, S. Y. & Chin, J. W.: Genetically encoding Nε-acetyllysine in recombinant proteins. Nat. Chem. Biol., 4, 232-234 (2008)[PubMed]
- Kaya, E., Vrabel, M., Deiml, C. et al.: A genetically encoded norbornene amino acid for the mild and selective modification of proteins in a copper-free click reaction. Angew. Chem. Int. Ed. Engl., 51, 4466-4469 (2012)[PubMed]
- Guo, L. T., Wang, Y. S., Nakamura, A. et al.: Polyspecific pyrrolysyl-tRNA synthetases from directed evolution. Proc. Natl. Acad. Sci. USA, 111, 16724-16729 (2014)[PubMed]
- Esvelt, K. M., Carlson, J. C. & Liu, D. R.: A system for the continuous directed evolution of biomolecules. Nature, 472, 499-503 (2011)[PubMed]
- Ostrov, N., Landon, M., Guell, M. et al.: Design, synthesis, and testing toward a 57-codon genome. Science, 353, 819-822 (2016)[PubMed]
- Wang, K., Fredens, J., Brunner, S. F. et al.: Defining synonymous codon compression schemes by genome recoding. Nature, 539, 59-64 (2016)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2014年 北海道大学大学院生命科学院博士課程 修了,同年より米国Yale大学Postdoctoral Associate.
研究テーマ:翻訳に関連するタンパク質の構造生物学.
関心事:立体構造の情報を利用したタンパク質の改変.合成生物学.
Dieter Söll
米国Yale大学Sterling Professor.
研究室URL:http://trna.research.yale.edu/
© 2017 鈴木干城・Dieter Söll Licensed under CC 表示 2.1 日本