小児のT細胞性急性リンパ性白血病における悪性度の高い新規のSPI1融合遺伝子の同定
関 正史・滝田順子
(東京大学大学院医学系研究科 小児科学分野)
email:関 正史
DOI: 10.7875/first.author.2017.082
Recurrent SPI1 (PU.1) fusions in high-risk pediatric T cell acute lymphoblastic leukemia.
Masafumi Seki, Shunsuke Kimura, Tomoya Isobe, Kenichi Yoshida, Hiroo Ueno, Yaeko Nakajima-Takagi, Changshan Wang, Lin Lin, Ayana Kon, Hiromichi Suzuki, Yusuke Shiozawa, Keisuke Kataoka, Yoichi Fujii, Yuichi Shiraishi, Kenichi Chiba, Hiroko Tanaka, Teppei Shimamura, Kyoko Masuda, Hiroshi Kawamoto, Kentaro Ohki, Motohiro Kato, Yuki Arakawa, Katsuyoshi Koh, Ryoji Hanada, Hiroshi Moritake, Masaharu Akiyama, Ryoji Kobayashi, Takao Deguchi, Yoshiko Hashii, Toshihiko Imamura, Atsushi Sato, Nobutaka Kiyokawa, Akira Oka, Yasuhide Hayashi, Masatoshi Takagi, Atsushi Manabe, Akira Ohara, Keizo Horibe, Masashi Sanada, Atsushi Iwama, Hiroyuki Mano, Satoru Miyano, Seishi Ogawa, Junko Takita
Nature Genetics, 49, 1274-1281 (2017)
小児のT細胞性急性リンパ性白血病は治療抵抗性の症例あるいは再発症例においては予後がきわめて不良であり,また,その遺伝学的な基盤は十分に解明されていない.筆者らは,小児のT細胞性急性リンパ性白血病の121の症例に対しトランスクリプトーム解析および塩基配列の決定による包括的なプロファイリングを行い,血球の分化にかかわる重要な転写因子であるPU.1をコードするSPI1遺伝子における新規の融合遺伝子を複数の例において同定した.SPI1融合遺伝子をもつ症例は解析した症例の3.9%をしめ,CD4陰性CD8陰性のダブルネガティブあるいはCD4陰性CD8陽性のシングルポジティブであり,一様に予後不良の経過を呈した.SPI1融合遺伝子をもつ症例は,T細胞へのコミットメント,アイデンティティの確立,分化に関与する遺伝子の発現,分化のプロファイルにおいて,既知のT細胞性急性リンパ性白血病とは独立してクラスタリングされた.SPI1融合遺伝子から翻訳されたタンパク質は転写活性を保持しており,マウスの造血幹細胞において恒常的に発現させたところ細胞の増殖を誘導し分化の停止をきたした.今回の結果により,リスクの高い小児のT細胞性急性リンパ性白血病におけるSPI1融合遺伝子の独特な機序が解明された.
小児においてT細胞性急性リンパ性白血病は急性リンパ性白血病の約15%をしめ,近年の化学療法の進歩にともないB前駆細胞性急性リンパ性白血病と遜色のない治療成績が得られるようになってきたが,治療抵抗性の症例や再発症例においていまだ救済は困難であり,また,病態に関連するゲノムの異常については十分に解明されていなかった.
小児のT細胞性急性リンパ性白血病の121の症例についてRNA-seq法によりトランスクリプトーム解析し,筆者らの研究グループで用いている解析プラットフォームであるGenomon 2.3.0を用いて融合遺伝子を検出した.その結果,既知の融合遺伝子にくわえ,血球の分化にかかわる重要な転写因子であるPU.1をコードするSPI1遺伝子における新規の融合遺伝子が5例において重複して検出され,これらは3’側にSPI1遺伝子,5’側にはSTMN1遺伝子あるいはTCF7遺伝子が同じ読み枠で融合していた.べつの60例のコホートについてRT-PCR法によりスクリーニングしたところ,TCF7-SPI1融合遺伝子が2例において同定され,合計で181例のうち7例においてSPI1融合遺伝子が検出された.イントロン領域を含めたキャプチャーによりすべての例においてゲノムの切断点を同定した.SPI1融合遺伝子をもつ症例はCD4陰性CD8陰性のダブルネガティブあるいはCD4陰性CD8陽性のシングルポジティブであり,いずれもCD2,CD3,CD5,CD7に陽性であった.SPI1融合遺伝子をもつ症例における特徴的な所見として,一般にはT細胞性急性リンパ性白血病において陽性になるとは知られていないHLA-DRがすべての例において陽性であった1).SPI1遺伝子の位置する領域における染色体の異常としては,STMN1-SPI1融合遺伝子をもつ1例にのみ染色体の転座が検出された.
SPI1遺伝子のコードするPU.1はETSファミリーに属する血球の分化にかかわる重要な転写因子であり,造血細胞の分化においてその発現は厳格に制御される.T細胞の分化において,コミットメントの前段階にかかわるフェーズ1遺伝子とよばれるLYL1遺伝子,LMO2遺伝子,GATA2遺伝子,MEIS1遺伝子,BCL11A遺伝子と同様に2),SPI1遺伝子は前胸腺前駆細胞や初期前駆T細胞において発現しており,DN2細胞からDN3細胞へ分化が進むと発現は低下し,フェーズ2遺伝子とよばれるTCF7遺伝子,NOTCH1遺伝子,RUNX1遺伝子,GATA3遺伝子,TCF3遺伝子,BCL11B遺伝子などの発現に置き換わり,そののち,フェーズ3遺伝子であるETS2遺伝子,LEF1遺伝子,ID3遺伝子などの発現が上昇する.SPI1融合遺伝子をもつ症例においては,いずれもSPI1遺伝子が高く発現していたが野生型のSPI1遺伝子の発現はほぼなく,これらの発現は5’側に融合したSTMN1遺伝子あるいはTCF7遺伝子のプロモーター活性により制御されていると考えられた.
融合した遺伝子によらずSPI1融合タンパク質はDNA結合領域であるETSドメインを保持していたが,C末端側の一部の領域は失っていた.SPI1融合タンパク質の転写因子としての活性について評価するため,PU.1との結合領域をもつCSF1R遺伝子のプロモーター配列を用いてルシフェラーゼアッセイを行った.その結果,STMN1-SPI1融合遺伝子あるいはTCF7-SPI1融合遺伝子を導入した細胞は野生型のSPI1遺伝子を導入した細胞と同等の活性を示したが,それぞれのETSドメインを欠失させると活性は完全に消失した.このことから,SPI1融合タンパク質はPU.1と同様に転写活性をもつことが示された.
in vitroの系においてSPI1融合遺伝子のT細胞の増殖への影響について検討した.マウスのダブルネガティブT細胞に野生型のSPI1遺伝子,STMN1-SPI1融合遺伝子,TCF7-SPI1融合遺伝子を導入したところ,SPI1融合遺伝子を導入した細胞において野生型のSPI1遺伝子を導入した細胞と同様の増殖が確認された.Tst4/DLL1共培養系を用いた分化の誘導の実験においては,野生型のSPI1遺伝子を導入した細胞と同様に,SPI1融合遺伝子を導入した細胞においてダブルポジティブ細胞,シングルポジティブ細胞,DN4細胞の減少,また,DN1細胞およびDN2細胞の増加が確認され,分化が停止していることが示された.また,in vitroの系において,マウスの造血幹細胞に野生型のSPI1遺伝子あるいはTCF7-SPI1融合遺伝子を導入したのちマウスへ移植したところ,ダブルネガティブ細胞の増加,CD4陽性CD8陰性のシングルポジティブ細胞およびCD4陰性CD8陽性のシングルポジティブ細胞の減少が確認された.
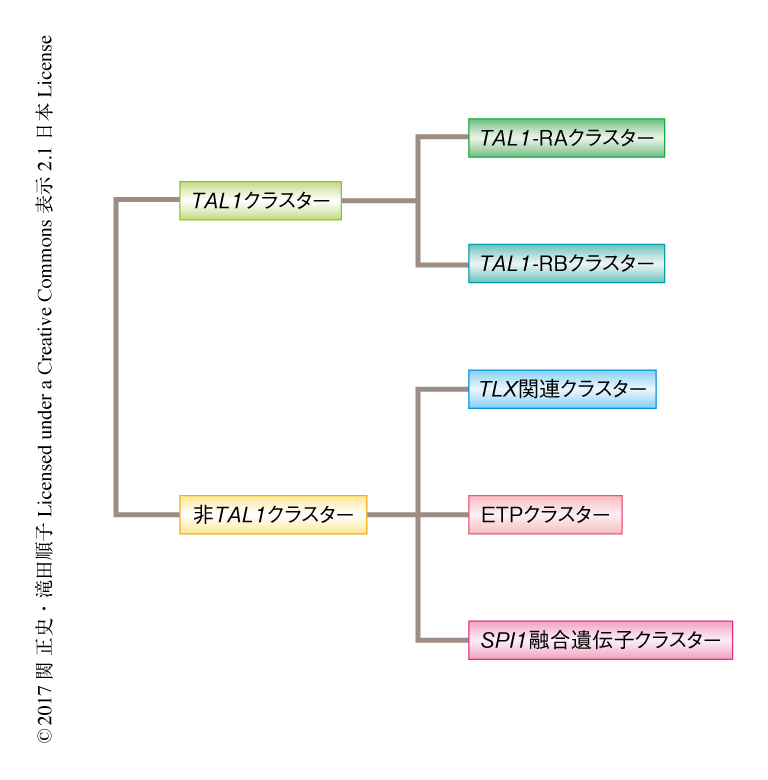
RNA-seq法によりトランスクリプトーム解析した121の症例にくわえ,べつのコホートにおいて同定され追加でトランスクリプトーム解析したTCF7-SPI1融合遺伝子をもつ2つの症例を含む123の症例について,発現プロファイルによりクラスタリングした.コンセンサスクラスタリングの手法を用い2段階でクラスタリングした(図1).まず,TAL1クラスターと非TAL1クラスターとに分類し,既報にある分類と同様に,TAL1クラスターはTAL1-RAクラスターとTAL1-RBクラスターの2つに,非TAL1クラスターは初期前駆T細胞と類似した発現プロファイルを示すETPクラスターとTLX1遺伝子あるいはTLX3遺伝子を高く発現するTLX関連クラスターに分類されたが3),ほかにSPI1融合遺伝子をもつ7つの症例のみで構成されるSPI1融合遺伝子クラスターも抽出され,あわせて5つにクラスタリングされた.SPI1融合遺伝子クラスターはDN3細胞あるいはDN4細胞に類似した転写因子の発現を呈しており,ほかのクラスターとは異なっていた.また,フェーズ1遺伝子の発現はSPI1融合遺伝子クラスターにおいては低下していたが,例外として,SPI1遺伝子とSPI1遺伝子のコードするPU.1により制御されるMEF2C遺伝子およびHHEX遺伝子は高く発現していた.そのほか,SPI1融合遺伝子クラスターにおいてはFLT3遺伝子およびKIT遺伝子も高く発現していたが,これらの遺伝子もPU.1により制御されているとする報告がある.TLX関連クラスター,TAL1-RAクラスター,TAL1-RBクラスターはダブルポジティブT細胞に相当すると考えられており,これらのクラスターにおいてみられたTLX1遺伝子,TLX3遺伝子,TAL1遺伝子,HOXA9遺伝子の高い発現は,SPI1融合遺伝子クラスターにおいてはまったくみられなかった.
おのおののクラスターとゲノムの異常との関連を調べるため,TARGETプロジェクトにより公開されている小児の急性リンパ性白血病におけるエキソーム解析の結果から抽出した158の遺伝子を標的となる領域として設計したカスタムベイトを用い,121の症例においてキャプチャーにより塩基配列を決定した.その結果,過去の報告と一致して,NOTCH1遺伝子およびCDKN2A遺伝子において高い頻度で異常が認められたが4-7),CDKN2A遺伝子の異常はETPクラスターにおいてはほかのクラスターと比べ頻度は有意に低かった.そのほか,10%以上の頻度で異常の認められた遺伝子として,PHF6遺伝子,FBXW7遺伝子,PTEN遺伝子,DNM2遺伝子,USP7遺伝子,NRAS遺伝子,JAK3遺伝子,WT1遺伝子,RUNX1遺伝子があり,USP7遺伝子以外は既知の異常として報告されていた.ETPクラスターはGATA3遺伝子,RUNX1遺伝子,NRAS遺伝子,ETV6遺伝子,さらに,エピジェネティック制御遺伝子の異常の頻度が高く,また,CDKN2A遺伝子の異常の頻度は低く,これは既報と一致する結果であった8,9).エピジェネティック制御遺伝子の異常の頻度はTLX関連クラスターにおいても高く,また,PHF6遺伝子およびWT1遺伝子の異常はTLX関連クラスターに特徴的なものと考えられた.TAL1-RAクラスターとTAL1-RBクラスターはPTEN遺伝子およびUSP7遺伝子の異常の頻度が高い点が共通していたが,TAL1-RBクラスターにおいて認められたNOTCH1遺伝子およびFBXW7遺伝子の異常,転写因子の異常,RAS経路の異常,エピジェネティック制御遺伝子の異常はTAL1-RAクラスターではほとんど認められなかった.USP7はPTENの脱ユビキチン化にかかわるが,USP7遺伝子の異常はT細胞性急性リンパ性白血病における新規の異常でありTAL1クラスターに集中して認められた.SPI1融合遺伝子クラスターは症例の数が少ないが,ほかのクラスターと同様に,NOTCH1遺伝子およびFBXW7遺伝子の異常,CDKN2A遺伝子の欠失の頻度が高く,そのほかの遺伝子の変異の頻度は低いが,RAS経路の異常の頻度が高かった.
SPI1融合遺伝子をもつ症例の予後について検討した.SPI1融合遺伝子をもたない症例と比較した場合,SPI1融合遺伝子をもつ症例は有意に生存期間が短く,生存期間の中央値は2.2年で,7例のうち6例は診断から3年以内に早期の再発により死亡していた.SPI1融合遺伝子をもつ症例をほかの融合遺伝子をもつ症例と比較した場合も,同様に生存期間は有意に短かった.多変量解析においてSPI1融合遺伝子は独立した予後因子として抽出され,SPI1融合遺伝子はアグレッシブな病型に関与することが示唆された.
小児のT細胞性急性リンパ性白血病において,SPI1融合遺伝子をもつ症例は特徴的な発現プロファイルをもち,きわめて予後の不良な臨床経過を呈した.小児のT細胞性急性リンパ性白血病においてこれほど不良な臨床像を示すゲノムの異常はこれまで報告されていなかったが,症例の数は少なく再現性の確認は必要である.SPI1融合遺伝子をもつ症例は既存の治療法では救済されないと思われ,より強力な治療法や代替となる治療法が必要であり,これらの症例を抽出する意義は大きい.RAS経路の異常が多く,また,FLT3遺伝子およびKIT遺伝子に異常に高い発現がみられたことから,今後,新規の治療の標的としての可能性があるだろう.
略歴:2016年 東京大学大学院医学系研究科にて博士号取得,同年より東京大学医学部附属病院 助教.
研究テーマ:小児の血液腫瘍および固形腫瘍.
関心事:転写因子.
滝田 順子(Junko Takita)
東京大学大学院医学系研究科 准教授.
© 2017 関 正史・滝田順子 Licensed under CC 表示 2.1 日本
(東京大学大学院医学系研究科 小児科学分野)
email:関 正史
DOI: 10.7875/first.author.2017.082
Recurrent SPI1 (PU.1) fusions in high-risk pediatric T cell acute lymphoblastic leukemia.
Masafumi Seki, Shunsuke Kimura, Tomoya Isobe, Kenichi Yoshida, Hiroo Ueno, Yaeko Nakajima-Takagi, Changshan Wang, Lin Lin, Ayana Kon, Hiromichi Suzuki, Yusuke Shiozawa, Keisuke Kataoka, Yoichi Fujii, Yuichi Shiraishi, Kenichi Chiba, Hiroko Tanaka, Teppei Shimamura, Kyoko Masuda, Hiroshi Kawamoto, Kentaro Ohki, Motohiro Kato, Yuki Arakawa, Katsuyoshi Koh, Ryoji Hanada, Hiroshi Moritake, Masaharu Akiyama, Ryoji Kobayashi, Takao Deguchi, Yoshiko Hashii, Toshihiko Imamura, Atsushi Sato, Nobutaka Kiyokawa, Akira Oka, Yasuhide Hayashi, Masatoshi Takagi, Atsushi Manabe, Akira Ohara, Keizo Horibe, Masashi Sanada, Atsushi Iwama, Hiroyuki Mano, Satoru Miyano, Seishi Ogawa, Junko Takita
Nature Genetics, 49, 1274-1281 (2017)
要 約
小児のT細胞性急性リンパ性白血病は治療抵抗性の症例あるいは再発症例においては予後がきわめて不良であり,また,その遺伝学的な基盤は十分に解明されていない.筆者らは,小児のT細胞性急性リンパ性白血病の121の症例に対しトランスクリプトーム解析および塩基配列の決定による包括的なプロファイリングを行い,血球の分化にかかわる重要な転写因子であるPU.1をコードするSPI1遺伝子における新規の融合遺伝子を複数の例において同定した.SPI1融合遺伝子をもつ症例は解析した症例の3.9%をしめ,CD4陰性CD8陰性のダブルネガティブあるいはCD4陰性CD8陽性のシングルポジティブであり,一様に予後不良の経過を呈した.SPI1融合遺伝子をもつ症例は,T細胞へのコミットメント,アイデンティティの確立,分化に関与する遺伝子の発現,分化のプロファイルにおいて,既知のT細胞性急性リンパ性白血病とは独立してクラスタリングされた.SPI1融合遺伝子から翻訳されたタンパク質は転写活性を保持しており,マウスの造血幹細胞において恒常的に発現させたところ細胞の増殖を誘導し分化の停止をきたした.今回の結果により,リスクの高い小児のT細胞性急性リンパ性白血病におけるSPI1融合遺伝子の独特な機序が解明された.
はじめに
小児においてT細胞性急性リンパ性白血病は急性リンパ性白血病の約15%をしめ,近年の化学療法の進歩にともないB前駆細胞性急性リンパ性白血病と遜色のない治療成績が得られるようになってきたが,治療抵抗性の症例や再発症例においていまだ救済は困難であり,また,病態に関連するゲノムの異常については十分に解明されていなかった.
1.新規のSPI1融合遺伝子の同定
小児のT細胞性急性リンパ性白血病の121の症例についてRNA-seq法によりトランスクリプトーム解析し,筆者らの研究グループで用いている解析プラットフォームであるGenomon 2.3.0を用いて融合遺伝子を検出した.その結果,既知の融合遺伝子にくわえ,血球の分化にかかわる重要な転写因子であるPU.1をコードするSPI1遺伝子における新規の融合遺伝子が5例において重複して検出され,これらは3’側にSPI1遺伝子,5’側にはSTMN1遺伝子あるいはTCF7遺伝子が同じ読み枠で融合していた.べつの60例のコホートについてRT-PCR法によりスクリーニングしたところ,TCF7-SPI1融合遺伝子が2例において同定され,合計で181例のうち7例においてSPI1融合遺伝子が検出された.イントロン領域を含めたキャプチャーによりすべての例においてゲノムの切断点を同定した.SPI1融合遺伝子をもつ症例はCD4陰性CD8陰性のダブルネガティブあるいはCD4陰性CD8陽性のシングルポジティブであり,いずれもCD2,CD3,CD5,CD7に陽性であった.SPI1融合遺伝子をもつ症例における特徴的な所見として,一般にはT細胞性急性リンパ性白血病において陽性になるとは知られていないHLA-DRがすべての例において陽性であった1).SPI1遺伝子の位置する領域における染色体の異常としては,STMN1-SPI1融合遺伝子をもつ1例にのみ染色体の転座が検出された.
2.SPI1融合タンパク質は野生型と同等の転写活性をもつ
SPI1遺伝子のコードするPU.1はETSファミリーに属する血球の分化にかかわる重要な転写因子であり,造血細胞の分化においてその発現は厳格に制御される.T細胞の分化において,コミットメントの前段階にかかわるフェーズ1遺伝子とよばれるLYL1遺伝子,LMO2遺伝子,GATA2遺伝子,MEIS1遺伝子,BCL11A遺伝子と同様に2),SPI1遺伝子は前胸腺前駆細胞や初期前駆T細胞において発現しており,DN2細胞からDN3細胞へ分化が進むと発現は低下し,フェーズ2遺伝子とよばれるTCF7遺伝子,NOTCH1遺伝子,RUNX1遺伝子,GATA3遺伝子,TCF3遺伝子,BCL11B遺伝子などの発現に置き換わり,そののち,フェーズ3遺伝子であるETS2遺伝子,LEF1遺伝子,ID3遺伝子などの発現が上昇する.SPI1融合遺伝子をもつ症例においては,いずれもSPI1遺伝子が高く発現していたが野生型のSPI1遺伝子の発現はほぼなく,これらの発現は5’側に融合したSTMN1遺伝子あるいはTCF7遺伝子のプロモーター活性により制御されていると考えられた.
融合した遺伝子によらずSPI1融合タンパク質はDNA結合領域であるETSドメインを保持していたが,C末端側の一部の領域は失っていた.SPI1融合タンパク質の転写因子としての活性について評価するため,PU.1との結合領域をもつCSF1R遺伝子のプロモーター配列を用いてルシフェラーゼアッセイを行った.その結果,STMN1-SPI1融合遺伝子あるいはTCF7-SPI1融合遺伝子を導入した細胞は野生型のSPI1遺伝子を導入した細胞と同等の活性を示したが,それぞれのETSドメインを欠失させると活性は完全に消失した.このことから,SPI1融合タンパク質はPU.1と同様に転写活性をもつことが示された.
in vitroの系においてSPI1融合遺伝子のT細胞の増殖への影響について検討した.マウスのダブルネガティブT細胞に野生型のSPI1遺伝子,STMN1-SPI1融合遺伝子,TCF7-SPI1融合遺伝子を導入したところ,SPI1融合遺伝子を導入した細胞において野生型のSPI1遺伝子を導入した細胞と同様の増殖が確認された.Tst4/DLL1共培養系を用いた分化の誘導の実験においては,野生型のSPI1遺伝子を導入した細胞と同様に,SPI1融合遺伝子を導入した細胞においてダブルポジティブ細胞,シングルポジティブ細胞,DN4細胞の減少,また,DN1細胞およびDN2細胞の増加が確認され,分化が停止していることが示された.また,in vitroの系において,マウスの造血幹細胞に野生型のSPI1遺伝子あるいはTCF7-SPI1融合遺伝子を導入したのちマウスへ移植したところ,ダブルネガティブ細胞の増加,CD4陽性CD8陰性のシングルポジティブ細胞およびCD4陰性CD8陽性のシングルポジティブ細胞の減少が確認された.
3.小児のT細胞性急性リンパ性白血病の症例の発現プロファイルによるクラスタリング
RNA-seq法によりトランスクリプトーム解析した121の症例にくわえ,べつのコホートにおいて同定され追加でトランスクリプトーム解析したTCF7-SPI1融合遺伝子をもつ2つの症例を含む123の症例について,発現プロファイルによりクラスタリングした.コンセンサスクラスタリングの手法を用い2段階でクラスタリングした(図1).まず,TAL1クラスターと非TAL1クラスターとに分類し,既報にある分類と同様に,TAL1クラスターはTAL1-RAクラスターとTAL1-RBクラスターの2つに,非TAL1クラスターは初期前駆T細胞と類似した発現プロファイルを示すETPクラスターとTLX1遺伝子あるいはTLX3遺伝子を高く発現するTLX関連クラスターに分類されたが3),ほかにSPI1融合遺伝子をもつ7つの症例のみで構成されるSPI1融合遺伝子クラスターも抽出され,あわせて5つにクラスタリングされた.SPI1融合遺伝子クラスターはDN3細胞あるいはDN4細胞に類似した転写因子の発現を呈しており,ほかのクラスターとは異なっていた.また,フェーズ1遺伝子の発現はSPI1融合遺伝子クラスターにおいては低下していたが,例外として,SPI1遺伝子とSPI1遺伝子のコードするPU.1により制御されるMEF2C遺伝子およびHHEX遺伝子は高く発現していた.そのほか,SPI1融合遺伝子クラスターにおいてはFLT3遺伝子およびKIT遺伝子も高く発現していたが,これらの遺伝子もPU.1により制御されているとする報告がある.TLX関連クラスター,TAL1-RAクラスター,TAL1-RBクラスターはダブルポジティブT細胞に相当すると考えられており,これらのクラスターにおいてみられたTLX1遺伝子,TLX3遺伝子,TAL1遺伝子,HOXA9遺伝子の高い発現は,SPI1融合遺伝子クラスターにおいてはまったくみられなかった.
4.クラスターとゲノムの異常との関連
おのおののクラスターとゲノムの異常との関連を調べるため,TARGETプロジェクトにより公開されている小児の急性リンパ性白血病におけるエキソーム解析の結果から抽出した158の遺伝子を標的となる領域として設計したカスタムベイトを用い,121の症例においてキャプチャーにより塩基配列を決定した.その結果,過去の報告と一致して,NOTCH1遺伝子およびCDKN2A遺伝子において高い頻度で異常が認められたが4-7),CDKN2A遺伝子の異常はETPクラスターにおいてはほかのクラスターと比べ頻度は有意に低かった.そのほか,10%以上の頻度で異常の認められた遺伝子として,PHF6遺伝子,FBXW7遺伝子,PTEN遺伝子,DNM2遺伝子,USP7遺伝子,NRAS遺伝子,JAK3遺伝子,WT1遺伝子,RUNX1遺伝子があり,USP7遺伝子以外は既知の異常として報告されていた.ETPクラスターはGATA3遺伝子,RUNX1遺伝子,NRAS遺伝子,ETV6遺伝子,さらに,エピジェネティック制御遺伝子の異常の頻度が高く,また,CDKN2A遺伝子の異常の頻度は低く,これは既報と一致する結果であった8,9).エピジェネティック制御遺伝子の異常の頻度はTLX関連クラスターにおいても高く,また,PHF6遺伝子およびWT1遺伝子の異常はTLX関連クラスターに特徴的なものと考えられた.TAL1-RAクラスターとTAL1-RBクラスターはPTEN遺伝子およびUSP7遺伝子の異常の頻度が高い点が共通していたが,TAL1-RBクラスターにおいて認められたNOTCH1遺伝子およびFBXW7遺伝子の異常,転写因子の異常,RAS経路の異常,エピジェネティック制御遺伝子の異常はTAL1-RAクラスターではほとんど認められなかった.USP7はPTENの脱ユビキチン化にかかわるが,USP7遺伝子の異常はT細胞性急性リンパ性白血病における新規の異常でありTAL1クラスターに集中して認められた.SPI1融合遺伝子クラスターは症例の数が少ないが,ほかのクラスターと同様に,NOTCH1遺伝子およびFBXW7遺伝子の異常,CDKN2A遺伝子の欠失の頻度が高く,そのほかの遺伝子の変異の頻度は低いが,RAS経路の異常の頻度が高かった.
5.小児のT細胞性急性リンパ性白血病におけるゲノムの異常と予後
SPI1融合遺伝子をもつ症例の予後について検討した.SPI1融合遺伝子をもたない症例と比較した場合,SPI1融合遺伝子をもつ症例は有意に生存期間が短く,生存期間の中央値は2.2年で,7例のうち6例は診断から3年以内に早期の再発により死亡していた.SPI1融合遺伝子をもつ症例をほかの融合遺伝子をもつ症例と比較した場合も,同様に生存期間は有意に短かった.多変量解析においてSPI1融合遺伝子は独立した予後因子として抽出され,SPI1融合遺伝子はアグレッシブな病型に関与することが示唆された.
おわりに
小児のT細胞性急性リンパ性白血病において,SPI1融合遺伝子をもつ症例は特徴的な発現プロファイルをもち,きわめて予後の不良な臨床経過を呈した.小児のT細胞性急性リンパ性白血病においてこれほど不良な臨床像を示すゲノムの異常はこれまで報告されていなかったが,症例の数は少なく再現性の確認は必要である.SPI1融合遺伝子をもつ症例は既存の治療法では救済されないと思われ,より強力な治療法や代替となる治療法が必要であり,これらの症例を抽出する意義は大きい.RAS経路の異常が多く,また,FLT3遺伝子およびKIT遺伝子に異常に高い発現がみられたことから,今後,新規の治療の標的としての可能性があるだろう.
文 献
- Babusikova, O., Stevulova, L. & Fajtova, M.: Immunophenotyping parameters as prognostic factors in T-acute leukemia patients. Neoplasma, 56, 508-513 (2009)[PubMed]
- Rothenberg, E. V., Moore, J. E. & Yui, M. A.: Launching the T-cell-lineage developmental programme. Nat. Rev. Immunol., 8, 9-21 (2008)[PubMed]
- Soulier, J., Clappier, E., Cayuela, J. M. et al.: HOXA genes are included in genetic and biologic networks defining human acute T-cell leukemia (T-ALL). Blood, 106, 274-286 (2005)[PubMed]
- Belver, L. & Ferrando, A.: The genetics and mechanisms of T cell acute lymphoblastic leukaemia. Nat. Rev. Cancer, 16, 494-507 (2016)[PubMed]
- Van Vlierberghe, P. & Ferrando, A.: The molecular basis of T cell acute lymphoblastic leukemia. J. Clin. Invest., 122, 3398-3406 (2012)[PubMed]
- Weng, A. P., Ferrando, A. A., Lee, W. et al.: Activating mutations of NOTCH1 in human T cell acute lymphoblastic leukemia. Science, 306, 269-271 (2004)[PubMed]
- Hebert, J., Cayuela, J. M., Berkeley, J. et al.: Candidate tumor-suppressor genes MTS1 (p16INK4A) and MTS2 (p15INK4B) display frequent homozygous deletions in primary cells from T- but not from B-cell lineage acute lymphoblastic leukemias. Blood, 84, 4038-4044 (1994)[PubMed]
- Zhang, J., Ding, L., Holmfeldt, L. et al.: The genetic basis of early T-cell precursor acute lymphoblastic leukaemia. Nature, 481, 157-163 (2012)[PubMed]
- Van Vlierberghe, P., Ambesi-Impiombato, A., Perez-Garcia, A. et al.: ETV6 mutations in early immature human T cell leukemias. J. Exp. Med., 208, 2571-2579 (2011)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2016年 東京大学大学院医学系研究科にて博士号取得,同年より東京大学医学部附属病院 助教.
研究テーマ:小児の血液腫瘍および固形腫瘍.
関心事:転写因子.
滝田 順子(Junko Takita)
東京大学大学院医学系研究科 准教授.
© 2017 関 正史・滝田順子 Licensed under CC 表示 2.1 日本