生細胞の超解像イメージングにより明らかにされたクロマチンドメインのダイナミックな構造
野崎 慎・前島一博
(国立遺伝学研究所 構造遺伝学研究センター生体高分子研究室)
email:野崎 慎,前島一博
DOI: 10.7875/first.author.2017.075
Dynamic organization of chromatin domains revealed by super-resolution live-cell imaging.
Tadasu Nozaki, Ryosuke Imai, Mai Tanbo, Ryosuke Nagashima, Sachiko Tamura, Tomomi Tani, Yasumasa Joti, Masaru Tomita, Kayo Hibino, Masato T. Kanemaki, Kerstin S. Wendt, Yasushi Okada, Takeharu Nagai, Kazuhiro Maeshima
Molecular Cell, 67, 282-293.e7 (2017)
ヒトのからだを構成する細胞のなかには全長2メートルにおよぶDNAが収められている.DNAはヒストンに巻きつくことによりヌクレオソームを形成し,さらに3次元的に折りたたまれることによりクロマチンを形成する.これまで長いあいだ,ヌクレオソームがらせん状に規則正しく折りたたまれてクロマチン線維ができ,これがさらにらせん状の構造を形成するという階層構造が定説とされてきた.しかし,2008年以降,筆者らは,生きた状態に近い細胞を観察することにより,規則正しく束ねられたクロマチン構造は存在せず,ヌクレオソームは細胞のなかに不規則に収納されることを明らかにしてきた.クロマチンは転写,複製,修復をはじめとする各種の生命反応にかかわるため,生きた細胞におけるクロマチンの実体のさらなる理解がもとめられている.この研究において,筆者らは,細胞のなかをタンパク質1分子のレベルで観察することのできる超解像顕微法を用いることにより,生細胞においてクロマチンを観察した.その結果,多数のヌクレオソームが集まって不規則に折りたたまれることにより“クロマチンドメイン”とよばれるコンパクトなかたまりが形成されることが明らかにされた.さらに,間期においては,このクロマチンドメインはヌクレオソームのあいだの相互作用やコヒーシンとよばれるクロマチンを束ねるリング状のタンパク質により維持されていた.クロマチンドメインはマウスのES細胞において分化をとおして変化した.また,細胞周期をつうじ分裂期の染色体においても観察されたことから,遺伝情報の検索,読み出し,維持に重要な染色体の機能単位としてはたらくことが示唆された.
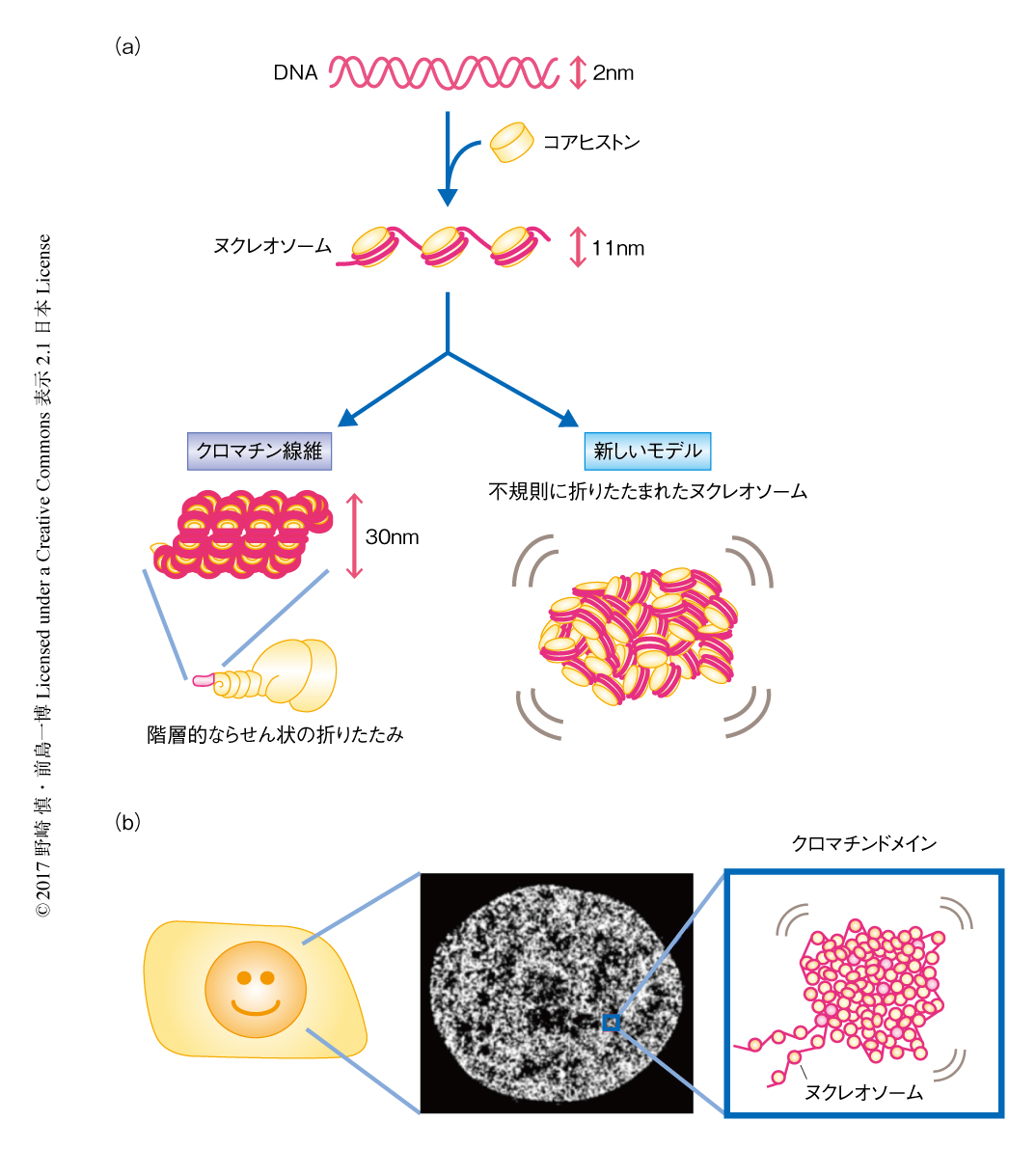
ヒトのからだは約40兆個の細胞からつくられている.それぞれの細胞には全長2メートルにもおよぶDNAがクロマチンを形成して収納されている.このDNAは直径2 nmのとても細い糸で,ヒストンという樽状のタンパク質に巻きつくことにより直径11 nmのヌクレオソームを形成する(図1a).いまから40年以上まえ,ヌクレオソームがらせん状に規則正しく折りたたまれることにより直径30 nmのクロマチン線維ができ,これがさらにらせん状に巻かれることにより階層構造が形成されるというモデルが提唱され,これまで,広く受け入れられてきた.しかしながら,2008年ごろより,筆者らは,クライオ電子顕微鏡やX線散乱法により,この定説のような規則正しいクロマチン線維は存在せず,ヌクレオソームは細胞のなかに不規則に収められていることを明らかにしてきた1,2)(図1a).そして,ヌクレオソームは不規則に折りたたまれることにより“クロマチンドメイン”とよばれるかたまりを形成するのではないかと推測してきた.また,最近では,DNAどうしの近接性を調べる3C(chromosome conformation capture)法やそれを応用したHi-C法により,TAD(topologically associating domain)とよばれる同様のクロマチンのかたまりの存在が示唆されている3).現在,生きた細胞のクロマチン,とりわけ,クロマチンドメインの実体についてさらなる理解がもとめられているが,その構造は数十nmから数百nmと非常に小さく,回折限界に起因する問題から従来の光学顕微鏡を用いて200 nm以下の構造を調べることは困難であった.
実際にクロマチンドメインを観察するためには,回折限界の壁をこえて,さらに,生きた細胞のクロマチンを観察する必要がある.そのため,生細胞におけるクロマチンの超解像イメージング法を確立することが必須であった.超解像イメージング法には複数の方法があるが,ヌクレオソームの1分子イメージングを軸にしたPALM(photoactivated localization microscopy)法を選択した4).これまで,PALM法によるクロマチンのイメージングは2つの問題から困難とされていた.1つ目は,DNAが折りたたまれている核や染色体は細胞がはりついているディッシュの底であるガラスの表面から高い位置にある点,2つ目は,微小な空間に多量のヌクレオソームが存在する点である.1つ目の問題は,従来の1分子イメージング法に用いられるガラスの表面を観察する全反射照明法に代えて,ガラスの表面から離れた領域を観察することのできる斜光照明法5) を用いることで解決した.2つ目の問題については,一般に光活性化の可能な蛍光タンパク質は波長405 nmのレーザーにより活性化するが,それに代えて,PA-mCherryに波長561 nmのレーザー光を直接に照射するとランダムかつ自発的に活性化し蛍光を発する性質を利用した.これらにより,ヌクレオソームが非常に多量で混雑している状態においても1分子レベルでの観察が可能になった.クロマチンの標識のためヒストンH2BとPA-mCherryの融合タンパク質を発現するHeLa細胞を作製し,超解像イメージングを実施した結果,生きたHeLa細胞の核の内部において約100個のヌクレオソームをフレームごとに撮影することに成功した.撮影した輝点が1段階の退色を示したことから,それぞれの輝点は1分子の蛍光タンパク質に起因することが確かめられた.おのおののフレームにおける輝点の重心を決定し,数万個の輝点を平面にマップすることにより,クロマチンの超解像画像を得ることができた(図1b).
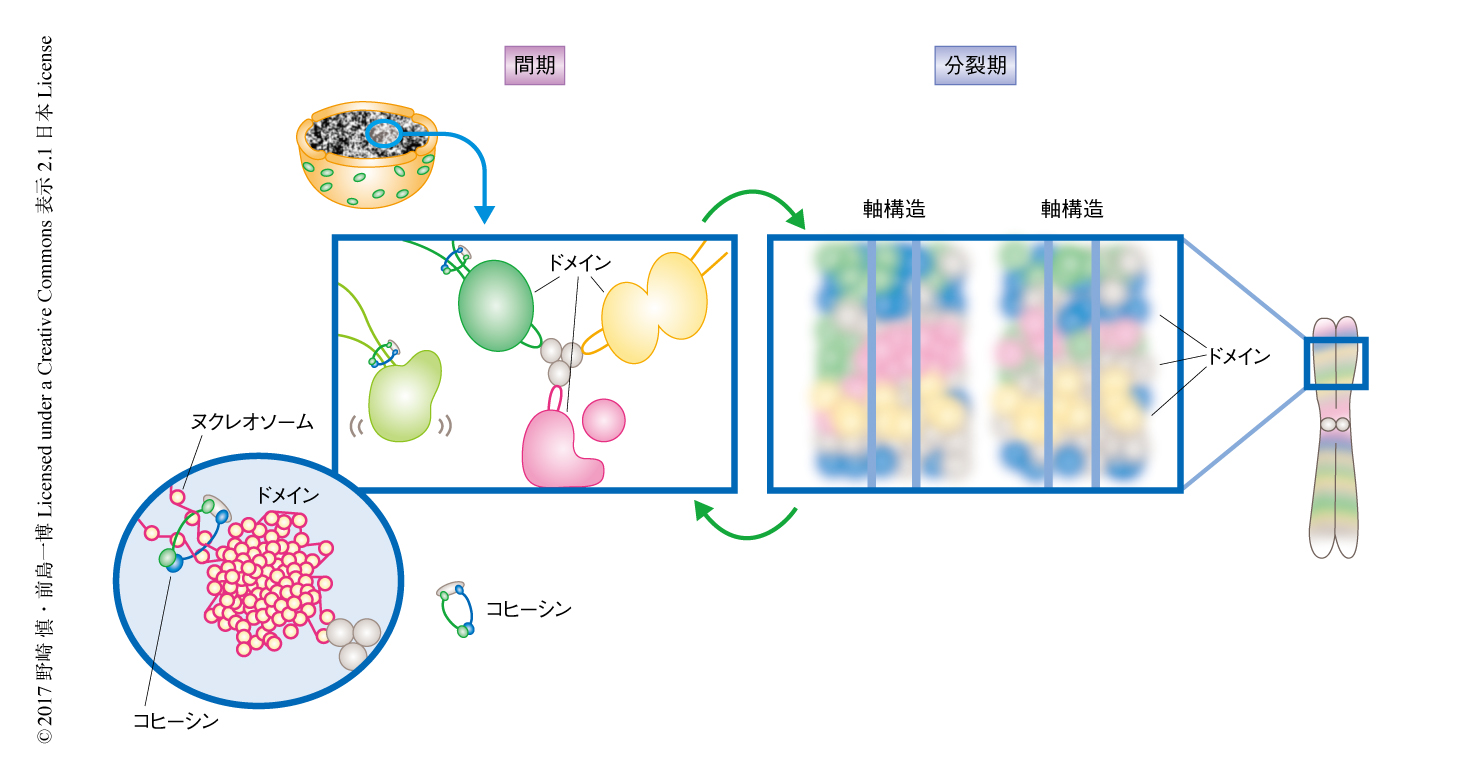
クロマチンの超解像イメージング法の確立により,一般的な光学系では観ることのできないクロマチンの非常に細やかな部分まで観察することができるようになった.クロマチンの超解像画像は,ヌクレオソームが不規則なかたまり,すなわち,クロマチンドメインを形成しているようにみえた(図1b).ヌクレオソームがどのようなかたまりを形成しているのか,その大きさや性質を定量的に調べるため,空間に配置された物体の分布について,かたまりの大きさおよびその度合いを評価することができるL関数とよばれる空間統計学の指標を導入した.その結果,クロマチンの超解像画像におけるヌクレオソームの分布はランダムではなくかたまりを形成しており,その大きさは直径160 nm程度であると推定された.これらのことから,生きた細胞の核において,ヌクレオソームが不規則に折りたたまれることにより形成されるクロマチンドメインが存在することが示された.そして,これらのクロマチンドメインは間期のあいだ維持されており,また,分裂期の染色体においても観察された(図2).このことから,クロマチンドメインは細胞周期をとおして維持され,レゴブロックのように染色体を形成するために非常に重要な構造としてはたらくことが示された.
これまで述べてきたPALM法によるイメージングと1分子ヌクレオソームイメージングによる動態解析を組み合わせ,ヌクレオソームの動きをフレームのあいだで追跡した結果,ヌクレオソームはとどまっているわけではなく,ゆらゆらとゆらいでおり,50 msのあいだに約60 nmも動いていることが示された(図1b).さらに,超解像画像を構築する際に,付近のヌクレオソームの動きを平均化して色分けすることによりクロマチンの動態に関するヒートマップを作成し,クロマチンのダイナミクスの局所性を評価した.その結果,クロマチンの動態は一様ではなく,核の内部においては大きな動きを示したが,核膜の付近や核小体の縁の付近などでは動きが抑えられていた.このことから,クロマチンの動態には局所性があり,とくに転写の活発な部分とそうではない部分とで動きに差のあることが示唆され,このような動態のばらつきはゲノムの機能と直結すると考えられた.
ここまで,超解像イメージング法によるクロマチンドメインの観察について記述してきたが,このクロマチンドメインの性質を構成するものは何だろうか.これを明らかにするため,クロマチンの状態を変化させるヒストン修飾およびクロマチン関連タンパク質に注目した.ヒストン修飾はエピジェネティックな制御の根幹を担い,クロマチンに大きく関与することが予想される.そこで,ヒストン脱アセチル化酵素の阻害剤であるトリコスタチンA 6) により処理したクロマチンに対し超解像イメージングを実施した.トリコスタチンAは核においてヒストンテールのアセチル化を大規模に促進し,このアセチル化はヌクレオソームのあいだの相互作用を弱めることが知られている.その結果,クロマチンは脱凝縮を起こし,それにともない動きも大きくなった.このことから,エピジェネティクな制御に強くかかわるヒストンのアセチル化によりクロマチンドメインの性質は大きく変化することが示された.
クロマチン構造にはクロマチン関連タンパク質が重要であることが過去の研究により示唆されている.そこで,クロマチン関連タンパク質の代表として,コヒーシン7),CTCF 7),コンデンシンII 8) の3つを選び,それらをノックダウンしたうえでクロマチンの超解像イメージングを実施した.コヒーシンのノックダウンによりクロマチンドメインは脱凝縮し,動きも大きくなった.コヒーシンをクロマチンにリクルートするNIPBLをノックダウンした場合にも同様の結果が得られたことから,コヒーシンはクロマチンドメインの形成に重要な役割をはたすと考えられた(図2).一方,CTCFあるいはコンデンシンIIをノックダウンしてもクロマチンドメインに変化はみられなかった.これらの結果から,ヒストンテールを介したヌクレオソームのあいだの相互作用,また,コヒーシンがクロマチンドメインの形成に重要であることが示された.
クロマチンドメインは核においてさまざまな性質をもち,また,ヒストンのアセチル化などにより変化したことから,転写や複製にかかわる機能をもつと考えられた.クロマチンドメインの機能について調べるため,マウスのES細胞において培養液からLIFを除去し,胚様体に分化する前後においてクロマチンドメインの性質を比較した.未分化マーカーであるSox2が強く発現している分化まえのES細胞においては,クロマチンドメインはすこし脱凝縮している状態で動きも大きかった.一方,胚様体に分化したのちの細胞においては,クロマチンドメインが明確になり動きも小さかった.このクロマチンドメインの変化はES細胞の分化にともなう発現の可能な遺伝子の限定に影響するのではないかと考えられた.もちろん,ここでのES細胞の分化は限定的なものであり,分化するさきも限定されていない.ES細胞をさまざまな誘導により分化させクロマチンを観察することにより,分化とクロマチンドメインとの関係性をさらに明らかにすることができると考えられる.
この研究において,ヌクレオソームが折りたたまれることにより形成されるクロマチンドメインが生細胞において存在することが示された.しかし,その機能についてはいまだ不明な部分が多い.分化によりその性質が変化することから,遺伝子発現の変化などに直接的な影響をおよぼすことが示唆される.筆者らは,生細胞におけるクロマチンの超解像イメージング法の強みはまだ完全には発揮されていないと考えている.このイメージング法はほかのタンパク質の1分子イメージングと同時に実施することができ,生きたクロマチンと生きたタンパク質との相互作用を直接的に観察することができる.転写因子,修復因子,複製因子とクロマチンを同時にイメージングすることにより,クロマチンドメインの機能を明らかにする研究がこれから進むと考えられる.そして,クロマチンを中心とした核におけるさまざまな生命反応の秘密が明らかにされていくと思われる.
筆者のひとり(野崎)は,以前,バイオインフォマティクスを専攻しており,DNAはATGCの集まりとしかとらえていなかった.しかし,細胞においてDNAは単なる1次元の文字列ではなく,その3次元的な構造やゆらぎにより新たな機能をもちうるとことを,生細胞におけるクロマチンのイメージングにより身をもって理解した.近年,Hi-C法の進歩によりクロマチンの直接的な観察を必要としないクロマチン構造の研究が増えているが,筆者らは,生きたクロマチンを観ることを大事にして研究を進めていきたい.
略歴:2016年 慶應義塾大学大学院政策・メディア研究科 修了,同年より国立遺伝学研究所 研究員.
研究テーマ:細胞におけるDNAのデータ構造およびその検索アルゴリズム.
関心事:細胞においてDNAの情報はどのように収納され検索されるのか.おいしいカレーうどんを作りたい.
前島 一博(Kazuhrio Maeshima)
国立遺伝学研究所 教授.
研究室URL:http://maeshima-lab.sakura.ne.jp/
? 2017 野崎 慎・前島一博 Licensed under CC 表示 2.1 日本
(国立遺伝学研究所 構造遺伝学研究センター生体高分子研究室)
email:野崎 慎,前島一博
DOI: 10.7875/first.author.2017.075
Dynamic organization of chromatin domains revealed by super-resolution live-cell imaging.
Tadasu Nozaki, Ryosuke Imai, Mai Tanbo, Ryosuke Nagashima, Sachiko Tamura, Tomomi Tani, Yasumasa Joti, Masaru Tomita, Kayo Hibino, Masato T. Kanemaki, Kerstin S. Wendt, Yasushi Okada, Takeharu Nagai, Kazuhiro Maeshima
Molecular Cell, 67, 282-293.e7 (2017)
この論文に出現する遺伝子・タンパク質のUniprot ID
要 約
ヒトのからだを構成する細胞のなかには全長2メートルにおよぶDNAが収められている.DNAはヒストンに巻きつくことによりヌクレオソームを形成し,さらに3次元的に折りたたまれることによりクロマチンを形成する.これまで長いあいだ,ヌクレオソームがらせん状に規則正しく折りたたまれてクロマチン線維ができ,これがさらにらせん状の構造を形成するという階層構造が定説とされてきた.しかし,2008年以降,筆者らは,生きた状態に近い細胞を観察することにより,規則正しく束ねられたクロマチン構造は存在せず,ヌクレオソームは細胞のなかに不規則に収納されることを明らかにしてきた.クロマチンは転写,複製,修復をはじめとする各種の生命反応にかかわるため,生きた細胞におけるクロマチンの実体のさらなる理解がもとめられている.この研究において,筆者らは,細胞のなかをタンパク質1分子のレベルで観察することのできる超解像顕微法を用いることにより,生細胞においてクロマチンを観察した.その結果,多数のヌクレオソームが集まって不規則に折りたたまれることにより“クロマチンドメイン”とよばれるコンパクトなかたまりが形成されることが明らかにされた.さらに,間期においては,このクロマチンドメインはヌクレオソームのあいだの相互作用やコヒーシンとよばれるクロマチンを束ねるリング状のタンパク質により維持されていた.クロマチンドメインはマウスのES細胞において分化をとおして変化した.また,細胞周期をつうじ分裂期の染色体においても観察されたことから,遺伝情報の検索,読み出し,維持に重要な染色体の機能単位としてはたらくことが示唆された.
はじめに
ヒトのからだは約40兆個の細胞からつくられている.それぞれの細胞には全長2メートルにもおよぶDNAがクロマチンを形成して収納されている.このDNAは直径2 nmのとても細い糸で,ヒストンという樽状のタンパク質に巻きつくことにより直径11 nmのヌクレオソームを形成する(図1a).いまから40年以上まえ,ヌクレオソームがらせん状に規則正しく折りたたまれることにより直径30 nmのクロマチン線維ができ,これがさらにらせん状に巻かれることにより階層構造が形成されるというモデルが提唱され,これまで,広く受け入れられてきた.しかしながら,2008年ごろより,筆者らは,クライオ電子顕微鏡やX線散乱法により,この定説のような規則正しいクロマチン線維は存在せず,ヌクレオソームは細胞のなかに不規則に収められていることを明らかにしてきた1,2)(図1a).そして,ヌクレオソームは不規則に折りたたまれることにより“クロマチンドメイン”とよばれるかたまりを形成するのではないかと推測してきた.また,最近では,DNAどうしの近接性を調べる3C(chromosome conformation capture)法やそれを応用したHi-C法により,TAD(topologically associating domain)とよばれる同様のクロマチンのかたまりの存在が示唆されている3).現在,生きた細胞のクロマチン,とりわけ,クロマチンドメインの実体についてさらなる理解がもとめられているが,その構造は数十nmから数百nmと非常に小さく,回折限界に起因する問題から従来の光学顕微鏡を用いて200 nm以下の構造を調べることは困難であった.
1.生細胞におけるクロマチンの超解像イメージング法の開発
実際にクロマチンドメインを観察するためには,回折限界の壁をこえて,さらに,生きた細胞のクロマチンを観察する必要がある.そのため,生細胞におけるクロマチンの超解像イメージング法を確立することが必須であった.超解像イメージング法には複数の方法があるが,ヌクレオソームの1分子イメージングを軸にしたPALM(photoactivated localization microscopy)法を選択した4).これまで,PALM法によるクロマチンのイメージングは2つの問題から困難とされていた.1つ目は,DNAが折りたたまれている核や染色体は細胞がはりついているディッシュの底であるガラスの表面から高い位置にある点,2つ目は,微小な空間に多量のヌクレオソームが存在する点である.1つ目の問題は,従来の1分子イメージング法に用いられるガラスの表面を観察する全反射照明法に代えて,ガラスの表面から離れた領域を観察することのできる斜光照明法5) を用いることで解決した.2つ目の問題については,一般に光活性化の可能な蛍光タンパク質は波長405 nmのレーザーにより活性化するが,それに代えて,PA-mCherryに波長561 nmのレーザー光を直接に照射するとランダムかつ自発的に活性化し蛍光を発する性質を利用した.これらにより,ヌクレオソームが非常に多量で混雑している状態においても1分子レベルでの観察が可能になった.クロマチンの標識のためヒストンH2BとPA-mCherryの融合タンパク質を発現するHeLa細胞を作製し,超解像イメージングを実施した結果,生きたHeLa細胞の核の内部において約100個のヌクレオソームをフレームごとに撮影することに成功した.撮影した輝点が1段階の退色を示したことから,それぞれの輝点は1分子の蛍光タンパク質に起因することが確かめられた.おのおののフレームにおける輝点の重心を決定し,数万個の輝点を平面にマップすることにより,クロマチンの超解像画像を得ることができた(図1b).
2.クロマチンドメインの観察および解析
クロマチンの超解像イメージング法の確立により,一般的な光学系では観ることのできないクロマチンの非常に細やかな部分まで観察することができるようになった.クロマチンの超解像画像は,ヌクレオソームが不規則なかたまり,すなわち,クロマチンドメインを形成しているようにみえた(図1b).ヌクレオソームがどのようなかたまりを形成しているのか,その大きさや性質を定量的に調べるため,空間に配置された物体の分布について,かたまりの大きさおよびその度合いを評価することができるL関数とよばれる空間統計学の指標を導入した.その結果,クロマチンの超解像画像におけるヌクレオソームの分布はランダムではなくかたまりを形成しており,その大きさは直径160 nm程度であると推定された.これらのことから,生きた細胞の核において,ヌクレオソームが不規則に折りたたまれることにより形成されるクロマチンドメインが存在することが示された.そして,これらのクロマチンドメインは間期のあいだ維持されており,また,分裂期の染色体においても観察された(図2).このことから,クロマチンドメインは細胞周期をとおして維持され,レゴブロックのように染色体を形成するために非常に重要な構造としてはたらくことが示された.
これまで述べてきたPALM法によるイメージングと1分子ヌクレオソームイメージングによる動態解析を組み合わせ,ヌクレオソームの動きをフレームのあいだで追跡した結果,ヌクレオソームはとどまっているわけではなく,ゆらゆらとゆらいでおり,50 msのあいだに約60 nmも動いていることが示された(図1b).さらに,超解像画像を構築する際に,付近のヌクレオソームの動きを平均化して色分けすることによりクロマチンの動態に関するヒートマップを作成し,クロマチンのダイナミクスの局所性を評価した.その結果,クロマチンの動態は一様ではなく,核の内部においては大きな動きを示したが,核膜の付近や核小体の縁の付近などでは動きが抑えられていた.このことから,クロマチンの動態には局所性があり,とくに転写の活発な部分とそうではない部分とで動きに差のあることが示唆され,このような動態のばらつきはゲノムの機能と直結すると考えられた.
3.クロマチンドメインの形成におけるヌクレオソームのあいだの相互作用およびコヒーシンの関与
ここまで,超解像イメージング法によるクロマチンドメインの観察について記述してきたが,このクロマチンドメインの性質を構成するものは何だろうか.これを明らかにするため,クロマチンの状態を変化させるヒストン修飾およびクロマチン関連タンパク質に注目した.ヒストン修飾はエピジェネティックな制御の根幹を担い,クロマチンに大きく関与することが予想される.そこで,ヒストン脱アセチル化酵素の阻害剤であるトリコスタチンA 6) により処理したクロマチンに対し超解像イメージングを実施した.トリコスタチンAは核においてヒストンテールのアセチル化を大規模に促進し,このアセチル化はヌクレオソームのあいだの相互作用を弱めることが知られている.その結果,クロマチンは脱凝縮を起こし,それにともない動きも大きくなった.このことから,エピジェネティクな制御に強くかかわるヒストンのアセチル化によりクロマチンドメインの性質は大きく変化することが示された.
クロマチン構造にはクロマチン関連タンパク質が重要であることが過去の研究により示唆されている.そこで,クロマチン関連タンパク質の代表として,コヒーシン7),CTCF 7),コンデンシンII 8) の3つを選び,それらをノックダウンしたうえでクロマチンの超解像イメージングを実施した.コヒーシンのノックダウンによりクロマチンドメインは脱凝縮し,動きも大きくなった.コヒーシンをクロマチンにリクルートするNIPBLをノックダウンした場合にも同様の結果が得られたことから,コヒーシンはクロマチンドメインの形成に重要な役割をはたすと考えられた(図2).一方,CTCFあるいはコンデンシンIIをノックダウンしてもクロマチンドメインに変化はみられなかった.これらの結果から,ヒストンテールを介したヌクレオソームのあいだの相互作用,また,コヒーシンがクロマチンドメインの形成に重要であることが示された.
4.分化によりクロマチンドメインは変化する
クロマチンドメインは核においてさまざまな性質をもち,また,ヒストンのアセチル化などにより変化したことから,転写や複製にかかわる機能をもつと考えられた.クロマチンドメインの機能について調べるため,マウスのES細胞において培養液からLIFを除去し,胚様体に分化する前後においてクロマチンドメインの性質を比較した.未分化マーカーであるSox2が強く発現している分化まえのES細胞においては,クロマチンドメインはすこし脱凝縮している状態で動きも大きかった.一方,胚様体に分化したのちの細胞においては,クロマチンドメインが明確になり動きも小さかった.このクロマチンドメインの変化はES細胞の分化にともなう発現の可能な遺伝子の限定に影響するのではないかと考えられた.もちろん,ここでのES細胞の分化は限定的なものであり,分化するさきも限定されていない.ES細胞をさまざまな誘導により分化させクロマチンを観察することにより,分化とクロマチンドメインとの関係性をさらに明らかにすることができると考えられる.
おわりに
この研究において,ヌクレオソームが折りたたまれることにより形成されるクロマチンドメインが生細胞において存在することが示された.しかし,その機能についてはいまだ不明な部分が多い.分化によりその性質が変化することから,遺伝子発現の変化などに直接的な影響をおよぼすことが示唆される.筆者らは,生細胞におけるクロマチンの超解像イメージング法の強みはまだ完全には発揮されていないと考えている.このイメージング法はほかのタンパク質の1分子イメージングと同時に実施することができ,生きたクロマチンと生きたタンパク質との相互作用を直接的に観察することができる.転写因子,修復因子,複製因子とクロマチンを同時にイメージングすることにより,クロマチンドメインの機能を明らかにする研究がこれから進むと考えられる.そして,クロマチンを中心とした核におけるさまざまな生命反応の秘密が明らかにされていくと思われる.
筆者のひとり(野崎)は,以前,バイオインフォマティクスを専攻しており,DNAはATGCの集まりとしかとらえていなかった.しかし,細胞においてDNAは単なる1次元の文字列ではなく,その3次元的な構造やゆらぎにより新たな機能をもちうるとことを,生細胞におけるクロマチンのイメージングにより身をもって理解した.近年,Hi-C法の進歩によりクロマチンの直接的な観察を必要としないクロマチン構造の研究が増えているが,筆者らは,生きたクロマチンを観ることを大事にして研究を進めていきたい.
文 献
- Maeshima, K., Ide, S., Hibino, K. et al.: Liquid-like behavior of chromatin. Curr. Opin. Genet. Dev., 37, 36-45 (2016)[PubMed]
- Maeshima, K., Imai, R., Tamura, S. et al.: Chromatin as dynamic 10-nm fibers. Chromosoma, 123, 225-237 (2014)[PubMed]
- Dekker, J. & Heard, E.: Structural and functional diversity of topologically associating domains. FEBS Lett., 589, 2877-2884 (2015)[PubMed]
- Betzig, E., Patterson, G. H., Sougrat, R. et al.: Imaging intracellular fluorescent proteins at nanometer resolution. Science, 313, 1642-1645 (2006)[PubMed]
- Tokunaga, M., Imamoto, N. & Sakata-Sogawa, K.: Highly inclined thin illumination enables clear single-molecule imaging in cells. Nat. Methods, 5, 159-161 (2008)[PubMed]
- Yoshida, M., Kijima, M., Akita, M. et al.: Potent and specific inhibition of mammalian histone deacetylase both in vivo and in vitro by trichostatin A. J. Biol. Chem., 265, 17174-17179 (1990)[PubMed]
- Wendt, K. S., Yoshida, K., Itoh, T. et al.: Cohesin mediates transcriptional insulation by CCCTC-binding factor. Nature, 451, 796-801 (2008)[PubMed]
- Ono, T., Yamashita, D. & Hirano, T.: Condensin II initiates sister chromatid resolution during S phase. J. Cell Biol., 200, 429-441 (2013)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2016年 慶應義塾大学大学院政策・メディア研究科 修了,同年より国立遺伝学研究所 研究員.
研究テーマ:細胞におけるDNAのデータ構造およびその検索アルゴリズム.
関心事:細胞においてDNAの情報はどのように収納され検索されるのか.おいしいカレーうどんを作りたい.
前島 一博(Kazuhrio Maeshima)
国立遺伝学研究所 教授.
研究室URL:http://maeshima-lab.sakura.ne.jp/
? 2017 野崎 慎・前島一博 Licensed under CC 表示 2.1 日本