転写因子T-betはTh1細胞においてI型インターフェロン応答を抑制する
菅野由香・岩田 慈・三上洋平
(米国NIH National Institute of Arthritis and Musculoskeletal and Skin Diseases,Molecular Immunology and Inflammation Branch)
email:菅野由香,岩田 慈,三上洋平
DOI: 10.7875/first.author.2017.061
The transcription factor T-bet limits amplification of type I IFN transcriptome and circuitry in T helper 1 cells.
Shigeru Iwata, Yohei Mikami, Hong-Wei Sun, Stephen R. Brooks, Dragana Jankovic, Kiyoshi Hirahara, Atsushi Onodera, Han-Yu Shih, Takeshi Kawabe, Kan Jiang, Toshinori Nakayama, Alan Sher, John J. O’Shea, Fred P. Davis, Yuka Kanno
Immunity, 46, 983-991.e4 (2017)
宿主の防御においては,インターフェロンγを産生するTh1細胞を含むCD4陽性ヘルパーT細胞への運命が適切に決定されることが必要である.インターフェロンγは抗病原性および抗腫瘍性の効果をもつインターフェロンファミリーのひとつとしてII型インターフェロンに分類され,Th1細胞を規定する転写因子であるT-betの発現を誘導し,インターフェロンγの産生をフィードフォワード制御により維持する.筆者らは,この研究において,Th1細胞がオートクラインしたインターフェロンを認識するうえでT-betのはたす新規の役割について明らかにした.T-betの非存在下ではインターフェロンγは異常なI型インターフェロン応答をひき起こした.通常,T-betはI型インターフェロンのオートクラインにより活性化される遺伝子および経路を優先的に抑制し,I型インターフェロンシグナル伝達系の異常な増幅を抑制する.したがって,T-betはTh1細胞への分化を積極的に誘導するだけでなく,Th1細胞においてI型インターフェロンの異常なオートクラインおよびその下流のシグナル伝達系を抑制することが明らかにされた.
多様な病原微生物に対する適切な応答において,CD4陽性ヘルパーT細胞が異なる感染症に適切に応答するサイトカインを選択的に産生する能力を獲得することが必要である1).CD4陽性ヘルパーT細胞のひとつであるTh1細胞およびTh1細胞の産生するII型インターフェロンであるインターフェロンγは,病原体,とくに細胞に侵入した細菌および寄生虫に対する生体防御機構に必須である1).対照的に,インターフェロンαおよびインターフェロンβを含むI型インターフェロンは多くの種類の細胞により産生され,とくにウイルスに対する生体防御機構にかかわるインターフェロン誘導遺伝子(interferon-stimulated gene:ISG)の発現を誘導する2-4).I型インターフェロンおよびII型インターフェロンの作用は感染症ごとにかなり異なると考えられている3).
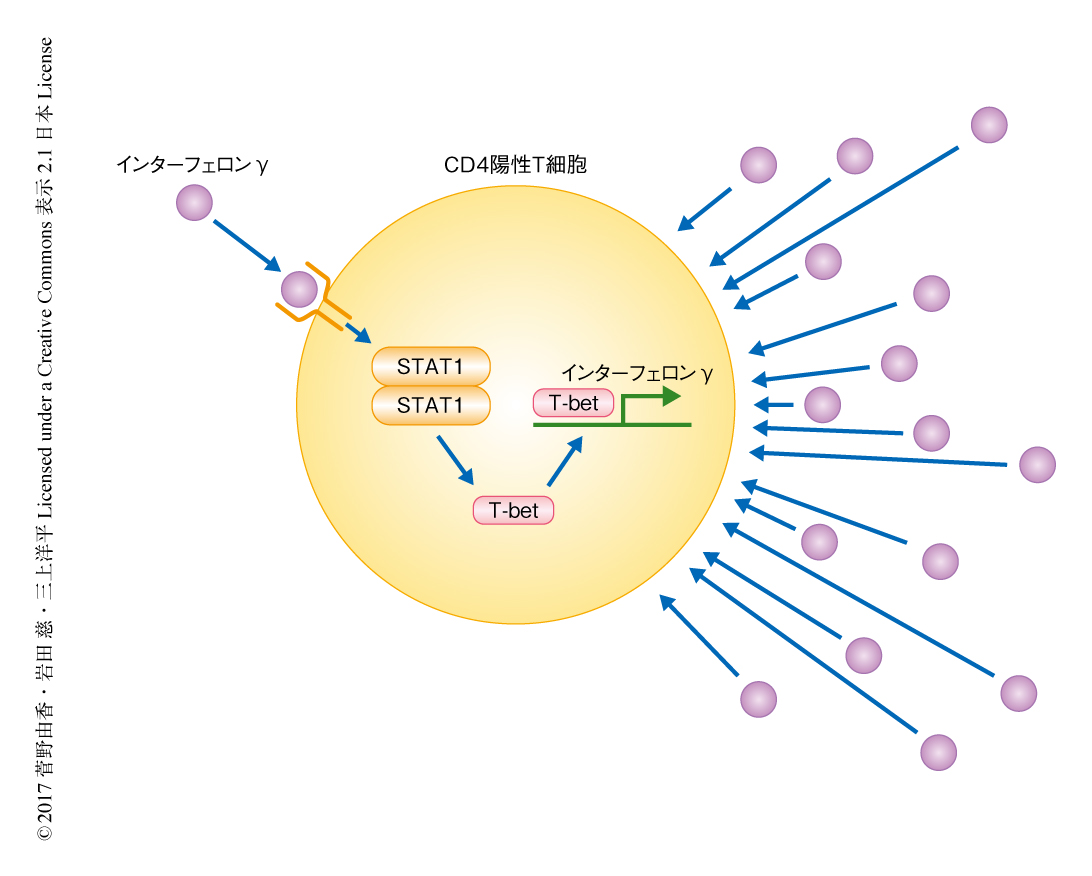
T細胞の分化における運命はシグナル依存性転写因子(signal-dependent transcription factor:SDTF)および系統決定的転写因子(lineage-determining transcription factor:LDTF)により決定される5)(図1).Th1細胞においてTbx21遺伝子にコードされる転写因子T-betは系統決定的転写因子としてインターフェロンγの産生を直接的に正にフィードフォワード制御する6).Th1細胞がサイトカインを産生し細胞の環境への適応能および増殖能を維持するためには,それらがつくりだす局所の環境,すなわち,高レベルのインターフェロンγに適応する必要がある7).インターフェロンγにより直接に活性化されるSTAT1のようなシグナル依存性転写因子とは対照的に,系統決定的転写因子としてのT-betの役割は,STAT1の標的であることのほか,インターフェロンγシグナル伝達系への関与については完全には説明されていない.この研究においては,RNA-seq法やChIP-seq法などを用いてTh1細胞の分化の促進におけるT-betの未知の作用について検討した.
CD4陽性T細胞におけるT-betに起因する包括的な転写の変化について検討するため,II型インターフェロンであるインターフェロンγにより処理した野生型のCD4陽性T細胞およびT-betを欠損したCD4陽性T細胞における遺伝子の発現をRNA-seq法を用いて比較した.その結果,T-betの有無により529個の遺伝子の発現が変化し,そのうち約半数は亢進し約半数は抑制された.対照的に,I型インターフェロンであるインターフェロンβに対する応答においてT-betの関与は軽微であった.インターフェロンγを処理したときにT-betの欠損の影響をうけた遺伝子座について,T-betにより活性化した遺伝子座の42%およびT-betにより抑制された遺伝子座の35%にT-betの結合することがChIP-seq法により明らかにされた.T-betにより制御され,かつ,遺伝子座に直接に結合する代表的な遺伝子として,活性化される遺伝子としてIfng遺伝子,抑制される遺伝子としてIsg15遺伝子があげられるが,Isg15遺伝子にくわえ,T-betにより抑制される遺伝子としてI型インターフェロンにより発現の誘導される遺伝子の発現が認められた.さらに,T-betにより抑制された遺伝子の遺伝子オントロジー解析により,I型インターフェロン応答および抗ウイルス性応答が有意に認められた.対照的に,T-betにより活性化される遺伝子の遺伝子オントロジー解析により解糖系の酵素を含む代謝遺伝子が認められた.インターフェロンγにより刺激したT-bet欠損CD4陽性T細胞においてI型インターフェロンにより発現の誘導される遺伝子の発現が亢進していたことから,T-betはそれらの転写を抑制することが示された.
インターフェロンβあるいはインターフェロンγにより制御される転写産物の転写への経時的な効果を比較したところCD4陽性T細胞においては転写制御の結果が大きく異なり,インターフェロンβおよびインターフェロンγの両方により制御される遺伝子は非常に少なかった.さらに,インターフェロンβは急速にインターフェロン誘導遺伝子の発現を誘導したが,インターフェロンγのおもな影響は晩期における代謝関連遺伝子の発現の誘導であった.インターフェロンγあるいはインターフェロンβの効果に対するT-betの影響について検討したところ,早期におけるインターフェロンβによる優先的なインターフェロン誘導遺伝子の発現の誘導はT-betとはほとんど無関係であった.しかし,後期においてT-betを欠損したCD4陽性T細胞においてはインターフェロンβおよびインターフェロンγはともにインターフェロン誘導遺伝子の発現を誘導した.このことから,ヘルパーT細胞はインターフェロンβシグナルおよびインターフェロンγシグナルに対しT-betに依存性に適切な応答をすることが示唆された.つまり,T-betの非存在下においてはインターフェロンγシグナルの特異性が維持されず,代わりに,インターフェロンβシグナルに近似することが示唆された.
T-betを欠損したCD4陽性T細胞においてみられた異常なインターフェロン応答の根底にある分子機構を明らかにするため,T-betがインターフェロンシグナル伝達系を変化させるかどうか検討した.I型インターフェロンはインターフェロン刺激応答配列(interferon-stimulated response element:ISRE)と結合するSTAT1,STAT2,IRF9からなるヘテロ三量体を活性化する8).野生型のCD4陽性T細胞において,インターフェロンβはSTAT2の発現およびリン酸化を誘導したが,インターフェロンγにはこのような効果は認められなかった.しかし,T-betを欠損したCD4陽性T細胞においてはインターフェロンγおよびインターフェロンβの両方がSTAT2の発現およびリン酸化を誘導した.これと一致して,T-betを欠損したCD4陽性T細胞のゲノムの全体におけるSTAT2の結合も野生型のCD4陽性T細胞と比較して増加した.さらに,T-betを欠損したCD4陽性T細胞においてインターフェロンγおよびインターフェロンβによりインターフェロン誘導遺伝子の近傍へのSTAT2の局在的な結合が増強され,活性化したエンハンサーの指標であるヒストンH3のLys27のアセチル化の増強と関連した.これらの結果から,T-betを欠損したCD4陽性T細胞においてはインターフェロンγがSTAT2を異常に活性化し,活性化されたSTAT2はインターフェロン誘導遺伝子の遺伝子座の近傍に結合し,その周辺のエンハンサーを活性化することが示された.T-betとSTAT2の結合部位の重複を比較することによりT-betがSTAT2の結合と直接的に干渉するかどうかを検討したところ,T-betの結合部位の24%がSTAT2の結合部位と重複していたことから,T-betとSTAT2が遺伝子座に競合的に結合する可能性が示唆された.
IRF7はI型インターフェロンにより発現が誘導され,I型インターフェロンの発現を制御する9).T-betを欠損したCD4陽性T細胞においてインターフェロンγに応答してIRF7のタンパク質レベルでの発現も異常に上昇した.T-betが存在しない場合,STAT2,IRF7,STAT1 2) のようなI型インターフェロンシグナル伝達系の重要な転写因子の,通常はT-betにより抑制される遺伝子座の近傍への結合が増強される可能性について考慮した.その結果,T-betはT-betにより抑制される遺伝子座に比べT-betにより活性化される遺伝子座への結合が多く認められた一方,STAT2,IRF7,STAT1は通常はT-betにより抑制される遺伝子座の周囲により多く結合した.さらに,さまざまな条件におけるそのほかの転写因子の結合を筆者らによるデータおよびこれまでに公開されたデータから検討したところ,T-betにより抑制される遺伝子座へのSTAT2,IRF7,STAT1の優先的な結合が,インターフェロンγによる刺激のもとSTAT2,IRF7,STAT1に選択的に認められた.T-betにより抑制される遺伝子座へのSTAT1の有意な優先的な結合はインターフェロンγを処理したT-betを欠損したCD4陽性T細胞においては観察されたがインターフェロンγを処理した野生型のCD4陽性T細胞においては観察されず,T-betの非存在下においてSTAT1の分布がゲノムワイドに変化することが示唆された.したがって,T-betの非存在下においては通常はT-betにより抑制される遺伝子座が,インターフェロンγの刺激に応答してSTAT2,IRF7,STAT1の優先的な標的になることが示唆された.
I型インターフェロンシグナル伝達系はインターフェロンのオートクラインを組み込んだフィードフォワードループから構成される9).したがって,I型インターフェロン応答を促進するSTAT2,IRF7,STAT1の増強はI型インターフェロンの産生の増強をひき起こすことが予想された.事実,インターフェロンγを処理したT-betを欠損したCD4陽性細胞において異常に高いレベルのI型インターフェロンが検出された.インターフェロンγを処理したT-betを欠損したCD4陽性T細胞におけるインターフェロンにより発現の誘導される遺伝子の発現から,このI型インターフェロンのオートクラインに起因するI型インターフェロンシグナル伝達系のフィードフォワードループの存在が示唆された.この可能性を確認するため,T-betを欠損したCD4陽性T細胞に抗インターフェロンα/β受容体抗体をインターフェロンγとともに添加したところ,用量に依存性にインターフェロンγの正常な発現が回復し,異常に亢進したインターフェロン誘導遺伝子の発現が減弱した.さらに,抗インターフェロンα/β受容体抗体はT-betを欠損したCD4陽性T細胞においてインターフェロンγにより異常に誘導されたSTAT2の活性を減弱させた.また,抗インターフェロンα/β受容体抗体はI型インターフェロンおよびIRF7のmRNAレベルでの異常な発現を改善したが,IRF3には影響をおよぼさなかった.したがって,インターフェロンγが豊富に存在する環境において,T-betはI型インターフェロンシグナル伝達系のフィードフォワードループの増幅を抑制するのに重要であることが明らかにされた(図2).T-betが存在しない場合,CD4陽性T細胞はインターフェロンγをI型インターフェロンであると誤認し,I型インターフェロン応答に近似する転写制御を開始する.しかし,インターフェロン誘導遺伝子座の26%はT-betと直接に結合しているため,インターフェロン誘導遺伝子座のうちのいくつかはT-betにより直接的に抑制される可能性がある.
in vitroの系において明らかにされた,T-betがインターフェロンγの下流に存在するI型インターフェロン伝達系のフィードフォワードループを抑制するという作用を,インターフェロンγの発現を誘導することが知られている寄生虫Toxoplasma gondiiの感染モデルを用いて検討した10).T-betノックアウトマウスは適切なTh1応答をすることができずT. gondiiの感染に耐えられないため11),野生型のCD4陽性T細胞およびT-betを欠損したCD4陽性T細胞の両方をRag2ノックアウトマウスに移入したのちT. gondiiを感染させた.このマウスはT. gondiiの感染に耐え,野生型CD4陽性T細胞およびT-betを欠損したCD4陽性T細胞は炎症環境にさらされる.その結果,T. gondiiに感染したマウスの腹腔浸出液から得た細胞において,T-betを欠損したCD4陽性T細胞は野生型のCD4陽性T細胞と比べSTAT1,STAT2,IRF7,IRF9の発現の顕著な上昇が認められ,in vitroの系において認められたようなI型インターフェロンシグナル伝達系のフィードフォワードループの増幅の起こる可能性が示唆された.T-betを欠損したCD4陽性T細胞におけるI型インターフェロンシグナル伝達系のフィードフォワードループの亢進は細胞に特有の現象である.対照的に,おもにI型インターフェロン応答がひき起こされるリンパ球性脈絡髄膜炎ウイルスの感染においては,STAT1,STAT2,IRF7,IRF9の発現に対しT-betの欠損は大きな影響を示さなかった.以上のことから,T-betはTh1細胞の分化において異常なI型インターフェロンシグナル伝達系のフィードフォワードループを抑制することが示された.
一般に,T-betのような系統決定的転写因子はヘルパーT細胞のサブセットの固定を促進しほかのサブセットへの脱分化を制限することが知られてきた1).この研究においては,系統決定的転写因子のそのほかの機能,つまり,細胞から分泌されたタンパク質の検知に系統決定的転写因子が影響をおよぼすことが示された.すなわち,T-betの非存在下においては,CD4陽性T細胞はI型インターフェロンとII型インターフェロンを異なる刺激として認識できなくなる.同様の機能をもつ系統決定的転写因子がどのくらい存在するのかは今後の検討課題のひとつである.さらに,多くの系統決定的転写因子は転写のアクティベーターまたはリプレッサーとよばれているが,ChIP-seq法により得られたデータから,T-betは約半分の遺伝子に対しアクティベーターとして,約半分の遺伝子に対しリプレッサーとして機能することが示された.T-betがある遺伝子座の近傍に結合した際に,その遺伝子の発現を促進するか抑制するかについては議論の余地があるが,おそらく,同じ遺伝子座と結合するほかの転写因子12,13),および,クロマチンのコンフォメーションあるいはループ14,15) により決定されると推測される.それでは,T-betは遺伝子のどこに結合するのか,どの転写因子とパートナーになるのかをどのように知るのだろうか? おそらく,この情報はゲノムにコードされていると推定されるが,遺伝子座に特異的な転写因子の結合の組合せが遺伝子にどのように設計されているかは今後の研究課題である.
米国NIH National Institute of Arthritis and Musculoskeletal and Skin Diseases常任研究員.
岩田 慈(Shigeru Iwata)
略歴:2011年 産業医科大学大学院医学研究科にて博士号取得,2013年 米国NIH National Institute of Arthritis and Musculoskeletal and Skin Diseases博士研究員を経て,2015年より産業医科大学.
三上 洋平(Yohei Mikami)
略歴:2012年 慶應義塾大学大学院医学研究科博士課程 修了,2013年より米国NIH National Institute of Arthritis and Musculoskeletal and Skin Diseases博士研究員.
研究テーマ:腸管粘膜免疫におけるエピゲノムの制御機構の解明および新規の治療法の開発.
© 2017 菅野由香・岩田 慈・三上洋平 Licensed under CC 表示 2.1 日本
(米国NIH National Institute of Arthritis and Musculoskeletal and Skin Diseases,Molecular Immunology and Inflammation Branch)
email:菅野由香,岩田 慈,三上洋平
DOI: 10.7875/first.author.2017.061
The transcription factor T-bet limits amplification of type I IFN transcriptome and circuitry in T helper 1 cells.
Shigeru Iwata, Yohei Mikami, Hong-Wei Sun, Stephen R. Brooks, Dragana Jankovic, Kiyoshi Hirahara, Atsushi Onodera, Han-Yu Shih, Takeshi Kawabe, Kan Jiang, Toshinori Nakayama, Alan Sher, John J. O’Shea, Fred P. Davis, Yuka Kanno
Immunity, 46, 983-991.e4 (2017)
この論文に出現する遺伝子・タンパク質のUniprot ID
T-bet(Q9JKD8), I型インターフェロン, type I IFN, インターフェロンγ(P01580), CD4(P06332), インターフェロン, II型インターフェロン, サイトカイン, インターフェロンα, インターフェロンβ(P01575), インターフェロン誘導遺伝子, interferon-stimulated gene, ISG, Tbx21(Q9JKD8), STAT1(P42225), Ifng(P01580), Isg15(Q64339), STAT2(Q9WVL2), IRF9(Q61179), ヒストンH3, IRF7(P70434), インターフェロンα/β受容体, IRF3(P70671), Rag2(P21784), クロマチン
要 約
宿主の防御においては,インターフェロンγを産生するTh1細胞を含むCD4陽性ヘルパーT細胞への運命が適切に決定されることが必要である.インターフェロンγは抗病原性および抗腫瘍性の効果をもつインターフェロンファミリーのひとつとしてII型インターフェロンに分類され,Th1細胞を規定する転写因子であるT-betの発現を誘導し,インターフェロンγの産生をフィードフォワード制御により維持する.筆者らは,この研究において,Th1細胞がオートクラインしたインターフェロンを認識するうえでT-betのはたす新規の役割について明らかにした.T-betの非存在下ではインターフェロンγは異常なI型インターフェロン応答をひき起こした.通常,T-betはI型インターフェロンのオートクラインにより活性化される遺伝子および経路を優先的に抑制し,I型インターフェロンシグナル伝達系の異常な増幅を抑制する.したがって,T-betはTh1細胞への分化を積極的に誘導するだけでなく,Th1細胞においてI型インターフェロンの異常なオートクラインおよびその下流のシグナル伝達系を抑制することが明らかにされた.
はじめに
多様な病原微生物に対する適切な応答において,CD4陽性ヘルパーT細胞が異なる感染症に適切に応答するサイトカインを選択的に産生する能力を獲得することが必要である1).CD4陽性ヘルパーT細胞のひとつであるTh1細胞およびTh1細胞の産生するII型インターフェロンであるインターフェロンγは,病原体,とくに細胞に侵入した細菌および寄生虫に対する生体防御機構に必須である1).対照的に,インターフェロンαおよびインターフェロンβを含むI型インターフェロンは多くの種類の細胞により産生され,とくにウイルスに対する生体防御機構にかかわるインターフェロン誘導遺伝子(interferon-stimulated gene:ISG)の発現を誘導する2-4).I型インターフェロンおよびII型インターフェロンの作用は感染症ごとにかなり異なると考えられている3).
T細胞の分化における運命はシグナル依存性転写因子(signal-dependent transcription factor:SDTF)および系統決定的転写因子(lineage-determining transcription factor:LDTF)により決定される5)(図1).Th1細胞においてTbx21遺伝子にコードされる転写因子T-betは系統決定的転写因子としてインターフェロンγの産生を直接的に正にフィードフォワード制御する6).Th1細胞がサイトカインを産生し細胞の環境への適応能および増殖能を維持するためには,それらがつくりだす局所の環境,すなわち,高レベルのインターフェロンγに適応する必要がある7).インターフェロンγにより直接に活性化されるSTAT1のようなシグナル依存性転写因子とは対照的に,系統決定的転写因子としてのT-betの役割は,STAT1の標的であることのほか,インターフェロンγシグナル伝達系への関与については完全には説明されていない.この研究においては,RNA-seq法やChIP-seq法などを用いてTh1細胞の分化の促進におけるT-betの未知の作用について検討した.
1.T-betはインターフェロンγを処理したCD4陽性T細胞においてI型インターフェロンにより発現の誘導される遺伝子の発現を抑制する
CD4陽性T細胞におけるT-betに起因する包括的な転写の変化について検討するため,II型インターフェロンであるインターフェロンγにより処理した野生型のCD4陽性T細胞およびT-betを欠損したCD4陽性T細胞における遺伝子の発現をRNA-seq法を用いて比較した.その結果,T-betの有無により529個の遺伝子の発現が変化し,そのうち約半数は亢進し約半数は抑制された.対照的に,I型インターフェロンであるインターフェロンβに対する応答においてT-betの関与は軽微であった.インターフェロンγを処理したときにT-betの欠損の影響をうけた遺伝子座について,T-betにより活性化した遺伝子座の42%およびT-betにより抑制された遺伝子座の35%にT-betの結合することがChIP-seq法により明らかにされた.T-betにより制御され,かつ,遺伝子座に直接に結合する代表的な遺伝子として,活性化される遺伝子としてIfng遺伝子,抑制される遺伝子としてIsg15遺伝子があげられるが,Isg15遺伝子にくわえ,T-betにより抑制される遺伝子としてI型インターフェロンにより発現の誘導される遺伝子の発現が認められた.さらに,T-betにより抑制された遺伝子の遺伝子オントロジー解析により,I型インターフェロン応答および抗ウイルス性応答が有意に認められた.対照的に,T-betにより活性化される遺伝子の遺伝子オントロジー解析により解糖系の酵素を含む代謝遺伝子が認められた.インターフェロンγにより刺激したT-bet欠損CD4陽性T細胞においてI型インターフェロンにより発現の誘導される遺伝子の発現が亢進していたことから,T-betはそれらの転写を抑制することが示された.
インターフェロンβあるいはインターフェロンγにより制御される転写産物の転写への経時的な効果を比較したところCD4陽性T細胞においては転写制御の結果が大きく異なり,インターフェロンβおよびインターフェロンγの両方により制御される遺伝子は非常に少なかった.さらに,インターフェロンβは急速にインターフェロン誘導遺伝子の発現を誘導したが,インターフェロンγのおもな影響は晩期における代謝関連遺伝子の発現の誘導であった.インターフェロンγあるいはインターフェロンβの効果に対するT-betの影響について検討したところ,早期におけるインターフェロンβによる優先的なインターフェロン誘導遺伝子の発現の誘導はT-betとはほとんど無関係であった.しかし,後期においてT-betを欠損したCD4陽性T細胞においてはインターフェロンβおよびインターフェロンγはともにインターフェロン誘導遺伝子の発現を誘導した.このことから,ヘルパーT細胞はインターフェロンβシグナルおよびインターフェロンγシグナルに対しT-betに依存性に適切な応答をすることが示唆された.つまり,T-betの非存在下においてはインターフェロンγシグナルの特異性が維持されず,代わりに,インターフェロンβシグナルに近似することが示唆された.
2.インターフェロンγにより活性化されたT-betを欠損したCD4陽性T細胞におけるSTAT2の異常な活性化
T-betを欠損したCD4陽性T細胞においてみられた異常なインターフェロン応答の根底にある分子機構を明らかにするため,T-betがインターフェロンシグナル伝達系を変化させるかどうか検討した.I型インターフェロンはインターフェロン刺激応答配列(interferon-stimulated response element:ISRE)と結合するSTAT1,STAT2,IRF9からなるヘテロ三量体を活性化する8).野生型のCD4陽性T細胞において,インターフェロンβはSTAT2の発現およびリン酸化を誘導したが,インターフェロンγにはこのような効果は認められなかった.しかし,T-betを欠損したCD4陽性T細胞においてはインターフェロンγおよびインターフェロンβの両方がSTAT2の発現およびリン酸化を誘導した.これと一致して,T-betを欠損したCD4陽性T細胞のゲノムの全体におけるSTAT2の結合も野生型のCD4陽性T細胞と比較して増加した.さらに,T-betを欠損したCD4陽性T細胞においてインターフェロンγおよびインターフェロンβによりインターフェロン誘導遺伝子の近傍へのSTAT2の局在的な結合が増強され,活性化したエンハンサーの指標であるヒストンH3のLys27のアセチル化の増強と関連した.これらの結果から,T-betを欠損したCD4陽性T細胞においてはインターフェロンγがSTAT2を異常に活性化し,活性化されたSTAT2はインターフェロン誘導遺伝子の遺伝子座の近傍に結合し,その周辺のエンハンサーを活性化することが示された.T-betとSTAT2の結合部位の重複を比較することによりT-betがSTAT2の結合と直接的に干渉するかどうかを検討したところ,T-betの結合部位の24%がSTAT2の結合部位と重複していたことから,T-betとSTAT2が遺伝子座に競合的に結合する可能性が示唆された.
3.T-betの非存在下においてT-betにより抑制される遺伝子座はSTAT2,IRF7,STAT1の標的になる
IRF7はI型インターフェロンにより発現が誘導され,I型インターフェロンの発現を制御する9).T-betを欠損したCD4陽性T細胞においてインターフェロンγに応答してIRF7のタンパク質レベルでの発現も異常に上昇した.T-betが存在しない場合,STAT2,IRF7,STAT1 2) のようなI型インターフェロンシグナル伝達系の重要な転写因子の,通常はT-betにより抑制される遺伝子座の近傍への結合が増強される可能性について考慮した.その結果,T-betはT-betにより抑制される遺伝子座に比べT-betにより活性化される遺伝子座への結合が多く認められた一方,STAT2,IRF7,STAT1は通常はT-betにより抑制される遺伝子座の周囲により多く結合した.さらに,さまざまな条件におけるそのほかの転写因子の結合を筆者らによるデータおよびこれまでに公開されたデータから検討したところ,T-betにより抑制される遺伝子座へのSTAT2,IRF7,STAT1の優先的な結合が,インターフェロンγによる刺激のもとSTAT2,IRF7,STAT1に選択的に認められた.T-betにより抑制される遺伝子座へのSTAT1の有意な優先的な結合はインターフェロンγを処理したT-betを欠損したCD4陽性T細胞においては観察されたがインターフェロンγを処理した野生型のCD4陽性T細胞においては観察されず,T-betの非存在下においてSTAT1の分布がゲノムワイドに変化することが示唆された.したがって,T-betの非存在下においては通常はT-betにより抑制される遺伝子座が,インターフェロンγの刺激に応答してSTAT2,IRF7,STAT1の優先的な標的になることが示唆された.
4.I型インターフェロンのオートクラインの阻害によりT-betを欠損したCD4陽性T細胞においてインターフェロンγによる正常な転写制御が回復する
I型インターフェロンシグナル伝達系はインターフェロンのオートクラインを組み込んだフィードフォワードループから構成される9).したがって,I型インターフェロン応答を促進するSTAT2,IRF7,STAT1の増強はI型インターフェロンの産生の増強をひき起こすことが予想された.事実,インターフェロンγを処理したT-betを欠損したCD4陽性細胞において異常に高いレベルのI型インターフェロンが検出された.インターフェロンγを処理したT-betを欠損したCD4陽性T細胞におけるインターフェロンにより発現の誘導される遺伝子の発現から,このI型インターフェロンのオートクラインに起因するI型インターフェロンシグナル伝達系のフィードフォワードループの存在が示唆された.この可能性を確認するため,T-betを欠損したCD4陽性T細胞に抗インターフェロンα/β受容体抗体をインターフェロンγとともに添加したところ,用量に依存性にインターフェロンγの正常な発現が回復し,異常に亢進したインターフェロン誘導遺伝子の発現が減弱した.さらに,抗インターフェロンα/β受容体抗体はT-betを欠損したCD4陽性T細胞においてインターフェロンγにより異常に誘導されたSTAT2の活性を減弱させた.また,抗インターフェロンα/β受容体抗体はI型インターフェロンおよびIRF7のmRNAレベルでの異常な発現を改善したが,IRF3には影響をおよぼさなかった.したがって,インターフェロンγが豊富に存在する環境において,T-betはI型インターフェロンシグナル伝達系のフィードフォワードループの増幅を抑制するのに重要であることが明らかにされた(図2).T-betが存在しない場合,CD4陽性T細胞はインターフェロンγをI型インターフェロンであると誤認し,I型インターフェロン応答に近似する転写制御を開始する.しかし,インターフェロン誘導遺伝子座の26%はT-betと直接に結合しているため,インターフェロン誘導遺伝子座のうちのいくつかはT-betにより直接的に抑制される可能性がある.
5.生体においてT-betはI型インターフェロンシグナル伝達系の重要な転写因子を選択的に抑制し正のフィードバックループを抑制する
in vitroの系において明らかにされた,T-betがインターフェロンγの下流に存在するI型インターフェロン伝達系のフィードフォワードループを抑制するという作用を,インターフェロンγの発現を誘導することが知られている寄生虫Toxoplasma gondiiの感染モデルを用いて検討した10).T-betノックアウトマウスは適切なTh1応答をすることができずT. gondiiの感染に耐えられないため11),野生型のCD4陽性T細胞およびT-betを欠損したCD4陽性T細胞の両方をRag2ノックアウトマウスに移入したのちT. gondiiを感染させた.このマウスはT. gondiiの感染に耐え,野生型CD4陽性T細胞およびT-betを欠損したCD4陽性T細胞は炎症環境にさらされる.その結果,T. gondiiに感染したマウスの腹腔浸出液から得た細胞において,T-betを欠損したCD4陽性T細胞は野生型のCD4陽性T細胞と比べSTAT1,STAT2,IRF7,IRF9の発現の顕著な上昇が認められ,in vitroの系において認められたようなI型インターフェロンシグナル伝達系のフィードフォワードループの増幅の起こる可能性が示唆された.T-betを欠損したCD4陽性T細胞におけるI型インターフェロンシグナル伝達系のフィードフォワードループの亢進は細胞に特有の現象である.対照的に,おもにI型インターフェロン応答がひき起こされるリンパ球性脈絡髄膜炎ウイルスの感染においては,STAT1,STAT2,IRF7,IRF9の発現に対しT-betの欠損は大きな影響を示さなかった.以上のことから,T-betはTh1細胞の分化において異常なI型インターフェロンシグナル伝達系のフィードフォワードループを抑制することが示された.
おわりに
一般に,T-betのような系統決定的転写因子はヘルパーT細胞のサブセットの固定を促進しほかのサブセットへの脱分化を制限することが知られてきた1).この研究においては,系統決定的転写因子のそのほかの機能,つまり,細胞から分泌されたタンパク質の検知に系統決定的転写因子が影響をおよぼすことが示された.すなわち,T-betの非存在下においては,CD4陽性T細胞はI型インターフェロンとII型インターフェロンを異なる刺激として認識できなくなる.同様の機能をもつ系統決定的転写因子がどのくらい存在するのかは今後の検討課題のひとつである.さらに,多くの系統決定的転写因子は転写のアクティベーターまたはリプレッサーとよばれているが,ChIP-seq法により得られたデータから,T-betは約半分の遺伝子に対しアクティベーターとして,約半分の遺伝子に対しリプレッサーとして機能することが示された.T-betがある遺伝子座の近傍に結合した際に,その遺伝子の発現を促進するか抑制するかについては議論の余地があるが,おそらく,同じ遺伝子座と結合するほかの転写因子12,13),および,クロマチンのコンフォメーションあるいはループ14,15) により決定されると推測される.それでは,T-betは遺伝子のどこに結合するのか,どの転写因子とパートナーになるのかをどのように知るのだろうか? おそらく,この情報はゲノムにコードされていると推定されるが,遺伝子座に特異的な転写因子の結合の組合せが遺伝子にどのように設計されているかは今後の研究課題である.
文 献
- Zhu, J., Yamane, H. & Paul, W. E.: Differentiation of effector CD4 T cell populations *. Annu. Rev. Immunol., 28, 445-489 (2010)[PubMed]
- Mostafavi, S., Yoshida, H., Moodley, D. et al.: Parsing the interferon transcriptional network and its disease associations. Cell, 164, 564-578 (2016)[PubMed]
- Schneider, W. M., Chevillotte, M. D. & Rice, C. M.: Interferon-stimulated genes: a complex web of host defenses. Annu. Rev. Immunol., 32, 513-545 (2014)[PubMed]
- Theofilopoulos, A. N., Baccala, R., Beutler, B. et al.: Type I interferons (α/β) in immunity and autoimmunity. Annu. Rev. Immunol., 23, 307-335 (2005)[PubMed]
- Heinz, S., Romanoski, C. E., Benner, C. et al.: The selection and function of cell type-specific enhancers. Nat. Rev. Mol. Cell Biol., 16, 144-154 (2015)[PubMed]
- Szabo, S. J., Kim, S. T., Costa, G. L. et al.: A novel transcription factor, T-bet, directs Th1 lineage commitment. Cell, 100, 655-669 (2000)[PubMed]
- Yosef, N. & Regev, A.: Writ large: genomic dissection of the effect of cellular environment on immune response. Science, 354, 64-68 (2016)[PubMed]
- Darnell, J. E. J., Kerr, I. M. & Stark, G. R.: Jak-STAT pathways and transcriptional activation in response to IFNs and other extracellular signaling proteins. Science, 264, 1415-1421 (1994)[PubMed]
- Honda, K., Takaoka, A. & Taniguchi, T.: Type I interferon gene induction by the interferon regulatory factor family of transcription factors. Immunity, 25, 349-360 (2006)[PubMed]
- Gazzinelli, R. T., Denkers, E. Y. & Sher, A.: Host resistance to Toxoplasma gondii: model for studying the selective induction of cell-mediated immunity by intracellular parasites. Infect. Agents Dis., 2, 139-149 (1993)[PubMed]
- Harms Pritchard, G., Hall, A. O., Christian, D. A. et al.: Diverse roles for T-bet in the effector responses required for resistance to infection. J. Immunol., 194, 1131-1140 (2015)[PubMed]
- Djuretic, I. M., Levanon, D., Negreanu, V. et al.: Transcription factors T-bet and Runx3 cooperate to activate Ifng and silence Il4 in T helper type 1 cells. Nat. Immunol., 8, 145-153 (2006)[PubMed]
- Dominguez, C. X., Amezquita, R. A., Guan, T. et al.: The transcription factors ZEB2 and T-bet cooperate to program cytotoxic T cell terminal differentiation in response to LCMV viral infection. J. Exp. Med., 212, 2041-2056 (2015)[PubMed]
- Liu, C. F., Brandt, G. S., Hoang, Q. Q. et al.: Crystal structure of the DNA binding domain of the transcription factor T-bet suggests simultaneous recognition of distant genome sites. Proc. Natl. Acad. Sci. USA, 113, E6572-E6581 (2016)[PubMed]
- Sekimata, M., PErez-Melgosa, M., Miller, S. A. et al.: CCCTC-binding factor and the transcription factor T-bet orchestrate T helper 1 cell-specific structure and function at the interferon-γ locus. Immunity, 31, 551-564 (2009)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
米国NIH National Institute of Arthritis and Musculoskeletal and Skin Diseases常任研究員.
岩田 慈(Shigeru Iwata)
略歴:2011年 産業医科大学大学院医学研究科にて博士号取得,2013年 米国NIH National Institute of Arthritis and Musculoskeletal and Skin Diseases博士研究員を経て,2015年より産業医科大学.
三上 洋平(Yohei Mikami)
略歴:2012年 慶應義塾大学大学院医学研究科博士課程 修了,2013年より米国NIH National Institute of Arthritis and Musculoskeletal and Skin Diseases博士研究員.
研究テーマ:腸管粘膜免疫におけるエピゲノムの制御機構の解明および新規の治療法の開発.
© 2017 菅野由香・岩田 慈・三上洋平 Licensed under CC 表示 2.1 日本