Cdc48-Rad23/Dsk2軸はプロテアソームに依存的なタンパク質分解における主要な経路である
土屋 光・大竹史明・田中啓二・佐伯 泰
(東京都医学総合研究所 蛋白質代謝研究室)
email:佐伯 泰
DOI: 10.7875/first.author.2017.055
In vivo ubiquitin linkage-type analysis reveals that the Cdc48-Rad23/Dsk2 axis contributes to K48-linked chain specificity of the proteasome.
Hikaru Tsuchiya, Fumiaki Ohtake, Naoko Arai, Ai Kaiho, Sayaka Yasuda, Keiji Tanaka, Yasushi Saeki
Molecular Cell, 66, 488-502.e7 (2017)
タンパク質のユビキチン化はプロテアソームに依存的なタンパク質分解のみならず,タンパク質の輸送やシグナル伝達など多彩な生命現象に関与する.ユビキチン結合タンパク質は構造の異なるユビキチン鎖を識別するデコーダー分子と想定されているが,細胞において実際にどの種類のユビキチン鎖を認識しているかについてはわかっていなかった.筆者らは,この研究において,出芽酵母のもつ主要な14種類のユビキチン結合タンパク質により認識されるユビキチン鎖を質量分析計を用いて網羅的に定量した.その結果,細胞におけるユビキチン鎖の使い分けが明確にされたとともに,ユビキチンに選択的な分子シャペロンCdc48とシャトルタンパク質であるRad23およびDsk2を介した間接的な経路がプロテアソームに依存的なタンパク質分解の主要な経路であることが明らかにされた.
ユビキチン化は,ユビキチン活性化酵素,ユビキチン連結酵素,ユビキチンリガーゼによる一連の酵素カスケードにより標的となるタンパク質にユビキチンが付加される翻訳後修飾である1).ユビキチン化がリン酸化やアセチル化など化学的な修飾と大きく異なる特徴として,構造に多様性をもつことがあげられる.Lysに1分子のユビキチンが付加されるモノユビキチン化は,標的となるタンパク質の局在や活性の制御に関与することが知られている.また,ユビキチンはそれ自体のもつ7箇所のLysおよびN末端のMetを介し,Lys6鎖,Lys11鎖,Lys27鎖,Lys29鎖,Lys33鎖,Lys48鎖,Lys63鎖,Met1鎖の構造の異なるユビキチン鎖を形成する.構造の異なるユビキチン鎖はそれぞれ異なる機能を発揮すると考えられている.Lys48を介して連結したLys48鎖はプロテアソームに依存的なタンパク質分解のシグナルとして,Lys63鎖はタンパク質の輸送,DNA修復,シグナル伝達などに関与することが知られている.また,Lys11鎖は細胞周期に特異的なタンパク質分解に,Met1鎖はNF-κBシグナルの活性化にかかわることが知られているが,そのほかのユビキチン鎖については不明な点が多い.さらに近年では,細胞にはユビキチンタンパク質それ自体へのリン酸化やアセチル化などの翻訳後修飾,また,異なる種類のユビキチン鎖が連結した混合鎖やひとつのユビキチンに複数のユビキチンが付加した分岐鎖など,複雑な構造をもつユビキチン鎖も存在することが明らかにされてきた.このようなユビキチン鎖の構造の多様性は“ユビキチンコード”と称されている2,3).
ユビキチン結合タンパク質は構造の異なるユビキチン鎖を特異的に認識し情報を下流に伝達するデコーダー分子と想定されている4).ユビキチンを識別するモチーフとしてUIM,UBA,UBANなど約20種類のユビキチン結合ドメインが同定されており,結晶構造解析やNMRの化学シフトによりユビキチンとの相互作用が原子レベルで明らかにされている.これまで,in vitroにおける解析により,NEMOのUBANドメインおよびUIMC1/RAP80のUIMドメインがそれぞれMet1鎖およびLys63鎖と特異的に結合することが報告されているが,そのほかの多くのユビキチン結合ドメインにはユビキチン鎖に対する厳密な選択性はみられない.出芽酵母には約50種類のユビキチン結合タンパク質が存在するが,細胞においてこれらはどのように使い分けられるのか,実際にどのユビキチンシグナルを識別するのか,など,ユビキチンシグナルの発動の機構についてはほとんどわかっていない.そのもっとも大きな理由として,ユビキチンシグナルが一過的かつ極微量であるため,従来の解析技術ではユビキチン結合タンパク質が認識するユビキチン鎖を直接に解析することが困難だったことがあげられる.
現在,ユビキチン鎖の種類を識別する手法としては質量分析計がもっとも有効とされている.以前に筆者らは,高分解能な質量分析計を用いたユビキチン鎖の超高感度な絶対定量法としてUb-AQUA/PRM(ubiquitin-absolute quantification/parallel reaction monitoring)法の開発に成功していたことから5),この方法によりユビキチン結合タンパク質が細胞において認識するユビキチン鎖の種類を明確にできると考えた.出芽酵母のもつ主要な14種類のユビキチン結合タンパク質にそれぞれ3×FLAGタグをノックインした株を作製し,細胞においてユビキチン鎖に対する選択性について網羅的に解析した.その結果,プロテアソーム分解系に関与するシャトルタンパク質であるRad23やDsk2など,ユビキチンに選択的な分子シャペロンCdc48のコファクターであるUfd1やNpl4は,Lys29鎖およびLys48鎖に対し高い選択性を示すこと,エンドサイトーシスに関与するEnt1やEnt2などはLys63鎖に対し高い選択性を示すことがわかった.一方,オートファジー経路にかかわるCue5およびMVB輸送経路にかかわるVps27は選択性が低く,Lys48鎖およびLys63鎖を認識した.
細胞にプロテアソームの阻害剤を処理することによりユビキチン鎖やユビキチン化基質の量が劇的に変動することが知られている6,7).そこで,プロテアソームの活性がおのおののユビキチン鎖に依存的な経路におよぼす影響について解析した.その結果,プロテアソーム阻害剤の処理によりシャトルタンパク質であるRad23と結合するユビキチン鎖の量は増加したが,ユビキチンに選択的な分子シャペロンCdc48のコファクターであるUfd1と結合するユビキチン鎖の量はほとんど変化しなかった.このことから,Cdc48-Ufd1-Npl4複合体がプロテアソーム分解系の律速になることが示唆された.エンドサイトーシスに関与するEnt2と結合するユビキチン鎖はプロテアソーム阻害剤により減少し,これはプロテアソーム阻害剤によるアミノ酸プールの減少にともなうアミノ酸トランスポーターの安定化によると示唆された.一方,オートファジー経路にかかわるCue5およびMVB輸送経路にかかわるVps27においては結合するLys48鎖の量が増加した.このことから,液胞(リソソーム)によるタンパク質分解はプロテアソーム分解系を補完することが示唆された.このように,プロテアソーム阻害剤の処理により解析したすべてのユビキチンに依存的な経路が変動したことから,プロテアソームの活性はユビキチンネットワークの全体に影響をおよぼすことが明らかにされた.
GSTを融合させたユビキチン結合タンパク質を用い,in vitroにおいて,Lys48鎖およびLys63鎖に対する結合能について解析した.その結果,従来の報告のとおり,プロテアソームのもつユビキチン受容体Rpn10やRad23などは,強度の違いはあるもののLys48鎖およびLys63鎖と結合した.一方,ユビキチンに選択的な分子シャペロンCdc48のコファクターについて検討したところ,Npl4がLys48鎖,とくに,6つ以上連結した長いユビキチン鎖に対し高い選択性をもつことが見い出された.バイオインフォマティクス解析により,Npl4はJAMMドメインと相同性のあるドメインをもつこと,変異により温度感受性になるGly323がこのJAMM様のドメインに含まれること,そして,このGly323の変異によりユビキチン鎖の結合能は消失することがわかった.Cdc48およびUfd1にはユビキチン鎖に対する結合能はなかったため,Cdc48-Ufd1-Npl4複合体はNpl4を介してLys48鎖と結合することが判明した.
プロテアソームはRpn10などのユビキチン受容体をもち,従来,プロテアソームはユビキチン化基質を直接に認識して分解すると考えられてきた.しかし,in vitroにおいてLys48鎖に対する選択性はみられず,Lys63鎖の結合した基質やモノユビキチン化された基質を分解した.そこで,細胞においてプロテアソームと相互作用するユビキチン鎖を絶対定量したところ,Lys48鎖に対した高い選択性を示した.Rad23とDsk2の二重欠損細胞において解析したところ,プロテアソームと結合するユビキチン鎖の量はいずれも10%以下にまで減少した.このことから,細胞においては大部分のユビキチン化基質はプロテアソームにより直接に認識されるのではなく,シャトルタンパク質Rad23とDsk2を介してプロテアソームに輸送されることが明らかにされた.
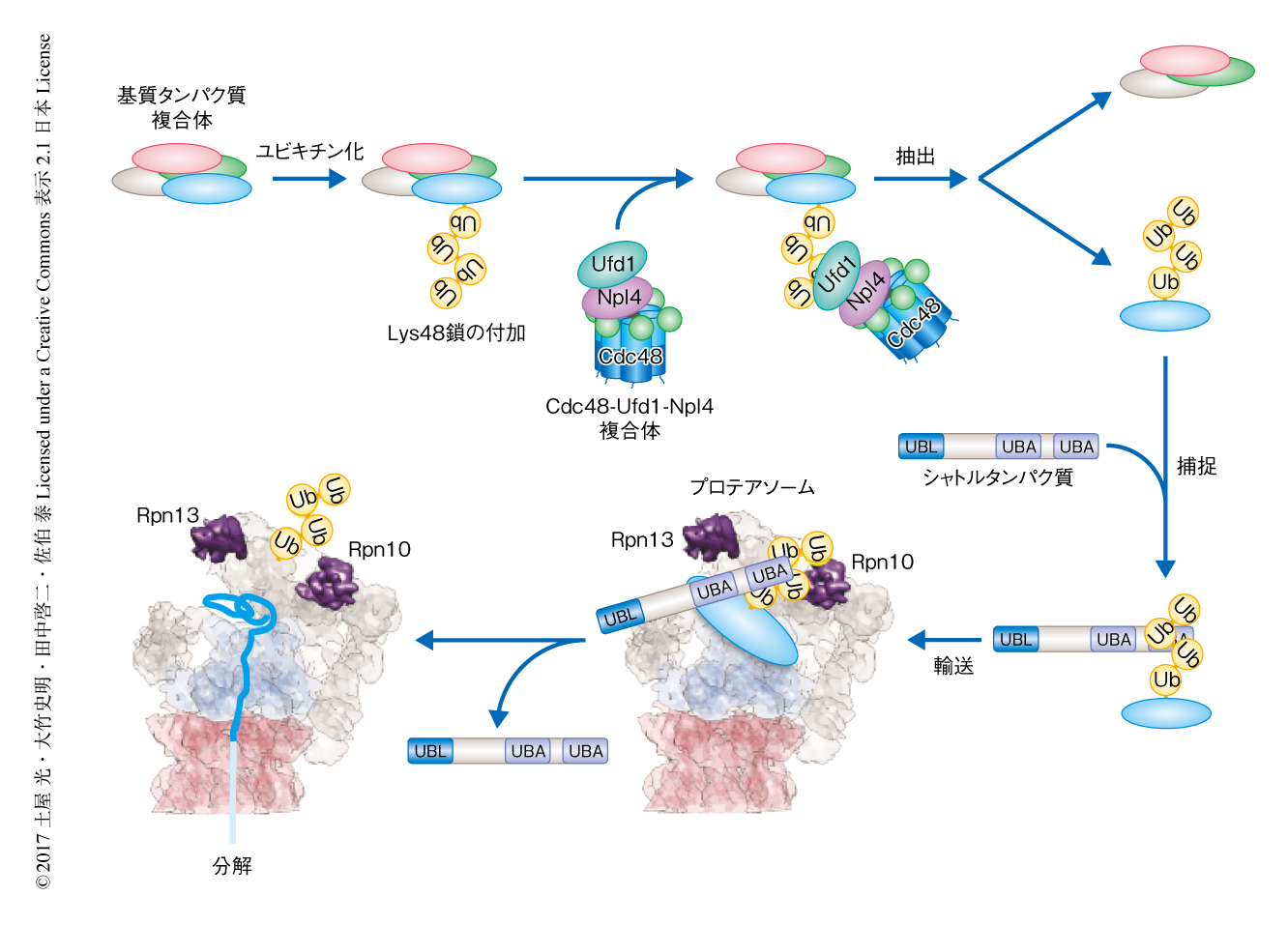
一方,Cdc48の不活性化された変異体においてプロテアソームと相互作用するユビキチン鎖を絶対定量したところ,さまざまな種類のユビキチン鎖がプロテアソームに大量に蓄積することがわかった.さらに,Rad23およびDsk2を欠損させるとその蓄積は完全にキャンセルされたことから,シャトルタンパク質Rad23およびDsk2はユビキチンに選択的な分子シャペロンCdc48の下流において機能し,Cdc48-Rad23/Dsk2軸が細胞におけるプロテアソーム分解系の主要な経路であることが示唆された(図1).
この研究により,細胞におけるユビキチン鎖の使い分けが明確にされるとともに,ユビキチンに選択的な分子シャペロンCdc48とシャトルタンパク質Rad23およびDsk2を介した間接的な経路がプロテアソームに依存的なタンパク質分解の主要な経路であることが明らかにされた.なぜ,プロテアソーム分解系にCdc48が必要なのか疑問が残ったが,この報告とほぼ同じ時期に,Cdc48/p97がユビキチン化タンパク質の解きほぐしの活性をもつことが報告された8,9).プロテアソームは硬い構造をもつタンパク質を分解することが苦手であり,Cdc48-Ufd1-Npl4複合体による基質となるタンパク質の解きほぐしが必要なのかもしれない.
また,ユビキチン鎖のうちLys6鎖,Lys11鎖,Lys27鎖,Lys33鎖,Met1鎖に対し選択性を示すユビキチン結合タンパク質は,解析した出芽酵母の14種類のなかには存在しなかった.NF-κB経路におけるMet1鎖に代表されるように,これら非典型的なユビキチン鎖のシグナル伝達における役割から考えると,デコーダーとなる未同定のタンパク質が存在することが推察される.さらに,ヒトのゲノムには100種類をこえるユビキチン結合タンパク質がコードされており,酵母からヒトへとユビキチンシグナルの多様性は増加していることが想定される.実際,ごく最近,8種類のユビキチン二量体をベイトとした大規模なプロテオーム解析がなされ,ヒトにおいてLys6鎖やLys27鎖と特異的に結合するユビキチン結合タンパク質が同定された10).今後,ヒトのユビキチン結合タンパク質についても細胞におけるユビキチン鎖に対する選択性をひとつずつ検証することにより,ユビキチンコードとそのデコーディングの機構の全貌が明らかにされると期待される.
東京都医学総合研究所 研究員.
大竹 史明(Fumiaki Ohtake)
東京都医学総合研究所 主席研究員.
田中 啓二(Keiji Tanaka)
東京都医学総合研究所 所長.
佐伯 泰(Yasushi Saeki)
東京都医学総合研究所 副参事研究員.
研究室URL:http://www.igakuken.or.jp/pro-meta/
© 2017 土屋 光・大竹史明・田中啓二・佐伯 泰 Licensed under CC 表示 2.1 日本
(東京都医学総合研究所 蛋白質代謝研究室)
email:佐伯 泰
DOI: 10.7875/first.author.2017.055
In vivo ubiquitin linkage-type analysis reveals that the Cdc48-Rad23/Dsk2 axis contributes to K48-linked chain specificity of the proteasome.
Hikaru Tsuchiya, Fumiaki Ohtake, Naoko Arai, Ai Kaiho, Sayaka Yasuda, Keiji Tanaka, Yasushi Saeki
Molecular Cell, 66, 488-502.e7 (2017)
要 約
タンパク質のユビキチン化はプロテアソームに依存的なタンパク質分解のみならず,タンパク質の輸送やシグナル伝達など多彩な生命現象に関与する.ユビキチン結合タンパク質は構造の異なるユビキチン鎖を識別するデコーダー分子と想定されているが,細胞において実際にどの種類のユビキチン鎖を認識しているかについてはわかっていなかった.筆者らは,この研究において,出芽酵母のもつ主要な14種類のユビキチン結合タンパク質により認識されるユビキチン鎖を質量分析計を用いて網羅的に定量した.その結果,細胞におけるユビキチン鎖の使い分けが明確にされたとともに,ユビキチンに選択的な分子シャペロンCdc48とシャトルタンパク質であるRad23およびDsk2を介した間接的な経路がプロテアソームに依存的なタンパク質分解の主要な経路であることが明らかにされた.
はじめに
ユビキチン化は,ユビキチン活性化酵素,ユビキチン連結酵素,ユビキチンリガーゼによる一連の酵素カスケードにより標的となるタンパク質にユビキチンが付加される翻訳後修飾である1).ユビキチン化がリン酸化やアセチル化など化学的な修飾と大きく異なる特徴として,構造に多様性をもつことがあげられる.Lysに1分子のユビキチンが付加されるモノユビキチン化は,標的となるタンパク質の局在や活性の制御に関与することが知られている.また,ユビキチンはそれ自体のもつ7箇所のLysおよびN末端のMetを介し,Lys6鎖,Lys11鎖,Lys27鎖,Lys29鎖,Lys33鎖,Lys48鎖,Lys63鎖,Met1鎖の構造の異なるユビキチン鎖を形成する.構造の異なるユビキチン鎖はそれぞれ異なる機能を発揮すると考えられている.Lys48を介して連結したLys48鎖はプロテアソームに依存的なタンパク質分解のシグナルとして,Lys63鎖はタンパク質の輸送,DNA修復,シグナル伝達などに関与することが知られている.また,Lys11鎖は細胞周期に特異的なタンパク質分解に,Met1鎖はNF-κBシグナルの活性化にかかわることが知られているが,そのほかのユビキチン鎖については不明な点が多い.さらに近年では,細胞にはユビキチンタンパク質それ自体へのリン酸化やアセチル化などの翻訳後修飾,また,異なる種類のユビキチン鎖が連結した混合鎖やひとつのユビキチンに複数のユビキチンが付加した分岐鎖など,複雑な構造をもつユビキチン鎖も存在することが明らかにされてきた.このようなユビキチン鎖の構造の多様性は“ユビキチンコード”と称されている2,3).
ユビキチン結合タンパク質は構造の異なるユビキチン鎖を特異的に認識し情報を下流に伝達するデコーダー分子と想定されている4).ユビキチンを識別するモチーフとしてUIM,UBA,UBANなど約20種類のユビキチン結合ドメインが同定されており,結晶構造解析やNMRの化学シフトによりユビキチンとの相互作用が原子レベルで明らかにされている.これまで,in vitroにおける解析により,NEMOのUBANドメインおよびUIMC1/RAP80のUIMドメインがそれぞれMet1鎖およびLys63鎖と特異的に結合することが報告されているが,そのほかの多くのユビキチン結合ドメインにはユビキチン鎖に対する厳密な選択性はみられない.出芽酵母には約50種類のユビキチン結合タンパク質が存在するが,細胞においてこれらはどのように使い分けられるのか,実際にどのユビキチンシグナルを識別するのか,など,ユビキチンシグナルの発動の機構についてはほとんどわかっていない.そのもっとも大きな理由として,ユビキチンシグナルが一過的かつ極微量であるため,従来の解析技術ではユビキチン結合タンパク質が認識するユビキチン鎖を直接に解析することが困難だったことがあげられる.
1.細胞におけるユビキチン結合タンパク質のユビキチン鎖に対する選択性
現在,ユビキチン鎖の種類を識別する手法としては質量分析計がもっとも有効とされている.以前に筆者らは,高分解能な質量分析計を用いたユビキチン鎖の超高感度な絶対定量法としてUb-AQUA/PRM(ubiquitin-absolute quantification/parallel reaction monitoring)法の開発に成功していたことから5),この方法によりユビキチン結合タンパク質が細胞において認識するユビキチン鎖の種類を明確にできると考えた.出芽酵母のもつ主要な14種類のユビキチン結合タンパク質にそれぞれ3×FLAGタグをノックインした株を作製し,細胞においてユビキチン鎖に対する選択性について網羅的に解析した.その結果,プロテアソーム分解系に関与するシャトルタンパク質であるRad23やDsk2など,ユビキチンに選択的な分子シャペロンCdc48のコファクターであるUfd1やNpl4は,Lys29鎖およびLys48鎖に対し高い選択性を示すこと,エンドサイトーシスに関与するEnt1やEnt2などはLys63鎖に対し高い選択性を示すことがわかった.一方,オートファジー経路にかかわるCue5およびMVB輸送経路にかかわるVps27は選択性が低く,Lys48鎖およびLys63鎖を認識した.
2.プロテアソームの活性がユビキチンネットワークにおよぼす影響
細胞にプロテアソームの阻害剤を処理することによりユビキチン鎖やユビキチン化基質の量が劇的に変動することが知られている6,7).そこで,プロテアソームの活性がおのおののユビキチン鎖に依存的な経路におよぼす影響について解析した.その結果,プロテアソーム阻害剤の処理によりシャトルタンパク質であるRad23と結合するユビキチン鎖の量は増加したが,ユビキチンに選択的な分子シャペロンCdc48のコファクターであるUfd1と結合するユビキチン鎖の量はほとんど変化しなかった.このことから,Cdc48-Ufd1-Npl4複合体がプロテアソーム分解系の律速になることが示唆された.エンドサイトーシスに関与するEnt2と結合するユビキチン鎖はプロテアソーム阻害剤により減少し,これはプロテアソーム阻害剤によるアミノ酸プールの減少にともなうアミノ酸トランスポーターの安定化によると示唆された.一方,オートファジー経路にかかわるCue5およびMVB輸送経路にかかわるVps27においては結合するLys48鎖の量が増加した.このことから,液胞(リソソーム)によるタンパク質分解はプロテアソーム分解系を補完することが示唆された.このように,プロテアソーム阻害剤の処理により解析したすべてのユビキチンに依存的な経路が変動したことから,プロテアソームの活性はユビキチンネットワークの全体に影響をおよぼすことが明らかにされた.
3.in vitroにおけるユビキチン結合タンパク質のユビキチン鎖に対する選択性
GSTを融合させたユビキチン結合タンパク質を用い,in vitroにおいて,Lys48鎖およびLys63鎖に対する結合能について解析した.その結果,従来の報告のとおり,プロテアソームのもつユビキチン受容体Rpn10やRad23などは,強度の違いはあるもののLys48鎖およびLys63鎖と結合した.一方,ユビキチンに選択的な分子シャペロンCdc48のコファクターについて検討したところ,Npl4がLys48鎖,とくに,6つ以上連結した長いユビキチン鎖に対し高い選択性をもつことが見い出された.バイオインフォマティクス解析により,Npl4はJAMMドメインと相同性のあるドメインをもつこと,変異により温度感受性になるGly323がこのJAMM様のドメインに含まれること,そして,このGly323の変異によりユビキチン鎖の結合能は消失することがわかった.Cdc48およびUfd1にはユビキチン鎖に対する結合能はなかったため,Cdc48-Ufd1-Npl4複合体はNpl4を介してLys48鎖と結合することが判明した.
4.プロテアソームへのユビキチン化基質の輸送経路
プロテアソームはRpn10などのユビキチン受容体をもち,従来,プロテアソームはユビキチン化基質を直接に認識して分解すると考えられてきた.しかし,in vitroにおいてLys48鎖に対する選択性はみられず,Lys63鎖の結合した基質やモノユビキチン化された基質を分解した.そこで,細胞においてプロテアソームと相互作用するユビキチン鎖を絶対定量したところ,Lys48鎖に対した高い選択性を示した.Rad23とDsk2の二重欠損細胞において解析したところ,プロテアソームと結合するユビキチン鎖の量はいずれも10%以下にまで減少した.このことから,細胞においては大部分のユビキチン化基質はプロテアソームにより直接に認識されるのではなく,シャトルタンパク質Rad23とDsk2を介してプロテアソームに輸送されることが明らかにされた.
一方,Cdc48の不活性化された変異体においてプロテアソームと相互作用するユビキチン鎖を絶対定量したところ,さまざまな種類のユビキチン鎖がプロテアソームに大量に蓄積することがわかった.さらに,Rad23およびDsk2を欠損させるとその蓄積は完全にキャンセルされたことから,シャトルタンパク質Rad23およびDsk2はユビキチンに選択的な分子シャペロンCdc48の下流において機能し,Cdc48-Rad23/Dsk2軸が細胞におけるプロテアソーム分解系の主要な経路であることが示唆された(図1).
おわりに
この研究により,細胞におけるユビキチン鎖の使い分けが明確にされるとともに,ユビキチンに選択的な分子シャペロンCdc48とシャトルタンパク質Rad23およびDsk2を介した間接的な経路がプロテアソームに依存的なタンパク質分解の主要な経路であることが明らかにされた.なぜ,プロテアソーム分解系にCdc48が必要なのか疑問が残ったが,この報告とほぼ同じ時期に,Cdc48/p97がユビキチン化タンパク質の解きほぐしの活性をもつことが報告された8,9).プロテアソームは硬い構造をもつタンパク質を分解することが苦手であり,Cdc48-Ufd1-Npl4複合体による基質となるタンパク質の解きほぐしが必要なのかもしれない.
また,ユビキチン鎖のうちLys6鎖,Lys11鎖,Lys27鎖,Lys33鎖,Met1鎖に対し選択性を示すユビキチン結合タンパク質は,解析した出芽酵母の14種類のなかには存在しなかった.NF-κB経路におけるMet1鎖に代表されるように,これら非典型的なユビキチン鎖のシグナル伝達における役割から考えると,デコーダーとなる未同定のタンパク質が存在することが推察される.さらに,ヒトのゲノムには100種類をこえるユビキチン結合タンパク質がコードされており,酵母からヒトへとユビキチンシグナルの多様性は増加していることが想定される.実際,ごく最近,8種類のユビキチン二量体をベイトとした大規模なプロテオーム解析がなされ,ヒトにおいてLys6鎖やLys27鎖と特異的に結合するユビキチン結合タンパク質が同定された10).今後,ヒトのユビキチン結合タンパク質についても細胞におけるユビキチン鎖に対する選択性をひとつずつ検証することにより,ユビキチンコードとそのデコーディングの機構の全貌が明らかにされると期待される.
文 献
- Hershko, A. & Ciechanover, A.: The ubiquitin system. Annu. Rev. Biochem., 67, 425-479 (1998)[PubMed]
- Swatek, K. N. & Komander, D.: Ubiquitin modifications. Cell Res., 26, 399-422 (2016)[PubMed]
- Yau, R. & Rape, M.: The increasing complexity of the ubiquitin code. Nat. Cell Biol., 18, 579-586 (2016)[PubMed]
- Husnjak, K. & Dikic, I.: Ubiquitin-binding proteins: decoders of ubiquitin-mediated cellular functions. Annu. Rev. Biochem., 81, 291-322 (2012)[PubMed]
- Tsuchiya, H., Tanaka, K. & Saeki, Y.: The parallel reaction monitoring method contributes to a highly sensitive polyubiquitin chain quantification. Biochem. Biophys. Res. Commun., 436, 223-229 (2013)[PubMed]
- Xu, P., Duong, D. M., Seyfried, N. T. et al.: Quantitative proteomics reveals the function of unconventional ubiquitin chains in proteasomal degradation. Cell, 137, 133-145 (2009)[PubMed]
- Kim, W., Bennett, E. J., Huttlin, E. L. et al.: Systematic and quantitative assessment of the ubiquitin-modified proteome. Mol. Cell, 44, 325-340 (2011)[PubMed]
- Bodnar, N. O. & Rapoport, T. A.: Molecular mechanism of substrate processing by the Cdc48 ATPase complex. Cell, 169, 722-735 e9 (2017)[PubMed]
- Blythe, E. E., Olson, K. C., Chau, V. et al.: Ubiquitin- and ATP-dependent unfoldase activity of P97/VCP・NPLOC4・UFD1L is enhanced by a mutation that causes multisystem proteinopathy. Proc. Natl. Acad. Sci. USA, 114, E4380-E4388 (2017)[PubMed]
- Zhang, X., Smits, A. H., van Tilburg, G. B. et al.: An interaction landscape of ubiquitin signaling. Mol. Cell, 65, 941-955.e8 (2017)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
東京都医学総合研究所 研究員.
大竹 史明(Fumiaki Ohtake)
東京都医学総合研究所 主席研究員.
田中 啓二(Keiji Tanaka)
東京都医学総合研究所 所長.
佐伯 泰(Yasushi Saeki)
東京都医学総合研究所 副参事研究員.
研究室URL:http://www.igakuken.or.jp/pro-meta/
© 2017 土屋 光・大竹史明・田中啓二・佐伯 泰 Licensed under CC 表示 2.1 日本