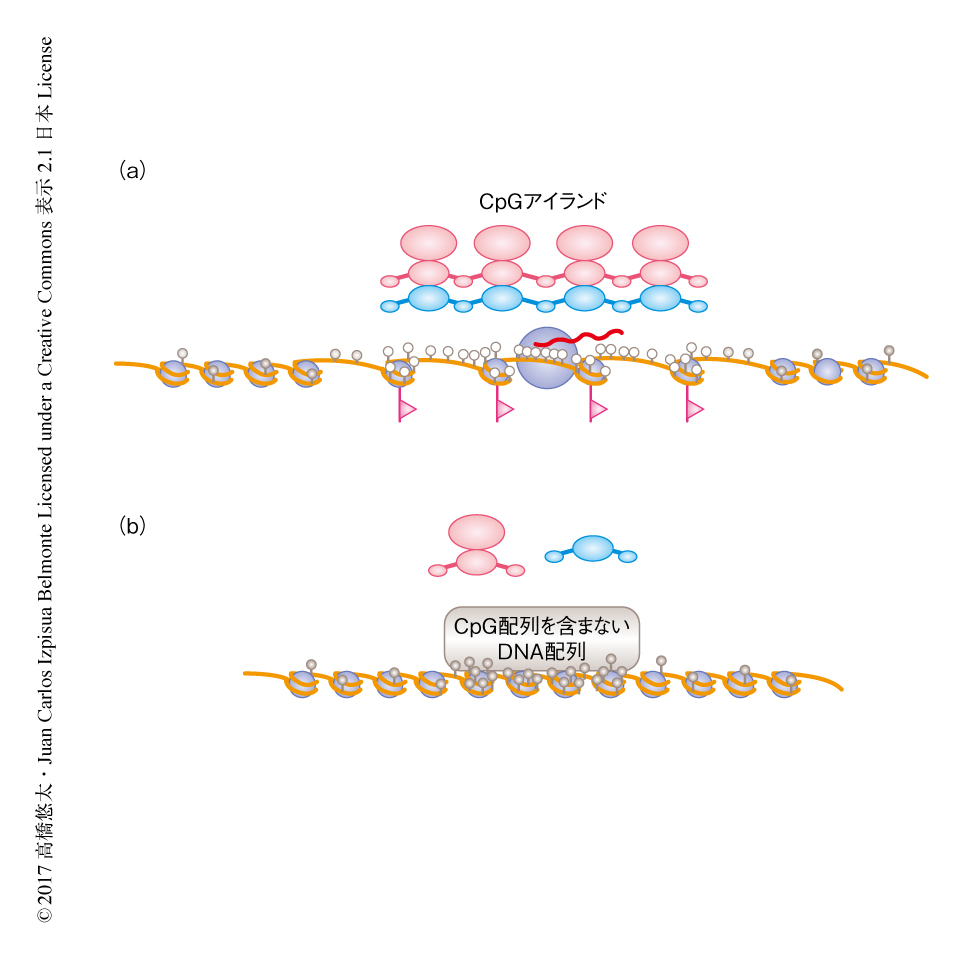
多能性幹細胞においてCpGアイランドにCpG配列を含まないDNA配列を挿入すると新規のDNAメチル化がひき起される
高橋悠太・Juan Carlos Izpisua Belmonte
(米国Salk Institute for Biological Studies,Gene Expression Laboratory)
email:高橋悠太
DOI: 10.7875/first.author.2017.045
Integration of CpG-free DNA induces de novo methylation of CpG islands in pluripotent stem cells.
Yuta Takahashi, Jun Wu, Keiichiro Suzuki, Paloma Martinez-Redondo, Mo Li, Hsin-Kai Liao, Min-Zu Wu, Reyna Hernández-Benítez, Tomoaki Hishida, Maxim Nikolaievich Shokhirev, Concepcion Rodriguez Esteban, Ignacio Sancho-Martinez, Juan Carlos Izpisua Belmonte
Science, 356, 503-508 (2017)
高度にDNAメチル化されている哺乳類のゲノムにおいてもCpGアイランドは非メチル化状態に維持されているが,その機構については不明な部分が多い.筆者らは,この研究において,多能性幹細胞においてCpGアイランドにCpG配列を含まないDNA配列を挿入することにより,その標的となるCpGアイランドの全体に新規のDNAメチル化がひき起こされることを見い出した.CpGアイランドのDNAメチル化は,CpG配列を含まないDNA配列の除去,長期にわたる培養,細胞の分化をへたのちにも安定に維持された.その方法を用い,DNAのミスマッチ修復に関与するMLH1遺伝子のCpGアイランドを標的とすることにより,がんに関係するエピゲノム変異をもつ多能性幹細胞株が樹立された.また,Angelman症候群の患者に特異的なiPS細胞においてインプリンティング異常が修復された.さらに,CpG配列を含まないDNA配列の挿入がCpGアイランドに新規のDNAメチル化をひき起こすしくみが明らかにされた.この研究は,生物の発生やヒトの疾患をより深く理解するために有用なエピゲノム編集技術の基盤になりうる.
近年の全ゲノムDNAメチル化解析により,哺乳類のゲノムにおいてCpG配列のシトシンの多くがメチル化されていることが明らかにされた1).その高メチル化状態は,発生の初期の胚盤胞期から着床までのあいだにDNAメチル化酵素DNMT3AおよびDNMT3Bにより確立される2,3).一方で,CpG配列が高い頻度で出現するCpGアイランドは,初期胚において新規のDNAメチル化をうけず,そののちも非メチル化状態に維持される2,4).CpGアイランドは遺伝子プロモーターの多くに存在し,そのCpGリッチな配列に依存して確立される保護機構により,新規のDNAメチル化をうけずに非メチル化状態に維持されると考えられている5).その保護機構においてはCXXCドメインをもつCXXCタンパク質が重要な役割をはたす.CXXCタンパク質はDNAメチル化されていないCpG配列にリクルートされるが,DNAメチル化されたCpG配列には結合できない.たとえば,CXXCドメインをもつCFP1はCpGアイランドに局在し,ヒストンH3の4番目のLysをメチル化するSet1複合体をCpGアイランドにリクルートする6).ヒストンH3の4番目のLysのトリメチル化は転写活性を示すマークであり,DNAメチル化に対し拮抗作用をもつ.実際に,遺伝子プロモーターをもたないCpGリッチなDNA配列のゲノムへの挿入はヒストンH3の4番目のLysのトリメチル化を新規にひき起こす7).しかし,同時に,CpGリッチなDNA配列のみでは安定した非メチル化状態を維持できないことも報告されている.それらにくわえ,転写活性や転写因子の結合もCpGアイランドの非メチル化状態の維持に寄与することが知られている8,9).しかしながら,これまで,CpGリッチなDNA配列に依存して確立される保護機構および転写により,どのようにCpGアイランドが新規のDNAメチル化をうけずに非メチル化状態が維持されるのかは解明されていなかった.
CpGリッチなDNA配列の阻害がCpGアイランドの非メチル化状態に対しどのような影響をおよぼすのかを検討するため,ヒトのES細胞において,DNAのミスマッチ修復に関与するMLH1遺伝子のプロモーターから第1イントロンに存在するCpGアイランドを標的とし,相同組換えを介してCpG配列を含まないDNA配列を挿入した.通常,MLH1遺伝子のCpGアイランドは非メチル化状態を維持しているが,エピゲノム変異とよばれる新規のDNAメチル化をうける場合があり,大腸における発がんに関与するといわれている.MLH1遺伝子の片方のアレルのCpGアイランドにCpG配列を含まないDNA配列を挿入しDNAメチル化の状態を解析したところ,CpG配列を含まないDNA配列が挿入されたCpGアイランドの全域において新規のDNAメチル化が観察された(図1).CpGアイランドのDNAメチル化は,Cre-loxP系を用いたCpG配列を含まないDNA配列の除去,および,それにつづく長期にわたる培養ののちにも安定に維持された.細胞におけるMLH1 mRNA量およびMLH1タンパク質量は半減し,新規のDNAメチル化によりMLH1遺伝子がサイレンシングされることが確認された.さらに,HSP90AB1遺伝子といったハウスキーピング遺伝子のCpGアイランドにおいても,CpG配列を含まないDNA配列の挿入により新規のDNAメチル化が観察された.
ES細胞とは異なり,線維芽細胞などの分化した細胞においてCpG配列を含まないDNA配列をMLH1遺伝子のCpGアイランドに挿入した場合には新規のDNAメチル化は観察されなかった.これは,分化した細胞におけるDNAメチル化酵素DNMT3Bの発現量の低さ,および,ゲノムに対する新規のDNAメチル化活性の低さに起因するのかもしれない.
Angelman症候群は,母性アレルのみから発現するユビキチンリガーゼUBE3Aがニューロンにおいて欠損することによりひき起こされる10).Angelman症候群の責任領域である第15染色体q11-q13においてSNURF遺伝子にCpGアイランドが存在し,健常人においては,母性アレルにおいて高メチル化状態,父性アレルにおいて非メチル化状態という,アレルに依存的なDNAメチル化のパターンを示す.アレルに依存的なDNAメチル化はSNURF遺伝子の下流にコードされるUBE3AアンチセンスRNAの発現を制御し,UBE3AアンチセンスRNAは同じアレルに存在するUBE3A遺伝子の発現を抑制する.つまり,SNURF遺伝子のCpGアイランドがDNAメチル化されていない父性アレルにおいてはUBE3AアンチセンスRNAが発現しUBE3Aの発現が抑制される.一方,母性アレルにおいてはDNAメチル化によりUBE3AアンチセンスRNAが発現せずUBE3Aが発現する.Angelman症候群の一部の患者は父親から両方の第15染色体を受け継いでおり,両方のアレルのSNURF遺伝子のCpGアイランドが非メチル化状態にある.それにより,ニューロンにおいてUBE3Aが欠損しAngelman症候群がひき起こされる.
父親から両方の第15染色体を受け継いだ型のAngelman症候群の患者の細胞からiPS細胞を樹立し,両方のアレルのSNURF遺伝子のCpGアイランドが非メチル化状態であることを確認した.Angelman症候群の患者に由来するiPS細胞において片方のアレルのSNURF遺伝子のCpGアイランドに相同組換えを介してCpG配列を含まないDNA配列を挿入しDNAメチル化の状態を解析したところ,野生型のiPS細胞と同じDNAメチル化のパターンを示し,新規のDNAメチル化はCpG配列を含まないDNA配列を除去したのちにも安定に維持された.それにともない,SNURF遺伝子のCpGアイランドにおけるCFP1の結合量およびヒストンH3の4番目のLysのトリメチル化の状態も野生型のiPS細胞と同じ程度までに回復し維持された.さらに,CpG配列を含まないDNA配列の挿入により修復されたSNURF遺伝子のCpGアイランドのDNAメチル化の状態は,iPS細胞をニューロンへと分化させたのちにも安定に維持された.分化したニューロンにおいて,UBE3AアンチセンスRNAの発現は新規のDNAメチル化により阻害されており,UBE3Aのタンパク質量は野生型のニューロンと同じ程度にまで回復していた.
CpG配列を含まないDNA配列の挿入にあたり,DNA二本鎖切断や相同組換えそれ自体は新規のDNAメチル化をひき起こさないことが確認された.また,用いたCpG配列を含まないDNA配列を断片化し,それぞれの断片をCpGアイランドに挿入したところ,1 kb以上の長さの複数の断片が新規のDNAメチル化をひき起こした.さらに,そのうちのひとつの断片に新たに10個のCpG配列を付加してCpGアイランドに挿入した場合,DNAメチル化はひき起こされなかった.つまり,新規のDNAメチル化をひき起こすには挿入されるDNA配列がCpG配列を含まないことが必須であった.
CpGリッチなDNA配列に依存して確立される保護機構のほか,転写および転写因子の結合がCpGアイランドを非メチル化状態に維持するために重要とされている.Angelman症候群の患者から得たiPS細胞において,転写因子の結合部位を多く含むSNURF遺伝子のプロモーター領域を除去したうえで,SNURF遺伝子のCpGアイランドにおけるDNAメチル化の状態を解析した.その結果,転写の阻害のみでは新規のDNAメチル化はひき起こされないことが示された.CpG配列を含まないDNA配列をSNURF遺伝子の転写開始点から530 bpほど下流に挿入してDNAメチル化の状態を解析した.転写開始点の周辺にCpG配列を含まないDNA配列を挿入した場合にはCpGアイランドの全域が新規のDNAメチル化をうけるが,転写開始点から離れた下流の領域に挿入した場合,プロモーター領域や転写開始点を含む上流の領域において新規のDNAメチル化は観察されなかった.この結果から,CpGリッチなDNA配列に依存して確立される保護機構の分断のみではCpGアイランドの全域に新規のDNAメチル化はひき起こされないことが示された.転写の阻害およびCpGリッチなDNA配列に依存して確立される保護機構の分断の両方により新規のDNAメチル化がひき起こされるかどうか検討した.SNURF遺伝子のプロモーターが削除されたアレルにおいて転写開始点から離れた下流の領域にCpG配列を含まないDNA配列を挿入したところ,CpGアイランドの全域において新規のDNAメチル化がひき起された.これらの結果から,転写の阻害,および,CpGリッチなDNA配列に依存して確立される保護機構の分断が新規のDNAメチル化をひき起こすのに必要十分であることが明らかにされた.
筆者らは,この研究において,多能性幹細胞においてCpGアイランドへのCpG配列を含まないDNA配列の挿入により,標的となるCpGアイランドの全体が新規にDNAメチル化されることを見い出した.さらに,その新規のDNAメチル化およびそれにともなうエピジェネティックなマークはCpG配列を含まないDNA配列の除去,長期にわたる培養,細胞の分化をへたのちにも安定に維持された.この手法を用いて,がんに関係するエピゲノム変異をもつ多能性幹細胞株が樹立された.また,Angelman症候群の患者に特異的なiPS細胞においてインプリンティング異常を修復することに成功し,このiPS細胞から分化させたニューロンにおいても原因遺伝子であるUBE3A遺伝子の発現は回復していた.さらに,CpG配列を含まないDNA配列の挿入が転写の阻害およびCpGリッチなDNA配列に依存して確立される保護機構の分断を起こし,新規のDNAメチル化をひき起こすことを見い出した.この研究は,DNAメチル化およびエピジェネティクスにより制御されるさまざまな生命現象を研究するうえで重要なエピゲノム編集技術の基盤になりうる.
略歴:2011年 筑波大学大学院生命環境科学研究科博士課程 修了,2012年 米国Salk Institute for Biological StudiesにてResearch Associate,2014年より筑波大学生命領域学際研究センター 国際テニュアトラック助教.
研究テーマ:幹細胞とエピゲノム.
Juan Carlos Izpisua Belmonte
米国Salk Institute for Biological StudiesにてProfessor.
研究室URL:http://www.salk.edu/labs/belmonte/
© 2017 高橋悠太・Juan Carlos Izpisua Belmonte Licensed under CC 表示 2.1 日本
(米国Salk Institute for Biological Studies,Gene Expression Laboratory)
email:高橋悠太
DOI: 10.7875/first.author.2017.045
Integration of CpG-free DNA induces de novo methylation of CpG islands in pluripotent stem cells.
Yuta Takahashi, Jun Wu, Keiichiro Suzuki, Paloma Martinez-Redondo, Mo Li, Hsin-Kai Liao, Min-Zu Wu, Reyna Hernández-Benítez, Tomoaki Hishida, Maxim Nikolaievich Shokhirev, Concepcion Rodriguez Esteban, Ignacio Sancho-Martinez, Juan Carlos Izpisua Belmonte
Science, 356, 503-508 (2017)
要 約
高度にDNAメチル化されている哺乳類のゲノムにおいてもCpGアイランドは非メチル化状態に維持されているが,その機構については不明な部分が多い.筆者らは,この研究において,多能性幹細胞においてCpGアイランドにCpG配列を含まないDNA配列を挿入することにより,その標的となるCpGアイランドの全体に新規のDNAメチル化がひき起こされることを見い出した.CpGアイランドのDNAメチル化は,CpG配列を含まないDNA配列の除去,長期にわたる培養,細胞の分化をへたのちにも安定に維持された.その方法を用い,DNAのミスマッチ修復に関与するMLH1遺伝子のCpGアイランドを標的とすることにより,がんに関係するエピゲノム変異をもつ多能性幹細胞株が樹立された.また,Angelman症候群の患者に特異的なiPS細胞においてインプリンティング異常が修復された.さらに,CpG配列を含まないDNA配列の挿入がCpGアイランドに新規のDNAメチル化をひき起こすしくみが明らかにされた.この研究は,生物の発生やヒトの疾患をより深く理解するために有用なエピゲノム編集技術の基盤になりうる.
はじめに
近年の全ゲノムDNAメチル化解析により,哺乳類のゲノムにおいてCpG配列のシトシンの多くがメチル化されていることが明らかにされた1).その高メチル化状態は,発生の初期の胚盤胞期から着床までのあいだにDNAメチル化酵素DNMT3AおよびDNMT3Bにより確立される2,3).一方で,CpG配列が高い頻度で出現するCpGアイランドは,初期胚において新規のDNAメチル化をうけず,そののちも非メチル化状態に維持される2,4).CpGアイランドは遺伝子プロモーターの多くに存在し,そのCpGリッチな配列に依存して確立される保護機構により,新規のDNAメチル化をうけずに非メチル化状態に維持されると考えられている5).その保護機構においてはCXXCドメインをもつCXXCタンパク質が重要な役割をはたす.CXXCタンパク質はDNAメチル化されていないCpG配列にリクルートされるが,DNAメチル化されたCpG配列には結合できない.たとえば,CXXCドメインをもつCFP1はCpGアイランドに局在し,ヒストンH3の4番目のLysをメチル化するSet1複合体をCpGアイランドにリクルートする6).ヒストンH3の4番目のLysのトリメチル化は転写活性を示すマークであり,DNAメチル化に対し拮抗作用をもつ.実際に,遺伝子プロモーターをもたないCpGリッチなDNA配列のゲノムへの挿入はヒストンH3の4番目のLysのトリメチル化を新規にひき起こす7).しかし,同時に,CpGリッチなDNA配列のみでは安定した非メチル化状態を維持できないことも報告されている.それらにくわえ,転写活性や転写因子の結合もCpGアイランドの非メチル化状態の維持に寄与することが知られている8,9).しかしながら,これまで,CpGリッチなDNA配列に依存して確立される保護機構および転写により,どのようにCpGアイランドが新規のDNAメチル化をうけずに非メチル化状態が維持されるのかは解明されていなかった.
1.ヒトのES細胞においてCpG配列を含まないDNA配列の挿入はCpGアイランドに新規のDNAメチル化をひき起こす
CpGリッチなDNA配列の阻害がCpGアイランドの非メチル化状態に対しどのような影響をおよぼすのかを検討するため,ヒトのES細胞において,DNAのミスマッチ修復に関与するMLH1遺伝子のプロモーターから第1イントロンに存在するCpGアイランドを標的とし,相同組換えを介してCpG配列を含まないDNA配列を挿入した.通常,MLH1遺伝子のCpGアイランドは非メチル化状態を維持しているが,エピゲノム変異とよばれる新規のDNAメチル化をうける場合があり,大腸における発がんに関与するといわれている.MLH1遺伝子の片方のアレルのCpGアイランドにCpG配列を含まないDNA配列を挿入しDNAメチル化の状態を解析したところ,CpG配列を含まないDNA配列が挿入されたCpGアイランドの全域において新規のDNAメチル化が観察された(図1).CpGアイランドのDNAメチル化は,Cre-loxP系を用いたCpG配列を含まないDNA配列の除去,および,それにつづく長期にわたる培養ののちにも安定に維持された.細胞におけるMLH1 mRNA量およびMLH1タンパク質量は半減し,新規のDNAメチル化によりMLH1遺伝子がサイレンシングされることが確認された.さらに,HSP90AB1遺伝子といったハウスキーピング遺伝子のCpGアイランドにおいても,CpG配列を含まないDNA配列の挿入により新規のDNAメチル化が観察された.
ES細胞とは異なり,線維芽細胞などの分化した細胞においてCpG配列を含まないDNA配列をMLH1遺伝子のCpGアイランドに挿入した場合には新規のDNAメチル化は観察されなかった.これは,分化した細胞におけるDNAメチル化酵素DNMT3Bの発現量の低さ,および,ゲノムに対する新規のDNAメチル化活性の低さに起因するのかもしれない.
2.Angelman症候群におけるインプリンティング異常の修復
Angelman症候群は,母性アレルのみから発現するユビキチンリガーゼUBE3Aがニューロンにおいて欠損することによりひき起こされる10).Angelman症候群の責任領域である第15染色体q11-q13においてSNURF遺伝子にCpGアイランドが存在し,健常人においては,母性アレルにおいて高メチル化状態,父性アレルにおいて非メチル化状態という,アレルに依存的なDNAメチル化のパターンを示す.アレルに依存的なDNAメチル化はSNURF遺伝子の下流にコードされるUBE3AアンチセンスRNAの発現を制御し,UBE3AアンチセンスRNAは同じアレルに存在するUBE3A遺伝子の発現を抑制する.つまり,SNURF遺伝子のCpGアイランドがDNAメチル化されていない父性アレルにおいてはUBE3AアンチセンスRNAが発現しUBE3Aの発現が抑制される.一方,母性アレルにおいてはDNAメチル化によりUBE3AアンチセンスRNAが発現せずUBE3Aが発現する.Angelman症候群の一部の患者は父親から両方の第15染色体を受け継いでおり,両方のアレルのSNURF遺伝子のCpGアイランドが非メチル化状態にある.それにより,ニューロンにおいてUBE3Aが欠損しAngelman症候群がひき起こされる.
父親から両方の第15染色体を受け継いだ型のAngelman症候群の患者の細胞からiPS細胞を樹立し,両方のアレルのSNURF遺伝子のCpGアイランドが非メチル化状態であることを確認した.Angelman症候群の患者に由来するiPS細胞において片方のアレルのSNURF遺伝子のCpGアイランドに相同組換えを介してCpG配列を含まないDNA配列を挿入しDNAメチル化の状態を解析したところ,野生型のiPS細胞と同じDNAメチル化のパターンを示し,新規のDNAメチル化はCpG配列を含まないDNA配列を除去したのちにも安定に維持された.それにともない,SNURF遺伝子のCpGアイランドにおけるCFP1の結合量およびヒストンH3の4番目のLysのトリメチル化の状態も野生型のiPS細胞と同じ程度までに回復し維持された.さらに,CpG配列を含まないDNA配列の挿入により修復されたSNURF遺伝子のCpGアイランドのDNAメチル化の状態は,iPS細胞をニューロンへと分化させたのちにも安定に維持された.分化したニューロンにおいて,UBE3AアンチセンスRNAの発現は新規のDNAメチル化により阻害されており,UBE3Aのタンパク質量は野生型のニューロンと同じ程度にまで回復していた.
3.新規のDNAメチル化をひき起こすには挿入されるDNA配列がCpG配列を含まないことが必須である
CpG配列を含まないDNA配列の挿入にあたり,DNA二本鎖切断や相同組換えそれ自体は新規のDNAメチル化をひき起こさないことが確認された.また,用いたCpG配列を含まないDNA配列を断片化し,それぞれの断片をCpGアイランドに挿入したところ,1 kb以上の長さの複数の断片が新規のDNAメチル化をひき起こした.さらに,そのうちのひとつの断片に新たに10個のCpG配列を付加してCpGアイランドに挿入した場合,DNAメチル化はひき起こされなかった.つまり,新規のDNAメチル化をひき起こすには挿入されるDNA配列がCpG配列を含まないことが必須であった.
4.転写の阻害および保護機構の分断の両方が新規のDNAメチル化に必要である
CpGリッチなDNA配列に依存して確立される保護機構のほか,転写および転写因子の結合がCpGアイランドを非メチル化状態に維持するために重要とされている.Angelman症候群の患者から得たiPS細胞において,転写因子の結合部位を多く含むSNURF遺伝子のプロモーター領域を除去したうえで,SNURF遺伝子のCpGアイランドにおけるDNAメチル化の状態を解析した.その結果,転写の阻害のみでは新規のDNAメチル化はひき起こされないことが示された.CpG配列を含まないDNA配列をSNURF遺伝子の転写開始点から530 bpほど下流に挿入してDNAメチル化の状態を解析した.転写開始点の周辺にCpG配列を含まないDNA配列を挿入した場合にはCpGアイランドの全域が新規のDNAメチル化をうけるが,転写開始点から離れた下流の領域に挿入した場合,プロモーター領域や転写開始点を含む上流の領域において新規のDNAメチル化は観察されなかった.この結果から,CpGリッチなDNA配列に依存して確立される保護機構の分断のみではCpGアイランドの全域に新規のDNAメチル化はひき起こされないことが示された.転写の阻害およびCpGリッチなDNA配列に依存して確立される保護機構の分断の両方により新規のDNAメチル化がひき起こされるかどうか検討した.SNURF遺伝子のプロモーターが削除されたアレルにおいて転写開始点から離れた下流の領域にCpG配列を含まないDNA配列を挿入したところ,CpGアイランドの全域において新規のDNAメチル化がひき起された.これらの結果から,転写の阻害,および,CpGリッチなDNA配列に依存して確立される保護機構の分断が新規のDNAメチル化をひき起こすのに必要十分であることが明らかにされた.
おわりに
筆者らは,この研究において,多能性幹細胞においてCpGアイランドへのCpG配列を含まないDNA配列の挿入により,標的となるCpGアイランドの全体が新規にDNAメチル化されることを見い出した.さらに,その新規のDNAメチル化およびそれにともなうエピジェネティックなマークはCpG配列を含まないDNA配列の除去,長期にわたる培養,細胞の分化をへたのちにも安定に維持された.この手法を用いて,がんに関係するエピゲノム変異をもつ多能性幹細胞株が樹立された.また,Angelman症候群の患者に特異的なiPS細胞においてインプリンティング異常を修復することに成功し,このiPS細胞から分化させたニューロンにおいても原因遺伝子であるUBE3A遺伝子の発現は回復していた.さらに,CpG配列を含まないDNA配列の挿入が転写の阻害およびCpGリッチなDNA配列に依存して確立される保護機構の分断を起こし,新規のDNAメチル化をひき起こすことを見い出した.この研究は,DNAメチル化およびエピジェネティクスにより制御されるさまざまな生命現象を研究するうえで重要なエピゲノム編集技術の基盤になりうる.
文 献
- Lister, R., Mukamel, E. A., Nery, J. R. et al.: Global epigenomic reconfiguration during mammalian brain development. Science, 341, 1237905 (2013)[PubMed]
- Guo, H., Zhu, P., Yan, L. et al.: The DNA methylation landscape of human early embryos. Nature, 511, 606-610 (2014)[PubMed]
- Okano, M., Bell, D. W., Haber, D. A. et al.: DNA methyltransferases Dnmt3a and Dnmt3b are essential for de novo methylation and mammalian development. Cell, 99, 247-257 (1999)[PubMed]
- Xie, W., Schultz, M. D., Lister, R. et al.: Epigenomic analysis of multilineage differentiation of human embryonic stem cells. Cell, 153, 1134-1148 (2013)[PubMed]
- Deaton, A. M. & Bird, A.: CpG islands and the regulation of transcription. Genes Dev., 25, 1010-1022 (2011)[PubMed]
- Clouaire, T., Webb, S., Skene, P. et al.: Cfp1 integrates both CpG content and gene activity for accurate H3K4me3 deposition in embryonic stem cells. Genes Dev., 26, 1714-1728 (2012)[PubMed]
- Thomson, J. P., Skene, P. J., Selfridge, J. et al.: CpG islands influence chromatin structure via the CpG-binding protein Cfp1. Nature, 464, 1082-1086 (2010)[PubMed]
- Brandeis, M., Frank, D., Keshet, I. et al.: Sp1 elements protect a CpG island from de novo methylation. Nature, 371, 435-438 (1994)[PubMed]
- Ginno, P. A., Lott, P. L., Christensen, H. C. et al.: R-loop formation is a distinctive characteristic of unmethylated human CpG island promoters. Mol. Cell, 45, 814-825 (2012)[PubMed]
- Lossie, A. C., Whitney, M. M., Amidon, D. et al.: Distinct phenotypes distinguish the molecular classes of Angelman syndrome. J. Med. Genet., 38, 834-845 (2001)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2011年 筑波大学大学院生命環境科学研究科博士課程 修了,2012年 米国Salk Institute for Biological StudiesにてResearch Associate,2014年より筑波大学生命領域学際研究センター 国際テニュアトラック助教.
研究テーマ:幹細胞とエピゲノム.
Juan Carlos Izpisua Belmonte
米国Salk Institute for Biological StudiesにてProfessor.
研究室URL:http://www.salk.edu/labs/belmonte/
© 2017 高橋悠太・Juan Carlos Izpisua Belmonte Licensed under CC 表示 2.1 日本