lncRNAから翻訳されるペプチドSPARはmTOR複合体1および筋の再生を制御する
松本有樹修・Pier Paolo Pandolfi
(米国Beth Israel Deaconess Medical Center,Department of Medicine)
email:松本有樹修
DOI: 10.7875/first.author.2017.003
mTORC1 and muscle regeneration are regulated by the LINC00961-encoded SPAR polypeptide.
Akinobu Matsumoto, Alessandra Pasut, Masaki Matsumoto, Riu Yamashita, Jacqueline Fung, Emanuele Monteleone, Alan Saghatelian, Keiichi I. Nakayama, John G. Clohessy, Pier Paolo Pandolfi
Nature, 541, 228-232 (2017)
lncRNAからタンパク質は翻訳されないと考えられているが,ショウジョウバエやゼブラフィッシュにおいて一部のlncRNAから機能性のペプチドが翻訳されることが明らかにされた(2017年1月22日 著者からの依頼により一部修正).しかし,哺乳類においてこれら機能性のペプチドはほとんど解析されていない.そこで,筆者らは,質量分析法を用いてlncRNAから翻訳されるペプチドを同定した.これらのうち,LINC00961から翻訳されるペプチドSPARはヒトやマウスなど哺乳類にのみ保存され,リソソームに局在してv-ATPase複合体と結合することによりアミノ酸に依存的なmTOR複合体1の活性化を抑制した.Sparノックアウトマウスを用いた解析により,筋が障害されるとSparの発現が低下し,mTOR複合体1の活性化が亢進され筋の再生が促進されることが明らかにされた.lncRNAの数は高等生物になるにつれ増加し,組織特異性も高いことから,これらのlncRNAと考えられていたRNAから翻訳されるペプチドは,進化とともに必要になってきた組織特異的なシステムの制御のため機能するものと考えられる.
2000年代からはじまったトランスクリプトーム解析により,ゲノムの約70~90%が転写されていることが明らかにされた.これらの転写産物にはlncRNA(long non-coding RNA,長鎖非コードRNA)という200塩基以上のタンパク質をコードしないmRNA様のRNAが大量に存在した.近年の解析により,核に存在するlncRNAがクロマチン修飾酵素を特定の遺伝子領域に輸送するといった新たな機能が明らかにされた.しかし,これらの機能は膨大に存在するlncRNAのごく一部にしかあてはまらず,多くのlncRNAの機能はいまだ不明である.
これらlncRNAは本当にタンパク質をコードしないのだろうか? 膨大に存在するlncRNAの多くは計算式によりnon-coding RNAとして分類されただけである.多くのlncRNAにおいて100アミノ酸残基以下の小さなORFが大量に推測されたが,これらは非常に小さく,おそらく翻訳されていないであろうとしてnon-coding RNAに分類された.だが,ショウジョウバエやゼブラフィッシュにおいてlncRNAと考えられていた一部のRNAからはペプチドが翻訳されており,それらが機能をもつことが明らかにされた1-3)(2017年1月22日 著者からの依頼により一部修正).しかし,哺乳類などの高等生物においてこれらペプチドの解析はほとんど進んでいなかった.そこで,筆者らは,ヒトにおいてlncRNAから翻訳される新規のペプチドを同定しそれらの機能を解析した.
質量分析法を用いてlncRNAから翻訳されるペプチドを同定する手法を確立した.この手法により新規のペプチドの同定に成功し,それらのうちLINC00961から翻訳される90アミノ酸残基のペプチドに焦点をあてた.LINC00961は通常のmRNAと同様にポリA鎖をもちリボソームに局在したことから,翻訳されている可能性が示唆された.LINC00961の配列からは3つのORFが推測された.それぞれのORFのC末端側にFLAG配列をノックインしたベクターを作製しウェスタンブロット法により発現を確認したところ,質量分析法により同定されたペプチドに対応する90アミノ酸残基のORFにFLAG配列をノックインした場合には発現が検出されたが,それ以外のORFの発現は検出されなかった.このことから,90アミノ酸残基のORFは翻訳されており,それ以外のORFは翻訳されていないことが示された.詳細な解析により,LINC00961からは90アミノ酸残基のペプチドのほか,N末端側の15アミノ酸残基の欠失した75アミノ酸残基のペプチドも翻訳されていることがわかった.どちらのペプチドもN末端側の領域に膜貫通ドメインをもち,リソソームに局在した.トポロジー解析によりペプチドの大部分をしめるC末端側の領域が細胞質の側に露出することがわかった.さらに,このペプチドに対する抗体を作製しウェスタンブロット法により内在性の発現を確認した.LINC00961をノックダウンすることによりこのペプチドの発現は低下したことから,LINC00961から翻訳されたペプチドであることが確認された.
LINC00961から翻訳されるペプチドの機能を明らかにするため,FLAG配列を付加したペプチドを過剰に発現し,免疫沈降法により結合タンパク質を同定した.その結果,このペプチドはv-ATPase複合体を構成するサブユニットの多くと結合することがわかった.また,このペプチドに対する抗体を用いて,内在性のペプチドもv-ATPase複合体と結合することが確認された.さらに,in vitroにおいて,このペプチドとv-ATPase複合体を構成するサブユニットであるATP6V0A1およびATP6V0A2とが直接的に結合することが確認された.v-ATPase複合体はプロトンポンプでありリソソームの酸性化に寄与することが知られている.そこで,このペプチドがv-ATPase複合体のプロトンポンプ活性に影響するかどうかを検討したが,LINC00961の過剰発現はv-ATPaseのプロトンポンプ活性にはまったく影響しなかった.
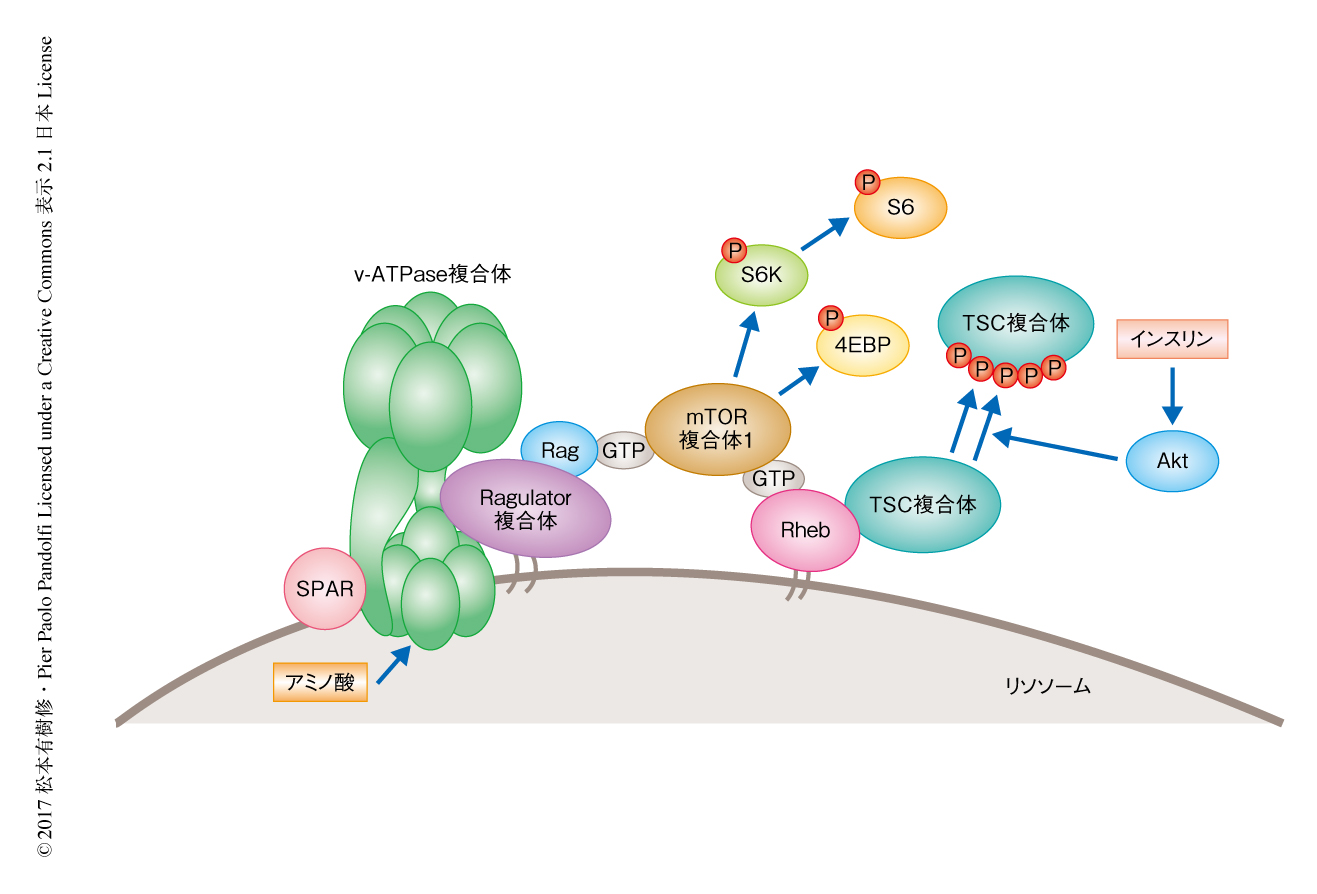
プロトンポンプとしての機能のほか,v-ATPase複合体はリソソームにおいてRagulator複合体と結合することによりアミノ酸に依存的なmTOR複合体1の活性化に寄与することが知られている4,5)(図1).アミノ酸はv-ATPase複合体およびRagulator複合体を活性化し,Ragulator複合体のGEF活性によりRagがGTP型に変換される.これが引き金となり,mTOR複合体1はリソソームへとリクルートされる.また,リソソームにはRhebが局在し,mTOR複合体1はRhebにより活性化される.Rhebはインスリンなどの刺激により活性化されるため,mTOR複合体1の活性化には,アミノ酸シグナルによるmTOR複合体1のリソソームへのリクルート,および,インスリンシグナルによるRhebの活性化の両方が必要である.そこで,LINC00961から翻訳されるペプチドはアミノ酸に依存的なmTOR複合体1の活性化に寄与すると仮定した.LINC00961を過剰に発現すると,アミノ酸に依存的なmTOR複合体1の活性化は劇的に抑制されたが,ペプチドを翻訳しないようにしたLINC00961の変異体を過剰に発現しても同様の表現型は観察されなかった.このことから,LINC00961のRNAとしての機能ではなく,翻訳されたペプチドの機能がこの表現型に寄与することがわかった.また,LINC00961をノックダウンすると,過剰発現とは逆にアミノ酸に依存的なmTOR複合体1の活性化はより亢進された.一方,LINC00961の過剰発現はインスリンに依存的なmTOR複合体1の活性化にはまったく影響しなかった.さらに,インスリンに依存的なAktのリン酸化やEGFに依存的なERKのリン酸化など,mTOR複合体1を経由しないシグナル伝達経路についても検討したが,LINC00961の過剰発現はまったく影響しなかった.以上のことより,LINC00961から翻訳されるペプチドはSPAR(small regulatory polypeptide of amino acids response)と名づけられた.
アミノ酸が枯渇した条件においてはv-ATPase複合体とRagulator複合体とはタイトな超複合体を形成するが,アミノ酸の添加によりこのタイトな構造はゆるみルーズな構造へと変化することが知られている4).この構造の変化が引き金となりRagulator複合体は活性化し,mTOR複合体1はリソソームへとひき寄せられる.そこで,SPARがこの超複合体の制御に関与するかどうか検討したところ,SPARを過剰に発現する細胞においてこのアミノ酸に依存的な超複合体の構造の変化が抑制された.このことより,SPARはv-ATPase複合体と結合することによりアミノ酸に依存的な超複合体の構造の変化を制御し,mTOR複合体1の活性化を抑制すると考えられた(図1).
ヒトのLINC00961にはマウスにホモログが存在する.マウスのSparはATGコドンを1つだけもち,その長さは75アミノ酸残基であり,ヒトの短いSPARと類似していた.アミノ酸配列の相同性は約65%であったが,マウスのSparをヒトの細胞であるHEK293T細胞において過剰に発現したところ同様のmTOR複合体1の抑制が観察されたことから,マウスのSparの機能は進化的に保存されていることがわかった.
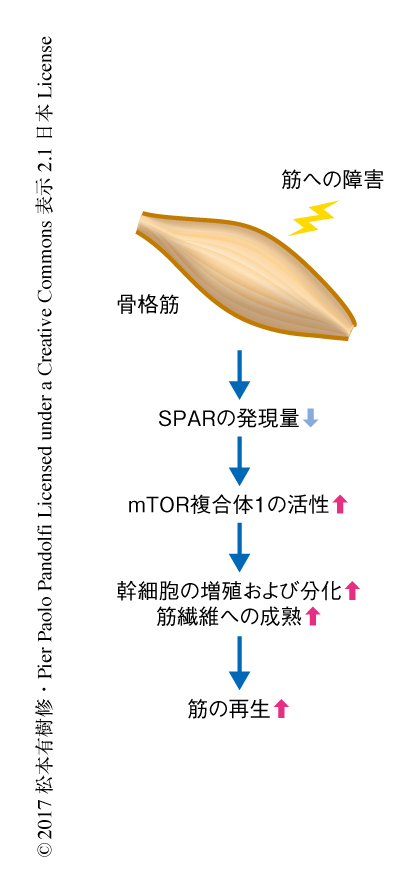
Sparの個体における役割を解析するため,CRISPR-Cas9系を用いてSparノックアウトマウスを作製した.また,遺伝子の全体を欠損させるのではなく開始コドンであるATGのみを欠損させることにより,RNAの発現はそのままでペプチドの発現のみを欠損させた.このSparノックアウトマウスはメンデル比にそって生まれ明らかな異常は示さなかったことから,Sparは発生には重要でないことがわかった.Sparは骨格筋においてとくに高く発現した.通常,骨格筋においてmTOR複合体1の活性は抑制されているが,筋が障害されたのちの筋の再生の過程において急激に活性化されることが知られている6).また,mTOR複合体1の阻害剤であるラパマイシンの投与により筋の再生は劇的に抑制された.筋が障害された直後にSparの発現は劇的に低下し,この発現の低下は筋の再生にともない回復した.そこで,Sparは筋が障害されたのちのmTOR複合体1の活性化を負に制御し筋の再生の過程を制御するのではないかと考えた.
筋が障害されたのちのmTOR複合体1の活性化にアミノ酸が必須であるかどうかを検討するため,筋を障害すると同時にロイシンを欠損した餌を投与した.その結果,対照となる餌を投与したマウスと比べ,筋が障害されたのちのmTOR複合体1の活性化が抑制され筋の再生も遅延した.このことから,筋が障害されたのちのmTOR複合体1の活性化および筋の再生はアミノ酸に依存的であることが示された.野生型のマウスおよびSparノックアウトマウスにおいて同様に解析したところ,Sparノックアウトマウスにおいて筋が障害されたのちのmTOR複合体1の活性化がより増強されており,筋の再生も有意に亢進した.また,Sparノックアウトマウスにおいては筋が障害されたのちの筋幹細胞の増殖および分化,筋繊維への成熟が亢進した.以上の結果より,Sparは筋の再生の過程において発現が低下することによりmTOR複合体1の活性化を増強し筋の再生を促進することがわかった(図2).
今回の解析により,lncRNAから翻訳されたペプチドが同定された.これらのうち,新規のペプチドであるSPARはヒトやマウスなど哺乳類において保存されており,アミノ酸に依存的なmTOR複合体1の活性化を抑制し筋の再生の過程を制御することが明らかにされた.また,この論文のリバイス中に,哺乳類における2つの機能性ペプチドについての論文がそれぞれ報告された7,8).これらのペプチドもlncRNAから翻訳されており,どちらもCa2+ポンプとして知られるSERCAと結合することによりその機能をそれぞれ抑制あるいは活性化した.これらの結果もあわせ,lncRNAと考えられていたRNAから翻訳されたペプチドがさまざまな生命現象において重要であることが示された.
これらペプチドの研究はまだはじまったばかりである.筆者らは質量分析法を用いていくつかの新規のペプチドの同定に成功したが,すべてを同定したとは考えていない.lncRNAは組織特異的に発現することが多く,発現量も少ないことが多いため,これらから翻訳されるペプチドをみのがしている可能性は高い.lncRNAの種類は膨大であることが知られているが,どれくらいの割合のlncRNAからペプチドが翻訳されているかはいまだ不明であり,今後,より効率的なペプチドの同定法の確立が必要である.lncRNAと考えられていたRNAから翻訳されるペプチドはこれまでみのがされてきており,今後,さまざまな疾患への関与が明らかにされると期待される.
略歴:2011年 九州大学大学院医学系学府博士課程 修了,同年 九州大学生体防御医学研究所 学術研究員を経て,2012年より米国Beth Israel Deaconess Medical Center博士研究員,2014年より科学技術振興機構 さきがけ研究員.
研究テーマ:lncRNAから翻訳される新規のペプチドの同定および機能の解析.
関心事:脳神経系.
Pier Paolo Pandolfi
米国Beth Israel Deaconess Medical Center教授.
研究室URL:http://www.bidmc.org/Research/Departments/Medicine/Divisions/Genetics/PandolfiLab.aspx
© 2017 松本有樹修・Pier Paolo Pandolfi Licensed under CC 表示 2.1 日本
(米国Beth Israel Deaconess Medical Center,Department of Medicine)
email:松本有樹修
DOI: 10.7875/first.author.2017.003
mTORC1 and muscle regeneration are regulated by the LINC00961-encoded SPAR polypeptide.
Akinobu Matsumoto, Alessandra Pasut, Masaki Matsumoto, Riu Yamashita, Jacqueline Fung, Emanuele Monteleone, Alan Saghatelian, Keiichi I. Nakayama, John G. Clohessy, Pier Paolo Pandolfi
Nature, 541, 228-232 (2017)
要 約
lncRNAからタンパク質は翻訳されないと考えられているが,ショウジョウバエやゼブラフィッシュにおいて一部のlncRNAから機能性のペプチドが翻訳されることが明らかにされた(2017年1月22日 著者からの依頼により一部修正).しかし,哺乳類においてこれら機能性のペプチドはほとんど解析されていない.そこで,筆者らは,質量分析法を用いてlncRNAから翻訳されるペプチドを同定した.これらのうち,LINC00961から翻訳されるペプチドSPARはヒトやマウスなど哺乳類にのみ保存され,リソソームに局在してv-ATPase複合体と結合することによりアミノ酸に依存的なmTOR複合体1の活性化を抑制した.Sparノックアウトマウスを用いた解析により,筋が障害されるとSparの発現が低下し,mTOR複合体1の活性化が亢進され筋の再生が促進されることが明らかにされた.lncRNAの数は高等生物になるにつれ増加し,組織特異性も高いことから,これらのlncRNAと考えられていたRNAから翻訳されるペプチドは,進化とともに必要になってきた組織特異的なシステムの制御のため機能するものと考えられる.
はじめに
2000年代からはじまったトランスクリプトーム解析により,ゲノムの約70~90%が転写されていることが明らかにされた.これらの転写産物にはlncRNA(long non-coding RNA,長鎖非コードRNA)という200塩基以上のタンパク質をコードしないmRNA様のRNAが大量に存在した.近年の解析により,核に存在するlncRNAがクロマチン修飾酵素を特定の遺伝子領域に輸送するといった新たな機能が明らかにされた.しかし,これらの機能は膨大に存在するlncRNAのごく一部にしかあてはまらず,多くのlncRNAの機能はいまだ不明である.
これらlncRNAは本当にタンパク質をコードしないのだろうか? 膨大に存在するlncRNAの多くは計算式によりnon-coding RNAとして分類されただけである.多くのlncRNAにおいて100アミノ酸残基以下の小さなORFが大量に推測されたが,これらは非常に小さく,おそらく翻訳されていないであろうとしてnon-coding RNAに分類された.だが,ショウジョウバエやゼブラフィッシュにおいてlncRNAと考えられていた一部のRNAからはペプチドが翻訳されており,それらが機能をもつことが明らかにされた1-3)(2017年1月22日 著者からの依頼により一部修正).しかし,哺乳類などの高等生物においてこれらペプチドの解析はほとんど進んでいなかった.そこで,筆者らは,ヒトにおいてlncRNAから翻訳される新規のペプチドを同定しそれらの機能を解析した.
1.lncRNAのひとつLINC00961はリソソームに局在する膜貫通ペプチドに翻訳される
質量分析法を用いてlncRNAから翻訳されるペプチドを同定する手法を確立した.この手法により新規のペプチドの同定に成功し,それらのうちLINC00961から翻訳される90アミノ酸残基のペプチドに焦点をあてた.LINC00961は通常のmRNAと同様にポリA鎖をもちリボソームに局在したことから,翻訳されている可能性が示唆された.LINC00961の配列からは3つのORFが推測された.それぞれのORFのC末端側にFLAG配列をノックインしたベクターを作製しウェスタンブロット法により発現を確認したところ,質量分析法により同定されたペプチドに対応する90アミノ酸残基のORFにFLAG配列をノックインした場合には発現が検出されたが,それ以外のORFの発現は検出されなかった.このことから,90アミノ酸残基のORFは翻訳されており,それ以外のORFは翻訳されていないことが示された.詳細な解析により,LINC00961からは90アミノ酸残基のペプチドのほか,N末端側の15アミノ酸残基の欠失した75アミノ酸残基のペプチドも翻訳されていることがわかった.どちらのペプチドもN末端側の領域に膜貫通ドメインをもち,リソソームに局在した.トポロジー解析によりペプチドの大部分をしめるC末端側の領域が細胞質の側に露出することがわかった.さらに,このペプチドに対する抗体を作製しウェスタンブロット法により内在性の発現を確認した.LINC00961をノックダウンすることによりこのペプチドの発現は低下したことから,LINC00961から翻訳されたペプチドであることが確認された.
2.LINC00961から翻訳されるペプチドSPARはアミノ酸に依存的なmTOR複合体1の活性化を抑制する
LINC00961から翻訳されるペプチドの機能を明らかにするため,FLAG配列を付加したペプチドを過剰に発現し,免疫沈降法により結合タンパク質を同定した.その結果,このペプチドはv-ATPase複合体を構成するサブユニットの多くと結合することがわかった.また,このペプチドに対する抗体を用いて,内在性のペプチドもv-ATPase複合体と結合することが確認された.さらに,in vitroにおいて,このペプチドとv-ATPase複合体を構成するサブユニットであるATP6V0A1およびATP6V0A2とが直接的に結合することが確認された.v-ATPase複合体はプロトンポンプでありリソソームの酸性化に寄与することが知られている.そこで,このペプチドがv-ATPase複合体のプロトンポンプ活性に影響するかどうかを検討したが,LINC00961の過剰発現はv-ATPaseのプロトンポンプ活性にはまったく影響しなかった.
プロトンポンプとしての機能のほか,v-ATPase複合体はリソソームにおいてRagulator複合体と結合することによりアミノ酸に依存的なmTOR複合体1の活性化に寄与することが知られている4,5)(図1).アミノ酸はv-ATPase複合体およびRagulator複合体を活性化し,Ragulator複合体のGEF活性によりRagがGTP型に変換される.これが引き金となり,mTOR複合体1はリソソームへとリクルートされる.また,リソソームにはRhebが局在し,mTOR複合体1はRhebにより活性化される.Rhebはインスリンなどの刺激により活性化されるため,mTOR複合体1の活性化には,アミノ酸シグナルによるmTOR複合体1のリソソームへのリクルート,および,インスリンシグナルによるRhebの活性化の両方が必要である.そこで,LINC00961から翻訳されるペプチドはアミノ酸に依存的なmTOR複合体1の活性化に寄与すると仮定した.LINC00961を過剰に発現すると,アミノ酸に依存的なmTOR複合体1の活性化は劇的に抑制されたが,ペプチドを翻訳しないようにしたLINC00961の変異体を過剰に発現しても同様の表現型は観察されなかった.このことから,LINC00961のRNAとしての機能ではなく,翻訳されたペプチドの機能がこの表現型に寄与することがわかった.また,LINC00961をノックダウンすると,過剰発現とは逆にアミノ酸に依存的なmTOR複合体1の活性化はより亢進された.一方,LINC00961の過剰発現はインスリンに依存的なmTOR複合体1の活性化にはまったく影響しなかった.さらに,インスリンに依存的なAktのリン酸化やEGFに依存的なERKのリン酸化など,mTOR複合体1を経由しないシグナル伝達経路についても検討したが,LINC00961の過剰発現はまったく影響しなかった.以上のことより,LINC00961から翻訳されるペプチドはSPAR(small regulatory polypeptide of amino acids response)と名づけられた.
アミノ酸が枯渇した条件においてはv-ATPase複合体とRagulator複合体とはタイトな超複合体を形成するが,アミノ酸の添加によりこのタイトな構造はゆるみルーズな構造へと変化することが知られている4).この構造の変化が引き金となりRagulator複合体は活性化し,mTOR複合体1はリソソームへとひき寄せられる.そこで,SPARがこの超複合体の制御に関与するかどうか検討したところ,SPARを過剰に発現する細胞においてこのアミノ酸に依存的な超複合体の構造の変化が抑制された.このことより,SPARはv-ATPase複合体と結合することによりアミノ酸に依存的な超複合体の構造の変化を制御し,mTOR複合体1の活性化を抑制すると考えられた(図1).
3.筋が障害されたのちのSparの発現の低下はmTOR複合体1の活性化を増強し筋の再生を促進する
ヒトのLINC00961にはマウスにホモログが存在する.マウスのSparはATGコドンを1つだけもち,その長さは75アミノ酸残基であり,ヒトの短いSPARと類似していた.アミノ酸配列の相同性は約65%であったが,マウスのSparをヒトの細胞であるHEK293T細胞において過剰に発現したところ同様のmTOR複合体1の抑制が観察されたことから,マウスのSparの機能は進化的に保存されていることがわかった.
Sparの個体における役割を解析するため,CRISPR-Cas9系を用いてSparノックアウトマウスを作製した.また,遺伝子の全体を欠損させるのではなく開始コドンであるATGのみを欠損させることにより,RNAの発現はそのままでペプチドの発現のみを欠損させた.このSparノックアウトマウスはメンデル比にそって生まれ明らかな異常は示さなかったことから,Sparは発生には重要でないことがわかった.Sparは骨格筋においてとくに高く発現した.通常,骨格筋においてmTOR複合体1の活性は抑制されているが,筋が障害されたのちの筋の再生の過程において急激に活性化されることが知られている6).また,mTOR複合体1の阻害剤であるラパマイシンの投与により筋の再生は劇的に抑制された.筋が障害された直後にSparの発現は劇的に低下し,この発現の低下は筋の再生にともない回復した.そこで,Sparは筋が障害されたのちのmTOR複合体1の活性化を負に制御し筋の再生の過程を制御するのではないかと考えた.
筋が障害されたのちのmTOR複合体1の活性化にアミノ酸が必須であるかどうかを検討するため,筋を障害すると同時にロイシンを欠損した餌を投与した.その結果,対照となる餌を投与したマウスと比べ,筋が障害されたのちのmTOR複合体1の活性化が抑制され筋の再生も遅延した.このことから,筋が障害されたのちのmTOR複合体1の活性化および筋の再生はアミノ酸に依存的であることが示された.野生型のマウスおよびSparノックアウトマウスにおいて同様に解析したところ,Sparノックアウトマウスにおいて筋が障害されたのちのmTOR複合体1の活性化がより増強されており,筋の再生も有意に亢進した.また,Sparノックアウトマウスにおいては筋が障害されたのちの筋幹細胞の増殖および分化,筋繊維への成熟が亢進した.以上の結果より,Sparは筋の再生の過程において発現が低下することによりmTOR複合体1の活性化を増強し筋の再生を促進することがわかった(図2).
おわりに
今回の解析により,lncRNAから翻訳されたペプチドが同定された.これらのうち,新規のペプチドであるSPARはヒトやマウスなど哺乳類において保存されており,アミノ酸に依存的なmTOR複合体1の活性化を抑制し筋の再生の過程を制御することが明らかにされた.また,この論文のリバイス中に,哺乳類における2つの機能性ペプチドについての論文がそれぞれ報告された7,8).これらのペプチドもlncRNAから翻訳されており,どちらもCa2+ポンプとして知られるSERCAと結合することによりその機能をそれぞれ抑制あるいは活性化した.これらの結果もあわせ,lncRNAと考えられていたRNAから翻訳されたペプチドがさまざまな生命現象において重要であることが示された.
これらペプチドの研究はまだはじまったばかりである.筆者らは質量分析法を用いていくつかの新規のペプチドの同定に成功したが,すべてを同定したとは考えていない.lncRNAは組織特異的に発現することが多く,発現量も少ないことが多いため,これらから翻訳されるペプチドをみのがしている可能性は高い.lncRNAの種類は膨大であることが知られているが,どれくらいの割合のlncRNAからペプチドが翻訳されているかはいまだ不明であり,今後,より効率的なペプチドの同定法の確立が必要である.lncRNAと考えられていたRNAから翻訳されるペプチドはこれまでみのがされてきており,今後,さまざまな疾患への関与が明らかにされると期待される.
文 献
- Kondo, T., Plaza, S., Zanet, J. et al.: Small peptides switch the transcriptional activity of Shavenbaby during Drosophila embryogenesis. Science, 329, 336-339 (2010)[PubMed]
- Magny, E. G., Pueyo, J. I., Pearl, F. M. et al.: Conserved regulation of cardiac calcium uptake by peptides encoded in small open reading frames. Science, 341, 1116-1120 (2013)[PubMed]
- Pauli, A., Norris, M. L., Valen, E. et al.: Toddler: an embryonic signal that promotes cell movement via Apelin receptors. Science, 343, 1248636 (2014)[PubMed]
- Zoncu, R., Bar-Peled, L., Efeyan, A. et al.: mTORC1 senses lysosomal amino acids through an inside-out mechanism that requires the vacuolar H+-ATPase. Science, 334, 678-683 (2011)[PubMed]
- Bar-Peled, L. & Sabatini, D. M.: Regulation of mTORC1 by amino acids. Trends Cell Biol., 24, 400-406 (2014)[PubMed]
- Ge, Y., Wu, A. L., Warnes, C. et al.: mTOR regulates skeletal muscle regeneration in vivo through kinase-dependent and kinase-independent mechanisms. Am. J. Physiol. Cell Physiol., 297, C1434-C1444 (2009)[PubMed]
- Anderson, D. M., Anderson, K. M., Chang, C. L. et al.: A micropeptide encoded by a putative long noncoding RNA regulates muscle performance. Cell, 160, 595-606 (2015)[PubMed]
- Nelson, B. R., Makarewich, C. A., Anderson, D. M. et al.: A peptide encoded by a transcript annotated as long noncoding RNA enhances SERCA activity in muscle. Science, 351, 271-275 (2016)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2011年 九州大学大学院医学系学府博士課程 修了,同年 九州大学生体防御医学研究所 学術研究員を経て,2012年より米国Beth Israel Deaconess Medical Center博士研究員,2014年より科学技術振興機構 さきがけ研究員.
研究テーマ:lncRNAから翻訳される新規のペプチドの同定および機能の解析.
関心事:脳神経系.
Pier Paolo Pandolfi
米国Beth Israel Deaconess Medical Center教授.
研究室URL:http://www.bidmc.org/Research/Departments/Medicine/Divisions/Genetics/PandolfiLab.aspx
© 2017 松本有樹修・Pier Paolo Pandolfi Licensed under CC 表示 2.1 日本