リボソーム,ArfA,翻訳終結因子RF2,開始tRNA,mRNAからなる複合体のクライオ電子顕微鏡による構造解析
栗田大輔・姫野俵太
(弘前大学農学生命科学部 分子生命科学科分子生物学研究室)
email:栗田大輔,姫野俵太
DOI: 10.7875/first.author.2016.132
Mechanistic insights into the alternative translation termination by ArfA and RF2.
Chengying Ma, Daisuke Kurita, Ningning Li, Yan Chen, Hyouta Himeno, Ning Gao
Nature, 541, 550-553 (2017)
細胞においてさまざまな理由により翻訳は停滞する.たとえば,リボソームが終止コドンをもたないノンストップmRNAにおいて翻訳をはじめると,終止コドンに到達することができないため翻訳は途中で停滞する.このような翻訳の停滞を解消する機構としてtmRNAによるトランス-トランスレーションが知られていた.しかし,最近になり,翻訳の停滞の解消にはたらく新たなタンパク質としてArfAが発見され,トランス-トランスレーションとは異なるリボソームレスキュー機構の存在が明らかにされた.筆者らは,70Sリボソーム,ArfA,翻訳終結因子RF2,開始tRNA,mRNAからなるノンストップ複合体をクライオ電子顕微鏡により解析し,3.0Åの分解能で構造を決定した.これにより,ArfAのN末端側の領域はリボソーム30Sサブユニットのデコーディング領域とRF2とにはさまれるかたちで結合する一方,ArfAのC末端側の領域はリボソームのmRNA結合部位に位置することが明らかにされた.この結果から,ArfAが空のmRNAエントリーチャネルを認識することにより翻訳の停滞した状態,すなわち,mRNAの3’末端の側において停滞するリボソームを見分けることが示唆された.
原核生物あるいは真核生物をとわず,翻訳の途中でリボソームが停滞することがある.このような異常な事態を解消する機構として,細菌にはtmRNAによるトランス-トランスレーションというリボソームレスキュー機構が存在する.tmRNAが必須である生物もいるが,大腸菌や枯草菌などの多くの細菌においてはtmRNAの欠損が通常の培養条件における生育に大きな影響をおよぼすことはない.このことから,トランス-トランスレーションに代わる機構の存在の可能性が示唆されていた.2010年,分子遺伝学的な手法を用いた研究により,翻訳の停滞の解消にはたらくタンパク質としてArfAの存在が明らかにされた1).ArfAは停滞したリボソームと結合し,翻訳終結因子RF2と協調してペプチジルtRNAを加水分解することによりリボソームの停滞を解消する2,3).通常の翻訳においてRF2は3種類の終止コドンのうちUAAあるいはUGAを認識してリボソームと結合するが,ArfAのかかわるリボソームレスキュー機構において終止コドンの関与は想定されていない.どのようにしてArfAおよびRF2は終止コドンの存在しないmRNAと結合したリボソームを認識しているのか,なぜもうひとつの翻訳終結因子RF1ではArfAは機能せずRF2でなければならないのか,など重要な問題が未解決のまま残されていた.
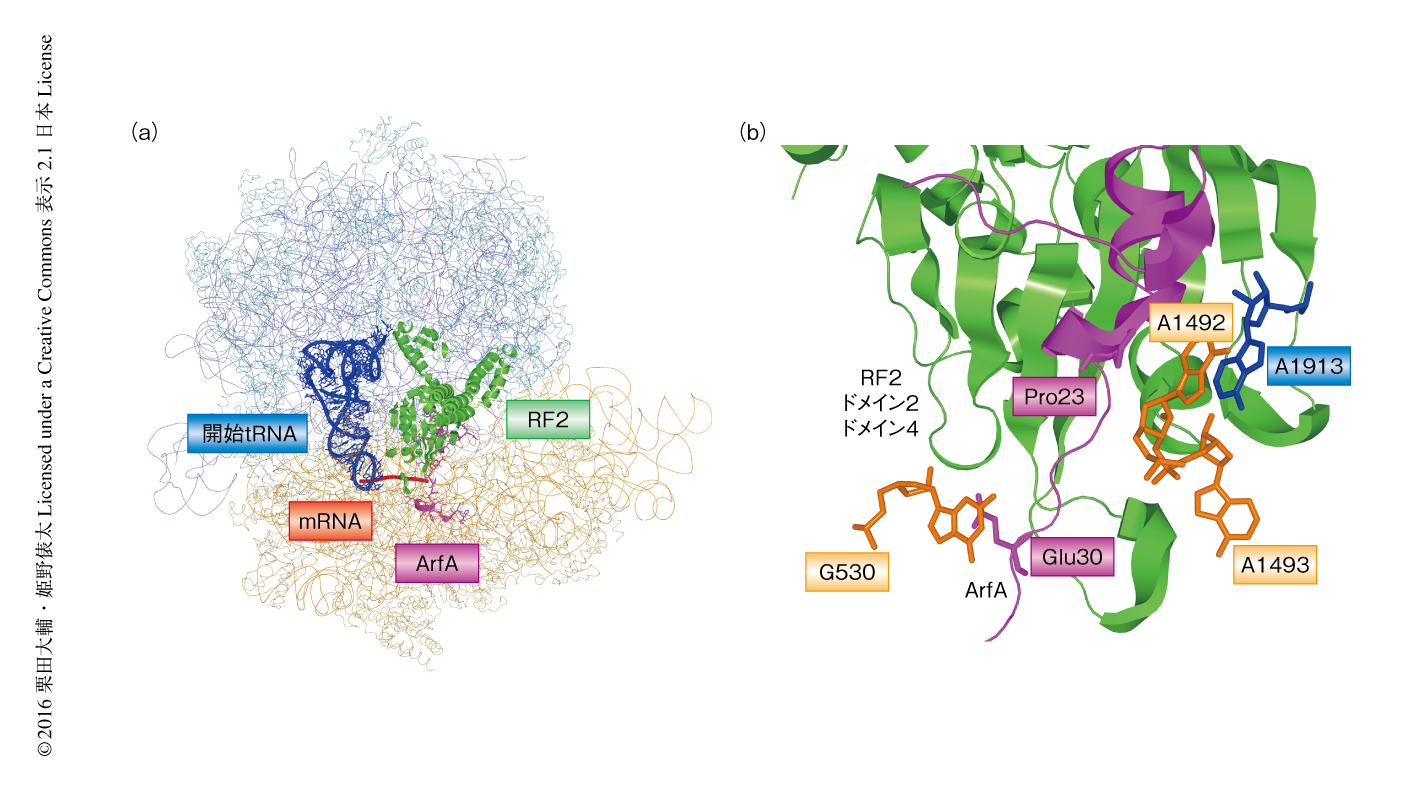
70Sリボソーム,ArfA,翻訳終結因子RF2,開始tRNA,mRNAからノンストップ複合体を形成させ,クライオ電子顕微鏡によりその構造を解析し,3.0Åの分解能で構造を決定した(図1a).その結果,ArfAのN末端側の領域はリボソームのアミノアシルtRNAの結合部位であるA部位のデコーディング領域の付近に位置しており,リボソーム30Sサブユニットのヘリックス44やリボソームタンパク質S12,リボソーム50Sサブユニットのヘリックス69のあいだに位置していた.一方,ArfAのC末端側の領域はリボソームにおけるmRNAの結合部位であるmRNAエントリーチャネルに位置していた.これは,筆者らが,2014年,部位特異的なラジカルプローブ法により明らかにした結果と一致し4),リボソームにおいてArfAのC末端側の領域の結合部位とmRNAの結合部位とが重複することを意味する.通常の翻訳の過程においては,このmRNAエントリーチャネルはmRNAにより占拠されているためArfAのC末端側の領域は入り込むことができないが,もしmRNAの3’末端側の領域が欠損していたならArfAのC末端側の領域はmRNAエントリーチャネルに入り込むことができる.
ノンストップ複合体のリボソームのデコーディング領域は通常の翻訳終結複合体のリボソームとは異なる構造をとっていた.デコーディング領域には16S rRNAの高度に保存された3つの塩基,G530,A1492,A1493が存在する.通常の翻訳終結複合体においては,G530は終止コドンUAAの3文字目のAとスタッキング相互作用する5).また,A1493は23S rRNAのA1913とのスタッキング相互作用により安定化されており,A1492はG530の方向に伸びるという特徴的な構造をしている.それに対して,ノンストップ複合体においては,G530の近くには終止コドンの代わりにArfAのGlu30が位置していた(図1b).そして,A1913とスタッキング相互作用するのはA1493ではなくA1492であり,さらに,A1492はリボソーム50Sサブユニットのヘリックス69とArfAのPro23とのあいだにはさまれるかたちで安定化されていた.ノンストップ複合体のデコーディング領域はこれまでに構造が報告されたリボソームのどの状態とも異なる構造をとっており6),おそらく,ArfAがリボソーム30Sサブユニットのヘリックスh44とリボソーム50Sサブユニットのヘリックス69を微調整しているものと考えられた.
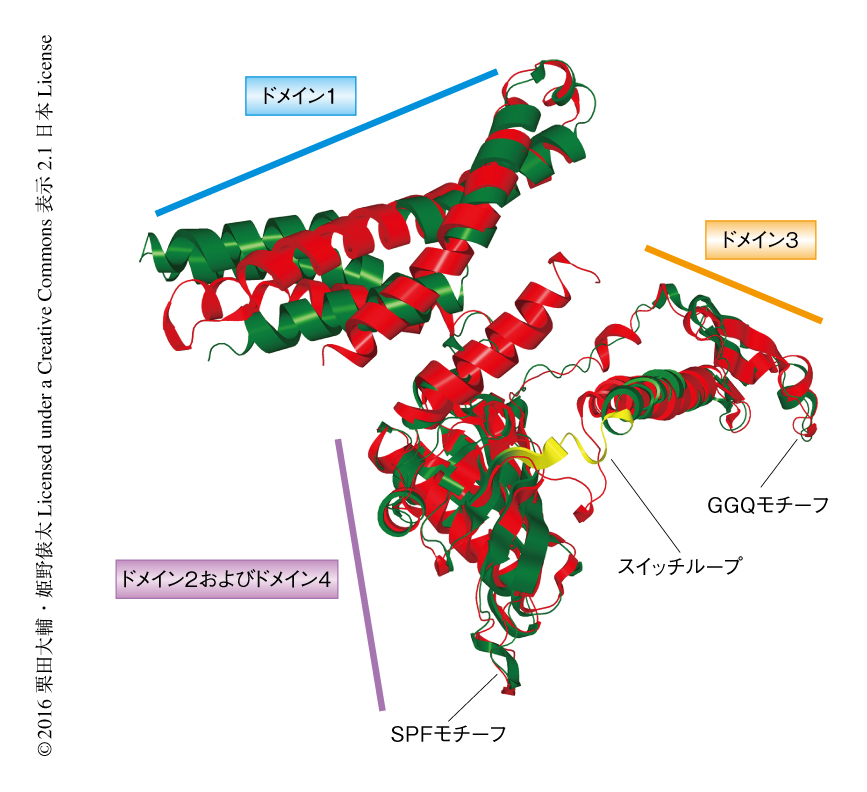
ノンストップ複合体と翻訳終結複合体とを比較したところ,デコーディング領域だけでなくRF2の構造にも違いがみられた.もっとも顕著な違いはスイッチループの構造であった(図2).RF2は4つのドメインからなり,スイッチループはドメイン3とドメイン4のあいだに存在する.これまでの研究により,RF2はリボソームとの結合の前後で構造変化を起こすことが示唆されており7),この構造変化はRF2が終止コドンを認識したのち,ペプチジルtRNAの加水分解の活性中心であるドメイン3の先端のGGQモチーフを,P部位のtRNAのCCA末端の近傍に位置させるために重要であると考えられてきた.ノンストップ複合体におけるスイッチループは翻訳終結複合体のスイッチループとは構造が異なり,疎水的な相互作用をとおしてArfAと接触していた.実際に,このスイッチループに変異を導入するとペプチジルtRNAの加水分解活性が完全に消失した.
RF1とRF2のアミノ酸配列および立体構造は全体的によく似ているが,スイッチループに着目すると両者の違いがみえてくる.RF2のスイッチループには疎水性のアミノ酸残基が多いのに対し,RF1では極性アミノ酸残基がめだつ.この違いがArfAとの相互作用の有無にかかわるのかもしれない.
ArfAが終止コドンを擬態しているのかという点は興味のもたれる問題である.翻訳終結複合体とノンストップ複合体とを比較したところ,翻訳終結複合体においてはRF2のSPFモチーフのSer205が終止コドンの2文字目のプリン塩基と相互作用するのに対し5),ノンストップ複合体においてはSPFモチーフとArfAとの直接的な相互作用は確認されなかった.また,リボソームにおけるArfAの結合部位は終止コドンの位置する部位とは微妙にずれていた.これらを考慮すると,ArfAによる終止コドンの擬態という考え方は構造的な意味からは否定された.しかし,RF2をリクルートするための足場を形成するという機能的な意味においては,ArfAは終止コドンの代理をつとめているといえるかもしれない.
大腸菌においては,ArfAおよびRF2による機構のほかにも少なくとも2つのリボソームレスキュー機構,すなわち,tmRNAおよびSmpBによるトランス-トランスレーション,および,ArfBによる機構が存在する.トランス-トランスレーションの場合はSmpBのC末端のテイルがリボソームのmRNAエントリーチャネルと結合すること,ArfBによる機構の場合もやはりC末端のテイルがmRNAエントリーチャネルと結合することが,結晶構造解析により明らかにされている8,9).これら3つのリボソームレスキュー機構を比較することにより,翻訳の停滞を解消するタンパク質がどのようにして停滞したリボソームを認識するのか,という疑問の答えになりうる,共通の分子機構がうかびあがってきた.それぞれのタンパク質はmRNAエントリーチャネルと結合することによりリボソームの状態,すなわち,mRNAの有無を見分けるのである.いい換えると,3つのリボソームレスキュー機構にかかわるタンパク質は,どれもC末端側の領域がリボソームの停滞の状態を見分けるためのセンサーとして機能する.
略歴:2009年 岩手大学大学院連合農学研究科 修了,同年 弘前大学農学生命科学部 特別研究員を経て,2012年より同 助教.
研究テーマ:翻訳の停滞を解消するシステムの分子機構.
姫野 俵太(Hyouta Himeno)
弘前大学農学生命科学部 教授.
研究室URL:http://hirosaki-rna.org/himeno/
© 2016 栗田大輔・姫野俵太 Licensed under CC 表示 2.1 日本
(弘前大学農学生命科学部 分子生命科学科分子生物学研究室)
email:栗田大輔,姫野俵太
DOI: 10.7875/first.author.2016.132
Mechanistic insights into the alternative translation termination by ArfA and RF2.
Chengying Ma, Daisuke Kurita, Ningning Li, Yan Chen, Hyouta Himeno, Ning Gao
Nature, 541, 550-553 (2017)
要 約
細胞においてさまざまな理由により翻訳は停滞する.たとえば,リボソームが終止コドンをもたないノンストップmRNAにおいて翻訳をはじめると,終止コドンに到達することができないため翻訳は途中で停滞する.このような翻訳の停滞を解消する機構としてtmRNAによるトランス-トランスレーションが知られていた.しかし,最近になり,翻訳の停滞の解消にはたらく新たなタンパク質としてArfAが発見され,トランス-トランスレーションとは異なるリボソームレスキュー機構の存在が明らかにされた.筆者らは,70Sリボソーム,ArfA,翻訳終結因子RF2,開始tRNA,mRNAからなるノンストップ複合体をクライオ電子顕微鏡により解析し,3.0Åの分解能で構造を決定した.これにより,ArfAのN末端側の領域はリボソーム30Sサブユニットのデコーディング領域とRF2とにはさまれるかたちで結合する一方,ArfAのC末端側の領域はリボソームのmRNA結合部位に位置することが明らかにされた.この結果から,ArfAが空のmRNAエントリーチャネルを認識することにより翻訳の停滞した状態,すなわち,mRNAの3’末端の側において停滞するリボソームを見分けることが示唆された.
はじめに
原核生物あるいは真核生物をとわず,翻訳の途中でリボソームが停滞することがある.このような異常な事態を解消する機構として,細菌にはtmRNAによるトランス-トランスレーションというリボソームレスキュー機構が存在する.tmRNAが必須である生物もいるが,大腸菌や枯草菌などの多くの細菌においてはtmRNAの欠損が通常の培養条件における生育に大きな影響をおよぼすことはない.このことから,トランス-トランスレーションに代わる機構の存在の可能性が示唆されていた.2010年,分子遺伝学的な手法を用いた研究により,翻訳の停滞の解消にはたらくタンパク質としてArfAの存在が明らかにされた1).ArfAは停滞したリボソームと結合し,翻訳終結因子RF2と協調してペプチジルtRNAを加水分解することによりリボソームの停滞を解消する2,3).通常の翻訳においてRF2は3種類の終止コドンのうちUAAあるいはUGAを認識してリボソームと結合するが,ArfAのかかわるリボソームレスキュー機構において終止コドンの関与は想定されていない.どのようにしてArfAおよびRF2は終止コドンの存在しないmRNAと結合したリボソームを認識しているのか,なぜもうひとつの翻訳終結因子RF1ではArfAは機能せずRF2でなければならないのか,など重要な問題が未解決のまま残されていた.
1.ノンストップ複合体におけるArfAの結合の位置
70Sリボソーム,ArfA,翻訳終結因子RF2,開始tRNA,mRNAからノンストップ複合体を形成させ,クライオ電子顕微鏡によりその構造を解析し,3.0Åの分解能で構造を決定した(図1a).その結果,ArfAのN末端側の領域はリボソームのアミノアシルtRNAの結合部位であるA部位のデコーディング領域の付近に位置しており,リボソーム30Sサブユニットのヘリックス44やリボソームタンパク質S12,リボソーム50Sサブユニットのヘリックス69のあいだに位置していた.一方,ArfAのC末端側の領域はリボソームにおけるmRNAの結合部位であるmRNAエントリーチャネルに位置していた.これは,筆者らが,2014年,部位特異的なラジカルプローブ法により明らかにした結果と一致し4),リボソームにおいてArfAのC末端側の領域の結合部位とmRNAの結合部位とが重複することを意味する.通常の翻訳の過程においては,このmRNAエントリーチャネルはmRNAにより占拠されているためArfAのC末端側の領域は入り込むことができないが,もしmRNAの3’末端側の領域が欠損していたならArfAのC末端側の領域はmRNAエントリーチャネルに入り込むことができる.
2.ノンストップ複合体と翻訳終結複合体におけるリボソームの構造の違い
ノンストップ複合体のリボソームのデコーディング領域は通常の翻訳終結複合体のリボソームとは異なる構造をとっていた.デコーディング領域には16S rRNAの高度に保存された3つの塩基,G530,A1492,A1493が存在する.通常の翻訳終結複合体においては,G530は終止コドンUAAの3文字目のAとスタッキング相互作用する5).また,A1493は23S rRNAのA1913とのスタッキング相互作用により安定化されており,A1492はG530の方向に伸びるという特徴的な構造をしている.それに対して,ノンストップ複合体においては,G530の近くには終止コドンの代わりにArfAのGlu30が位置していた(図1b).そして,A1913とスタッキング相互作用するのはA1493ではなくA1492であり,さらに,A1492はリボソーム50Sサブユニットのヘリックス69とArfAのPro23とのあいだにはさまれるかたちで安定化されていた.ノンストップ複合体のデコーディング領域はこれまでに構造が報告されたリボソームのどの状態とも異なる構造をとっており6),おそらく,ArfAがリボソーム30Sサブユニットのヘリックスh44とリボソーム50Sサブユニットのヘリックス69を微調整しているものと考えられた.
3.ノンストップ複合体におけるRF2の構造
ノンストップ複合体と翻訳終結複合体とを比較したところ,デコーディング領域だけでなくRF2の構造にも違いがみられた.もっとも顕著な違いはスイッチループの構造であった(図2).RF2は4つのドメインからなり,スイッチループはドメイン3とドメイン4のあいだに存在する.これまでの研究により,RF2はリボソームとの結合の前後で構造変化を起こすことが示唆されており7),この構造変化はRF2が終止コドンを認識したのち,ペプチジルtRNAの加水分解の活性中心であるドメイン3の先端のGGQモチーフを,P部位のtRNAのCCA末端の近傍に位置させるために重要であると考えられてきた.ノンストップ複合体におけるスイッチループは翻訳終結複合体のスイッチループとは構造が異なり,疎水的な相互作用をとおしてArfAと接触していた.実際に,このスイッチループに変異を導入するとペプチジルtRNAの加水分解活性が完全に消失した.
RF1とRF2のアミノ酸配列および立体構造は全体的によく似ているが,スイッチループに着目すると両者の違いがみえてくる.RF2のスイッチループには疎水性のアミノ酸残基が多いのに対し,RF1では極性アミノ酸残基がめだつ.この違いがArfAとの相互作用の有無にかかわるのかもしれない.
4.ArfAは終止コドンの擬態タンパク質か
ArfAが終止コドンを擬態しているのかという点は興味のもたれる問題である.翻訳終結複合体とノンストップ複合体とを比較したところ,翻訳終結複合体においてはRF2のSPFモチーフのSer205が終止コドンの2文字目のプリン塩基と相互作用するのに対し5),ノンストップ複合体においてはSPFモチーフとArfAとの直接的な相互作用は確認されなかった.また,リボソームにおけるArfAの結合部位は終止コドンの位置する部位とは微妙にずれていた.これらを考慮すると,ArfAによる終止コドンの擬態という考え方は構造的な意味からは否定された.しかし,RF2をリクルートするための足場を形成するという機能的な意味においては,ArfAは終止コドンの代理をつとめているといえるかもしれない.
おわりに
大腸菌においては,ArfAおよびRF2による機構のほかにも少なくとも2つのリボソームレスキュー機構,すなわち,tmRNAおよびSmpBによるトランス-トランスレーション,および,ArfBによる機構が存在する.トランス-トランスレーションの場合はSmpBのC末端のテイルがリボソームのmRNAエントリーチャネルと結合すること,ArfBによる機構の場合もやはりC末端のテイルがmRNAエントリーチャネルと結合することが,結晶構造解析により明らかにされている8,9).これら3つのリボソームレスキュー機構を比較することにより,翻訳の停滞を解消するタンパク質がどのようにして停滞したリボソームを認識するのか,という疑問の答えになりうる,共通の分子機構がうかびあがってきた.それぞれのタンパク質はmRNAエントリーチャネルと結合することによりリボソームの状態,すなわち,mRNAの有無を見分けるのである.いい換えると,3つのリボソームレスキュー機構にかかわるタンパク質は,どれもC末端側の領域がリボソームの停滞の状態を見分けるためのセンサーとして機能する.
文 献
- Chadani, Y., Ono, K., Ozawa, S. et al.: Ribosome rescue by Escherichia coli ArfA (YhdL) in the absence of trans-translation system. Mol. Microbiol., 78, 796-808 (2010)[PubMed]
- Chadani, Y., Ito, K., Kutsukake, K. et al.: ArfA recruits release factor 2 to rescue stalled ribosomes by peptidyl-tRNA hydrolysis in Escherichia coli. Mol. Microbiol., 86, 37-50 (2012)[PubMed]
- Shimizu, Y.: ArfA recruits RF2 into stalled ribosomes. J. Mol. Biol., 423, 624-631 (2012)[PubMed]
- Kurita, D., Chadani, Y., Muto, A. et al.: ArfA recognizes the lack of mRNA in the mRNA channel after RF2 binding for ribosome rescue. Nucleic Acids Res., 42, 13339-13352 (2014)[PubMed]
- Korostelev, A., Asahara, H., Lancaster, L. et al.: Crystal structure of a translation termination complex formed with release factor RF2. Proc. Natl. Acad. Sci. USA, 105, 19684-19689 (2008)[PubMed]
- Zhou, J., Korostelev, A., Lancaster, L. et al.: Crystal structures of 70S ribosomes bound to release factors RF1, RF2 and RF3. Curr. Opin. Struct. Biol., 22, 733-742 (2012)[PubMed]
- Vestergaard, B., Van, L. B., Andersen, G. R. et al.: Bacterial polypeptide release factor RF2 is structurally distinct from eukaryotic eRF1. Mol. Cell, 8, 1375-1382 (2001)[PubMed]
- Neubauer, C., Gillet, R., Kelley, A. C. et al.: Decoding in the absence of a codon by tmRNA and SmpB in the ribosome. Science, 335, 1366-1369 (2012)[PubMed]
- Gagnon, M. G., Seetharaman, S. V., Bulkley, D. et al.: Structural basis for the rescue of stalled ribosomes: structure of YaeJ bound to the ribosome. Science, 335, 1370-1372 (2012)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2009年 岩手大学大学院連合農学研究科 修了,同年 弘前大学農学生命科学部 特別研究員を経て,2012年より同 助教.
研究テーマ:翻訳の停滞を解消するシステムの分子機構.
姫野 俵太(Hyouta Himeno)
弘前大学農学生命科学部 教授.
研究室URL:http://hirosaki-rna.org/himeno/
© 2016 栗田大輔・姫野俵太 Licensed under CC 表示 2.1 日本