タンパク質の結び目構造からのシグナルによるtRNAのメチル化修飾
坂口怜子・Ya-Ming Hou
(米国Thomas Jefferson大学Department of Biochemistry and Molecular Biology)
email:坂口怜子
DOI: 10.7875/first.author.2016.097
Methyl transfer by substrate signaling from a knotted protein fold.
Thomas Christian, Reiko Sakaguchi, Agata P. Perlinska, Georges Lahoud, Takuhiro Ito, Erika A. Taylor, Shigeyuki Yokoyama, Joanna I. Sulkowska, Ya-Ming Hou
Nature Structural & Molecular Biology, 23, 941-948 (2016)
結び目構造をもつタンパク質は構造の自由度が低い.そして,結び目をもつタンパク質が空間的にへだてられた部位とのあいだでシグナルを伝達するのに十分なダイナミクスをもつのかどうかは知られていない.結び目構造をもつタンパク質のひとつに,S-アデノシルメチオニンを基質としてtRNAの37位のGにメチル基を転移する酵素TrmDがある.TrmDによりメチル化修飾されたtRNAはリボソームにおけるフレームシフトの防止において重要である.この研究において,構造生物学,反応速度論,シミュレーションの3つの観点から,構造的に束縛の大きいTrmDの結び目構造はその酵素活性に必須であることが示された.予想に反して,結び目構造に含まれるアミノ酸残基はS-アデノシルメチオニンとの結合にともない複雑な挙動を示すことがわかった.S-アデノシルメチオニンとの結合にともなう自由エネルギーの変化がシグナルとして伝達し,tRNAとの結合を安定化し活性中心が形成されていた.この研究により,タンパク質の結び目構造は基質との結合により生じる自由エネルギーをとらえ酵素活性を促進する巧妙な構造であることが示された.
タンパク質の生合成においてtRNAは重要な役割をはたす.tRNAはその3’末端にて対応するアミノ酸と共有結合したのち,mRNAのもつコドンに対応してリボソームに取り込まれ,tRNAと結合したアミノ酸は新生するペプチド鎖に連結される.tRNAが正常に機能するためには,転写されたのちさまざまに修飾される必要がある.tRNAにはこれまで100をこえる修飾が知られており,37位のGのN1位におけるメチル化修飾はタンパク質の生合成において正確性を保つために非常に重要である.tRNAの37位はmRNAと結合するアンチコドン領域のすぐとなりに位置し,そのメチル化修飾は多くの機能をはたす.これまでの研究により,古細菌においてはtRNACysのアミノアシル化を促進すること1),出芽酵母においてはtRNAAspがArgとまちがって結合するのをふせぐこと2) が知られている.そして,もっとも重要な役割は,リボソームにおいてtRNAがmRNAと結合する際にフレームシフトを防止することである.GのN1位におけるメチル化はWatson-Crick塩基対の形成を阻害するので,アンチコドン領域のすぐとなりの37位の塩基がmRNAのコドンのとなりの塩基と結合しなくなり,4つの塩基があやまって1つのコドンとして読まれることがふせがれる.これまでに,tRNAの37位のGのN1位におけるメチル化修飾が存在しない場合はフレームシフトが起こりやすくなることが示されている3).このメチル化修飾は全生物界において保存されており,既知のtRNA修飾酵素のうち,37位のGのN1位におけるメチル化修飾を担う酵素の欠損のみが細菌および出芽酵母において致死性を示す4,5).tRNAの37位のGにメチル基を転移する酵素は,真正細菌においてはTrmD,真核生物および古細菌においてはTrm5である.TrmDとTrm5は,S-アデノシルメチオニンをメチル基の供与体として用いてまったく同じ化学反応を触媒するにもかかわらず,1次構造および3次構造に相同性はなく,それぞれ異なる祖先から進化したものと考えられている(図1).
細菌においてTrmDは高度に保存されており,N末端側にS-アデノシルメチオニン結合ドメイン,C末端側にtRNA認識ドメインをもつ単量体からなるホモ二量体として機能する6).一方で,Trm5は比較的最近になり同定され,その結晶構造も報告された7,8).Trm5は単量体として機能する.TrmDは細菌にしか存在せず,その欠損は細菌にとり致死性であることから,副作用の心配のない新規の抗生物質の標的として魅力的である.TrmDのみを選択的に阻害する物質を探索するためには,TrmDとTrm5の基質認識機構の違いを理解することが重要である.
最近,TrmD,tRNA,S-アデノシルメチオニンの不活性アナログであるsinefunginからなる三者複合体の結晶構造が報告された9).TrmDはホモ二量体を形成し,その結合面に活性中心を形成していた.二量体の一方(A)と結合したS-アデノシルメチオニンが他方(B)と結合したtRNAと反応し,このとき,Bと結合したS-アデノシルメチオニンは不活性化していた.しかし,2箇所の活性部位がどのようなしくみより互いにシグナルを伝達し,不要なほうのS-アデノシルメチオニンを不活性化するのかは明らかではなかった.この研究においては,この機構の解明のため,構造生物学,反応速度論,コンピューターシミュレーションと複数のアプローチにより解析した.
TrmD,tRNA,sinefunginからなる三者複合体の結晶構造をもとに,結び目構造の付近のダイナミクスを計算した.分子動力学シミュレーション法を用いて結び目構造と結合したS-アデノシルメチオニンの構造ゆらぎを比較したところ,活性化しているほうはもう一方と比べゆらぎが格段に小さかったことから,TrmDホモ二量体のそれぞれの結び目構造は非対称であることがわかった.また,活性化しているS-アデノシルメチオニンの構造ゆらぎはTrm5の活性部位と比較しても小さかった.
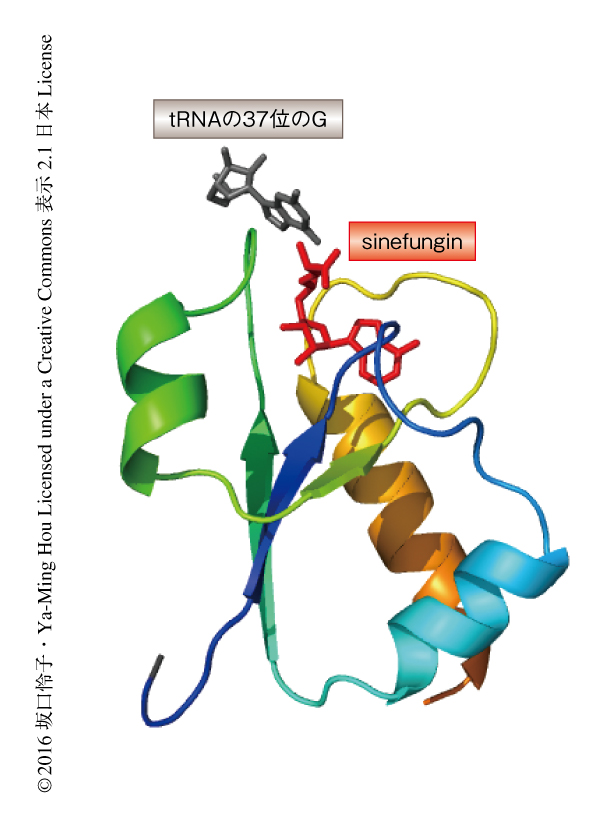
さらに,シミュレーションの結果から,S-アデノシルメチオニンが折れ曲がって結合した場合,そのメチル基と標的であるtRNAの37位のGのN1位とのあいだの距離は3.5Åとメチル基の転移に望ましい距離であったのに対し,S-アデノシルメチオニンがまっすぐな状態では4.3Åと離れてしまっており,しかも,α炭素原子はtRNAと立体障害を生じていた.したがって,TrmD三者複合体の結晶構造と同様に,TrmDによるtRNAへのメチル基の転移においてはS-アデノシルメチオニンは折れ曲がった構造をとることが必須であることがわかった(図2,PDB ID:4YVI).
TrmDファミリーにおいて高度に保存されている15個のアミノ酸残基を選び,これをAlaに置換した変異体を作製して酵素活性を速度論的に解析した.S-アデノシルメチオニンおよびtRNAに対する結合定数,および,メチル基の転移における速度定数を評価し,その活性の低下を自由エネルギーの損失として算出しTrmDにマッピングした.その結果,S-アデノシルメチオニンとの結合に重要なアミノ酸残基とtRNAとの結合に重要なアミノ酸残基とのあいだにはある程度の相関があり,tRNAとの結合においてもっとも重要なアミノ酸残基はメチル基の転移にも直接に関与することがわかった.この結果から,S-アデノシルメチオニンとの結合にともなうエネルギーの変化が順に伝達していくと考えられ,S-アデノシルメチオニンとの結合→tRNAとの結合→メチル基の転移,とシグナルが伝達するようすが明らかにされた.
tRNAとの結合能およびメチル基の転移活性が顕著に損なわれたTyr115をAlaに置換したTrmD変異体を用いて,基質との結合のシグナル伝達について検討した.このTrmD変異体について分子動力学シミュレーションをしたところ,C末端ドメインの構造が変化しS-アデノシルメチオニンとの水素結合が減弱していることが示唆された.くわえて,メチル基の転移に重要なArg54とtRNAの37位のGとの距離が大きくなり,S-アデノシルメチオニンからのメチル基の転移が阻害されていた.そして,結び目部位における自由エネルギーの分布において,野生型のTrmDにおいてはエネルギーの極小が2つあったのに対し,Tyr115をAlaに置換したTrmD変異体ではエネルギーの極小は1つであった.以上の結果から,Tyr115をAlaに置換したTrmD変異体においてホモ二量体が非対称ではなくなっていると考えられた.一方で,TrmDのTyr115の変異により1つのTrmDホモ二量体に対し2つのtRNAが結合するようになることが実験的に確認され,この可能性が裏づけられた.これらの結果から,TrmDホモ二量体を構成するおのおのの単量体のわずかな構造の違いから生じる非対称性がシグナルの伝達を担うと考えられた.
TrmDのさまざまなAla置換変異体における酵素活性の低下について詳細に検討した.Gly55をAlaに置換したTrmD変異体においてはtRNAとの結合能が顕著に低下しており,S-アデノシルメチオニンとは直接に相互作用していないにもかかわらずS-アデノシルメチオニンとの結合能も低下していた.この変異の影響をシミュレーションしたところ,S-アデノシルメチオニンと直接に結合するアミノ酸残基との相互作用が減弱していた.この結果から,TrmDとtRNAとの接触を阻害するとS-アデノシルメチオニンとの結合能が低下することが示された.つぎに,S-アデノシルメチオニンのメチオニン部位と相互作用するアミノ酸残基に着目した.なかでも,Ser170をAlaに置換したTrmD変異体においてS-アデノシルメチオニンとの結合能,tRNAとの結合能,メチル基の転移活性のすべてが低下していた.169番目のアミノ酸残基はメチル基の転移にかかわる一般塩基としてはたらき,171番目のアミノ酸残基はS-アデノシルメチオニンとの結合にともない構造を安定化することから,S-アデノシルメチオニンとの結合が活性中心の形成の引き金になることが示唆された.
TrmDのAsp169はtRNAの37位のGのN1位,TrmDのArg154はtRNAの37位のGのO6位とそれぞれ相互作用しtRNAの37位のGを安定化する.Arg154をAlaに置換したTrmD変異体およびAsp169をAlaに置換したTrmD変異体は,tRNAとの結合能およびメチル基の転移活性が大幅に低下していたが,S-アデノシルメチオニンとの結合能に影響はなかった.これらの結果から,tRNAの37位のGと結合することがtRNA全体の安定化に寄与し,ひとたびtRNAの37位のGが安定化すればS-アデノシルメチオニンの位置をさらに微調整することなくメチル基の転移は進むと考えられた.
筆者らは,現在まで,ヒトそのほか哺乳類に影響のない抗生物質の開発をめざしてTrmDおよびTrm5の特性を評価してきた10,11).この研究における特筆すべき点は,TrmDのみを特異的に標的とする戦略に新たな知見をあたえたことである.S-アデノシルメチオニンがTrmDの結び目構造と結合することがシグナル伝達の起点であったことから,TrmDの理想的な阻害剤は,結び目構造を標的とし,折れ曲がったS-アデノシルメチオニンの代わりにピッタリと入り込むようなものであると考えられる.このような阻害の様式は,S-アデノシルメチオニンを基質とするTrm5を含むほかの酵素への影響が少ないと予想される.筆者らの研究室においては,別途,TrmDを選択的に阻害する物質をスクリーニングしており,これらの知見が総合的に新規の抗生物質の開発に資することを目標としている.
略歴:2009年 京都大学大学院エネルギー科学研究科 修了,同年 米国Thomas Jefferson大学 博士研究員を経て,2013年より京都大学物質-細胞統合システム拠点 特定助教.
研究テーマ:細胞におけるシグナル伝達物質およびシグナル伝達経路の可視化.
抱負:健康第一で研究に取り組んでいます.
Ya-Ming Hou
米国Thomas Jefferson大学 教授.
研究室URL:http://www.houlaboratory.org/
© 2016 坂口怜子・Ya-Ming Hou Licensed under CC 表示 2.1 日本
(米国Thomas Jefferson大学Department of Biochemistry and Molecular Biology)
email:坂口怜子
DOI: 10.7875/first.author.2016.097
Methyl transfer by substrate signaling from a knotted protein fold.
Thomas Christian, Reiko Sakaguchi, Agata P. Perlinska, Georges Lahoud, Takuhiro Ito, Erika A. Taylor, Shigeyuki Yokoyama, Joanna I. Sulkowska, Ya-Ming Hou
Nature Structural & Molecular Biology, 23, 941-948 (2016)
要 約
結び目構造をもつタンパク質は構造の自由度が低い.そして,結び目をもつタンパク質が空間的にへだてられた部位とのあいだでシグナルを伝達するのに十分なダイナミクスをもつのかどうかは知られていない.結び目構造をもつタンパク質のひとつに,S-アデノシルメチオニンを基質としてtRNAの37位のGにメチル基を転移する酵素TrmDがある.TrmDによりメチル化修飾されたtRNAはリボソームにおけるフレームシフトの防止において重要である.この研究において,構造生物学,反応速度論,シミュレーションの3つの観点から,構造的に束縛の大きいTrmDの結び目構造はその酵素活性に必須であることが示された.予想に反して,結び目構造に含まれるアミノ酸残基はS-アデノシルメチオニンとの結合にともない複雑な挙動を示すことがわかった.S-アデノシルメチオニンとの結合にともなう自由エネルギーの変化がシグナルとして伝達し,tRNAとの結合を安定化し活性中心が形成されていた.この研究により,タンパク質の結び目構造は基質との結合により生じる自由エネルギーをとらえ酵素活性を促進する巧妙な構造であることが示された.
はじめに
タンパク質の生合成においてtRNAは重要な役割をはたす.tRNAはその3’末端にて対応するアミノ酸と共有結合したのち,mRNAのもつコドンに対応してリボソームに取り込まれ,tRNAと結合したアミノ酸は新生するペプチド鎖に連結される.tRNAが正常に機能するためには,転写されたのちさまざまに修飾される必要がある.tRNAにはこれまで100をこえる修飾が知られており,37位のGのN1位におけるメチル化修飾はタンパク質の生合成において正確性を保つために非常に重要である.tRNAの37位はmRNAと結合するアンチコドン領域のすぐとなりに位置し,そのメチル化修飾は多くの機能をはたす.これまでの研究により,古細菌においてはtRNACysのアミノアシル化を促進すること1),出芽酵母においてはtRNAAspがArgとまちがって結合するのをふせぐこと2) が知られている.そして,もっとも重要な役割は,リボソームにおいてtRNAがmRNAと結合する際にフレームシフトを防止することである.GのN1位におけるメチル化はWatson-Crick塩基対の形成を阻害するので,アンチコドン領域のすぐとなりの37位の塩基がmRNAのコドンのとなりの塩基と結合しなくなり,4つの塩基があやまって1つのコドンとして読まれることがふせがれる.これまでに,tRNAの37位のGのN1位におけるメチル化修飾が存在しない場合はフレームシフトが起こりやすくなることが示されている3).このメチル化修飾は全生物界において保存されており,既知のtRNA修飾酵素のうち,37位のGのN1位におけるメチル化修飾を担う酵素の欠損のみが細菌および出芽酵母において致死性を示す4,5).tRNAの37位のGにメチル基を転移する酵素は,真正細菌においてはTrmD,真核生物および古細菌においてはTrm5である.TrmDとTrm5は,S-アデノシルメチオニンをメチル基の供与体として用いてまったく同じ化学反応を触媒するにもかかわらず,1次構造および3次構造に相同性はなく,それぞれ異なる祖先から進化したものと考えられている(図1).
細菌においてTrmDは高度に保存されており,N末端側にS-アデノシルメチオニン結合ドメイン,C末端側にtRNA認識ドメインをもつ単量体からなるホモ二量体として機能する6).一方で,Trm5は比較的最近になり同定され,その結晶構造も報告された7,8).Trm5は単量体として機能する.TrmDは細菌にしか存在せず,その欠損は細菌にとり致死性であることから,副作用の心配のない新規の抗生物質の標的として魅力的である.TrmDのみを選択的に阻害する物質を探索するためには,TrmDとTrm5の基質認識機構の違いを理解することが重要である.
最近,TrmD,tRNA,S-アデノシルメチオニンの不活性アナログであるsinefunginからなる三者複合体の結晶構造が報告された9).TrmDはホモ二量体を形成し,その結合面に活性中心を形成していた.二量体の一方(A)と結合したS-アデノシルメチオニンが他方(B)と結合したtRNAと反応し,このとき,Bと結合したS-アデノシルメチオニンは不活性化していた.しかし,2箇所の活性部位がどのようなしくみより互いにシグナルを伝達し,不要なほうのS-アデノシルメチオニンを不活性化するのかは明らかではなかった.この研究においては,この機構の解明のため,構造生物学,反応速度論,コンピューターシミュレーションと複数のアプローチにより解析した.
1.TrmDの結び目構造と結合したS-アデノシルメチオニンの向きの重要性
TrmD,tRNA,sinefunginからなる三者複合体の結晶構造をもとに,結び目構造の付近のダイナミクスを計算した.分子動力学シミュレーション法を用いて結び目構造と結合したS-アデノシルメチオニンの構造ゆらぎを比較したところ,活性化しているほうはもう一方と比べゆらぎが格段に小さかったことから,TrmDホモ二量体のそれぞれの結び目構造は非対称であることがわかった.また,活性化しているS-アデノシルメチオニンの構造ゆらぎはTrm5の活性部位と比較しても小さかった.
さらに,シミュレーションの結果から,S-アデノシルメチオニンが折れ曲がって結合した場合,そのメチル基と標的であるtRNAの37位のGのN1位とのあいだの距離は3.5Åとメチル基の転移に望ましい距離であったのに対し,S-アデノシルメチオニンがまっすぐな状態では4.3Åと離れてしまっており,しかも,α炭素原子はtRNAと立体障害を生じていた.したがって,TrmD三者複合体の結晶構造と同様に,TrmDによるtRNAへのメチル基の転移においてはS-アデノシルメチオニンは折れ曲がった構造をとることが必須であることがわかった(図2,PDB ID:4YVI).
2.変異体の解析による基質との結合のシグナル伝達の解明
TrmDファミリーにおいて高度に保存されている15個のアミノ酸残基を選び,これをAlaに置換した変異体を作製して酵素活性を速度論的に解析した.S-アデノシルメチオニンおよびtRNAに対する結合定数,および,メチル基の転移における速度定数を評価し,その活性の低下を自由エネルギーの損失として算出しTrmDにマッピングした.その結果,S-アデノシルメチオニンとの結合に重要なアミノ酸残基とtRNAとの結合に重要なアミノ酸残基とのあいだにはある程度の相関があり,tRNAとの結合においてもっとも重要なアミノ酸残基はメチル基の転移にも直接に関与することがわかった.この結果から,S-アデノシルメチオニンとの結合にともなうエネルギーの変化が順に伝達していくと考えられ,S-アデノシルメチオニンとの結合→tRNAとの結合→メチル基の転移,とシグナルが伝達するようすが明らかにされた.
tRNAとの結合能およびメチル基の転移活性が顕著に損なわれたTyr115をAlaに置換したTrmD変異体を用いて,基質との結合のシグナル伝達について検討した.このTrmD変異体について分子動力学シミュレーションをしたところ,C末端ドメインの構造が変化しS-アデノシルメチオニンとの水素結合が減弱していることが示唆された.くわえて,メチル基の転移に重要なArg54とtRNAの37位のGとの距離が大きくなり,S-アデノシルメチオニンからのメチル基の転移が阻害されていた.そして,結び目部位における自由エネルギーの分布において,野生型のTrmDにおいてはエネルギーの極小が2つあったのに対し,Tyr115をAlaに置換したTrmD変異体ではエネルギーの極小は1つであった.以上の結果から,Tyr115をAlaに置換したTrmD変異体においてホモ二量体が非対称ではなくなっていると考えられた.一方で,TrmDのTyr115の変異により1つのTrmDホモ二量体に対し2つのtRNAが結合するようになることが実験的に確認され,この可能性が裏づけられた.これらの結果から,TrmDホモ二量体を構成するおのおのの単量体のわずかな構造の違いから生じる非対称性がシグナルの伝達を担うと考えられた.
3.S-アデノシルメチオニンとの結合を引き金とした活性中心の形成およびtRNAの安定化
TrmDのさまざまなAla置換変異体における酵素活性の低下について詳細に検討した.Gly55をAlaに置換したTrmD変異体においてはtRNAとの結合能が顕著に低下しており,S-アデノシルメチオニンとは直接に相互作用していないにもかかわらずS-アデノシルメチオニンとの結合能も低下していた.この変異の影響をシミュレーションしたところ,S-アデノシルメチオニンと直接に結合するアミノ酸残基との相互作用が減弱していた.この結果から,TrmDとtRNAとの接触を阻害するとS-アデノシルメチオニンとの結合能が低下することが示された.つぎに,S-アデノシルメチオニンのメチオニン部位と相互作用するアミノ酸残基に着目した.なかでも,Ser170をAlaに置換したTrmD変異体においてS-アデノシルメチオニンとの結合能,tRNAとの結合能,メチル基の転移活性のすべてが低下していた.169番目のアミノ酸残基はメチル基の転移にかかわる一般塩基としてはたらき,171番目のアミノ酸残基はS-アデノシルメチオニンとの結合にともない構造を安定化することから,S-アデノシルメチオニンとの結合が活性中心の形成の引き金になることが示唆された.
TrmDのAsp169はtRNAの37位のGのN1位,TrmDのArg154はtRNAの37位のGのO6位とそれぞれ相互作用しtRNAの37位のGを安定化する.Arg154をAlaに置換したTrmD変異体およびAsp169をAlaに置換したTrmD変異体は,tRNAとの結合能およびメチル基の転移活性が大幅に低下していたが,S-アデノシルメチオニンとの結合能に影響はなかった.これらの結果から,tRNAの37位のGと結合することがtRNA全体の安定化に寄与し,ひとたびtRNAの37位のGが安定化すればS-アデノシルメチオニンの位置をさらに微調整することなくメチル基の転移は進むと考えられた.
おわりに
筆者らは,現在まで,ヒトそのほか哺乳類に影響のない抗生物質の開発をめざしてTrmDおよびTrm5の特性を評価してきた10,11).この研究における特筆すべき点は,TrmDのみを特異的に標的とする戦略に新たな知見をあたえたことである.S-アデノシルメチオニンがTrmDの結び目構造と結合することがシグナル伝達の起点であったことから,TrmDの理想的な阻害剤は,結び目構造を標的とし,折れ曲がったS-アデノシルメチオニンの代わりにピッタリと入り込むようなものであると考えられる.このような阻害の様式は,S-アデノシルメチオニンを基質とするTrm5を含むほかの酵素への影響が少ないと予想される.筆者らの研究室においては,別途,TrmDを選択的に阻害する物質をスクリーニングしており,これらの知見が総合的に新規の抗生物質の開発に資することを目標としている.
文 献
- Hauenstein, S. I. & Perona, J. J.: Redundant synthesis of cysteinyl-tRNACys in Methanosarcina mazei. J. Biol. Chem., 283, 22007-22017 (2008)[PubMed]
- Perret, V., Garcia, A., Grosjean, H. et al.: Relaxation of a transfer RNA specificity by removal of modified nucleotides. Nature, 344, 787-789 (1990)[PubMed]
- Urbonavicius, J., Qian, Q., Durand, J. M. et al.: Improvement of reading frame maintenance is a common function for several tRNA modifications. EMBO J., 20, 4863-4873 (2001)[PubMed]
- Persson, B. C., Bylund, G. O., Berg, D. E. et al.: Functional analysis of the ffh-trmD region of the Escherichia coli chromosome by using reverse genetics. J. Bacteriol., 177, 5554-5560 (1995)[PubMed]
- O'Dwyer, K., Watts, J. M., Biswas, S. et al.: Characterization of Streptococcus pneumoniae TrmD, a tRNA methyltransferase essential for growth. J. Bacteriol., 186, 2346-2354 (2004)[PubMed]
- Elkins, P. A., Watts, J. M., Zalacain, M. et al.: Insights into catalysis by a knotted TrmD tRNA methyltransferase. J. Mol. Biol., 333, 931-949 (2003)[PubMed]
- Goto-Ito, S., Ito, T., Ishii, R. et al.: Crystal structure of archaeal tRNA(m1G37)methyltransferase aTrm5. Proteins, 72, 1274-1289 (2008)[PubMed]
- Christian, T., Evilia, C., Williams, S. et al.: Distinct origins of tRNA(m1G37) methyltransferase. J. Mol. Biol., 339, 707-719 (2004)[PubMed]
- Ito, T., Masuda, I., Yoshida, K. et al.: Structural basis for methyl-donor-dependent and sequence-specific binding to tRNA substrates by knotted methyltransferase TrmD. Proc. Natl. Acad. Sci. USA, 112, E4197-E4205 (2015)[PubMed]
- Sakaguchi, R., Giessing, A., Dai, Q. et al.: Recognition of guanosine by dissimilar tRNA methyltransferases. RNA, 18, 1687-1701 (2012)[PubMed]
- Sakaguchi, R., Lahoud, G., Christian, T. et al.: A divalent metal ion-dependent N1-methyl transfer to G37-tRNA. Chem. Biol., 21, 1351-1360 (2014)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2009年 京都大学大学院エネルギー科学研究科 修了,同年 米国Thomas Jefferson大学 博士研究員を経て,2013年より京都大学物質-細胞統合システム拠点 特定助教.
研究テーマ:細胞におけるシグナル伝達物質およびシグナル伝達経路の可視化.
抱負:健康第一で研究に取り組んでいます.
Ya-Ming Hou
米国Thomas Jefferson大学 教授.
研究室URL:http://www.houlaboratory.org/
© 2016 坂口怜子・Ya-Ming Hou Licensed under CC 表示 2.1 日本