CRISPR-Cas9系を応用した特定のゲノム領域をDNA脱メチル化する手法の開発
森田純代・堀居拓郎・畑田出穂
(群馬大学生体調節研究所 附属生体情報ゲノムリソースセンターゲノム科学リソース分野)
email:畑田出穂
DOI: 10.7875/first.author.2016.095
Targeted DNA demethylation in vivo using dCas9-peptide repeat and scFv-TET1 catalytic domain fusions.
Sumiyo Morita, Hirofumi Noguchi, Takuro Horii, Kazuhiko Nakabayashi, Mika Kimura, Kohji Okamura, Atsuhiko Sakai, Hideyuki Nakashima, Kenichiro Hata, Kinichi Nakashima, Izuho Hatada
Nature Biotechnology, 34, 1060-1065 (2016)
DNAメチル化やヒストンの修飾などのエピジェネティックな変化は,発生や分化,さらには,がんや生活習慣病といったさまざまな疾患の基盤になることが明らかにされてきたが,特定のゲノム領域のエピジェネティックな状態を操作する技術はまだ発展の途上にある.今回,筆者らは,ゲノム編集技術に用いられるCRISPR-Cas9系を応用し,SunTag系と組み合わせることにより,特定のゲノム領域を効率的にDNA脱メチル化する手法を開発した.SunTag系は5アミノ酸残基のリンカーにより分割された10コピーのGCN4から構成されるが,リンカーの長さを22アミノ酸残基に改変することによりDNA脱メチル化の効率は上昇し,9つの遺伝子座において試験した結果,DNA脱メチル化の効率は7つの遺伝子座において50%以上,そのうち4つの遺伝子座において90%をこえた.さらに,この系をin vivoに適用することにも成功した.
エピジェネティックな修飾のひとつであるDNAメチル化は発生や分化における遺伝子の発現制御において重要な役割をもち,さらには,がんや生活習慣病といったさまざまな疾患の基盤になることが明らかにされてきた.しかしながら,これまで特定のゲノム領域におけるDNAメチル化あるいはDNA脱メチル化を操作する技術はなかったため,特定の遺伝子だけをDNAメチル化あるいはDNA脱メチル化させたモデル動物を作製して詳細に解析することは困難であった.また,アザシチジンやデシタビンといったDNAメチラーゼの阻害剤は骨髄異型性症候群などの治療において非常に効果があるが,広範囲にDNA脱メチル化が起こるため副作用のリスクが指摘されていた.
近年,ZFN系1) あるいはTALEN系2) につづくゲノム編集技術としてCRISPR-Cas9系3-5) が注目をあつめている.これは,従来のゲノム編集技術と比較して標的配列のデザインが簡便であり,配列ごとに異なるタンパク質を合成する必要がないという特徴がある.このゲノム編集技術を応用し,エピジェネティックな修飾の状態のみを改変する“エピゲノム編集”技術の開発が待望されていた.今回,筆者らは,CRISPR-Cas9系を応用し,SunTag系と組み合わせることにより,特定のゲノム領域を効率的にDNA脱メチル化する手法を開発することに成功した.
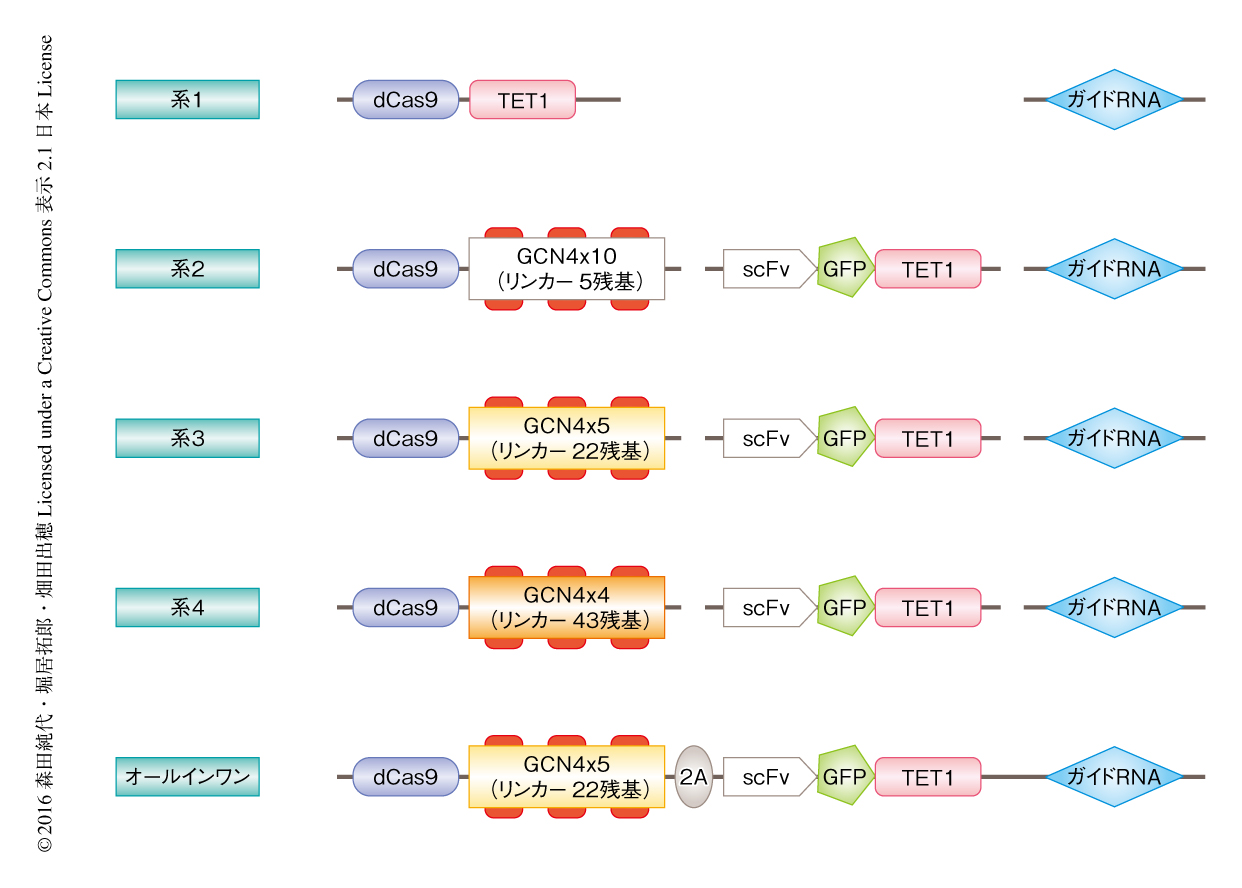
標的配列を含むガイドRNAと複合体を形成して標的配列と結合するがDNA切断活性のないdCas9と,DNA脱メチル化酵素であるTET1とを融合させたものを系1として(図1),マウスのES細胞においてGfap遺伝子のプロモーター領域のDNA脱メチル化を試みた.Gfap遺伝子はアストロサイトの分化に重要な遺伝子であり,胎生中期の神経前駆細胞においてそのプロモーター領域は高度にDNAメチル化されているが,胎生後期においてこの領域にDNA脱メチル化が起こり神経前駆細胞はアストロサイトに分化することが可能になる6).マウスのES細胞においてもGfap遺伝子のプロモーター領域は高度にDNAメチル化されていた.この系1の細胞への導入ではDNA脱メチル化の効率は約10%にとどまり,さらなる改善の余地があると考えられた.
ひとつのdCas9に対し複数のTET1が結合すればDNA脱メチル化の効率も上昇するのではないかと考え,SunTag系7) を組み合わせた(図2a).SunTag系は5アミノ酸残基のリンカーにより分割された10コピーのGCN4から構成され,ミニ抗体によりこのGCN4が認識される.そこで,dCas9の下流にSunTag系をつないだものと,GCN4を認識して結合するミニ抗体にTET1を融合したものを系2として(図1),これらを同時に細胞に導入し,DNA脱メチル化の効率を調べた.しかし,予想に反し,系1と比較してDNA脱メチル化の効率は上昇しなかった.その原因として,リンカーが短いためTET1が機能するだけの空間がなく,TET1どうしが立体障害を起こしてうまく機能しないのではないかと考えた(図2b).そこで,リンカーを22アミノ酸残基としたものを系3,および,43アミノ酸残基としたものを系4とした(図1).系3あるいは系4の細胞への導入において,DNA脱メチル化の効率はそれぞれ約40%あるいは約30%にまで上昇した.また,これらのDNA脱メチル化はTET1の過剰な発現によるものでないことが確認された.
系3を導入した細胞を,ミニ抗体に組み込まれたGFPを指標にして蛍光セルソーターにより分別したところ,DNA脱メチル化の効率は90%をこえた.さらに効率を上昇させる目的で,系3をもとにオールインワンベクターを作製し(図1),これを導入した細胞を同様に分別したところ,DNA脱メチル化の効率は90%をこえた.これらのことから,トランスフェクションするベクターが複数に分かれていても,この系においてはGFPを指標として分別することで十分であると考えられた.また,DNA脱メチル化は少なくともgRNAの標的配列から200 bpほど離れた領域にまで及んでいた.
この系において非特異的なDNA脱メチル化が起こっていないか調べるため,全ゲノムバイサルファイトシーケンス法8) によりDNAメチル化について解析した.その結果,gRNAの標的配列と同じ程度にDNA脱メチル化されているゲノム領域はなかった.またRNA-seq法により遺伝子の発現を解析したところ,変化はほとんどみられなかった.
以上は,マウスのES細胞においてGfap遺伝子のプロモーター領域をDNA脱メチル化したときの結果であるが,この系はマウスの神経前駆細胞やヒトの細胞にも応用が可能であった.9つの遺伝子座において検討した結果,7つの遺伝子座においてDNA脱メチル化の効率は50%以上であり,そのうち4つの遺伝子座において90%以上であった.さらに,DNA脱メチル化によりこれらの遺伝子の発現が誘導された.また,複数のgRNAを導入することにより広いゲノム領域をDNA脱メチル化することも可能であった.
この系がin vivoにおいても機能するかどうか検討するため,マウスの胎仔の脳においてGfap遺伝子のプロモーター領域のDNA脱メチル化を試みた.さきに述べたように,胎生中期の神経前駆細胞においてGfap遺伝子のプロモーター領域は高度にDNAメチル化されているが,胎生後期においてこの領域にDNA脱メチル化が起こり,Gfap遺伝子の発現は上昇しアストロサイトに分化する.この領域がDNA脱メチル化されない神経前駆細胞はニューロンへの分化が可能になる.胎生14日目において母体から胎仔を取り出し,オールインワンベクターを脳室帯にエレクトロポレーションした.胎仔を母体にもどして1日後にはGfap遺伝子のプロモーター領域はDNA脱メチル化され,4日後にはGFAPのタンパク質レベルでの発現が上昇していた.したがって,in vivoにおいても,この系は特定のゲノム領域をDNA脱メチル化することが明らかにされた.
筆者らは,CRISPR-Cas9系を応用し,dCas9とSunTag系を組み合わせることにより,特定のゲノム領域をきわめて効率的にDNA脱メチル化する系を開発した.さらに,この系をin vivoに適用することにも成功した.
すでにZFN系やTALEN系などによるゲノム編集技術を応用したDNA脱メチル化の手法について報告されているが9,10),筆者らの開発した系と比較して,そのDNA脱メチル化の効率はあまり高くなく,DNA脱メチル化される領域も30 bp程度とせまい.その理由として,2つのことが考えられる.ひとつは,Cas9はZFNやTALENと比較してDNAメチル化されたゲノム領域と結合しやすいことが考えられる11-13).つまり,Cas9はDNAの修飾に影響されることなく標的配列と結合する.もうひとつは,筆者らの開発した系は複数のTET1をリクルートするため,標的配列を含むより広い範囲で効率的にDNA脱メチル化が起こることが考えられる.この技術を用いて,将来的にはDNA脱メチル化を必要とする遺伝子治療への応用が可能ではないかと考えている.
略歴:2000年 東京大学大学院医学系研究科 修了,同年より群馬大学生体調節研究所 研究員.
研究テーマ:エピジェネティクス.
堀居 拓郎(Takuro Horii)
群馬大学生体調節研究所 助教.
畑田 出穂(Izuho Hatada)
群馬大学生体調節研究所 教授.
研究室URL:http://epigenome.dept.showa.gunma-u.ac.jp/~hatada/
© 2016 森田純代・堀居拓郎・畑田出穂 Licensed under CC 表示 2.1 日本
(群馬大学生体調節研究所 附属生体情報ゲノムリソースセンターゲノム科学リソース分野)
email:畑田出穂
DOI: 10.7875/first.author.2016.095
Targeted DNA demethylation in vivo using dCas9-peptide repeat and scFv-TET1 catalytic domain fusions.
Sumiyo Morita, Hirofumi Noguchi, Takuro Horii, Kazuhiko Nakabayashi, Mika Kimura, Kohji Okamura, Atsuhiko Sakai, Hideyuki Nakashima, Kenichiro Hata, Kinichi Nakashima, Izuho Hatada
Nature Biotechnology, 34, 1060-1065 (2016)
要 約
DNAメチル化やヒストンの修飾などのエピジェネティックな変化は,発生や分化,さらには,がんや生活習慣病といったさまざまな疾患の基盤になることが明らかにされてきたが,特定のゲノム領域のエピジェネティックな状態を操作する技術はまだ発展の途上にある.今回,筆者らは,ゲノム編集技術に用いられるCRISPR-Cas9系を応用し,SunTag系と組み合わせることにより,特定のゲノム領域を効率的にDNA脱メチル化する手法を開発した.SunTag系は5アミノ酸残基のリンカーにより分割された10コピーのGCN4から構成されるが,リンカーの長さを22アミノ酸残基に改変することによりDNA脱メチル化の効率は上昇し,9つの遺伝子座において試験した結果,DNA脱メチル化の効率は7つの遺伝子座において50%以上,そのうち4つの遺伝子座において90%をこえた.さらに,この系をin vivoに適用することにも成功した.
はじめに
エピジェネティックな修飾のひとつであるDNAメチル化は発生や分化における遺伝子の発現制御において重要な役割をもち,さらには,がんや生活習慣病といったさまざまな疾患の基盤になることが明らかにされてきた.しかしながら,これまで特定のゲノム領域におけるDNAメチル化あるいはDNA脱メチル化を操作する技術はなかったため,特定の遺伝子だけをDNAメチル化あるいはDNA脱メチル化させたモデル動物を作製して詳細に解析することは困難であった.また,アザシチジンやデシタビンといったDNAメチラーゼの阻害剤は骨髄異型性症候群などの治療において非常に効果があるが,広範囲にDNA脱メチル化が起こるため副作用のリスクが指摘されていた.
近年,ZFN系1) あるいはTALEN系2) につづくゲノム編集技術としてCRISPR-Cas9系3-5) が注目をあつめている.これは,従来のゲノム編集技術と比較して標的配列のデザインが簡便であり,配列ごとに異なるタンパク質を合成する必要がないという特徴がある.このゲノム編集技術を応用し,エピジェネティックな修飾の状態のみを改変する“エピゲノム編集”技術の開発が待望されていた.今回,筆者らは,CRISPR-Cas9系を応用し,SunTag系と組み合わせることにより,特定のゲノム領域を効率的にDNA脱メチル化する手法を開発することに成功した.
1.dCas9とTET1との融合タンパク質では効率的なDNA脱メチル化は起こらない
標的配列を含むガイドRNAと複合体を形成して標的配列と結合するがDNA切断活性のないdCas9と,DNA脱メチル化酵素であるTET1とを融合させたものを系1として(図1),マウスのES細胞においてGfap遺伝子のプロモーター領域のDNA脱メチル化を試みた.Gfap遺伝子はアストロサイトの分化に重要な遺伝子であり,胎生中期の神経前駆細胞においてそのプロモーター領域は高度にDNAメチル化されているが,胎生後期においてこの領域にDNA脱メチル化が起こり神経前駆細胞はアストロサイトに分化することが可能になる6).マウスのES細胞においてもGfap遺伝子のプロモーター領域は高度にDNAメチル化されていた.この系1の細胞への導入ではDNA脱メチル化の効率は約10%にとどまり,さらなる改善の余地があると考えられた.
2.SunTag系を組み合わせることによりDNA脱メチル化の効率は上昇する
ひとつのdCas9に対し複数のTET1が結合すればDNA脱メチル化の効率も上昇するのではないかと考え,SunTag系7) を組み合わせた(図2a).SunTag系は5アミノ酸残基のリンカーにより分割された10コピーのGCN4から構成され,ミニ抗体によりこのGCN4が認識される.そこで,dCas9の下流にSunTag系をつないだものと,GCN4を認識して結合するミニ抗体にTET1を融合したものを系2として(図1),これらを同時に細胞に導入し,DNA脱メチル化の効率を調べた.しかし,予想に反し,系1と比較してDNA脱メチル化の効率は上昇しなかった.その原因として,リンカーが短いためTET1が機能するだけの空間がなく,TET1どうしが立体障害を起こしてうまく機能しないのではないかと考えた(図2b).そこで,リンカーを22アミノ酸残基としたものを系3,および,43アミノ酸残基としたものを系4とした(図1).系3あるいは系4の細胞への導入において,DNA脱メチル化の効率はそれぞれ約40%あるいは約30%にまで上昇した.また,これらのDNA脱メチル化はTET1の過剰な発現によるものでないことが確認された.
系3を導入した細胞を,ミニ抗体に組み込まれたGFPを指標にして蛍光セルソーターにより分別したところ,DNA脱メチル化の効率は90%をこえた.さらに効率を上昇させる目的で,系3をもとにオールインワンベクターを作製し(図1),これを導入した細胞を同様に分別したところ,DNA脱メチル化の効率は90%をこえた.これらのことから,トランスフェクションするベクターが複数に分かれていても,この系においてはGFPを指標として分別することで十分であると考えられた.また,DNA脱メチル化は少なくともgRNAの標的配列から200 bpほど離れた領域にまで及んでいた.
この系において非特異的なDNA脱メチル化が起こっていないか調べるため,全ゲノムバイサルファイトシーケンス法8) によりDNAメチル化について解析した.その結果,gRNAの標的配列と同じ程度にDNA脱メチル化されているゲノム領域はなかった.またRNA-seq法により遺伝子の発現を解析したところ,変化はほとんどみられなかった.
以上は,マウスのES細胞においてGfap遺伝子のプロモーター領域をDNA脱メチル化したときの結果であるが,この系はマウスの神経前駆細胞やヒトの細胞にも応用が可能であった.9つの遺伝子座において検討した結果,7つの遺伝子座においてDNA脱メチル化の効率は50%以上であり,そのうち4つの遺伝子座において90%以上であった.さらに,DNA脱メチル化によりこれらの遺伝子の発現が誘導された.また,複数のgRNAを導入することにより広いゲノム領域をDNA脱メチル化することも可能であった.
3.この系はin vivoにおいても機能する
この系がin vivoにおいても機能するかどうか検討するため,マウスの胎仔の脳においてGfap遺伝子のプロモーター領域のDNA脱メチル化を試みた.さきに述べたように,胎生中期の神経前駆細胞においてGfap遺伝子のプロモーター領域は高度にDNAメチル化されているが,胎生後期においてこの領域にDNA脱メチル化が起こり,Gfap遺伝子の発現は上昇しアストロサイトに分化する.この領域がDNA脱メチル化されない神経前駆細胞はニューロンへの分化が可能になる.胎生14日目において母体から胎仔を取り出し,オールインワンベクターを脳室帯にエレクトロポレーションした.胎仔を母体にもどして1日後にはGfap遺伝子のプロモーター領域はDNA脱メチル化され,4日後にはGFAPのタンパク質レベルでの発現が上昇していた.したがって,in vivoにおいても,この系は特定のゲノム領域をDNA脱メチル化することが明らかにされた.
おわりに
筆者らは,CRISPR-Cas9系を応用し,dCas9とSunTag系を組み合わせることにより,特定のゲノム領域をきわめて効率的にDNA脱メチル化する系を開発した.さらに,この系をin vivoに適用することにも成功した.
すでにZFN系やTALEN系などによるゲノム編集技術を応用したDNA脱メチル化の手法について報告されているが9,10),筆者らの開発した系と比較して,そのDNA脱メチル化の効率はあまり高くなく,DNA脱メチル化される領域も30 bp程度とせまい.その理由として,2つのことが考えられる.ひとつは,Cas9はZFNやTALENと比較してDNAメチル化されたゲノム領域と結合しやすいことが考えられる11-13).つまり,Cas9はDNAの修飾に影響されることなく標的配列と結合する.もうひとつは,筆者らの開発した系は複数のTET1をリクルートするため,標的配列を含むより広い範囲で効率的にDNA脱メチル化が起こることが考えられる.この技術を用いて,将来的にはDNA脱メチル化を必要とする遺伝子治療への応用が可能ではないかと考えている.
文 献
- Porteus, M. H. & Carroll, D.: Gene targeting using zinc finger nucleases. Nat. Biotechnol., 23, 967-973 (2005)[PubMed]
- Miller, J. C., Tan, S., Qiao, G. et al.: A TALE nuclease architecture for efficient genome editing. Nat. Biotechnol., 29, 143-148 (2011)[PubMed]
- Jinek, M., Chylinski, K., Fonfara, I. et al.: A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 337, 816-821 (2012)[PubMed]
- Mali, P., Yang, L., Esvelt, K. M. et al.: RNA-guided human genome engineering via Cas9. Science, 339, 823-826 (2013)[PubMed]
- Cong, L., Ran, F. A., Cox, D. et al.: Multiplex genome engineering using CRISPR/Cas systems. Science, 339, 819-823 (2013)[PubMed]
- Takizawa, T., Nakashima, K., Namihira, M. et al.: DNA methylation is a critical cell-intrinsic determinant of astrocyte differentiation in the fetal brain. Dev. Cell, 1, 749-758 (2001)[PubMed]
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S. et al.: A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell, 159, 635-646 (2014)[PubMed]
- Miura, F., Enomoto, Y., Dairiki, R. et al.: Amplification-free whole-genome bisulfite sequencing by post-bisulfite adaptor tagging. Nucleic Acids Res., 40, e136 (2012)[PubMed]
- Chen, H., Kazemier, H. G., de Groote, M. L. et al.: Induced DNA demethylation by targeting Ten-Eleven Translocation 2 to the human ICAM-1 promoter. Nucleic Acids Res., 42, 1563-1574 (2014)[PubMed]
- Maeder, M. L., Angstman, J. F., Richardson, M. E. et al.: Targeted DNA demethylation and activation of endogenous genes using programmable TALE-TET1 fusion proteins. Nat. Biotechnol., 31, 1137-1142 (2013)[PubMed]
- Hsu, P. D., Scott, D. A., Weinstein, J. A. et al.: DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol., 31, 827-832 (2013)[PubMed]
- Bultmann, S., Morbitzer, R., Schmidt, C. S. et al.: Targeted transcriptional activation of silent oct4 pluripotency gene by combining designer TALEs and inhibition of epigenetic modifiers. Nucleic Acids Res., 40, 5368-5377 (2012)[PubMed]
- Chen, S., Oikonomou, G., Chiu, C. N. et al.: A large-scale in vivo analysis reveals that TALENs are significantly more mutagenic than ZFNs generated using context-dependent assembly. Nucleic Acids Res., 41, 2769-2778 (2013)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2000年 東京大学大学院医学系研究科 修了,同年より群馬大学生体調節研究所 研究員.
研究テーマ:エピジェネティクス.
堀居 拓郎(Takuro Horii)
群馬大学生体調節研究所 助教.
畑田 出穂(Izuho Hatada)
群馬大学生体調節研究所 教授.
研究室URL:http://epigenome.dept.showa.gunma-u.ac.jp/~hatada/
© 2016 森田純代・堀居拓郎・畑田出穂 Licensed under CC 表示 2.1 日本