ゲノムワイド関連解析を用いたイネにおける農業形質にかかわる新規の遺伝子の同定
矢野憲司・松岡 信
(名古屋大学生物機能開発利用研究センター 植物分子育種分野)
email:矢野憲司,松岡 信
DOI: 10.7875/first.author.2016.073
Genome-wide association study using whole-genome sequencing rapidly identifies new genes influencing agronomic traits in rice.
Kenji Yano, Eiji Yamamoto, Koichiro Aya, Hideyuki Takeuchi, Pei-ching Lo, Li Hu, Masanori Yamasaki, Shinya Yoshida, Hidemi Kitano, Ko Hirano, Makoto Matsuoka
Nature Genetics, 48, 927-934 (2016)
作物の育種において,有用な遺伝子の同定は収量の増加やそれにかかわる遺伝的な機構を解明するためにきわめて重要である.これまで,ゲノムワイド関連解析はヒトの疾患にかかわる遺伝子の同定や疾患の機構の解明に重要な貢献をはたしてきたが,作物を材料とした遺伝子の同定はまれであった.そこで,筆者らは,ゲノムワイド関連解析に用いる集団の選択や解析の手法を改良することにより,イネにおいて有用な形質にかかわる遺伝子の同定を試み,開花やひとつの穂あたりの粒の数などにかかわる複数の遺伝子を同定することに成功した.この結果から,作物においてもゲノムワイド関連解析が迅速な遺伝子の同定に有効であることが示された.さらに,この研究で用いた手法はほかの作物にも応用することが可能であり,作物において新規の遺伝子の同定が加速することが期待される.
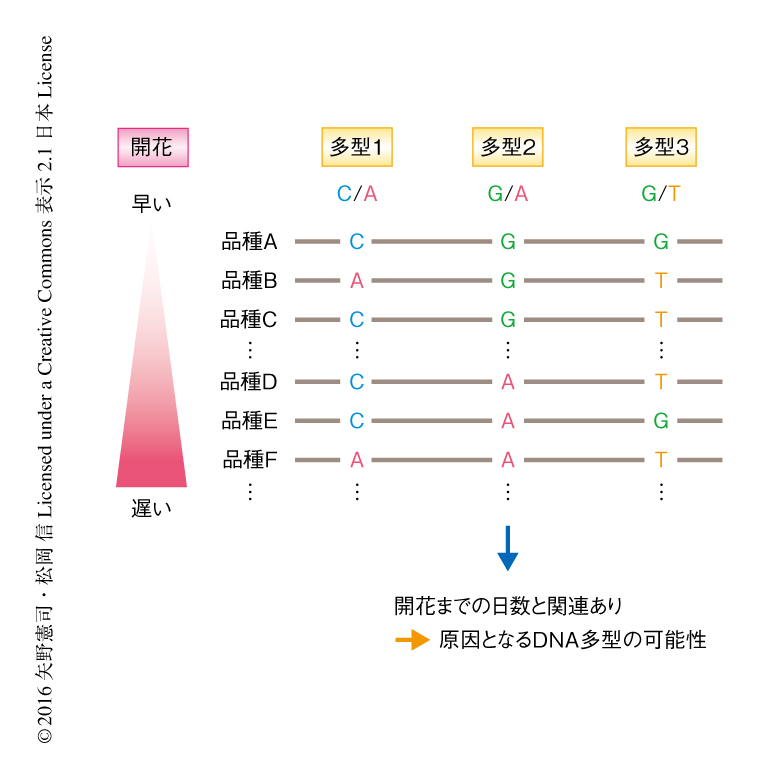
ゲノムワイド関連解析(genome-wide association study:GWAS)とは,ある集団を設定し,集団に存在する個体のあいだの形質の違いとDNA配列の違いとの関連をゲノム全体にわたり調べることにより,対象とする形質と関連するDNA多型を統計的に検出する手法である(図1).作物において品種ごとに異なる収量や病気抵抗性などの形質は,おのおのの品種のもつゲノムの遺伝的な変異に由来する.作物の育種の過程において,ある遺伝的な変異が有益な形質をもたらした場合にはその変異は選抜されるが,選抜の目的や環境により有益な表現型は異なるため,育種された品種のゲノムは多様性をもつことになる.このような集団に対するゲノムワイド関連解析により,ゲノム全体のなかから標的とした形質にかかわるDNA多型を効率的にみつけだすことが期待できる.
近年,次世代シークエンサーの発達にともない,多数の個体のゲノム全体にわたるDNA多型を検出できるようになり,高精度なゲノムワイド関連解析が可能になった1).一方,これまでにも作物に対するゲノムワイド関連解析は報告されているが,イネなどの自殖性の作物においてゲノムワイド関連解析により原因遺伝子の特定までいたったケースは必ずしも多くない.筆者らは,この研究において,遺伝的な多様性が低いにもかかわらず表現型の多様性が確保された日本において育成されたイネの集団に対しゲノムワイド関連解析を適用することにより,効率的な遺伝子の同定が可能であることを示した.
イネに対するゲノムワイド関連解析により遺伝子の同定にいたったケースがほとんどなかった要因はさまざま議論されるが,なかでも,ゲノムワイド関連解析に用いた集団の遺伝的な多様性が高すぎるため,形質とは関連のないDNA多型が偽陽性として検出されたことが指摘されていた1,2).そこで,遺伝的な多様性が少ないと予想された日本において育成された温帯ジャポニカ種176品種に着目した.実際に,全ゲノム情報を用いて系統樹を作成したところ,世界各地で生育するイネの集団と比較して,この集団の遺伝的な多様性は非常に限定的であることが確認された.その一方で,この集団のもつ開花までの日数,草丈,穂の長さ,葉の幅など形質における多様性は,ジャポニカ種およびインディカ種を含む集団とほぼ同じ程度であった.この結果から,日本において育成されたイネの集団においては遺伝的な多様性が低いにもかかわらず表現型の高い多様性が維持されており,ゲノムワイド関連解析に適した集団であることが示された.
これまでの研究により,イネの開花にかかわる遺伝子やその原因となるDNA多型は詳細に調べられている3).そこで,日本において育成されたイネの集団を用いたゲノムワイド関連解析の有効性を確認するため,開花にかかわる既知の遺伝子が同定されるかどうか検証した.その結果,開花に対するゲノムワイド関連解析により,第1染色体,第3染色体,第6染色体,第7染色体,第11染色体の5箇所に開花と関連があると予想されるDNA多型が検出された.既知の遺伝子のゲノムにおける位置と検出されたDNA多型の位置を比較したところ,第3染色体に座乗するHd6遺伝子および第7染色体に座乗するHd2遺伝子がゲノムワイド関連解析により検出されたDNA多型の位置と一致した.この領域のDNA配列をくわしく調べた結果,それぞれの領域において複数のDNA多型が見い出された.
イネなど自家受粉する種は,トウモロコシなど他家受粉する種に比べ異型接合体のあいだの組換えが少ないため,原因となる変異に隣接したDNA多型がつぎの世代へいっしょに伝達される.その結果,ゲノムワイド関連解析において原因となる変異に隣接した複数のDNA多型と表現型との関連が検出されるため,表現型に違いをもたらすようなDNA多型をみつけにくいことが指摘されていた4).そこで,表現型に違いをもたらすDNA多型を直接的に検出する手はじめとして,候補となる領域に座乗する遺伝子におけるDNA多型に着目した.候補となる領域のすべてのDNA多型の位置と遺伝子の座乗する位置とを比較して,遺伝子の機能におよぼす効果について予想した.たとえば,第3染色体に検出された複数のDNA多型の場合,アミノ酸配列に変異をもたらすようなDNA多型はHd6遺伝子の翻訳領域において終止コドンへの置換をもたらすひとつのみであった.以前の報告により,同様のDNA多型がHd6遺伝子の機能に影響をおよぼし開花を早めることが知られており5),このDNA多型により176のイネの集団を分類して開花を比較したところ,終止コドンをもたらすDNA多型をもつグループは開花の早いことが確認された.さらに,第7染色体に検出された複数のDNA多型についても同様に解析したところ,アミノ酸配列に変異をもたらすようなDNA多型はHd2遺伝子に見い出されたひとつのみであった.このDNA多型についても以前に開花にかかわることが報告されていたことから6),開花に影響するDNA多型の有力な候補と考えられた.以上の結果から,遺伝子の座乗する領域に生じたDNA多型を網羅的に調べる手法は,形質に影響をおよぼすDNA多型を探しだす手段として有効であることが示された.
ゲノムワイド関連解析により開花ともっとも強い関連を示した第1染色体のDNA多型の検出を試みた.第1染色体の候補となる領域には開花と関連する91のDNA多型が存在し,このうち,アミノ酸配列に変異をもたらすDNA多型は8つであり,そのうち7つはトランスポゾンをコードする遺伝子に存在し,残りのひとつはヌクレオチジルトランスフェラーゼ遺伝子に存在した.このイネのヌクレオチジルトランスフェラーゼ遺伝子と高い配列相同性をもつシロイヌナズナのHESO1遺伝子は,開花に影響することが報告されていた7).そこで,この遺伝子が実際にイネの開花に関係するかどうかを確認するため,異なるDNA多型をもつこの遺伝子をイネに導入して形質転換させたところ,そのうちひとつのDNA多型をもつ遺伝子を導入したイネにおいて開花が遅延した.この結果から,イネにおいてもこのヌクレオチジルトランスフェラーゼ遺伝子が開花を制御することが確認された.さらに,同様の解析により,第11染色体に開花,草丈,穂の長さにかかわる転写因子をコードする新規の遺伝子,第4染色体に1株あたりの穂の数,1穂あたりの種子の数,葉の幅にかかわる遺伝子が同定された.以上の結果から,今回のゲノムワイド関連解析を用いた手法は新規の遺伝子の同定にも有効であることが確認された.
ゲノムワイド関連解析により開花に関連するとして第6染色体に検出されたDNA多型の近傍には,育種の過程において開花期の制御に重要な役割をはたすHd1遺伝子が位置し8),この領域に検出された開花との関連はHd1遺伝子に起因することが予想された.しかし,開花に関連を示したDNA多型の位置とHd1遺伝子の座乗する位置を詳細に比較したところ約1 Mbpの距離があり,同一の連鎖ブロックには座乗しないことが判明した.一方,DNA多型によりHd1遺伝子を分類したところ11種類になった.通常のゲノムワイド関連解析においてはすべてのDNA多型と表現型との関連を機械的に調べるため,ひとつの遺伝子に表現型を変化させる複数のDNA多型があった場合,表現型との関連が検出されにくくなると想定される9).この問題を改善するため,ひとつの遺伝子に生じた複数のDNA多型については遺伝子ごとにマーカーをわりあて,ゲノムワイド関連解析にこのマーカーセットを用いた結果,Hd1遺伝子と開花との強い関連が示された.
そこで,同様の手法により,芒(のぎ)の長さに関連するDNA多型の検出を試みた.芒は収穫のさまたげとなるため無芒化は育種の重要な目標である.また,用いたイネの集団における無芒化は,育種の系譜から少なくとも独立に2回にわたり起こったことが示されており,同一の遺伝子に複数の変異の存在する可能性が予想された.そこで,遺伝子ごとのマーカーセットを用いたゲノムワイド関連解析により,芒の長さに関連するDNA多型を検出したところ,通常のマーカーセットを用いたゲノムワイド関連解析においては検出されなかった強い関連を示す遺伝子が検出された.この遺伝子はEPFLと名づけられたタンパク質をコードしていた.この遺伝子について,機能獲得型と予想された対立遺伝子を芒のないイネの品種に導入したところ,芒が形成された.これらの結果から,原因遺伝子に複数のハプロタイプの存在が想定される場合には,遺伝子ごとに変換したマーカーを用いたゲノムワイド関連解析が有効であると結論された.
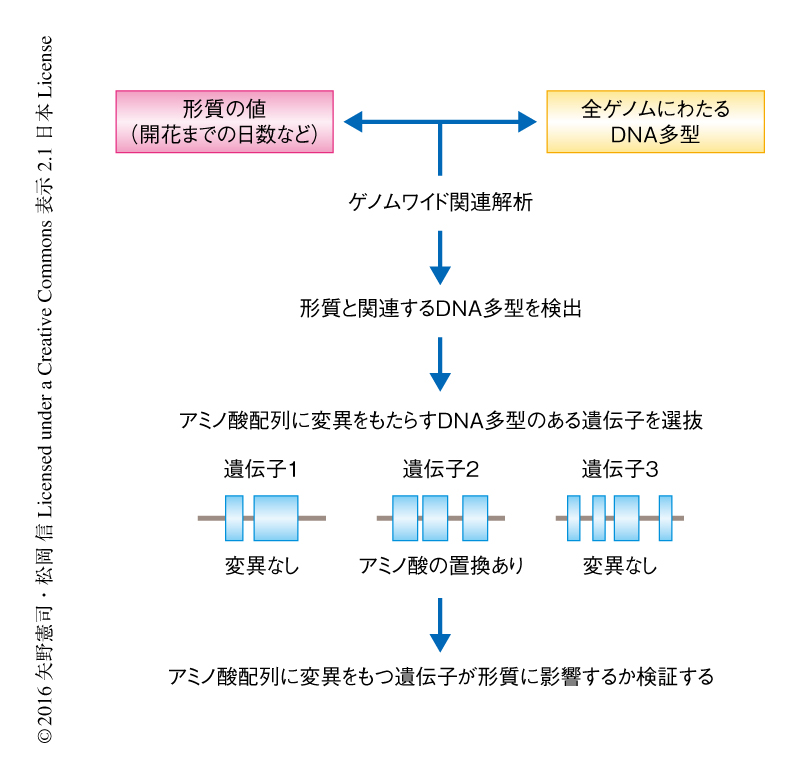
今回の研究により,ゲノムワイド関連解析に用いる集団の選択や解析の手法をくふうすることにより,作物においても効率的に新規の遺伝子を同定することが可能であることが確認された.しかし,今回の手法は,作物に対するゲノムワイド関連解析において,すべてのケースに適用できるわけではない.この研究において,候補となるDNA多型を選抜する際には,遺伝子の機能に影響する可能性の高いことからアミノ酸配列の変異をもたらすようなDNA多型に着目した(図2).しかし,遺伝子の機能の相違はアミノ酸配列の変異だけでなく,発現のレベルにおいてももたらされる.実際,最近になり報告された,イネの種子の大きさを対象としたゲノムワイド関連解析においては,複数の候補遺伝子のなかから原因遺伝子を選抜する際に遺伝子の発現情報にもとづく手法が有効であることが示された10).今後は,アミノ酸配列の変異および発現レベルの両方の情報にもとづき候補遺伝子を選抜することで,より汎用的な遺伝子の同定が可能であると考えられる.
略歴:2016年 名古屋大学大学院生命農学研究科博士課程 修了,同年より東京大学大学院農学生命科学研究科 特別研究員.
研究テーマ:ゲノム情報を利用した有用な遺伝子の同定および分子育種.
抱負:作物の栄養生理や養分吸収にかかわる分子機能を解明したい.
松岡 信(Makoto Matsuoka)
名古屋大学生物機能開発利用研究センター 教授.
研究室URL:http://bbc.agr.nagoya-u.ac.jp/~pmb/
© 2016 矢野憲司・松岡 信 Licensed under CC 表示 2.1 日本
(名古屋大学生物機能開発利用研究センター 植物分子育種分野)
email:矢野憲司,松岡 信
DOI: 10.7875/first.author.2016.073
Genome-wide association study using whole-genome sequencing rapidly identifies new genes influencing agronomic traits in rice.
Kenji Yano, Eiji Yamamoto, Koichiro Aya, Hideyuki Takeuchi, Pei-ching Lo, Li Hu, Masanori Yamasaki, Shinya Yoshida, Hidemi Kitano, Ko Hirano, Makoto Matsuoka
Nature Genetics, 48, 927-934 (2016)
この論文に出現する遺伝子・タンパク質のUniprot ID
要 約
作物の育種において,有用な遺伝子の同定は収量の増加やそれにかかわる遺伝的な機構を解明するためにきわめて重要である.これまで,ゲノムワイド関連解析はヒトの疾患にかかわる遺伝子の同定や疾患の機構の解明に重要な貢献をはたしてきたが,作物を材料とした遺伝子の同定はまれであった.そこで,筆者らは,ゲノムワイド関連解析に用いる集団の選択や解析の手法を改良することにより,イネにおいて有用な形質にかかわる遺伝子の同定を試み,開花やひとつの穂あたりの粒の数などにかかわる複数の遺伝子を同定することに成功した.この結果から,作物においてもゲノムワイド関連解析が迅速な遺伝子の同定に有効であることが示された.さらに,この研究で用いた手法はほかの作物にも応用することが可能であり,作物において新規の遺伝子の同定が加速することが期待される.
はじめに
ゲノムワイド関連解析(genome-wide association study:GWAS)とは,ある集団を設定し,集団に存在する個体のあいだの形質の違いとDNA配列の違いとの関連をゲノム全体にわたり調べることにより,対象とする形質と関連するDNA多型を統計的に検出する手法である(図1).作物において品種ごとに異なる収量や病気抵抗性などの形質は,おのおのの品種のもつゲノムの遺伝的な変異に由来する.作物の育種の過程において,ある遺伝的な変異が有益な形質をもたらした場合にはその変異は選抜されるが,選抜の目的や環境により有益な表現型は異なるため,育種された品種のゲノムは多様性をもつことになる.このような集団に対するゲノムワイド関連解析により,ゲノム全体のなかから標的とした形質にかかわるDNA多型を効率的にみつけだすことが期待できる.
近年,次世代シークエンサーの発達にともない,多数の個体のゲノム全体にわたるDNA多型を検出できるようになり,高精度なゲノムワイド関連解析が可能になった1).一方,これまでにも作物に対するゲノムワイド関連解析は報告されているが,イネなどの自殖性の作物においてゲノムワイド関連解析により原因遺伝子の特定までいたったケースは必ずしも多くない.筆者らは,この研究において,遺伝的な多様性が低いにもかかわらず表現型の多様性が確保された日本において育成されたイネの集団に対しゲノムワイド関連解析を適用することにより,効率的な遺伝子の同定が可能であることを示した.
1.ゲノムワイド関連解析に用いた集団
イネに対するゲノムワイド関連解析により遺伝子の同定にいたったケースがほとんどなかった要因はさまざま議論されるが,なかでも,ゲノムワイド関連解析に用いた集団の遺伝的な多様性が高すぎるため,形質とは関連のないDNA多型が偽陽性として検出されたことが指摘されていた1,2).そこで,遺伝的な多様性が少ないと予想された日本において育成された温帯ジャポニカ種176品種に着目した.実際に,全ゲノム情報を用いて系統樹を作成したところ,世界各地で生育するイネの集団と比較して,この集団の遺伝的な多様性は非常に限定的であることが確認された.その一方で,この集団のもつ開花までの日数,草丈,穂の長さ,葉の幅など形質における多様性は,ジャポニカ種およびインディカ種を含む集団とほぼ同じ程度であった.この結果から,日本において育成されたイネの集団においては遺伝的な多様性が低いにもかかわらず表現型の高い多様性が維持されており,ゲノムワイド関連解析に適した集団であることが示された.
2.ゲノムワイド関連解析による遺伝子の迅速な同定
これまでの研究により,イネの開花にかかわる遺伝子やその原因となるDNA多型は詳細に調べられている3).そこで,日本において育成されたイネの集団を用いたゲノムワイド関連解析の有効性を確認するため,開花にかかわる既知の遺伝子が同定されるかどうか検証した.その結果,開花に対するゲノムワイド関連解析により,第1染色体,第3染色体,第6染色体,第7染色体,第11染色体の5箇所に開花と関連があると予想されるDNA多型が検出された.既知の遺伝子のゲノムにおける位置と検出されたDNA多型の位置を比較したところ,第3染色体に座乗するHd6遺伝子および第7染色体に座乗するHd2遺伝子がゲノムワイド関連解析により検出されたDNA多型の位置と一致した.この領域のDNA配列をくわしく調べた結果,それぞれの領域において複数のDNA多型が見い出された.
イネなど自家受粉する種は,トウモロコシなど他家受粉する種に比べ異型接合体のあいだの組換えが少ないため,原因となる変異に隣接したDNA多型がつぎの世代へいっしょに伝達される.その結果,ゲノムワイド関連解析において原因となる変異に隣接した複数のDNA多型と表現型との関連が検出されるため,表現型に違いをもたらすようなDNA多型をみつけにくいことが指摘されていた4).そこで,表現型に違いをもたらすDNA多型を直接的に検出する手はじめとして,候補となる領域に座乗する遺伝子におけるDNA多型に着目した.候補となる領域のすべてのDNA多型の位置と遺伝子の座乗する位置とを比較して,遺伝子の機能におよぼす効果について予想した.たとえば,第3染色体に検出された複数のDNA多型の場合,アミノ酸配列に変異をもたらすようなDNA多型はHd6遺伝子の翻訳領域において終止コドンへの置換をもたらすひとつのみであった.以前の報告により,同様のDNA多型がHd6遺伝子の機能に影響をおよぼし開花を早めることが知られており5),このDNA多型により176のイネの集団を分類して開花を比較したところ,終止コドンをもたらすDNA多型をもつグループは開花の早いことが確認された.さらに,第7染色体に検出された複数のDNA多型についても同様に解析したところ,アミノ酸配列に変異をもたらすようなDNA多型はHd2遺伝子に見い出されたひとつのみであった.このDNA多型についても以前に開花にかかわることが報告されていたことから6),開花に影響するDNA多型の有力な候補と考えられた.以上の結果から,遺伝子の座乗する領域に生じたDNA多型を網羅的に調べる手法は,形質に影響をおよぼすDNA多型を探しだす手段として有効であることが示された.
3.ゲノムワイド関連解析による新規の遺伝子の同定
ゲノムワイド関連解析により開花ともっとも強い関連を示した第1染色体のDNA多型の検出を試みた.第1染色体の候補となる領域には開花と関連する91のDNA多型が存在し,このうち,アミノ酸配列に変異をもたらすDNA多型は8つであり,そのうち7つはトランスポゾンをコードする遺伝子に存在し,残りのひとつはヌクレオチジルトランスフェラーゼ遺伝子に存在した.このイネのヌクレオチジルトランスフェラーゼ遺伝子と高い配列相同性をもつシロイヌナズナのHESO1遺伝子は,開花に影響することが報告されていた7).そこで,この遺伝子が実際にイネの開花に関係するかどうかを確認するため,異なるDNA多型をもつこの遺伝子をイネに導入して形質転換させたところ,そのうちひとつのDNA多型をもつ遺伝子を導入したイネにおいて開花が遅延した.この結果から,イネにおいてもこのヌクレオチジルトランスフェラーゼ遺伝子が開花を制御することが確認された.さらに,同様の解析により,第11染色体に開花,草丈,穂の長さにかかわる転写因子をコードする新規の遺伝子,第4染色体に1株あたりの穂の数,1穂あたりの種子の数,葉の幅にかかわる遺伝子が同定された.以上の結果から,今回のゲノムワイド関連解析を用いた手法は新規の遺伝子の同定にも有効であることが確認された.
4.複雑なゲノムの構造によりもたらされるゲノムワイド関連解析における偽陽性
ゲノムワイド関連解析により開花に関連するとして第6染色体に検出されたDNA多型の近傍には,育種の過程において開花期の制御に重要な役割をはたすHd1遺伝子が位置し8),この領域に検出された開花との関連はHd1遺伝子に起因することが予想された.しかし,開花に関連を示したDNA多型の位置とHd1遺伝子の座乗する位置を詳細に比較したところ約1 Mbpの距離があり,同一の連鎖ブロックには座乗しないことが判明した.一方,DNA多型によりHd1遺伝子を分類したところ11種類になった.通常のゲノムワイド関連解析においてはすべてのDNA多型と表現型との関連を機械的に調べるため,ひとつの遺伝子に表現型を変化させる複数のDNA多型があった場合,表現型との関連が検出されにくくなると想定される9).この問題を改善するため,ひとつの遺伝子に生じた複数のDNA多型については遺伝子ごとにマーカーをわりあて,ゲノムワイド関連解析にこのマーカーセットを用いた結果,Hd1遺伝子と開花との強い関連が示された.
そこで,同様の手法により,芒(のぎ)の長さに関連するDNA多型の検出を試みた.芒は収穫のさまたげとなるため無芒化は育種の重要な目標である.また,用いたイネの集団における無芒化は,育種の系譜から少なくとも独立に2回にわたり起こったことが示されており,同一の遺伝子に複数の変異の存在する可能性が予想された.そこで,遺伝子ごとのマーカーセットを用いたゲノムワイド関連解析により,芒の長さに関連するDNA多型を検出したところ,通常のマーカーセットを用いたゲノムワイド関連解析においては検出されなかった強い関連を示す遺伝子が検出された.この遺伝子はEPFLと名づけられたタンパク質をコードしていた.この遺伝子について,機能獲得型と予想された対立遺伝子を芒のないイネの品種に導入したところ,芒が形成された.これらの結果から,原因遺伝子に複数のハプロタイプの存在が想定される場合には,遺伝子ごとに変換したマーカーを用いたゲノムワイド関連解析が有効であると結論された.
おわりに
今回の研究により,ゲノムワイド関連解析に用いる集団の選択や解析の手法をくふうすることにより,作物においても効率的に新規の遺伝子を同定することが可能であることが確認された.しかし,今回の手法は,作物に対するゲノムワイド関連解析において,すべてのケースに適用できるわけではない.この研究において,候補となるDNA多型を選抜する際には,遺伝子の機能に影響する可能性の高いことからアミノ酸配列の変異をもたらすようなDNA多型に着目した(図2).しかし,遺伝子の機能の相違はアミノ酸配列の変異だけでなく,発現のレベルにおいてももたらされる.実際,最近になり報告された,イネの種子の大きさを対象としたゲノムワイド関連解析においては,複数の候補遺伝子のなかから原因遺伝子を選抜する際に遺伝子の発現情報にもとづく手法が有効であることが示された10).今後は,アミノ酸配列の変異および発現レベルの両方の情報にもとづき候補遺伝子を選抜することで,より汎用的な遺伝子の同定が可能であると考えられる.
文 献
- Myles, S., Peiffer, J., Brown, P. J. et al.: Association mapping: critical considerations shift from genotyping to experimental design. Plant Cell, 21, 2194-2202 (2009)[PubMed]
- Hamblin, M. T., Buckler, E. S. & Jannink, J. L.: Population genetics of genomics-based crop improvement methods. Trends Genet., 27, 98-106 (2011)[PubMed]
- Matsubara, K., Hori, K., Ogiso-Tanaka, E. et al.: Cloning of quantitative trait genes from rice reveals conservation and divergence of photoperiod flowering pathways in Arabidopsis and rice. Front. Plant Sci., 5, 193 (2014)[PubMed]
- Zhao, K., Tung, C. W., Eizenga, G. C. et al.: Genome-wide association mapping reveals a rich genetic architecture of complex traits in Oryza sativa. Nat. Commun., 2, 467 (2011)[PubMed]
- Takahashi, Y., Shomura, A., Sasaki, T. et al.: Hd6, a rice quantitative trait locus involved in photoperiod sensitivity, encodes the α subunit of protein kinase CK2. Proc. Natl. Acad. Sci. USA, 98, 7922-7927 (2001)[PubMed]
- Koo, B. H., Yoo, S. C., Park, J. W. et al.: Natural variation in OsPRR37 regulates heading date and contributes to rice cultivation at a wide range of latitudes. Mol. Plant, 6, 1877-1888 (2013)[PubMed]
- Ren, G., Chen, X. & Yu, B.: Uridylation of miRNAs by HEN1 SUPPRESSOR1 in Arabidopsis. Curr. Biol., 22, 695-700 (2012)[PubMed]
- Yano, M., Katayose, Y., Ashikari, M. et al.: Hd1, a major photoperiod sensitivity quantitative trait locus in rice, is closely related to the Arabidopsis flowering time gene CONSTANS. Plant Cell, 12, 2473-2484 (2000)[PubMed]
- Atwell, S., Huang, Y. S., Vilhjalmsson, B. J. et al.: Genome-wide association study of 107 phenotypes in Arabidopsis thaliana inbred lines. Nature, 465, 627-631 (2010)[PubMed]
- Si, L., Chen, J., Huang, X. et al.: OsSPL13 controls grain size in cultivated rice. Nat. Genet., 48, 447-456 (2016)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2016年 名古屋大学大学院生命農学研究科博士課程 修了,同年より東京大学大学院農学生命科学研究科 特別研究員.
研究テーマ:ゲノム情報を利用した有用な遺伝子の同定および分子育種.
抱負:作物の栄養生理や養分吸収にかかわる分子機能を解明したい.
松岡 信(Makoto Matsuoka)
名古屋大学生物機能開発利用研究センター 教授.
研究室URL:http://bbc.agr.nagoya-u.ac.jp/~pmb/
© 2016 矢野憲司・松岡 信 Licensed under CC 表示 2.1 日本