小分子rocaglateは翻訳開始因子eIF4AをRNA配列に対し選択的な翻訳抑制因子に変える
岩崎 信太郎
(米国California大学Berkeley校Department of Molecular and Cell Biology)
email:岩崎信太郎
DOI: 10.7875/first.author.2016.072
Rocaglates convert DEAD-box protein eIF4A into a sequence-selective translational repressor.
Shintaro Iwasaki, Stephen N. Floor, Nicholas T. Ingolia
Nature, 534, 558-561 (2016)
rocaglateはがん細胞を特異的に死滅させることのできる非常に有用な小分子であり,真核生物翻訳開始因子のひとつであるeIF4Aを標的として翻訳を抑制することが知られていたが,その分子機構は不明であった.この研究において,次世代シークエンサーを用いた網羅的な解析および生化学的な手法により,rocaglateとeIF4Aとの複合体はAやGの連続したポリプリン配列と選択的に結合することが明らかにされた.また,mRNAと安定に結合したrocaglateとeIF4Aとの複合体がリボソーム小サブユニットによるスキャニングの立体障害になることにより,ポリプリン配列をもつmRNAに対し選択的に翻訳を抑制することが明らかにされた.この研究により,抗がん剤の作用機構が明らかにされただけでなく,小分子によりRNA結合タンパク質が新規にRNA配列に対する選択性を獲得する例が示された.
タンパク質の生合成反応である翻訳は時空間的に緻密に制御され細胞の運命をつかさどる.細胞の増殖とタンパク質の生合成とは密接に結びついているため,翻訳制御の異常はがん化やがん細胞の増殖につながることが知られている1).実際に,翻訳因子あるいはその制御機構を標的とした抗がん剤がさかんに研究されている.
真核生物における翻訳は真核生物翻訳開始因子(eukaryotic translation initiation factor:eIF)とよばれるタンパク質がmRNAのうえにリボソーム小サブユニットをリクルートすることによりはじまる.そののち,リボソーム小サブユニットはmRNAの5’側非翻訳領域を3’方向に移動しながら開始コドンをみつけだす.この反応をスキャニングとよぶ.真核生物翻訳開始因子のひとつeIF4AはDEADボックス型RNAヘリカーゼであり,5’側非翻訳領域においてRNAの2次構造を解きほぐすことによりリボソーム小サブユニットによるスキャニングを促進すると考えられている2).
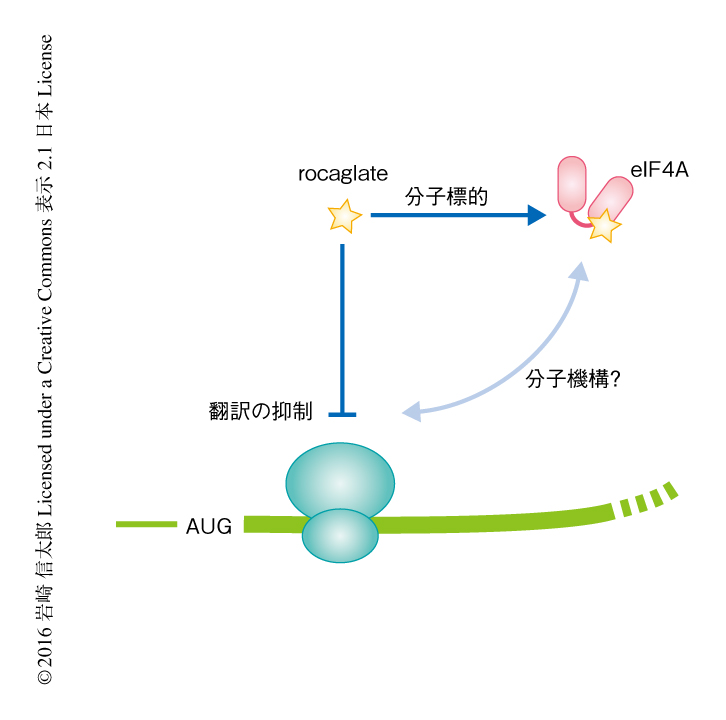
eIF4Aはさまざまな生物に由来する天然小分子の標的になっており,それらの小分子には抗がん作用のあることが知られている.rocaglateはその代表的なものであり,アグライアとよばれる東南アジアに自生する植物から単離された天然小分子の総称である.rocaglateは通常の細胞には影響せずにがん細胞を特異的に死滅させるという特徴をもち3),eIF4Aを標的として翻訳を抑制し抗がん作用を発揮すると考えられている.しかしながら,rocaglateがどのようなmRNAの翻訳を抑制するのか,また,どのような分子機構により翻訳を抑制するのかといったことは不明であった(図1).この研究においては,これらの点を明らかにすべく,代表的なrocaglateとしてrocaglamide A(RocA)を用い,次世代シークエンサーを用いた網羅的な手法および生化学的な手法により解析した.
RocAが細胞においてどのmRNAの翻訳を抑制するかを網羅的に検証するため,リボソームプロファイリング法とよばれる手法を用いた4).翻訳中のリボソームはmRNAと非常に強固に結合するため,リボソームが直接的に結合している部分はRNaseによる分解から保護され,いわゆる“フットプリント”に相当するRNAの断片を生じる.これを回収し次世代シークエンサーを用いて解読することにより,細胞においてリボソームがどのmRNAのどのコドンを翻訳していたかを定量的かつ網羅的に解析することができる.RocAを処理したHEK293細胞をリボソームプロファイリング法により解析したところ,mRNAのあいだでRocAに対する感受性が最大で30倍ほど異なることが明らかにされた.これは,RocAが一部のmRNAに対し強く翻訳を抑制し,ほかの一部のmRNAは抑制されにくいことを示した.このようなmRNAに対する選択性はこれまで解析された翻訳阻害剤においては知られておらず,RocAが翻訳阻害剤として特徴的な性質をもつことを示した.eIF4Aはリボソーム小サブユニットによるスキャニングを促進することが知られていたことから,RocAによるmRNAの選択的な翻訳の抑制は5’側非翻訳領域の配列に依存することが予想された.実際に,リボソームプロファイリング法において翻訳の強い抑制のみられたmRNAの5’側非翻訳領域は,レポーターmRNAからの翻訳をより強く抑制した.
RocAはどのように翻訳を抑制すべきmRNAを選択しているのであろうか? 先行研究により,RocAはeIF4AとRNAとの結合を強めるはたらきのあることが示されていた5).そこで,eIF4AにはどのようなmRNAが結合しているのか,また,eIF4Aと結合するmRNAはRocAによりどう変化するかについて,RIP-Seq(RNA-immunoprecipitation sequencing,RNA免疫沈降-シークエンシング)法を用いて網羅的に解析した.その結果,RocAにより全体としてmRNAとeIF4Aとの結合は強固になったが,強固になる程度はmRNAのあいだで大幅に異なり,最大で約15倍の差が観察された.これは,RocAによりeIF4AとmRNAとの結合は選択的に強化されることを意味した.また,リボソームプロファイリング法による結果とRIP-Seq法による結果を直接に比較したところ,RocAによりeIF4Aと強固に結合するmRNAほど,より強く翻訳が抑制されることが明らかにされた.以上の結果から,RocAとeIF4Aとの複合体がmRNAに対し選択的に結合することが,RocAによる選択的な翻訳の抑制の機構であることが示された.
RocAとeIF4Aとの複合体はどのような配列をもつmRNAを選択するのだろうか? このことを網羅的かつ生化学的に解析するため,Bind-n-Seq法とよばれる手法を用いた6).Bind-n-Seq法においては,完全にランダムな配列をもつ合成RNAとRNA結合タンパク質とを混ぜ,RNA結合タンパク質と結合したRNAのみを精製し次世代シークエンサーを用いて解析する.ランダムなRNAを用いるため,いちどの実験によりすべてのRNA配列の組合せを検証することができ,RNA結合タンパク質がどのような選択性をもつのかを解析することができる.この手法を応用し,RocAによりeIF4AにおいてRNA配列に対するどのような選択性の変化が生じるかを検証した.その結果,AとGが連続するポリプリン配列をもつRNAがRocAとeIF4Aとの複合体と選択的に結合することが示された.この結果は,リボソームプロファイリング法による結果およびRIP-Seq法による結果を非常によく説明した.5’側非翻訳領域に多くのポリプリン配列をもつmRNAはRIP-Seq法による解析においてもRocAとeIFAとの複合体と強固に結合し,また,リボソームプロファイリング法による解析においても翻訳が強く抑制されることが示されたのである.このことは,ポリプリン配列をレポーターmRNAの5’側非翻訳領域に挿入すると,RocAに対する感受性が40倍ほど高まったことからも確認された.
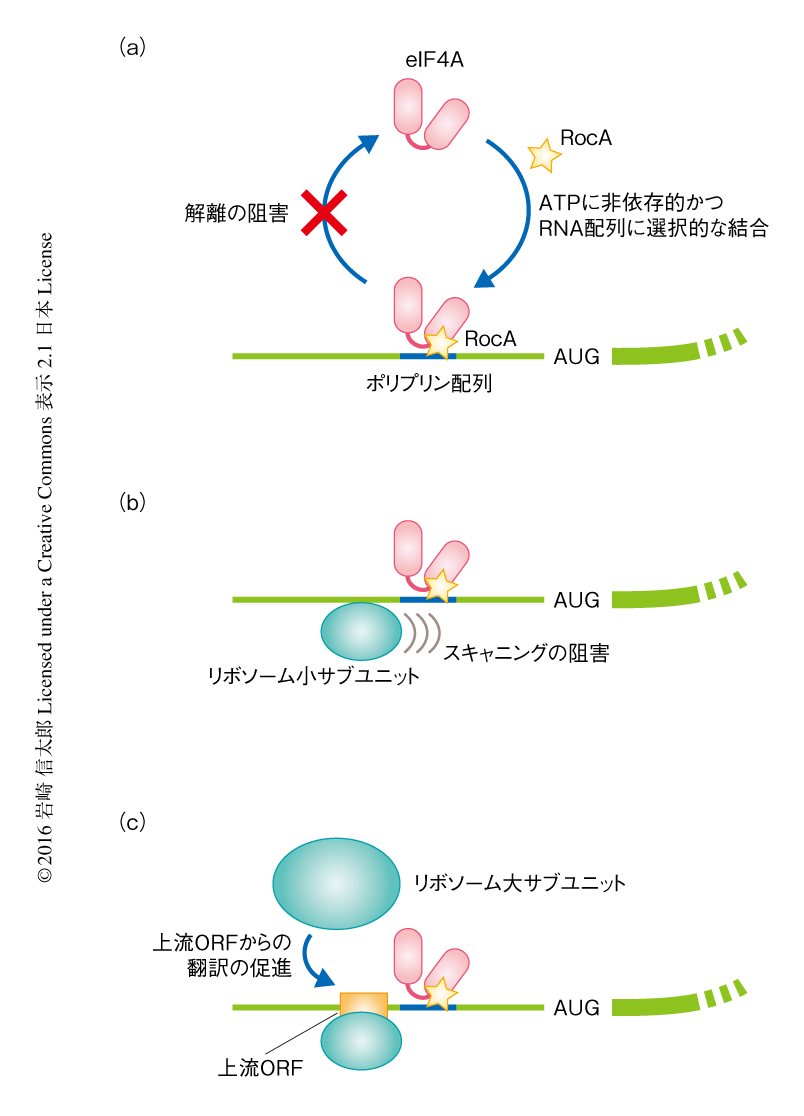
一般に,eIF4Aを含むDEADボックス型RNAヘリカーゼはATPに依存的なRNA結合タンパク質であると理解されている.eIF4AはATPとともにRNAと結合し,eIF4AのもつATPase活性によるATPの加水分解とともにRNAから解離する.このATPによるRNAとの結合および解離のサイクルは,eIF4Aの翻訳における機能において非常に重要であることが知られている.RocAがこのRNAとの結合および解離のサイクルにどのように影響するかを検証するため,RNA-タンパク質結合実験を行ったところ,RocAをくわえることによりeIF4AはATPに対する依存性をいっさい示すことなくRNAと結合することが明らかにされた.その一方で,ATPに非依存的にeIF4Aと結合するRNAはポリプリン配列をもつRNAにかぎられた.また,eIF4AとRocAとの複合体はポリプリン配列をもつRNAから解離できなくなっていた.以上の結果は,RocAとeIF4Aとの複合体はATPを加水分解したのちにもポリプリン配列をもつRNAと特異的に結合しつづけることを意味した(図2a).
RNAのうえに安定に形成されたRocAとeIF4Aとの複合体は,どのように翻訳を抑制するのだろうか? ウサギ網状赤血球抽出液を用いたin vitro翻訳系,および,開始コドンのうえのリボソーム小サブユニットを検出することのできるトウプリント法による解析により,RocAとeIF4Aとの複合体が5’側非翻訳領域に安定に結合しつづけることにより,リボソーム小サブユニットによるスキャニングに対し立体障害となり翻訳が阻害されることが明らかになった(図2b).以上の結果が示すように,RocAは単純に翻訳に利用可能なeIF4Aの量を減少させるわけではない.たとえば,in vitro翻訳系にさらに組換えeIF4AをくわえてもRocAによる翻訳抑制は解除されず,むしろ強くなった.これは,くわえたeIF4AがRocAと結合することにより,mRNAのうえにRocAとeIF4Aとの複合体をさらに形成してしまうためと考えられた.
mRNAの多くは通常のORFとは別に5’側非翻訳領域に上流ORF(upstream ORF:uORF)とよばれる領域をもつ7).上流ORFは通常のリボソーム小サブユニットによるスキャニングにおいては,いわゆるKozak配列などの配列をもたないことにより読み飛ばされる.RNA結合タンパク質やRNAの2次構造が立体障害となってリボソーム小サブユニットのスキャニングを障害するような場合には,その上流に存在する上流ORFからの翻訳が促進されることが知られている8,9).これは,リボソーム小サブユニットが上流ORFの開始コドンの付近にとどまる時間が長くなることにより,上流ORFの開始コドンの認識される頻度が高くなるためであると考えられている.実際に,RocAとeIF4Aとの複合体の結合したmRNAにおいて,上流ORFからの翻訳が促進された(図2c).リボソームプロファイリング法においてはリボソームのフットプリントがmRNAのどの位置に由来するかを検証することができるが,RocAの存在のもとではRocAに対し高い感受性を示すmRNAの5’側非翻訳領域に由来するリボソームのフットプリントが蓄積した.また,レポーターmRNAを用いたin vitro翻訳系において,RocAとeIF4Aとの複合体の結合するポリプリン配列の上流に設計した上流ORFからの翻訳が促進された.一般に,上流ORFはスキャニングしているリボソーム小サブユニットをとらえるため,下流のORFからの翻訳は抑制される10).RocAは上流ORFからの翻訳を促進させることから,ポリプリン配列にくわえ上流ORFをもつmRNAはさらにRocAにより翻訳が強く抑制されることが予想された.網羅的かつ定量的な手法により,上流ORFの存在によりmRNAはRocAにより翻訳が強く抑制されることが確認された.
この研究において,小分子によりRNA結合タンパク質に新たにRNA配列に対する特異性の付与されるはじめての例が示された.RocAはどのような分子機構によりeIF4AにRNA配列に対する特異性をあたえるのだろうか? これまで解析されたDEADボックス型RNAヘリカーゼの結晶構造において,DEADボックス型RNAヘリカーゼはRNAのリン酸基およびリボース骨格と結合するが,塩基とはいっさい結合しない.筆者らは,2つの可能性を考えている.第1に,RocAが結合することによりeIF4Aの構造が変化し新たに塩基を認識する残基の生じる可能性,第2に,RocAがeIF4Aと結合しつつ同時にRNAとも結合して塩基を認識する可能性である.これらの可能性を検証するため,現在,eIF4A,RocA,ポリプリン配列をもつRNAからなる三者複合体の結晶構造解析を進めている.
RocAがなぜがん細胞を特異的に死滅させることができるかについては,2つの仮説を考えている.そのひとつは,がん遺伝子が偶然にもRocAの標的になるようなポリプリン配列をもち,それらの発現が抑制されることによりがん細胞が死滅するという仮説である(がん遺伝子抑制モデル).また,RocAは異数性とよばれる染色体の数の異常によりひき起こされるがん細胞に対しより効果を発揮する.異数性をもつ細胞はタンパク質のストイキオメトリーの変化によりタンパク質毒性の状態にあることが知られている.RocAはmRNAに対し選択的に翻訳を抑制することによりさらにタンパク質のストイキオメトリーを変化させ,タンパク質毒性を高めることが考えられる.これらのことから,もうひとつ,異数性に由来するがん細胞は二重のタンパク質毒性のストレスに耐えられずに死滅するという仮説を考えている(タンパク質毒性モデル).これらの仮説の検証にはさらに詳細な解析が必要である.
古典的には,eIF4AはRNAヘリカーゼであることからRNAの2次構造を解きほぐすことによりリボソーム小サブユニットによるスキャニングを促進すると考えられてきた.この仮定にもとづき,RocAによる翻訳の阻害は5’側非翻訳領域の2次構造と相関すると考えられてきたが11),そもそも,RocAは単純にeIF4Aを不活性化するわけではなく,また,リボソームプロファイリング法による結果からRNAの2次構造との相関はみられなかった.さらに,eIF4Aの典型的な阻害剤であるヒプリスタノールを用いたリボソームプロファイリング法による結果においても,やはり,翻訳の阻害と5’側非翻訳領域の2次構造との相関はみられなかった.これらの結果から,5’側非翻訳領域の2次構造を解きほぐすという機能がeIF4Aの役割ではないことが示唆される.実際に,酵母においても同様の結果が示されている12).むしろ,Ded1(ヒトにおいては,DDX3)とよばれる別のDEADボックス型RNAヘリカーゼの変異体において翻訳の阻害とRNAの2次構造との相関がみられたことから,Ded1が翻訳における真のRNAヘリカーゼであることが示唆される.eIF4Aの翻訳における真の機能についてはさらなる解析が待たれる.セントラルドグマという基本的な生物学の研究においても不明な点が多く残されており,それが抗がん剤の開発という重要な応用の可能性をひめている.
略歴:2011年 東京大学大学院新領域創成科学研究科 修了,同年 東京大学分子細胞生物学研究所 助教,2013年 米国Carnegie Institution for Scienceポスドクフェローを経て,同年より米国California大学Berkeley校.
研究テーマ:RNAのかかわる現象を網羅的かつ生化学的な手法により理解する.
抱負:Hope for the best, prepare for the worst.
© 2016 岩崎 信太郎 Licensed under CC 表示 2.1 日本
(米国California大学Berkeley校Department of Molecular and Cell Biology)
email:岩崎信太郎
DOI: 10.7875/first.author.2016.072
Rocaglates convert DEAD-box protein eIF4A into a sequence-selective translational repressor.
Shintaro Iwasaki, Stephen N. Floor, Nicholas T. Ingolia
Nature, 534, 558-561 (2016)
要 約
rocaglateはがん細胞を特異的に死滅させることのできる非常に有用な小分子であり,真核生物翻訳開始因子のひとつであるeIF4Aを標的として翻訳を抑制することが知られていたが,その分子機構は不明であった.この研究において,次世代シークエンサーを用いた網羅的な解析および生化学的な手法により,rocaglateとeIF4Aとの複合体はAやGの連続したポリプリン配列と選択的に結合することが明らかにされた.また,mRNAと安定に結合したrocaglateとeIF4Aとの複合体がリボソーム小サブユニットによるスキャニングの立体障害になることにより,ポリプリン配列をもつmRNAに対し選択的に翻訳を抑制することが明らかにされた.この研究により,抗がん剤の作用機構が明らかにされただけでなく,小分子によりRNA結合タンパク質が新規にRNA配列に対する選択性を獲得する例が示された.
はじめに
タンパク質の生合成反応である翻訳は時空間的に緻密に制御され細胞の運命をつかさどる.細胞の増殖とタンパク質の生合成とは密接に結びついているため,翻訳制御の異常はがん化やがん細胞の増殖につながることが知られている1).実際に,翻訳因子あるいはその制御機構を標的とした抗がん剤がさかんに研究されている.
真核生物における翻訳は真核生物翻訳開始因子(eukaryotic translation initiation factor:eIF)とよばれるタンパク質がmRNAのうえにリボソーム小サブユニットをリクルートすることによりはじまる.そののち,リボソーム小サブユニットはmRNAの5’側非翻訳領域を3’方向に移動しながら開始コドンをみつけだす.この反応をスキャニングとよぶ.真核生物翻訳開始因子のひとつeIF4AはDEADボックス型RNAヘリカーゼであり,5’側非翻訳領域においてRNAの2次構造を解きほぐすことによりリボソーム小サブユニットによるスキャニングを促進すると考えられている2).
eIF4Aはさまざまな生物に由来する天然小分子の標的になっており,それらの小分子には抗がん作用のあることが知られている.rocaglateはその代表的なものであり,アグライアとよばれる東南アジアに自生する植物から単離された天然小分子の総称である.rocaglateは通常の細胞には影響せずにがん細胞を特異的に死滅させるという特徴をもち3),eIF4Aを標的として翻訳を抑制し抗がん作用を発揮すると考えられている.しかしながら,rocaglateがどのようなmRNAの翻訳を抑制するのか,また,どのような分子機構により翻訳を抑制するのかといったことは不明であった(図1).この研究においては,これらの点を明らかにすべく,代表的なrocaglateとしてrocaglamide A(RocA)を用い,次世代シークエンサーを用いた網羅的な手法および生化学的な手法により解析した.
1.RocAとeIF4Aとの複合体は選択的にmRNAと結合し選択的に翻訳を阻害する
RocAが細胞においてどのmRNAの翻訳を抑制するかを網羅的に検証するため,リボソームプロファイリング法とよばれる手法を用いた4).翻訳中のリボソームはmRNAと非常に強固に結合するため,リボソームが直接的に結合している部分はRNaseによる分解から保護され,いわゆる“フットプリント”に相当するRNAの断片を生じる.これを回収し次世代シークエンサーを用いて解読することにより,細胞においてリボソームがどのmRNAのどのコドンを翻訳していたかを定量的かつ網羅的に解析することができる.RocAを処理したHEK293細胞をリボソームプロファイリング法により解析したところ,mRNAのあいだでRocAに対する感受性が最大で30倍ほど異なることが明らかにされた.これは,RocAが一部のmRNAに対し強く翻訳を抑制し,ほかの一部のmRNAは抑制されにくいことを示した.このようなmRNAに対する選択性はこれまで解析された翻訳阻害剤においては知られておらず,RocAが翻訳阻害剤として特徴的な性質をもつことを示した.eIF4Aはリボソーム小サブユニットによるスキャニングを促進することが知られていたことから,RocAによるmRNAの選択的な翻訳の抑制は5’側非翻訳領域の配列に依存することが予想された.実際に,リボソームプロファイリング法において翻訳の強い抑制のみられたmRNAの5’側非翻訳領域は,レポーターmRNAからの翻訳をより強く抑制した.
RocAはどのように翻訳を抑制すべきmRNAを選択しているのであろうか? 先行研究により,RocAはeIF4AとRNAとの結合を強めるはたらきのあることが示されていた5).そこで,eIF4AにはどのようなmRNAが結合しているのか,また,eIF4Aと結合するmRNAはRocAによりどう変化するかについて,RIP-Seq(RNA-immunoprecipitation sequencing,RNA免疫沈降-シークエンシング)法を用いて網羅的に解析した.その結果,RocAにより全体としてmRNAとeIF4Aとの結合は強固になったが,強固になる程度はmRNAのあいだで大幅に異なり,最大で約15倍の差が観察された.これは,RocAによりeIF4AとmRNAとの結合は選択的に強化されることを意味した.また,リボソームプロファイリング法による結果とRIP-Seq法による結果を直接に比較したところ,RocAによりeIF4Aと強固に結合するmRNAほど,より強く翻訳が抑制されることが明らかにされた.以上の結果から,RocAとeIF4Aとの複合体がmRNAに対し選択的に結合することが,RocAによる選択的な翻訳の抑制の機構であることが示された.
2.RocAはeIF4AをRNA配列に選択的なRNA結合タンパク質に変える
RocAとeIF4Aとの複合体はどのような配列をもつmRNAを選択するのだろうか? このことを網羅的かつ生化学的に解析するため,Bind-n-Seq法とよばれる手法を用いた6).Bind-n-Seq法においては,完全にランダムな配列をもつ合成RNAとRNA結合タンパク質とを混ぜ,RNA結合タンパク質と結合したRNAのみを精製し次世代シークエンサーを用いて解析する.ランダムなRNAを用いるため,いちどの実験によりすべてのRNA配列の組合せを検証することができ,RNA結合タンパク質がどのような選択性をもつのかを解析することができる.この手法を応用し,RocAによりeIF4AにおいてRNA配列に対するどのような選択性の変化が生じるかを検証した.その結果,AとGが連続するポリプリン配列をもつRNAがRocAとeIF4Aとの複合体と選択的に結合することが示された.この結果は,リボソームプロファイリング法による結果およびRIP-Seq法による結果を非常によく説明した.5’側非翻訳領域に多くのポリプリン配列をもつmRNAはRIP-Seq法による解析においてもRocAとeIFAとの複合体と強固に結合し,また,リボソームプロファイリング法による解析においても翻訳が強く抑制されることが示されたのである.このことは,ポリプリン配列をレポーターmRNAの5’側非翻訳領域に挿入すると,RocAに対する感受性が40倍ほど高まったことからも確認された.
一般に,eIF4Aを含むDEADボックス型RNAヘリカーゼはATPに依存的なRNA結合タンパク質であると理解されている.eIF4AはATPとともにRNAと結合し,eIF4AのもつATPase活性によるATPの加水分解とともにRNAから解離する.このATPによるRNAとの結合および解離のサイクルは,eIF4Aの翻訳における機能において非常に重要であることが知られている.RocAがこのRNAとの結合および解離のサイクルにどのように影響するかを検証するため,RNA-タンパク質結合実験を行ったところ,RocAをくわえることによりeIF4AはATPに対する依存性をいっさい示すことなくRNAと結合することが明らかにされた.その一方で,ATPに非依存的にeIF4Aと結合するRNAはポリプリン配列をもつRNAにかぎられた.また,eIF4AとRocAとの複合体はポリプリン配列をもつRNAから解離できなくなっていた.以上の結果は,RocAとeIF4Aとの複合体はATPを加水分解したのちにもポリプリン配列をもつRNAと特異的に結合しつづけることを意味した(図2a).
3.RocAとeIF4Aとの複合体はリボソーム小サブユニットによるスキャニングの立体障壁になり翻訳を阻害する
RNAのうえに安定に形成されたRocAとeIF4Aとの複合体は,どのように翻訳を抑制するのだろうか? ウサギ網状赤血球抽出液を用いたin vitro翻訳系,および,開始コドンのうえのリボソーム小サブユニットを検出することのできるトウプリント法による解析により,RocAとeIF4Aとの複合体が5’側非翻訳領域に安定に結合しつづけることにより,リボソーム小サブユニットによるスキャニングに対し立体障害となり翻訳が阻害されることが明らかになった(図2b).以上の結果が示すように,RocAは単純に翻訳に利用可能なeIF4Aの量を減少させるわけではない.たとえば,in vitro翻訳系にさらに組換えeIF4AをくわえてもRocAによる翻訳抑制は解除されず,むしろ強くなった.これは,くわえたeIF4AがRocAと結合することにより,mRNAのうえにRocAとeIF4Aとの複合体をさらに形成してしまうためと考えられた.
4.RocAとeIF4Aとの複合体は上流ORFからの翻訳を促進する
mRNAの多くは通常のORFとは別に5’側非翻訳領域に上流ORF(upstream ORF:uORF)とよばれる領域をもつ7).上流ORFは通常のリボソーム小サブユニットによるスキャニングにおいては,いわゆるKozak配列などの配列をもたないことにより読み飛ばされる.RNA結合タンパク質やRNAの2次構造が立体障害となってリボソーム小サブユニットのスキャニングを障害するような場合には,その上流に存在する上流ORFからの翻訳が促進されることが知られている8,9).これは,リボソーム小サブユニットが上流ORFの開始コドンの付近にとどまる時間が長くなることにより,上流ORFの開始コドンの認識される頻度が高くなるためであると考えられている.実際に,RocAとeIF4Aとの複合体の結合したmRNAにおいて,上流ORFからの翻訳が促進された(図2c).リボソームプロファイリング法においてはリボソームのフットプリントがmRNAのどの位置に由来するかを検証することができるが,RocAの存在のもとではRocAに対し高い感受性を示すmRNAの5’側非翻訳領域に由来するリボソームのフットプリントが蓄積した.また,レポーターmRNAを用いたin vitro翻訳系において,RocAとeIF4Aとの複合体の結合するポリプリン配列の上流に設計した上流ORFからの翻訳が促進された.一般に,上流ORFはスキャニングしているリボソーム小サブユニットをとらえるため,下流のORFからの翻訳は抑制される10).RocAは上流ORFからの翻訳を促進させることから,ポリプリン配列にくわえ上流ORFをもつmRNAはさらにRocAにより翻訳が強く抑制されることが予想された.網羅的かつ定量的な手法により,上流ORFの存在によりmRNAはRocAにより翻訳が強く抑制されることが確認された.
おわりに
この研究において,小分子によりRNA結合タンパク質に新たにRNA配列に対する特異性の付与されるはじめての例が示された.RocAはどのような分子機構によりeIF4AにRNA配列に対する特異性をあたえるのだろうか? これまで解析されたDEADボックス型RNAヘリカーゼの結晶構造において,DEADボックス型RNAヘリカーゼはRNAのリン酸基およびリボース骨格と結合するが,塩基とはいっさい結合しない.筆者らは,2つの可能性を考えている.第1に,RocAが結合することによりeIF4Aの構造が変化し新たに塩基を認識する残基の生じる可能性,第2に,RocAがeIF4Aと結合しつつ同時にRNAとも結合して塩基を認識する可能性である.これらの可能性を検証するため,現在,eIF4A,RocA,ポリプリン配列をもつRNAからなる三者複合体の結晶構造解析を進めている.
RocAがなぜがん細胞を特異的に死滅させることができるかについては,2つの仮説を考えている.そのひとつは,がん遺伝子が偶然にもRocAの標的になるようなポリプリン配列をもち,それらの発現が抑制されることによりがん細胞が死滅するという仮説である(がん遺伝子抑制モデル).また,RocAは異数性とよばれる染色体の数の異常によりひき起こされるがん細胞に対しより効果を発揮する.異数性をもつ細胞はタンパク質のストイキオメトリーの変化によりタンパク質毒性の状態にあることが知られている.RocAはmRNAに対し選択的に翻訳を抑制することによりさらにタンパク質のストイキオメトリーを変化させ,タンパク質毒性を高めることが考えられる.これらのことから,もうひとつ,異数性に由来するがん細胞は二重のタンパク質毒性のストレスに耐えられずに死滅するという仮説を考えている(タンパク質毒性モデル).これらの仮説の検証にはさらに詳細な解析が必要である.
古典的には,eIF4AはRNAヘリカーゼであることからRNAの2次構造を解きほぐすことによりリボソーム小サブユニットによるスキャニングを促進すると考えられてきた.この仮定にもとづき,RocAによる翻訳の阻害は5’側非翻訳領域の2次構造と相関すると考えられてきたが11),そもそも,RocAは単純にeIF4Aを不活性化するわけではなく,また,リボソームプロファイリング法による結果からRNAの2次構造との相関はみられなかった.さらに,eIF4Aの典型的な阻害剤であるヒプリスタノールを用いたリボソームプロファイリング法による結果においても,やはり,翻訳の阻害と5’側非翻訳領域の2次構造との相関はみられなかった.これらの結果から,5’側非翻訳領域の2次構造を解きほぐすという機能がeIF4Aの役割ではないことが示唆される.実際に,酵母においても同様の結果が示されている12).むしろ,Ded1(ヒトにおいては,DDX3)とよばれる別のDEADボックス型RNAヘリカーゼの変異体において翻訳の阻害とRNAの2次構造との相関がみられたことから,Ded1が翻訳における真のRNAヘリカーゼであることが示唆される.eIF4Aの翻訳における真の機能についてはさらなる解析が待たれる.セントラルドグマという基本的な生物学の研究においても不明な点が多く残されており,それが抗がん剤の開発という重要な応用の可能性をひめている.
文 献
- Ruggero, D.: Translational control in cancer etiology. Cold Spring Harb. Perspect. Biol., 5, a012336 (2013)[PubMed]
- Sonenberg, N. & Hinnebusch, A. G.: Regulation of translation initiation in eukaryotes: mechanisms and biological targets. Cell, 136, 731-745 (2009)[PubMed]
- Santagata, S., Mendillo, M. L., Tang, Y. C. et al.: Tight coordination of protein translation and HSF1 activation supports the anabolic malignant state. Science, 341, 1238303 (2013)[PubMed]
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. et al.: Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science, 324, 218-223 (2009)[PubMed]
- Sadlish, H., Galicia-Vazquez, G., Paris, C. G. et al.: Evidence for a functionally relevant rocaglamide binding site on the eIF4A-RNA complex. ACS Chem. Biol., 8, 1519-1527 (2013)[PubMed]
- Lambert, N., Robertson, A., Jangi, M. et al.: RNA Bind-n-Seq: quantitative assessment of the sequence and structural binding specificity of RNA binding proteins. Mol. Cell, 54, 887-900 (2014)[PubMed]
- Lee, S., Liu, B., Lee, S. et al.: Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proc. Natl. Acad. Sci. USA, 109, E2424-E2432 (2012)[PubMed]
- Kozak, M.: Context effects and inefficient initiation at non-AUG codons in eucaryotic cell-free translation systems. Mol. Cell. Biol., 9, 5073-5080 (1989)[PubMed]
- Medenbach, J., Seiler, M. & Hentze, M. W.: Translational control via protein-regulated upstream open reading frames. Cell, 145, 902-913 (2011)[PubMed]
- Calvo, S. E., Pagliarini, D. J. & Mootha, V. K.: Upstream open reading frames cause widespread reduction of protein expression and are polymorphic among humans. Proc. Natl. Acad. Sci. USA, 106, 7507-7512 (2009)[PubMed]
- Wolfe, A. L., Singh, K., Zhong, Y. et al.: RNA G-quadruplexes cause eIF4A-dependent oncogene translation in cancer. Nature, 513, 65-70 (2014)[PubMed]
- Sen, N. D., Zhou, F., Ingolia, N. T. et al.: Genome-wide analysis of translational efficiency reveals distinct but overlapping functions of yeast DEAD-box RNA helicases Ded1 and eIF4A. Genome Res., 25, 1196-1205 (2015)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2011年 東京大学大学院新領域創成科学研究科 修了,同年 東京大学分子細胞生物学研究所 助教,2013年 米国Carnegie Institution for Scienceポスドクフェローを経て,同年より米国California大学Berkeley校.
研究テーマ:RNAのかかわる現象を網羅的かつ生化学的な手法により理解する.
抱負:Hope for the best, prepare for the worst.
© 2016 岩崎 信太郎 Licensed under CC 表示 2.1 日本