骨芽細胞の分化の決定における転写因子Sp7の作動様式の解明
北條宏徳・大庭伸介・Andrew P. McMahon
(Southern California大学Keck School of Medicine,Eli and Edythe Broad CIRM Center for Regenerative Medicine and Stem Cell Research)
email:北條宏徳
DOI: 10.7875/first.author.2016.043
Sp7/Osterix is restricted to bone-forming vertebrates where it acts as a Dlx co-factor in osteoblast specification.
Hironori Hojo, Shinsuke Ohba, Xinjun He, Lick Pui Lai, Andrew P. McMahon
Developmental Cell, 37, 238-253 (2016)
Spファミリーに属する転写因子Sp7は骨形成に必須である.筆者らは,マウスの骨芽細胞におけるChIP-seq法による解析により,Sp7が作用するゲノム領域の同定およびその作動様式の解明をめざした.その結果,Sp7の作用するゲノム領域は骨芽細胞において高発現する遺伝子を標的とするエンハンサー活性をもつことが示唆され,そのエンハンサー活性はいくつかのゲノム領域について細胞株およびマウスにおいて確認された.Spファミリーのメンバーは特異的なDNA配列であるGCボックスと結合してその機能を発揮すると考えられていた.しかしながら,この研究においては,Sp7は異なる作動様式,すなわち,ATリッチモチーフにおいてホメオボックス転写因子と複合体を形成することによりその機能を発揮するというモデルが提唱された.さらに,生物種のあいだの比較ゲノム解析により,Sp7の出現は骨組織をもつ脊椎動物の進化と密接に関連する可能性が示唆された.
骨形成は進化の過程において脊椎動物が獲得した機能のひとつで,骨組織はミネラルの貯蔵および身体の支持にはたらく.骨芽細胞は骨形成を担う主要な細胞である.マウスにおける遺伝学により,転写因子Sp7は骨芽細胞に必須であることが明らかにされている1).Sp7ノックアウトマウスにおいて骨はまったく形成されない.また,ヒトにおいて,Sp7をコードする遺伝子は骨形成不全症の原因遺伝子のひとつとして知られている2).
骨形成におけるその機能の重要性にもかかわらず,Sp7の標的となる遺伝子については十分には明らかにされていない.過去の報告において,骨芽細胞の分化に関与するいくつかの遺伝子の近傍においてSp7が作用することが示唆されているが,いまだにゲノムワイドな解析はなされていない.さらに,Sp7の作動様式についてもその理解は不十分である.Sp7はSpファミリーメンバーに属する.このファミリーはジンクフィンガードメインをもつことを特徴とし,メンバーのひとつSp1は,このジンクフィンガードメインを介してGCリッチなDNA配列であるGCボックスと結合することが知られている3).ジンクフィンガードメインはSpファミリーのあいだで高度に保存されていることから,Spファミリーのほかのメンバーも同様にGCボックスと結合すると考えられており,近年,いくつかのメンバーのGCボックスとの結合がゲノムワイドな解析により確認されている4).Sp7に関しては,in vitroにおける解析により同様の作動機序をもつことが複数の論文において示唆されている一方,GCボックスとの結合能をもたないとする報告もあり,in vivoにおける作用機序の解析が待たれていた.そこで,この研究においては,マウスの骨芽細胞を用いて,ChIP-seq法およびRNA-seq法により,in vivoにおいてSp7の標的となるゲノム領域の同定およびその作動様式の解明をめざした.
in vivoにおいて骨芽細胞のゲノムにおけるSp7の相互作用を検討する目的で,ビオチンタグとFLAGタグの融合ペプチドをコードする塩基配列をSp7遺伝子座にノックインしたマウスを作製した.このマウスの骨組織において,この融合ペプチドタグを付加したSp7は内在性のSp7と同様に発現しており,骨形成に大きな変化は認められなかった.Sp7の標的となるゲノム領域を同定する目的で,このマウスの新生仔の頭頂骨から骨芽細胞を回収し,抗FLAGタグ抗体を用いてChIP-seq法により解析した.バイオインフォマティクス解析により,Sp7とDNAとの相互作用が有意に検出されたゲノム領域2112箇所を同定し,Sp7作用領域とした.さらに,Sp7を発現する骨芽細胞における転写産物のプロファイルを得る目的で,Sp7-GFPレポーターマウス5) を用いて,FACSによりGFP陽性細胞を単離してRNA-seq法により解析し,この細胞において高発現する遺伝子を同定した.
エンハンサーとは,転写因子が作用することにより活性化され,その標的となる遺伝子の転写を正に制御するゲノム領域である.その多くは種のあいだで高度に保存されており,細胞の運命決定や分化にはたらくことが知られている.ChIP-seq法およびRNA-seq法により得られたデータから,骨芽細胞のゲノムにおいてSp7の制御のもとで機能するエンハンサーを同定するため,1)Sp7作用領域のなかでChIP-seq法において高いシグナルの認められたゲノム領域,2)Sp7作用領域の近傍における遺伝子の発現が骨芽細胞において高い領域,3)種のあいだで塩基配列が高度に保存されているゲノム領域,の3つの条件によりその候補となるゲノム領域を絞り込んだ.解析の結果,Sp7の制御のもとで機能するエンハンサーの候補として194箇所があげられた.そのなかには,これまでに骨芽細胞に特異的なエンハンサーとして報告のあるゲノム領域も含まれていた.骨芽細胞における新規のエンハンサーを同定する目的で,候補のうちの11箇所のゲノム領域に対しin vitroにおいてレポーター解析を行った.線維芽細胞株および骨芽細胞株を用いた結果,検討したすべてのゲノム領域において,レポーター活性は骨芽細胞株において高かった.そのうちのひとつ,Notch2遺伝子のイントロンにおけるエンハンサー活性をin vivoにおいて検討した.Notch2は骨芽細胞前駆細胞の増殖および分化にかかわることが知られている6).このゲノム領域を含むGFP-LacZレポーターマウスを作出し,新生仔のマウスにおけるホールマウントLacZ染色を行ったところ,骨組織においてレポーター活性が認められた.組織学的な解析の結果,骨組織においてレポーター活性の認められた細胞はすべてSp7を発現する骨芽細胞であった.以上より,Sp7は骨芽細胞に特異的なNotch2遺伝子のエンハンサーを介してNotch2の発現を制御することが示された.
in vivoにおいて骨芽細胞のゲノムにおけるSp7の作動様式を検討する目的で,得られたSp7作用領域の塩基配列を用いてモチーフ解析を行った.さきに述べたとおり,Spファミリーのメンバーは高度に保存されたジンクフィンガードメインを介してGCボックスと結合すると考えられてきた.しかしながら,ChIP-seq法による解析から予測されたSp7の結合モチーフにはGCボックスは有意に存在せず,ホメオボックス転写因子との結合配列を含むATリッチモチーフが大部分であった.複数のATリッチモチーフを並列したレポーターを作製しin vitroにおいてレポーター活性を測定したところ,骨芽細胞株において強いレポーター活性が認められたのに対し,線維芽細胞株においてレポーター活性は認められなかった.骨芽細胞株におけるレポーター活性はSp7のノックダウンによりほぼ完全に消失した.以上より,Sp7はin vivoにおいて,過去の報告とは異なる作動様式,すなわち,ATリッチモチーフとの相互作用を介して標的となる遺伝子の転写の活性を制御する可能性が示唆された.
Sp7がATリッチモチーフと直接的に結合するかどうかを検討する目的でゲルシフトアッセイを行った.その結果,直接的な結合はまったく認められず,Sp7はパートナーとなるほかの転写因子を介してATリッチモチーフと間接的に結合する可能性が示唆された.パートナーとなる転写因子は,1)ATリッチモチーフと直接に結合する,2)骨芽細胞において高発現する,3)Sp7と直接的に結合する,の3つの条件をみたすと考えられた.転写因子-DNA結合配列のデータベース4),過去の文献7),および,RNA-seq法により得られたデータを統合的に解析した結果,ホメオボックス転写因子であるDlx3,Dlx5,Dlx6が候補にあげられた.Dlx3,Dlx5,Dlx6のDNA結合モチーフはSp7に対するChIP-seq法による解析により得られたATリッチモチーフの一部と完全に一致すること,in vitroにおいてDlx3,Dlx5,Dlx6の発現は骨芽細胞の分化を誘導すること,Dlx5の長管骨における発現パターンはSp7の発現パターンと似ていること,Dlx5とDlx6のダブルノックアウトマウスにおいて骨形成が顕著に抑制されること8),最近の報告によりDlx5とSp7との直接的な結合が明らかにされたこと9),が裏づけとなった.Dlx3,Dlx5,Dlx6のうち,Dlx5が骨芽細胞においてもっとも高発現していたので,これを用いてSp7の作動様式のさらなる解析を試みた.
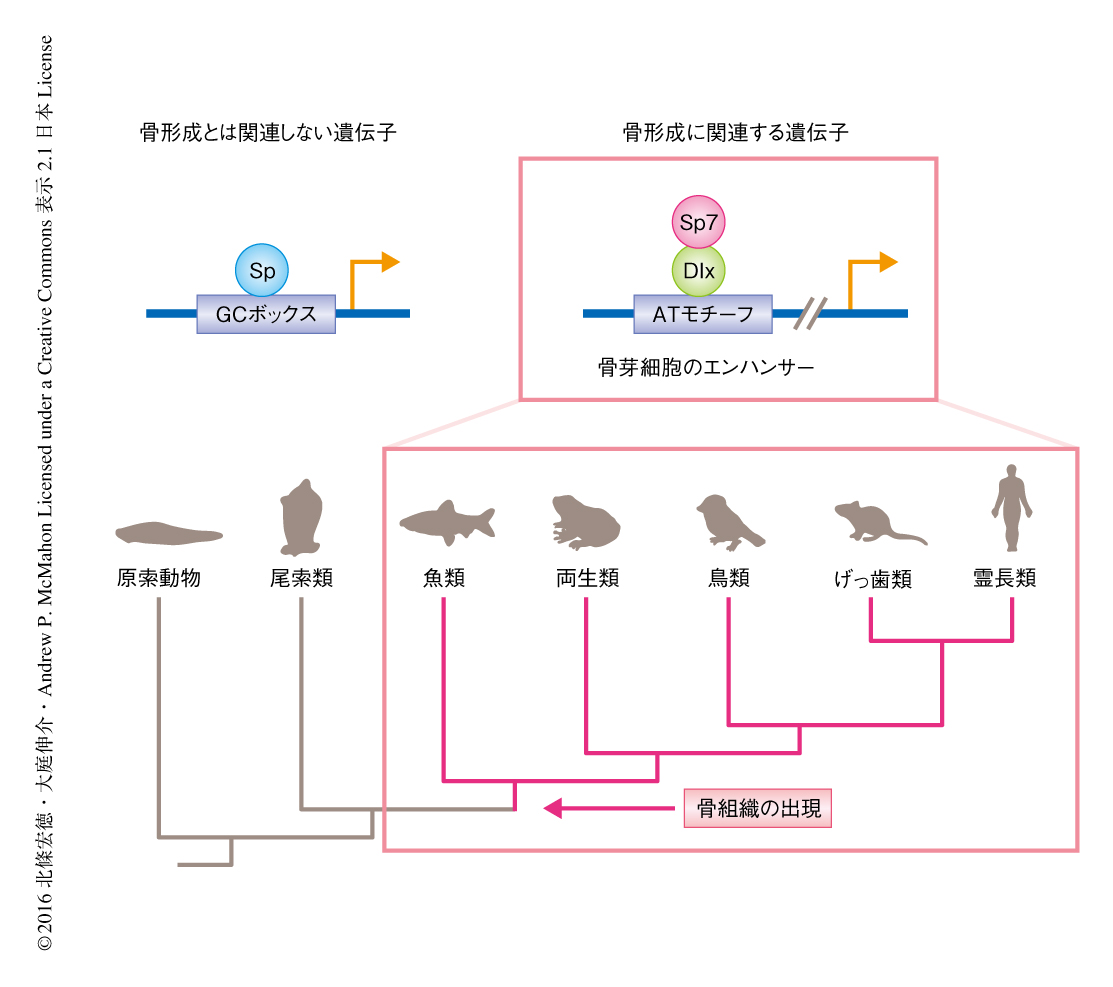
共免疫沈降-ウェスタンブロット法により,Sp7とDlx5との直接的な結合にはSp7のジンクフィンガードメインおよびDlx5のN末端ドメインが必須であることが明らかにされた.Sp7とDlx5のゲノムにおける相互作用を検討する目的で,骨芽細胞株を用いてChIP-seq法により解析したところ,Sp7およびDlx5は共通するゲノム領域に高頻度で作用すること,それらのゲノム領域にはATリッチモチーフが高頻度に存在すること,Sp7およびDlx5のChIP-seq法におけるシグナルには正の相関関係が認められること,が明らかにされた.in vitroにおけるレポーター解析の結果,Sp7およびDlx5はATリッチモチーフを介してその転写活性を相乗的に上昇させたのに対し,骨芽細胞株におけるDlx3,Dlx5,Dlx6のノックダウンはSp7の標的となるゲノム領域への作用を抑制した.以上より,Sp7は骨芽細胞において,ジンクフィンガードメインを介してホメオボックス転写因子Dlx5,Dlx3,Dlx6と転写複合体を形成し,ATリッチモチーフと作用することにより遺伝子の発現を制御する可能性が示唆された(図1).
Sp7の作動様式がSp7に特異的なものか,あるいは,Spファミリーのほかのメンバーと共通したものなのかを検討する目的で,骨芽細胞株においてSp1に対してChIP-seq法により解析した.Sp7に対するChIP-seq法により得られたデータと比較した結果,骨芽細胞のゲノムにおけるSp1の作用領域の大部分はSp7の作用領域とは異なっていた.Sp1に特異的な作用領域はおもに転写開始点の近傍に位置し,骨形成とは無関係の遺伝子の近傍に多く存在した一方,Sp7に特異的な作用領域はそのほとんどが転写開始点から遠位に存在し,骨形成と関連する遺伝子の近傍に多く存在した.モチーフ解析の結果,Sp1に特異的な作用領域にはGCボックスが有意に存在することが確認された.以上より,Sp1とSp7は,ゲノムにおいて異なる作用領域,異なるDNA結合モチーフ,異なる標的遺伝子をもつことが明らかにされた.
この違いはどこから生じたのだろうか? Sp1およびSp7のGCボックスとの結合能に注目し,ゲルシフトアッセイにより詳細に解析した.野生型のSp1およびSp7,および,互いにジンクフィンガードメインを入れ替えたSp1およびSp7の変異体を用いた結果,Sp1のもつジンクフィンガードメインはGCボックスとの結合に寄与したのに対し,Sp7のもつジンクフィンガードメインにはGCボックスとの結合能がほとんど認められなかった.Spファミリーのもつジンクフィンガードメインには3つのジンクフィンガーが存在し,それぞれのもつαへリックスがDNAとの結合に寄与することが知られている10).そこで,Spファミリーのメンバーのあいだでこのαへリックスのアミノ酸配列を比較したところ,Sp7に特異的なアミノ酸配列が見い出された.このSp7に特異的なアミノ酸残基をSp1の対応するアミノ酸残基と置換したところ,野生型のSp7においてはほとんど認められなかったGCボックスとの結合能が検出された.以上より,Sp7のもつジンクフィンガードメインに存在する特異的なアミノ酸配列がSp7のユニークな作動様式に寄与する可能性が示唆された.
このSp7の構造的な特徴に由来する生物学的な作用の進化的な意義について検証するため,脊索動物において,Sp7のもつジンクフィンガードメインのアミノ酸配列,あるいは,それともっとも相同性の高いアミノ酸配列を比較した.その結果,骨組織をもつ脊椎動物のみがSp7に特異的なアミノ酸配列をもっていた(図1).したがって,脊椎動物の進化において,Spファミリーのほかのメンバーとは異なる作動様式をもつSp7は,骨組織を形成する骨芽細胞の出現にともない獲得されたことが示唆された.
この研究により,in vivoにおいて骨芽細胞における転写因子Sp7の標的となるゲノム領域およびその作動様式が明らかにされた.Sp7の標的となるゲノム領域には,骨形成においてその機能が知られている転写因子,シグナル伝達タンパク質,細胞外基質をコードする遺伝子が存在し,Sp7により制御される転写ネットワークの一端が明らかにされた.今回,見い出されたSp7の作動様式はまったく予想外のものであった.Spファミリーのほかのメンバーとはまったく異なるDNA配列と相互作用すること,および,DNAとの相互作用にはパートナーとなる転写因子が必要だったことから,従来のin vitroにおける解析では検出することの困難な生命現象が,ChIP-seq法による解析によりはじめて明らかにされた例だといえる.この研究により,ホメオボックス転写因子であるDlx3,Dlx5,Dlx6がSp7のパートナーとなる転写因子であることが明らかにされたが,パートナーとなる転写因子はほかにも存在するのか,骨芽細胞以外の細胞でも同様の作動様式が存在するのかについては,今後の研究課題である.
略歴:2011年 東京大学大学院医学系研究科 修了,同年 東京大学医学部附属病院 博士研究員,2012年 Southern California大学Keck School of Medicine博士研究員を経て,2016年より東京大学大学院工学系研究科 博士研究員.
研究テーマ:骨形成における転写ネットワーク.
大庭 伸介(Shinsuke Ohba)
東京大学大学院工学系研究科 特任准教授.
Andrew P. McMahon
Southern California大学Keck School of MedicineにてProfessor.
研究室URL:http://mcmahonlab.usc.edu/
© 2016 北條宏徳・大庭伸介・Andrew P. McMahon Licensed under CC 表示 2.1 日本
(Southern California大学Keck School of Medicine,Eli and Edythe Broad CIRM Center for Regenerative Medicine and Stem Cell Research)
email:北條宏徳
DOI: 10.7875/first.author.2016.043
Sp7/Osterix is restricted to bone-forming vertebrates where it acts as a Dlx co-factor in osteoblast specification.
Hironori Hojo, Shinsuke Ohba, Xinjun He, Lick Pui Lai, Andrew P. McMahon
Developmental Cell, 37, 238-253 (2016)
要 約
Spファミリーに属する転写因子Sp7は骨形成に必須である.筆者らは,マウスの骨芽細胞におけるChIP-seq法による解析により,Sp7が作用するゲノム領域の同定およびその作動様式の解明をめざした.その結果,Sp7の作用するゲノム領域は骨芽細胞において高発現する遺伝子を標的とするエンハンサー活性をもつことが示唆され,そのエンハンサー活性はいくつかのゲノム領域について細胞株およびマウスにおいて確認された.Spファミリーのメンバーは特異的なDNA配列であるGCボックスと結合してその機能を発揮すると考えられていた.しかしながら,この研究においては,Sp7は異なる作動様式,すなわち,ATリッチモチーフにおいてホメオボックス転写因子と複合体を形成することによりその機能を発揮するというモデルが提唱された.さらに,生物種のあいだの比較ゲノム解析により,Sp7の出現は骨組織をもつ脊椎動物の進化と密接に関連する可能性が示唆された.
はじめに
骨形成は進化の過程において脊椎動物が獲得した機能のひとつで,骨組織はミネラルの貯蔵および身体の支持にはたらく.骨芽細胞は骨形成を担う主要な細胞である.マウスにおける遺伝学により,転写因子Sp7は骨芽細胞に必須であることが明らかにされている1).Sp7ノックアウトマウスにおいて骨はまったく形成されない.また,ヒトにおいて,Sp7をコードする遺伝子は骨形成不全症の原因遺伝子のひとつとして知られている2).
骨形成におけるその機能の重要性にもかかわらず,Sp7の標的となる遺伝子については十分には明らかにされていない.過去の報告において,骨芽細胞の分化に関与するいくつかの遺伝子の近傍においてSp7が作用することが示唆されているが,いまだにゲノムワイドな解析はなされていない.さらに,Sp7の作動様式についてもその理解は不十分である.Sp7はSpファミリーメンバーに属する.このファミリーはジンクフィンガードメインをもつことを特徴とし,メンバーのひとつSp1は,このジンクフィンガードメインを介してGCリッチなDNA配列であるGCボックスと結合することが知られている3).ジンクフィンガードメインはSpファミリーのあいだで高度に保存されていることから,Spファミリーのほかのメンバーも同様にGCボックスと結合すると考えられており,近年,いくつかのメンバーのGCボックスとの結合がゲノムワイドな解析により確認されている4).Sp7に関しては,in vitroにおける解析により同様の作動機序をもつことが複数の論文において示唆されている一方,GCボックスとの結合能をもたないとする報告もあり,in vivoにおける作用機序の解析が待たれていた.そこで,この研究においては,マウスの骨芽細胞を用いて,ChIP-seq法およびRNA-seq法により,in vivoにおいてSp7の標的となるゲノム領域の同定およびその作動様式の解明をめざした.
1.Sp7のゲノムワイドな解析による骨芽細胞におけるエンハンサーの同定
in vivoにおいて骨芽細胞のゲノムにおけるSp7の相互作用を検討する目的で,ビオチンタグとFLAGタグの融合ペプチドをコードする塩基配列をSp7遺伝子座にノックインしたマウスを作製した.このマウスの骨組織において,この融合ペプチドタグを付加したSp7は内在性のSp7と同様に発現しており,骨形成に大きな変化は認められなかった.Sp7の標的となるゲノム領域を同定する目的で,このマウスの新生仔の頭頂骨から骨芽細胞を回収し,抗FLAGタグ抗体を用いてChIP-seq法により解析した.バイオインフォマティクス解析により,Sp7とDNAとの相互作用が有意に検出されたゲノム領域2112箇所を同定し,Sp7作用領域とした.さらに,Sp7を発現する骨芽細胞における転写産物のプロファイルを得る目的で,Sp7-GFPレポーターマウス5) を用いて,FACSによりGFP陽性細胞を単離してRNA-seq法により解析し,この細胞において高発現する遺伝子を同定した.
エンハンサーとは,転写因子が作用することにより活性化され,その標的となる遺伝子の転写を正に制御するゲノム領域である.その多くは種のあいだで高度に保存されており,細胞の運命決定や分化にはたらくことが知られている.ChIP-seq法およびRNA-seq法により得られたデータから,骨芽細胞のゲノムにおいてSp7の制御のもとで機能するエンハンサーを同定するため,1)Sp7作用領域のなかでChIP-seq法において高いシグナルの認められたゲノム領域,2)Sp7作用領域の近傍における遺伝子の発現が骨芽細胞において高い領域,3)種のあいだで塩基配列が高度に保存されているゲノム領域,の3つの条件によりその候補となるゲノム領域を絞り込んだ.解析の結果,Sp7の制御のもとで機能するエンハンサーの候補として194箇所があげられた.そのなかには,これまでに骨芽細胞に特異的なエンハンサーとして報告のあるゲノム領域も含まれていた.骨芽細胞における新規のエンハンサーを同定する目的で,候補のうちの11箇所のゲノム領域に対しin vitroにおいてレポーター解析を行った.線維芽細胞株および骨芽細胞株を用いた結果,検討したすべてのゲノム領域において,レポーター活性は骨芽細胞株において高かった.そのうちのひとつ,Notch2遺伝子のイントロンにおけるエンハンサー活性をin vivoにおいて検討した.Notch2は骨芽細胞前駆細胞の増殖および分化にかかわることが知られている6).このゲノム領域を含むGFP-LacZレポーターマウスを作出し,新生仔のマウスにおけるホールマウントLacZ染色を行ったところ,骨組織においてレポーター活性が認められた.組織学的な解析の結果,骨組織においてレポーター活性の認められた細胞はすべてSp7を発現する骨芽細胞であった.以上より,Sp7は骨芽細胞に特異的なNotch2遺伝子のエンハンサーを介してNotch2の発現を制御することが示された.
2.Sp7はホメオボックス転写因子を介してATリッチモチーフと間接的に作用する
in vivoにおいて骨芽細胞のゲノムにおけるSp7の作動様式を検討する目的で,得られたSp7作用領域の塩基配列を用いてモチーフ解析を行った.さきに述べたとおり,Spファミリーのメンバーは高度に保存されたジンクフィンガードメインを介してGCボックスと結合すると考えられてきた.しかしながら,ChIP-seq法による解析から予測されたSp7の結合モチーフにはGCボックスは有意に存在せず,ホメオボックス転写因子との結合配列を含むATリッチモチーフが大部分であった.複数のATリッチモチーフを並列したレポーターを作製しin vitroにおいてレポーター活性を測定したところ,骨芽細胞株において強いレポーター活性が認められたのに対し,線維芽細胞株においてレポーター活性は認められなかった.骨芽細胞株におけるレポーター活性はSp7のノックダウンによりほぼ完全に消失した.以上より,Sp7はin vivoにおいて,過去の報告とは異なる作動様式,すなわち,ATリッチモチーフとの相互作用を介して標的となる遺伝子の転写の活性を制御する可能性が示唆された.
Sp7がATリッチモチーフと直接的に結合するかどうかを検討する目的でゲルシフトアッセイを行った.その結果,直接的な結合はまったく認められず,Sp7はパートナーとなるほかの転写因子を介してATリッチモチーフと間接的に結合する可能性が示唆された.パートナーとなる転写因子は,1)ATリッチモチーフと直接に結合する,2)骨芽細胞において高発現する,3)Sp7と直接的に結合する,の3つの条件をみたすと考えられた.転写因子-DNA結合配列のデータベース4),過去の文献7),および,RNA-seq法により得られたデータを統合的に解析した結果,ホメオボックス転写因子であるDlx3,Dlx5,Dlx6が候補にあげられた.Dlx3,Dlx5,Dlx6のDNA結合モチーフはSp7に対するChIP-seq法による解析により得られたATリッチモチーフの一部と完全に一致すること,in vitroにおいてDlx3,Dlx5,Dlx6の発現は骨芽細胞の分化を誘導すること,Dlx5の長管骨における発現パターンはSp7の発現パターンと似ていること,Dlx5とDlx6のダブルノックアウトマウスにおいて骨形成が顕著に抑制されること8),最近の報告によりDlx5とSp7との直接的な結合が明らかにされたこと9),が裏づけとなった.Dlx3,Dlx5,Dlx6のうち,Dlx5が骨芽細胞においてもっとも高発現していたので,これを用いてSp7の作動様式のさらなる解析を試みた.
共免疫沈降-ウェスタンブロット法により,Sp7とDlx5との直接的な結合にはSp7のジンクフィンガードメインおよびDlx5のN末端ドメインが必須であることが明らかにされた.Sp7とDlx5のゲノムにおける相互作用を検討する目的で,骨芽細胞株を用いてChIP-seq法により解析したところ,Sp7およびDlx5は共通するゲノム領域に高頻度で作用すること,それらのゲノム領域にはATリッチモチーフが高頻度に存在すること,Sp7およびDlx5のChIP-seq法におけるシグナルには正の相関関係が認められること,が明らかにされた.in vitroにおけるレポーター解析の結果,Sp7およびDlx5はATリッチモチーフを介してその転写活性を相乗的に上昇させたのに対し,骨芽細胞株におけるDlx3,Dlx5,Dlx6のノックダウンはSp7の標的となるゲノム領域への作用を抑制した.以上より,Sp7は骨芽細胞において,ジンクフィンガードメインを介してホメオボックス転写因子Dlx5,Dlx3,Dlx6と転写複合体を形成し,ATリッチモチーフと作用することにより遺伝子の発現を制御する可能性が示唆された(図1).
3.ジンクフィンガードメインに存在する特異的なアミノ酸配列がSp7の作動様式に寄与する
Sp7の作動様式がSp7に特異的なものか,あるいは,Spファミリーのほかのメンバーと共通したものなのかを検討する目的で,骨芽細胞株においてSp1に対してChIP-seq法により解析した.Sp7に対するChIP-seq法により得られたデータと比較した結果,骨芽細胞のゲノムにおけるSp1の作用領域の大部分はSp7の作用領域とは異なっていた.Sp1に特異的な作用領域はおもに転写開始点の近傍に位置し,骨形成とは無関係の遺伝子の近傍に多く存在した一方,Sp7に特異的な作用領域はそのほとんどが転写開始点から遠位に存在し,骨形成と関連する遺伝子の近傍に多く存在した.モチーフ解析の結果,Sp1に特異的な作用領域にはGCボックスが有意に存在することが確認された.以上より,Sp1とSp7は,ゲノムにおいて異なる作用領域,異なるDNA結合モチーフ,異なる標的遺伝子をもつことが明らかにされた.
この違いはどこから生じたのだろうか? Sp1およびSp7のGCボックスとの結合能に注目し,ゲルシフトアッセイにより詳細に解析した.野生型のSp1およびSp7,および,互いにジンクフィンガードメインを入れ替えたSp1およびSp7の変異体を用いた結果,Sp1のもつジンクフィンガードメインはGCボックスとの結合に寄与したのに対し,Sp7のもつジンクフィンガードメインにはGCボックスとの結合能がほとんど認められなかった.Spファミリーのもつジンクフィンガードメインには3つのジンクフィンガーが存在し,それぞれのもつαへリックスがDNAとの結合に寄与することが知られている10).そこで,Spファミリーのメンバーのあいだでこのαへリックスのアミノ酸配列を比較したところ,Sp7に特異的なアミノ酸配列が見い出された.このSp7に特異的なアミノ酸残基をSp1の対応するアミノ酸残基と置換したところ,野生型のSp7においてはほとんど認められなかったGCボックスとの結合能が検出された.以上より,Sp7のもつジンクフィンガードメインに存在する特異的なアミノ酸配列がSp7のユニークな作動様式に寄与する可能性が示唆された.
4.Sp7に特異的なアミノ酸配列は骨組織をもつ脊椎動物において保存されている
このSp7の構造的な特徴に由来する生物学的な作用の進化的な意義について検証するため,脊索動物において,Sp7のもつジンクフィンガードメインのアミノ酸配列,あるいは,それともっとも相同性の高いアミノ酸配列を比較した.その結果,骨組織をもつ脊椎動物のみがSp7に特異的なアミノ酸配列をもっていた(図1).したがって,脊椎動物の進化において,Spファミリーのほかのメンバーとは異なる作動様式をもつSp7は,骨組織を形成する骨芽細胞の出現にともない獲得されたことが示唆された.
おわりに
この研究により,in vivoにおいて骨芽細胞における転写因子Sp7の標的となるゲノム領域およびその作動様式が明らかにされた.Sp7の標的となるゲノム領域には,骨形成においてその機能が知られている転写因子,シグナル伝達タンパク質,細胞外基質をコードする遺伝子が存在し,Sp7により制御される転写ネットワークの一端が明らかにされた.今回,見い出されたSp7の作動様式はまったく予想外のものであった.Spファミリーのほかのメンバーとはまったく異なるDNA配列と相互作用すること,および,DNAとの相互作用にはパートナーとなる転写因子が必要だったことから,従来のin vitroにおける解析では検出することの困難な生命現象が,ChIP-seq法による解析によりはじめて明らかにされた例だといえる.この研究により,ホメオボックス転写因子であるDlx3,Dlx5,Dlx6がSp7のパートナーとなる転写因子であることが明らかにされたが,パートナーとなる転写因子はほかにも存在するのか,骨芽細胞以外の細胞でも同様の作動様式が存在するのかについては,今後の研究課題である.
文 献
- Nakashima, K., Zhou, X., Kunkel, G. et al.: The novel zinc finger-containing transcription factor osterix is required for osteoblast differentiation and bone formation. Cell, 108, 17-29 (2002)[PubMed]
- Lapunzina, P., Aglan, M., Temtamy, S. et al.: Identification of a frameshift mutation in Osterix in a patient with recessive osteogenesis imperfecta. Am. J. Hum. Genet., 87, 110-114 (2010)[PubMed]
- Kadonaga, J. T., Jones, K. A. & Tjian, R.: Promoter-specific activation of RNA polymerase II transcription by Sp1. Trends Biochem. Sci., 11, 20-23 (1986)
- Wingender, E., Schoeps, T. & Donitz, J.: TFClass: an expandable hierarchical classification of human transcription factors. Nucleic Acids Res., 41, D165-D170 (2013)[PubMed]
- Rodda, S. J. & McMahon, A. P.: Distinct roles for Hedgehog and canonical Wnt signaling in specification, differentiation and maintenance of osteoblast progenitors. Development, 133, 3231-3244 (2006)[PubMed]
- Long, F.: Building strong bones: molecular regulation of the osteoblast lineage. Nat. Rev. Mol. Cell Biol.,. 13, 27-38 (2012)[PubMed]
- Berger, M. F., Badis, G., Gehrke, A. R. et al.: Variation in homeodomain DNA binding revealed by high-resolution analysis of sequence preferences. Cell, 133, 1266-1276 (2008)[PubMed]
- Robledo, R. F., Rajan, L., Li, X. et al.: The Dlx5 and Dlx6 homeobox genes are essential for craniofacial, axial, and appendicular skeletal development. Genes Dev., 16, 1089-1101 (2002)[PubMed]
- Kawane, T., Komori, H., Liu, W. et al.: Dlx5 and mef2 regulate a novel runx2 enhancer for osteoblast-specific expression. J. Bone Miner. Res., 29, 1960-1969 (2014)[PubMed]
- Suske, G.: The Sp-family of transcription factors. Gene, 238, 291-300 (1999)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2011年 東京大学大学院医学系研究科 修了,同年 東京大学医学部附属病院 博士研究員,2012年 Southern California大学Keck School of Medicine博士研究員を経て,2016年より東京大学大学院工学系研究科 博士研究員.
研究テーマ:骨形成における転写ネットワーク.
大庭 伸介(Shinsuke Ohba)
東京大学大学院工学系研究科 特任准教授.
Andrew P. McMahon
Southern California大学Keck School of MedicineにてProfessor.
研究室URL:http://mcmahonlab.usc.edu/
© 2016 北條宏徳・大庭伸介・Andrew P. McMahon Licensed under CC 表示 2.1 日本