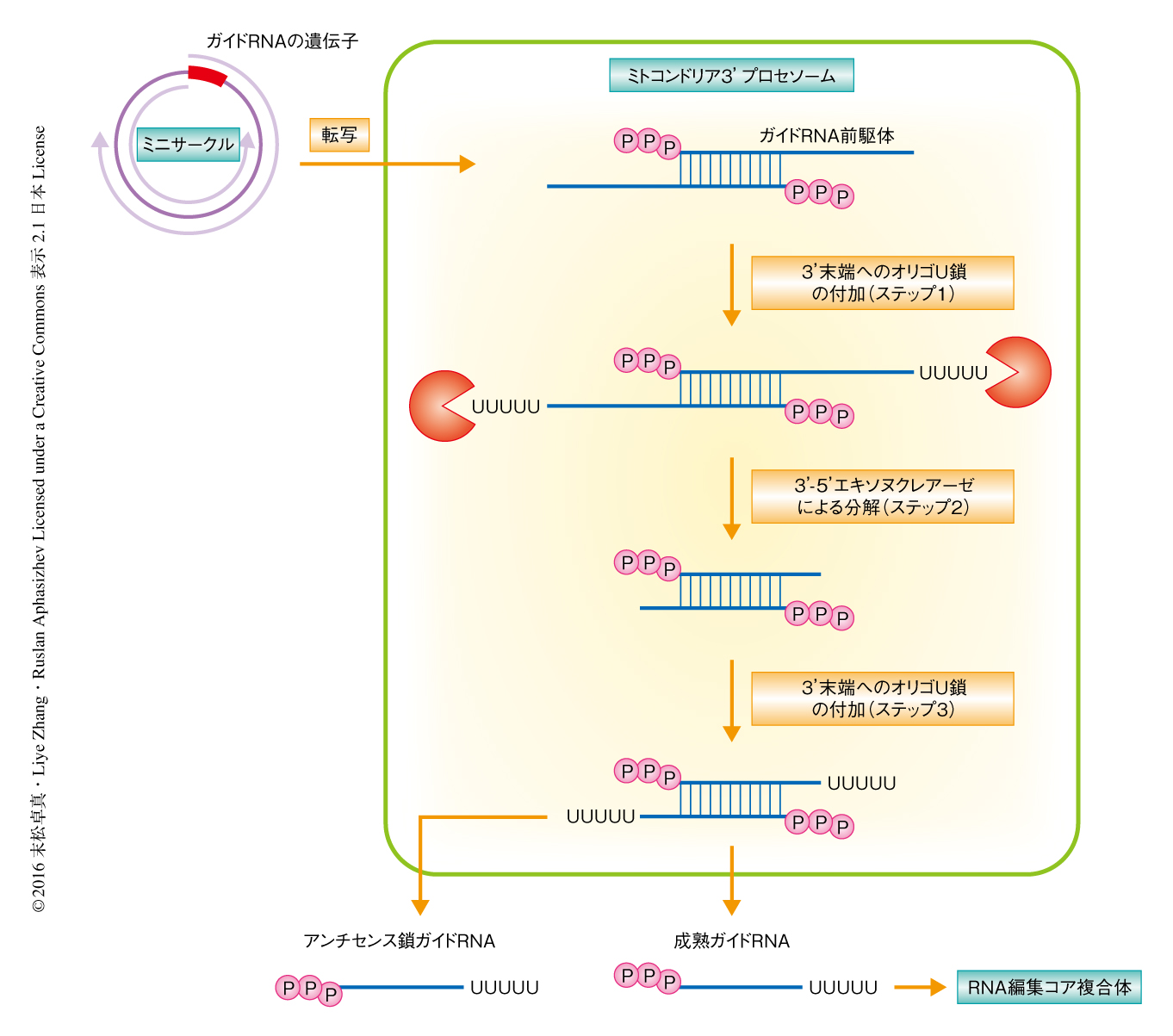
ミトコンドリア3'プロセソームとアンチセンスRNAによるガイドRNAの産生の機構
末松卓真・Liye Zhang・Ruslan Aphasizhev
(米国Boston大学School of Dental Medicine,Department of Molecular and Cell Biology)
email:末松卓真
DOI: 10.7875/first.author.2016.012
Antisense transcripts delimit exonucleolytic activity of the mitochondrial 3' processome to generate guide RNAs.
Takuma Suematsu, Liye Zhang, Inna Aphasizheva, Stefano Monti, Lan Huang, Qi Wang, Catherine E. Costello, Ruslan Aphasizhev
Molecular Cell, 61, 364-378 (2016)
トリパノソーマのミトコンドリアmRNAは,ガイドRNAとよばれる小分子RNAの配列にしたがいウリジンが挿入および除去されることにより翻訳の可能なmRNAへと編集される.ガイドRNAは,5'末端に1次転写産物の特徴である三リン酸,3'末端にオリゴU鎖をもつ.筆者らは,3'末端ウリジル転移酵素であるRET1,3'-5'エキソヌクレアーゼであるDSS1,機能未知の3つのタンパク質からなる複合体としてミトコンドリア3'プロセソームを同定した.このミトコンドリア3'プロセソームはガイドRNAの産生において,長鎖前駆体の3'末端へのオリゴU鎖の付加,3'末端からの40~60塩基までの分解,2次的な3'末端へのオリゴU鎖の付加に必須であった.また,ガイドRNAをコードする遺伝子は両方のDNA鎖から転写され,ガイドRNAを含む5'側の領域において2本鎖RNAを形成し,この2本鎖RNAの領域において3'-5'エキソヌクレアーゼは停止した.以上から,アンチセンスRNAと,RET1およびDSS1を含むミトコンドリア3'プロセソームによるガイドRNAの産生の機構が提唱された.
アフリカ睡眠病の病原体であるトリパノソーマ(Trypanosoma brucei)は鞭毛虫類に属する寄生性の原虫であり,そのミトコンドリアはいくつかのマキシサークルと数千のミニサークルから構成された巨大なDNA構造をもつ.マキシサークルには18のタンパク質および2つのrRNAの遺伝子がコードされているが,このうち12のmRNAは転写ののちウリジンの挿入および欠失によるRNA編集の過程をへて翻訳の可能なmRNAになる.このトリパノソーマにユニークな現象であるウリジン挿入欠失RNA編集においては,RNA編集コア複合体とよばれる複合体がmRNAの切断,ウリジンの挿入および除去といった酵素反応を担う1,2).この過程においては,ガイドRNAとよばれるミニサークルにコードされる40~60塩基の小分子RNAと編集前mRNAが不完全な2本鎖RNAを形成し,ガイドRNAの配列にしたがいウリジンが挿入および除去される.成熟ガイドRNAは5'末端に1次転写産物の特徴である三リン酸,3'末端に鋳型に非依存的に形成されるオリゴU鎖をもつ.このオリゴU鎖の付加は3'末端ウリジル転移酵素であるRET1により触媒される3,4).また,ガイドRNA前駆体は800~1200塩基という不均一な長さで転写され,これはミニサークルのほぼ全長をカバーする長さである.おのおののミニサークルは異なる配列をもちいくつかのガイドRNAをコードしているが,ポリシストロニックにガイドRNAを含む長鎖前駆体からどのように3'末端にオリゴU鎖をもつガイドRNAが産生されるかは不明であった.以前に,筆者らは,RET1のノックダウンおよびドミナントネガティブ型のRET1の強制発現により,成熟ガイドRNAが減少しガイドRNA前駆体が蓄積することを発見した4).しかし,RET1は3'末端のオリゴU鎖の伸長反応を触媒するがRNA分解活性はもたない.そこで,RET1を含むタンパク質複合体の存在を仮定し,その複合体の同定および機能解析からガイドRNAの産生経路の解明を試みた.
3'末端ウリジル転移酵素であるRET1をノックダウンによりガイドRNA前駆体の蓄積および成熟ガイドRNAの減少が観察されたという結果から4),RET1が未知のほかのタンパク質と複合体を形成してガイドRNAの産生に関与しているのではないかと仮定した.実際に,ミトコンドリアの抽出液をグリセロール密度勾配遠心法により分画したところ,RET1は約900 kDaの複合体に存在した.さらに,抗RET1抗体を用いた免疫沈降法および質量分析法により,RET1に相互作用するタンパク質としてDSS1および機能未知の3つのタンパク質が同定された.この複合体をミトコンドリア3'プロセソームと命名し,機能未知の3つのタンパク質をMPSS1,MPSS2,MPSS3と名づけた.DSS1は細菌のRNase IIあるいはRNase R,真核生物のRRP44やDIS3といったRNRスーパーファミリーに属する3'-5'エキソヌクレアーゼの相同タンパク質であった5).
ミトコンドリア3'プロセソームの構成タンパク質が生存に必須であるか,さらに,ミトコンドリアmRNAのプロセシングの過程における役割を明らかにするため,DSS1,MPSS1,MPSS2,MPSS3をノックダウンし,生存およびミトコンドリアmRNAの量的な変化を観察した.また,ドミナントネガティブ型のDSS1を強制発現させて同様に解析した.すると,いずれもノックダウンによる生存阻害を示した.定量RT-PCR法によりノックダウンののちのミトコンドリアmRNAの量的な変化について調べたところ,編集後mRNAはノックダウンにより減少したのに対し,編集前mRNAおよび編集をうけないmRNAはノックダウンにより増加する傾向を示した.以上から,ミトコンドリア3'プロセソームの構成タンパク質は生存に必須であり,また,ミトコンドリアmRNAのプロセシングに関与していることが明らかにされた.
ミトコンドリア3'プロセソームの構成タンパク質のノックダウンにより編集後mRNAの量が減少した一方,ミトコンドリア3'プロセソームとRNA編集コア複合体とのあいだの強固な相互作用を示唆するデータは得られなかった.したがって,ミトコンドリア3'プロセソームはRNA編集に関与するのではなく,ガイドRNAの産生経路に関与することが示唆された.そこで,ノーザンブロット法により既知の2つのガイドRNAについてノックダウンののちの経時的な変化について調べたところ,RET1のノックダウンと同様に,ミトコンドリア3'プロセソームの構成タンパク質のノックダウンによっても成熟ガイドRNAが減少しガイドRNA前駆体が蓄積した.ドミナントネガティブ型のDSS1を強制発現させた際にも同様にガイドRNA前駆体の蓄積が観察された.以上から,ミトコンドリア3'プロセソームのもつ酵素活性として期待される,RET1に由来する3'末端ウリジル転移酵素活性およびDSS1に由来する3'-5'エキソヌクレアーゼ活性が,ガイドRNA前駆体のプロセシングに必須であることが示唆された.
DSS1がミトコンドリア3'プロセソームにおいてRNA分解活性を担うことは,RNRスーパーファミリーの相同タンパク質であることや,さきのノックダウン実験から示唆された.また,ほかの研究グループによる先行研究において単体のDSS1からはRNA分解活性が検出されなかったことや5),DSS1はRNA認識モチーフをもたないことも含め,DSS1のRNA分解活性にはRET1を含むミトコンドリア3'プロセソームのほかの構成タンパク質が必要であると推測された.一方で,RET1のノックダウンおよびドミナントネガティブ型RET1の強制発現によってもガイドRNA前駆体の蓄積が観察されたが4),RET1のノックダウンによる複合体の不安定化からこの結果を説明することができても,ドミナントネガティブ型RET1の強制発現の結果をミトコンドリア3'プロセソームの構造的な変化から説明することはむすかしかった.そこで,RET1によるガイドRNA前駆体の3'末端へのオリゴU鎖の付加が,DSS1によるガイドRNA前駆体の3'末端からの分解に必要なステップではないかと仮定し,600~1200塩基の範囲に含まれるガイドRNA前駆体の配列を網羅的に解析した.すると,野生型において同定されたガイドRNA前駆体は3'末端に約12塩基のオリゴU鎖をもち,さらに,DSS1あるいはRET1のノックダウンにおいて同定されたガイドRNA前駆体のオリゴU鎖は4~5塩基に短縮していた.これらの結果は,ガイドRNA前駆体は実際に3'末端にオリゴU鎖の付加をうけ,このオリゴU鎖がDSS1によるガイドRNA前駆体の3'末端からの分解に必須であることを示唆し,さらに,DSS1あるいはRET1のノックダウンによりオリゴU鎖が短縮したことから,RET1とDSS1の酵素活性はミトコンドリア3'プロセソームにおいて相互依存的であることも示唆された.
トリパノソーマのミニサークルにはそれぞれいくつかガイドRNAがコードされていることから,長鎖前駆体はポリシストロニックにガイドRNAを含むと考えられる.一方で,小分子のミトコンドリアRNAの大部分は5'末端に三リン酸をもつことから6),5'末端の領域のみがガイドRNAになると考えられる.これらの示唆から,どのように長鎖前駆体が40~60塩基のガイドRNAとして成熟するか疑問であった.以前に,筆者らは,少量ではあるがガイドRNAのアンチセンス鎖の存在を報告した4).したがって,ガイドRNAをコードする遺伝子およびそのアンチセンス鎖がそれぞれ転写され,それらの5'末端の領域が2本鎖RNAを形成し,その2本鎖RNAの領域がミトコンドリア3'プロセソームのもつ3'-5'エキソヌクレアーゼ活性を阻害することにより,長鎖前駆体の5'末端の領域を残したガイドRNAが産生されるのではないか,と仮説をたてた.そこで,ドミナントネガティブ型RET1あるいはドミナントネガティブ型DSS1を強制発現させてノーザンブロット法によりガイドRNAのアンチセンスRNAを解析したところ,ガイドRNAと同様にアンチセンスRNAも減少し,その長鎖前駆体が蓄積していた.
2本鎖RNAの候補を調べるため,5'末端に三リン酸をもつ30~75塩基の小分子のミトコンドリアRNAのトランスクリプトーム解析を行った.ここで,ガイドRNAを編集されたmRNAの配列にマッピングされる小分子RNA,非ガイドRNAをマッピングされない小分子RNAと定義すると,10万以上の小分子RNAのうち,半数以上のRNAがガイドRNAとして同定された.これまで,ガイドRNAは3'末端に10~15塩基のオリゴU鎖をもつと考えられていたが7),今回の解析により,オリゴU鎖をもたない,あるいは1塩基のウリジンのみが付加した小分子RNAが多く発見された.また,ガイドRNAに比べ非ガイドRNAのオリゴU鎖は短い傾向がみられた.2本鎖RNAの候補としては1000種以上が同定された.同定された小分子RNAをミニサークルのゲノムにマッピングしたところ,ゲノムの両方のDNA鎖に広汎に転写が起こっていることもわかった.また,ミニサークルごとにマッピングされる小分子RNAの数は大きく異なり,ミニサークルごとに転写ユニットの数が多様であることも明らかにされたが,センス鎖とアンチセンス鎖は一貫して近傍に位置していた.
逆遺伝学的な手法およびミトコンドリアRNAのトランスクリプトーム解析により,ミトコンドリア3'プロセソームがガイドRNA前駆体の3'末端にオリゴU鎖を付加したのち,5'末端の領域に形成されたセンス鎖とアンチセンス鎖からなる2本鎖RNAの領域までガイドRNA前駆体を分解する,という仮説を立証したが,さらに,この仮説の試験管内再構築系における立証を試みた.精製された野生型のDSS1を含むミトコンドリア3'プロセソームと,ドミナントネガティブ型のDSS1を含むミトコンドリア3'プロセソームとで,1本鎖RNAに対する3'-5'エキソヌクレアーゼ活性を比較した.すると,野生型DSS1を含むミトコンドリア3'プロセソームは4~5塩基まで基質RNAを分解したのに対し,ドミナントネガティブ型DSS1を含むミトコンドリア3'プロセソームではRNA分解活性が失われていた.RNA分解活性に対する3'末端のオリゴU鎖の影響を調べたところ,ミトコンドリア3'プロセソームはオリゴU鎖をもつ基質RNAを効率よく分解することが見い出された.さらに,5'側の領域に25塩基対の2本鎖RNAの領域をもちセンス鎖およびアンチセンス鎖の両方の3'末端に約120塩基の1本鎖RNAの領域をもつ基質RNAに対するミトコンドリア3'プロセソームのRNA分解活性について調べたところ,効率よく1本鎖RNAの領域を分解するだけでなく,2本鎖RNAの領域を巻きもどし分解することを発見した.この巻きもどし活性はドミナントネガティブ型DSS1においては検出されなかったことから,RNA分解活性に依存した酵素活性であった.この巻きもどし活性には少なくとも10塩基以上の3'末端の突出が必要であり,一方で,2本鎖RNAの領域の長さには依存しないことも確認された.以上の知見をふまえ,センス鎖のみに3'末端の突出した領域をもつ基質RNAに対するミトコンドリア3'プロセソームのRNA分解活性について調べたところ,2本鎖RNAの領域より10~12塩基ほど下流においてミトコンドリア3'プロセソームが停止することが見い出された.この結果は,さきに同定された小分子RNAのペアがもつ3'末端の突出した領域の平均長に一致した.2本鎖RNAの領域に相互作用するようなほかのタンパク質により,より効率よくミトコンドリア3'プロセソームによる3'末端の分解が停止している可能性は否定できないが,少なくとも,アンチセンスRNAと形成された2本鎖RNAの領域によりミトコンドリア3'プロセソームによるガイドRNA前駆体の分解の停止位置が決定されるという仮説が実証された.
トリパノソーマのミトコンドリアのガイドRNAは,1990年に発見されて以来8),ウリジン挿入欠失RNA編集の機構においてmRNAへの情報伝達を担う小分子非コードRNAとしてその重要性が研究されてきた.しかし,RNA編集の過程の研究と比べると,ガイドRNAの産生経路への理解はだいぶ遅れていた.この研究においては,3'末端ウリジル転移酵素であるRET1をガイドRNAの産生経路の鍵と位置づけ,RET1を中心としたプロテオミクス解析,逆遺伝学的な解析,トランスクリプトーム解析,試験管内再構築系にもとづく生化学的な解析により,ガイドRNAの産生経路を解明した.その結果をふまえ,3'末端ウリジル転移酵素と3'-5'エキソヌクレアーゼという,酵素の性質としては正反対のタンパク質が同一の複合体を形成しガイドRNAの産生を担うというモデルが提唱された(図1).この機構は,miRNAやsiRNAのように,これまで知られていたRNase III型エンドヌクレアーゼを介した小分子RNAの産生経路とはまったく異なる機構であった.また,3'末端ウリジル転移酵素とエキソヌクレアーゼが協調したRNA分解経路も知られていたが9),この2つの酵素がRNAの産生にかかわる最初の例ともいえる.この研究においては,ガイドRNA前駆体のプロセシングの過程を解明したが,プロセシングののち,どのようにRNA編集コア複合体へ輸送され機能を発揮するかなど,ガイドRNAには興味深い点がまだまだ多い.
略歴:2008年 東京大学大学院医学系研究科博士課程 修了,同 特任助教,米国California大学Irvine校 博士研究員を経て,2013年より米国Boston大学School of Dental Medicine博士研究員.
研究テーマ:トリパノソーマのミトコンドリアにおける小分子RNAの産生の機構.
関心事:寄生虫のRNA科学.
Liye Zhang
米国Boston大学School of Dental Medicine博士研究員.
Ruslan Aphasizhev
米国Boston大学School of Dental Medicine教授.
© 2016 末松卓真・Liye Zhang・Ruslan Aphasizhev Licensed under CC 表示 2.1 日本
(米国Boston大学School of Dental Medicine,Department of Molecular and Cell Biology)
email:末松卓真
DOI: 10.7875/first.author.2016.012
Antisense transcripts delimit exonucleolytic activity of the mitochondrial 3' processome to generate guide RNAs.
Takuma Suematsu, Liye Zhang, Inna Aphasizheva, Stefano Monti, Lan Huang, Qi Wang, Catherine E. Costello, Ruslan Aphasizhev
Molecular Cell, 61, 364-378 (2016)
要 約
トリパノソーマのミトコンドリアmRNAは,ガイドRNAとよばれる小分子RNAの配列にしたがいウリジンが挿入および除去されることにより翻訳の可能なmRNAへと編集される.ガイドRNAは,5'末端に1次転写産物の特徴である三リン酸,3'末端にオリゴU鎖をもつ.筆者らは,3'末端ウリジル転移酵素であるRET1,3'-5'エキソヌクレアーゼであるDSS1,機能未知の3つのタンパク質からなる複合体としてミトコンドリア3'プロセソームを同定した.このミトコンドリア3'プロセソームはガイドRNAの産生において,長鎖前駆体の3'末端へのオリゴU鎖の付加,3'末端からの40~60塩基までの分解,2次的な3'末端へのオリゴU鎖の付加に必須であった.また,ガイドRNAをコードする遺伝子は両方のDNA鎖から転写され,ガイドRNAを含む5'側の領域において2本鎖RNAを形成し,この2本鎖RNAの領域において3'-5'エキソヌクレアーゼは停止した.以上から,アンチセンスRNAと,RET1およびDSS1を含むミトコンドリア3'プロセソームによるガイドRNAの産生の機構が提唱された.
はじめに
アフリカ睡眠病の病原体であるトリパノソーマ(Trypanosoma brucei)は鞭毛虫類に属する寄生性の原虫であり,そのミトコンドリアはいくつかのマキシサークルと数千のミニサークルから構成された巨大なDNA構造をもつ.マキシサークルには18のタンパク質および2つのrRNAの遺伝子がコードされているが,このうち12のmRNAは転写ののちウリジンの挿入および欠失によるRNA編集の過程をへて翻訳の可能なmRNAになる.このトリパノソーマにユニークな現象であるウリジン挿入欠失RNA編集においては,RNA編集コア複合体とよばれる複合体がmRNAの切断,ウリジンの挿入および除去といった酵素反応を担う1,2).この過程においては,ガイドRNAとよばれるミニサークルにコードされる40~60塩基の小分子RNAと編集前mRNAが不完全な2本鎖RNAを形成し,ガイドRNAの配列にしたがいウリジンが挿入および除去される.成熟ガイドRNAは5'末端に1次転写産物の特徴である三リン酸,3'末端に鋳型に非依存的に形成されるオリゴU鎖をもつ.このオリゴU鎖の付加は3'末端ウリジル転移酵素であるRET1により触媒される3,4).また,ガイドRNA前駆体は800~1200塩基という不均一な長さで転写され,これはミニサークルのほぼ全長をカバーする長さである.おのおののミニサークルは異なる配列をもちいくつかのガイドRNAをコードしているが,ポリシストロニックにガイドRNAを含む長鎖前駆体からどのように3'末端にオリゴU鎖をもつガイドRNAが産生されるかは不明であった.以前に,筆者らは,RET1のノックダウンおよびドミナントネガティブ型のRET1の強制発現により,成熟ガイドRNAが減少しガイドRNA前駆体が蓄積することを発見した4).しかし,RET1は3'末端のオリゴU鎖の伸長反応を触媒するがRNA分解活性はもたない.そこで,RET1を含むタンパク質複合体の存在を仮定し,その複合体の同定および機能解析からガイドRNAの産生経路の解明を試みた.
1.3'末端ウリジル転移酵素,3'-5'エキソヌクレアーゼ,機能未知の3つのタンパク質からミトコンドリア3'プロセソームが形成される
3'末端ウリジル転移酵素であるRET1をノックダウンによりガイドRNA前駆体の蓄積および成熟ガイドRNAの減少が観察されたという結果から4),RET1が未知のほかのタンパク質と複合体を形成してガイドRNAの産生に関与しているのではないかと仮定した.実際に,ミトコンドリアの抽出液をグリセロール密度勾配遠心法により分画したところ,RET1は約900 kDaの複合体に存在した.さらに,抗RET1抗体を用いた免疫沈降法および質量分析法により,RET1に相互作用するタンパク質としてDSS1および機能未知の3つのタンパク質が同定された.この複合体をミトコンドリア3'プロセソームと命名し,機能未知の3つのタンパク質をMPSS1,MPSS2,MPSS3と名づけた.DSS1は細菌のRNase IIあるいはRNase R,真核生物のRRP44やDIS3といったRNRスーパーファミリーに属する3'-5'エキソヌクレアーゼの相同タンパク質であった5).
2.ミトコンドリア3'プロセソームの構成タンパク質は生存およびミトコンドリアmRNAのプロセシングに必須である
ミトコンドリア3'プロセソームの構成タンパク質が生存に必須であるか,さらに,ミトコンドリアmRNAのプロセシングの過程における役割を明らかにするため,DSS1,MPSS1,MPSS2,MPSS3をノックダウンし,生存およびミトコンドリアmRNAの量的な変化を観察した.また,ドミナントネガティブ型のDSS1を強制発現させて同様に解析した.すると,いずれもノックダウンによる生存阻害を示した.定量RT-PCR法によりノックダウンののちのミトコンドリアmRNAの量的な変化について調べたところ,編集後mRNAはノックダウンにより減少したのに対し,編集前mRNAおよび編集をうけないmRNAはノックダウンにより増加する傾向を示した.以上から,ミトコンドリア3'プロセソームの構成タンパク質は生存に必須であり,また,ミトコンドリアmRNAのプロセシングに関与していることが明らかにされた.
3.ミトコンドリア3'プロセソームはガイドRNAのプロセシングに必須である
ミトコンドリア3'プロセソームの構成タンパク質のノックダウンにより編集後mRNAの量が減少した一方,ミトコンドリア3'プロセソームとRNA編集コア複合体とのあいだの強固な相互作用を示唆するデータは得られなかった.したがって,ミトコンドリア3'プロセソームはRNA編集に関与するのではなく,ガイドRNAの産生経路に関与することが示唆された.そこで,ノーザンブロット法により既知の2つのガイドRNAについてノックダウンののちの経時的な変化について調べたところ,RET1のノックダウンと同様に,ミトコンドリア3'プロセソームの構成タンパク質のノックダウンによっても成熟ガイドRNAが減少しガイドRNA前駆体が蓄積した.ドミナントネガティブ型のDSS1を強制発現させた際にも同様にガイドRNA前駆体の蓄積が観察された.以上から,ミトコンドリア3'プロセソームのもつ酵素活性として期待される,RET1に由来する3'末端ウリジル転移酵素活性およびDSS1に由来する3'-5'エキソヌクレアーゼ活性が,ガイドRNA前駆体のプロセシングに必須であることが示唆された.
4.3'末端のオリゴU鎖によりミトコンドリア3'プロセソームの3'-5'エキソヌクレアーゼ活性が促進される
DSS1がミトコンドリア3'プロセソームにおいてRNA分解活性を担うことは,RNRスーパーファミリーの相同タンパク質であることや,さきのノックダウン実験から示唆された.また,ほかの研究グループによる先行研究において単体のDSS1からはRNA分解活性が検出されなかったことや5),DSS1はRNA認識モチーフをもたないことも含め,DSS1のRNA分解活性にはRET1を含むミトコンドリア3'プロセソームのほかの構成タンパク質が必要であると推測された.一方で,RET1のノックダウンおよびドミナントネガティブ型RET1の強制発現によってもガイドRNA前駆体の蓄積が観察されたが4),RET1のノックダウンによる複合体の不安定化からこの結果を説明することができても,ドミナントネガティブ型RET1の強制発現の結果をミトコンドリア3'プロセソームの構造的な変化から説明することはむすかしかった.そこで,RET1によるガイドRNA前駆体の3'末端へのオリゴU鎖の付加が,DSS1によるガイドRNA前駆体の3'末端からの分解に必要なステップではないかと仮定し,600~1200塩基の範囲に含まれるガイドRNA前駆体の配列を網羅的に解析した.すると,野生型において同定されたガイドRNA前駆体は3'末端に約12塩基のオリゴU鎖をもち,さらに,DSS1あるいはRET1のノックダウンにおいて同定されたガイドRNA前駆体のオリゴU鎖は4~5塩基に短縮していた.これらの結果は,ガイドRNA前駆体は実際に3'末端にオリゴU鎖の付加をうけ,このオリゴU鎖がDSS1によるガイドRNA前駆体の3'末端からの分解に必須であることを示唆し,さらに,DSS1あるいはRET1のノックダウンによりオリゴU鎖が短縮したことから,RET1とDSS1の酵素活性はミトコンドリア3'プロセソームにおいて相互依存的であることも示唆された.
5.ガイドRNAをコードする遺伝子のアンチセンス鎖RNAがプロセシング中間体の構造を規定する
トリパノソーマのミニサークルにはそれぞれいくつかガイドRNAがコードされていることから,長鎖前駆体はポリシストロニックにガイドRNAを含むと考えられる.一方で,小分子のミトコンドリアRNAの大部分は5'末端に三リン酸をもつことから6),5'末端の領域のみがガイドRNAになると考えられる.これらの示唆から,どのように長鎖前駆体が40~60塩基のガイドRNAとして成熟するか疑問であった.以前に,筆者らは,少量ではあるがガイドRNAのアンチセンス鎖の存在を報告した4).したがって,ガイドRNAをコードする遺伝子およびそのアンチセンス鎖がそれぞれ転写され,それらの5'末端の領域が2本鎖RNAを形成し,その2本鎖RNAの領域がミトコンドリア3'プロセソームのもつ3'-5'エキソヌクレアーゼ活性を阻害することにより,長鎖前駆体の5'末端の領域を残したガイドRNAが産生されるのではないか,と仮説をたてた.そこで,ドミナントネガティブ型RET1あるいはドミナントネガティブ型DSS1を強制発現させてノーザンブロット法によりガイドRNAのアンチセンスRNAを解析したところ,ガイドRNAと同様にアンチセンスRNAも減少し,その長鎖前駆体が蓄積していた.
2本鎖RNAの候補を調べるため,5'末端に三リン酸をもつ30~75塩基の小分子のミトコンドリアRNAのトランスクリプトーム解析を行った.ここで,ガイドRNAを編集されたmRNAの配列にマッピングされる小分子RNA,非ガイドRNAをマッピングされない小分子RNAと定義すると,10万以上の小分子RNAのうち,半数以上のRNAがガイドRNAとして同定された.これまで,ガイドRNAは3'末端に10~15塩基のオリゴU鎖をもつと考えられていたが7),今回の解析により,オリゴU鎖をもたない,あるいは1塩基のウリジンのみが付加した小分子RNAが多く発見された.また,ガイドRNAに比べ非ガイドRNAのオリゴU鎖は短い傾向がみられた.2本鎖RNAの候補としては1000種以上が同定された.同定された小分子RNAをミニサークルのゲノムにマッピングしたところ,ゲノムの両方のDNA鎖に広汎に転写が起こっていることもわかった.また,ミニサークルごとにマッピングされる小分子RNAの数は大きく異なり,ミニサークルごとに転写ユニットの数が多様であることも明らかにされたが,センス鎖とアンチセンス鎖は一貫して近傍に位置していた.
6.アンチセンスRNAにもとづいた3'末端の決定
逆遺伝学的な手法およびミトコンドリアRNAのトランスクリプトーム解析により,ミトコンドリア3'プロセソームがガイドRNA前駆体の3'末端にオリゴU鎖を付加したのち,5'末端の領域に形成されたセンス鎖とアンチセンス鎖からなる2本鎖RNAの領域までガイドRNA前駆体を分解する,という仮説を立証したが,さらに,この仮説の試験管内再構築系における立証を試みた.精製された野生型のDSS1を含むミトコンドリア3'プロセソームと,ドミナントネガティブ型のDSS1を含むミトコンドリア3'プロセソームとで,1本鎖RNAに対する3'-5'エキソヌクレアーゼ活性を比較した.すると,野生型DSS1を含むミトコンドリア3'プロセソームは4~5塩基まで基質RNAを分解したのに対し,ドミナントネガティブ型DSS1を含むミトコンドリア3'プロセソームではRNA分解活性が失われていた.RNA分解活性に対する3'末端のオリゴU鎖の影響を調べたところ,ミトコンドリア3'プロセソームはオリゴU鎖をもつ基質RNAを効率よく分解することが見い出された.さらに,5'側の領域に25塩基対の2本鎖RNAの領域をもちセンス鎖およびアンチセンス鎖の両方の3'末端に約120塩基の1本鎖RNAの領域をもつ基質RNAに対するミトコンドリア3'プロセソームのRNA分解活性について調べたところ,効率よく1本鎖RNAの領域を分解するだけでなく,2本鎖RNAの領域を巻きもどし分解することを発見した.この巻きもどし活性はドミナントネガティブ型DSS1においては検出されなかったことから,RNA分解活性に依存した酵素活性であった.この巻きもどし活性には少なくとも10塩基以上の3'末端の突出が必要であり,一方で,2本鎖RNAの領域の長さには依存しないことも確認された.以上の知見をふまえ,センス鎖のみに3'末端の突出した領域をもつ基質RNAに対するミトコンドリア3'プロセソームのRNA分解活性について調べたところ,2本鎖RNAの領域より10~12塩基ほど下流においてミトコンドリア3'プロセソームが停止することが見い出された.この結果は,さきに同定された小分子RNAのペアがもつ3'末端の突出した領域の平均長に一致した.2本鎖RNAの領域に相互作用するようなほかのタンパク質により,より効率よくミトコンドリア3'プロセソームによる3'末端の分解が停止している可能性は否定できないが,少なくとも,アンチセンスRNAと形成された2本鎖RNAの領域によりミトコンドリア3'プロセソームによるガイドRNA前駆体の分解の停止位置が決定されるという仮説が実証された.
おわりに
トリパノソーマのミトコンドリアのガイドRNAは,1990年に発見されて以来8),ウリジン挿入欠失RNA編集の機構においてmRNAへの情報伝達を担う小分子非コードRNAとしてその重要性が研究されてきた.しかし,RNA編集の過程の研究と比べると,ガイドRNAの産生経路への理解はだいぶ遅れていた.この研究においては,3'末端ウリジル転移酵素であるRET1をガイドRNAの産生経路の鍵と位置づけ,RET1を中心としたプロテオミクス解析,逆遺伝学的な解析,トランスクリプトーム解析,試験管内再構築系にもとづく生化学的な解析により,ガイドRNAの産生経路を解明した.その結果をふまえ,3'末端ウリジル転移酵素と3'-5'エキソヌクレアーゼという,酵素の性質としては正反対のタンパク質が同一の複合体を形成しガイドRNAの産生を担うというモデルが提唱された(図1).この機構は,miRNAやsiRNAのように,これまで知られていたRNase III型エンドヌクレアーゼを介した小分子RNAの産生経路とはまったく異なる機構であった.また,3'末端ウリジル転移酵素とエキソヌクレアーゼが協調したRNA分解経路も知られていたが9),この2つの酵素がRNAの産生にかかわる最初の例ともいえる.この研究においては,ガイドRNA前駆体のプロセシングの過程を解明したが,プロセシングののち,どのようにRNA編集コア複合体へ輸送され機能を発揮するかなど,ガイドRNAには興味深い点がまだまだ多い.
文 献
- Blum, B., Bakalara, N. & Simpson, L.: A model for RNA editing in kinetoplastid mitochondria: "guide" RNA molecules transcribed from maxicircle DNA provide the edited information. Cell, 60, 189-198 (1990)[PubMed]
- Aphasizhev, R., Aphasizheva, I., Nelson, R. E. et al.: Isolation of a U-insertion/deletion editing complex from Leishmania tarentolae mitochondria. EMBO J., 22, 913-924 (2003)[PubMed]
- Aphasizhev, R., Sbicego, S., Peris, M. et al.: Trypanosome mitochondrial 3' terminal uridylyl transferase (TUTase): the key enzyme in U-insertion/deletion RNA editing. Cell, 108, 637-648 (2002)[PubMed]
- Aphasizheva, I. & Aphasizhev, R.: RET1-catalyzed uridylylation shapes the mitochondrial transcriptome in Trypanosoma brucei. Mol. Cell. Biol., 30, 1555-1567 (2010)[PubMed]
- Mattiacio, J. L. & Read, L. K.: Roles for TbDSS-1 in RNA surveillance and decay of maturation by-products from the 12S rRNA locus. Nucleic Acids Res., 36, 319-329 (2008)[PubMed]
- Aphasizheva, I., Zhang, L., Wang, X. et al.: RNA binding and core complexes constitute the U-insertion/deletion editosome. Mol. Cell. Biol., 34, 4329-4342 (2014)[PubMed]
- Thiemann, O. H. & Simpson, L.: Analysis of the 3' uridylylation sites of guide RNAs from Leishmania tarentolae. Mol. Biochem. Parasitol., 79, 229-234 (1996)[PubMed]
- Blum, B. & Simpson, L.: Guide RNAs in kinetoplastid mtochondria have a nonencoded 3' oligo(U) tail involved in recogntion of the preedited region. Cell, 62, 391-397 (1990)[PubMed]
- Chang, H. M., Triboulet, R., Thornton, J. E. et al.: A role for the Perlman syndrome exonuclease Dis3l2 in the Lin28-let-7 pathway. Nature, 497, 244-248 (2013)[PubMed]
著者プロフィール
略歴:2008年 東京大学大学院医学系研究科博士課程 修了,同 特任助教,米国California大学Irvine校 博士研究員を経て,2013年より米国Boston大学School of Dental Medicine博士研究員.
研究テーマ:トリパノソーマのミトコンドリアにおける小分子RNAの産生の機構.
関心事:寄生虫のRNA科学.
Liye Zhang
米国Boston大学School of Dental Medicine博士研究員.
Ruslan Aphasizhev
米国Boston大学School of Dental Medicine教授.
© 2016 末松卓真・Liye Zhang・Ruslan Aphasizhev Licensed under CC 表示 2.1 日本